É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Microbiota de jardins de formigas Attine: visualizando uma paisagem microbiana por microscopia eletrônica de varredura
Neste Artigo
Resumo
Propomos um protocolo otimizado de Microscopia Eletrônica de Varredura para visualização de amostras altamente heterogêneas e delicadas contendo biomassa vegetal e fúngica, juntamente com microbiota e biofilme. Este protocolo permite descrever as dimensões espaciais da organização da microbiota.
Resumo
Em ecossistemas de macroescala, como florestas tropicais ou recifes de coral, a localização espacial dos organismos é a base de nossa compreensão da ecologia da comunidade. No mundo microbiano, da mesma forma, os ecossistemas em microescala estão longe de ser uma mistura aleatória e homogênea de organismos e habitats. Acessar a distribuição espacial dos micróbios é fundamental para entender o funcionamento e a ecologia da microbiota, pois as espécies coabitantes são mais propensas a interagir e influenciar a fisiologia umas das outras.
Um ecossistema microbiano entre reinos está no centro das colônias de formigas produtoras de fungos, que cultivam fungos basidiomicetos como recurso nutricional. As formigas Attine procuram diversos substratos (principalmente à base de plantas), metabolizados pelo fungo cultivado enquanto formam uma estrutura esponjosa, um "jardim microbiano" que atua como um intestino externo. O jardim é uma malha entrelaçada de hifas fúngicas que crescem metabolizando o substrato, abrindo nichos para que uma microbiota característica e adaptada se estabeleça. Acredita-se que a microbiota contribua para a degradação do substrato e o crescimento de fungos, embora sua organização espacial ainda não tenha sido determinada.
Aqui, descrevemos como empregamos a Microscopia Eletrônica de Varredura (MEV) para investigar, com detalhes sem precedentes, a organização espacial da microbiota e do biofilme em diferentes sistemas de fungicultura de formigas produtoras de fungos. A imagem de MEV forneceu uma descrição da estrutura e organização espacial da microbiota. O MEV revelou que a microbiota comumente se reúne em biofilmes, uma estrutura generalizada das paisagens microbianas na fungicultura. Apresentamos os protocolos empregados para fixar, desidratar, secar, revestir por pulverização catódica e criar imagens de uma comunidade tão complexa. Esses protocolos foram otimizados para lidar com amostras delicadas e heterogêneas, compreendendo biomassa vegetal e fúngica, bem como a microbiota e o biofilme.
Introdução
Os ecossistemas são compostos por organismos interconectados por processos em uma localização geográfica específica (ou seja, o meio ambiente). Os organismos interagem com seu ambiente ao longo do tempo, a partir do qual emergem padrões espaciais complexos e heterogêneos. A padronização espacial determina a diversidade e a estabilidade ecológica e, em última análise, o funcionamento do ecossistema 1,2,3,4. Em ecossistemas de macroescala, como pântanos, savanas, recifes de coral e ecossistemas áridos, os padrões espaciais estão correlacionados com o fluxo e a concentração de recursos. Permitir a otimização de recursos, a heterogeneidade espacial e a padronização resultam em ecossistemas mais resilientes do que os homogêneos2. A localização espacial dos organismos na base da ecologia da comunidade também é traduzida para o mundo microbiano.
Os ecossistemas microbianos, longe de organismos misturados de forma aleatória e homogênea em microhabitats, exibem padrões espaciais que definem grande parte de seu funcionamento 5,6,7. Das colunas de Winogradsky à microbiota associada ao ambiente e ao hospedeiro, esses ecossistemas são organizados de forma heterogênea no espaço, com arranjos espaciais que provocam diferentes respostas fenotípicas. As espécies coabitantes são mais propensas a interagir e influenciar a fisiologia umas das outras. Assim, a organização espacial da comunidade, mais do que sua composição per se, delimita propriedades ecossistêmicas e nichos ecológicos 5,7,8. Ilustrando esses conceitos, as mudanças na padronização espacial parecem correlacionadas à progressão patológica de placas dentárias, cárie, doenças gengivais 9,10, doença inflamatória intestinal11, infecções pulmonares de fibrose cística, infecções crônicas de feridas12,13, câncer colorretal e adenomas14.
No âmbito da biogeografia microbiana (o estudo da distribuição e padronização da biodiversidade no espaço e no tempo em microescala), o conhecimento dos ecossistemas microbianos é amplamente beneficiado pela compreensão de seus padrões espaciais 6,13,15,16,17. Analisamos os padrões espaciais de um ecossistema microbiano construído por insetos, encontrado no centro das carismáticas colônias de formigas attine (Hymenoptera: Formicidae: Myrmicinae: Attini: Attina). Lá reside um "jardim microbiano", centrado em torno de um fungo basidiomiceto na tribo Leucocoprinae (Basidiomycota: Agaricaceae) ou na família Pterulaceae (Basidiomycota: Agaricales) 18 , 19 , 20 , 21 , 22 . O jardim é uma estrutura esponjosa que emerge de uma malha entrelaçada de hifas que cresce metabolizando o substrato principalmente vegetal incorporado pelas formigas (Figura 1). Estes podem incluir, de acordo com os gêneros atinos: partes secas de plantas, excrementos e carcaças de insetos, folhas recém-cortadas, sementes e partes florais23,24. Análogo a um intestino herbívoro externo, o jardim converte enzimaticamente e quimicamente polímeros recalcitrantes em recursos nutricionais lábeis, fornecendo às formigas aminoácidos essenciais, lipídios e açúcares solúveis 21,25,26,27,28.
Análises ultraestruturais, enzimáticas e transcriptômicas realizadas para jardins dos gêneros de corte de folhas Atta e Acromyrmex sugerem que esses ambientes estruturam um continuum de degradação do substrato e manchas nutricionais 26,29,30,31,32. As partes jovens do jardim tendem a ser mais escuras devido ao substrato recém-incorporado após serem fragmentadas. Esses substratos adicionados recentemente são frequentemente colonizados pelas bordas, que foram cortadas por formigas operárias e inoculadas com aglomerados miceliais. Irradiando das bordas cortadas, as hifas fúngicas se espalham sobre o substrato 29,32,33. A abundância de hifas aumenta à medida que a degradação do substrato progride, resultando em regiões esbranquiçadas e metabolicamente ativas 30,31,32. Regiões mais antigas, com substrato mais degradado e microbiota abundante29,32, tendem a apresentar tons acastanhados e maior umidade. Os trabalhadores removem fragmentos dessa região, separando-os em pilhas de resíduos, onde também retiram substratos que prejudicam o simbionte fúngico 34,35,36. As pilhas de resíduos, embora fisicamente separadas do jardim, são um local de degradação contínua do substrato e ciclagem de nutrientes pela abundante microbiota habitante 29,32,37,38,39.
Uma microbiota composta principalmente por Enterobacter, Klebsiella, Pantoea, Pseudomonas e Serratia, também habita o jardim, aparentemente sendo compartilhada por diversos sistemas de fungicultura attine. Codificando vias metabólicas que poderiam complementar o metabolismo fúngico, a microbiota participa potencialmente das respostas fisiológicas do jardim 40,41,42,43,44. Não apenas os dados metagenômicos indicaram que a microbiota estava lá41,42, mas também a análise por microscopia eletrônica de varredura (MEV) da fungicultura de formigas cortadeiras mostrou principalmente bactérias em forma de bastonete sobre o substrato da planta32. Embora as bactérias (incluindo cepas celulolíticas) tenham sido isoladas de todo o jardim, elas foram visualizadas apenas em partes mais antigas do jardim e em pilhas de resíduos, bem como no pellet inicial carregado pelas rainhas fundadoras 29,32. Também era incerto se a microbiota poderia formar biofilmes in vivo (ou seja, no jardim e nos resíduos), conforme sugerido por sua capacidade metabólica42 e observado in vitro44.
Aqui, empregamos o SEM para compreender melhor a organização espacial da microbiota nas regiões do jardim, detalhando as interações físicas microbiota-substrato e microbiota-hifas. Ao fornecer imagens com maior profundidade focal, o MEV permite observações de estruturas microscópicas tridimensionais em alta resolução, permitindo uma análise completa dos padrões espaciais da microbiota do jardim. Detalhamos as etapas para fixar, desidratar, secar, revestir e criar imagens de amostras fúngicas heterogêneas e delicadas. Ao remover a etapa de pós-fixação com tetróxido de ósmio (OsO4) e reduzir o tempo de desidratação, simplificamos os protocolos 32,33,45 para preparação de amostras de jardim e resíduos para análise de MEV. Este protocolo adaptado preserva os padrões estruturais das hifas, bem como a organização espacial da microbiota e do biofilme, e pode ser aplicado a outros ecossistemas microbianos delicados e biofilmes.
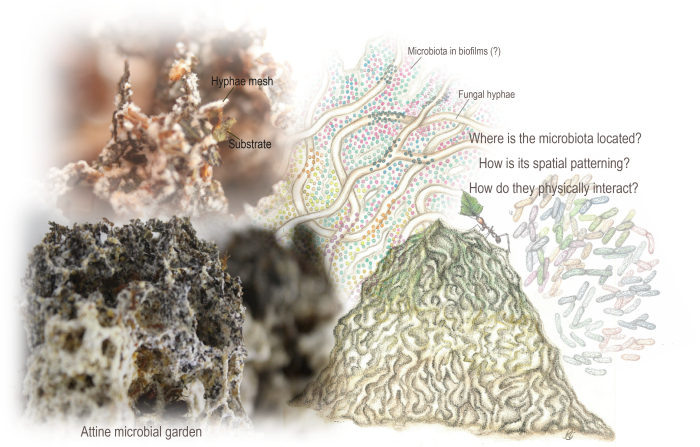
Figura 1: Jardins microbianos Attine. O jardim é uma estrutura esponjosa resultante de uma malha entrelaçada de hifas que cresce metabolizando o substrato principalmente vegetal incorporado pelas formigas. Também habitando o jardim está a microbiota, que codifica vias metabólicas que podem complementar o metabolismo fúngico. Dados metagenômicos e análises prévias de Microscopia Eletrônica de Varredura indicaram sua presença, embora tivéssemos pouco conhecimento de sua organização espacial e interações físicas com o substrato e as hifas fúngicas. Empregamos o MEV para desvendar a organização e padronização espacial da microbiota e do biofilme. Ilustrações de Mariana Barcoto (jardim e microbiota adaptados de Barcoto e Rodrigues 94), e fotos de Mariana Barcoto e Enzo Sorrentino. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Amostragem de colônias de campo
NOTA: Ao coletar colônias de formigas, certifique-se de que todas as permissões exigidas pela legislação local sejam obtidas antes da coleta. No nosso caso, o alvará de coleta #74585 foi emitido pelo Instituto Chico Mendes de Conservação e Biodiversidade (ICMBio). Quando as amostras vierem de uma colônia de laboratório, vá para a seção 2.
- Localize e marque a colônia. Escave uma trincheira ao redor da área do ninho até que a câmara do jardim fique exposta (Figura 2A).
NOTA: Algumas espécies de attine podem construir suas colônias sob a serapilheira ou dentro de troncos podres. Nesses casos, gire cuidadosamente a cama ou quebre cuidadosamente as toras para coletar as amostras. Para obter informações detalhadas sobre localização, coleta e manutenção de colônias vivas de diversas espécies de formigas attinas, consulte Sosa-Calvo et al.46. - Abra a câmara do jardim lateralmente para evitar que o solo caia na superfície do jardim. Colete cuidadosamente amostras de jardim usando uma pinça entomológica, uma colher ou uma escumadeira de cozinha, dependendo do tamanho do jardim.
NOTA: Certifique-se de esterilizar as ferramentas antes de recolhê-las. Ao coletar jardins de formigas cortadeiras, use luvas de tecido grosso para evitar (ou pelo menos atenuar) as picadas das operárias. Para outras espécies de attine, luvas de tecido são opcionais. - Transfira as amostras do jardim para um recipiente de plástico limpo contendo uma camada de gesso para equilibrar a umidade do jardim. Depois de transferir o jardim e as formigas, feche hermeticamente o recipiente para evitar a secagem da amostra. Armazene amostras de jardim a 23-25 °C até o processamento.
- Feche a vala com solo removido anteriormente.
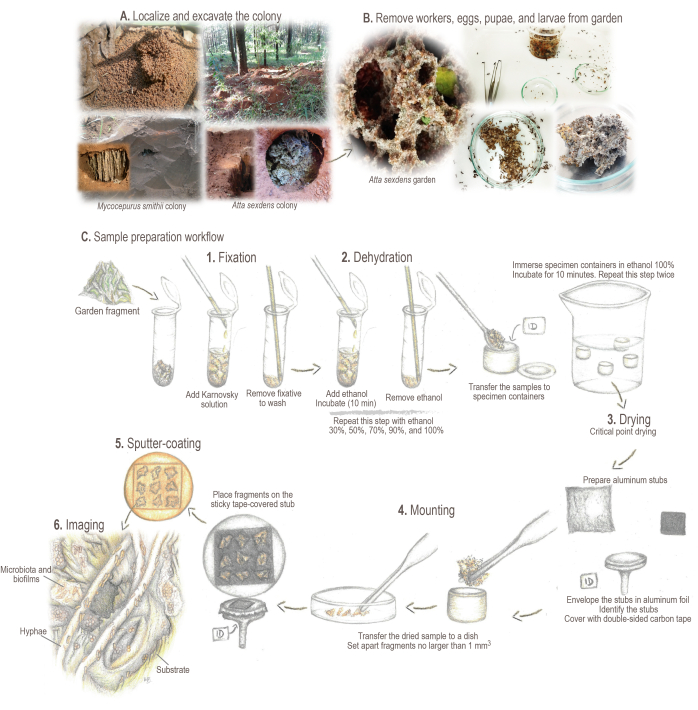
Figura 2: Protocolo de preparo da amostra. (A) Amostragem de colônias de campo. (B) Processamento de amostras. (C) Breves fundamentos e fluxo de trabalho para preparação de amostras: 1. Fixação: para fortalecer e preservar a estrutura da amostra. 2. Desidratação: o teor de água das amostras é trocado por etanol. 3. Secagem do ponto crítico: o CO2 líquido substitui o etanol e é evaporado. 4. Montagem: amostra exibida para análise. 5. Revestimento por pulverização catódica com ouro: evite o carregamento da amostra. 6. Imagem. Ilustrações e fotos de Mariana Barcoto. Clique aqui para ver uma versão maior desta figura.
2. Reagentes
NOTA: Lembre-se de que as seguintes soluções devem ser preparadas com antecedência.
- Prepare o tampão de cacodilato de sódio 0,2 M. Para isso, dissolver 42,8 g de cacodilato de sódio em 800 mL de água destilada, mexendo até a dissolução e estabilização do pH em 7,2 (se necessário, ajustar o pH com ácido clorídrico). Completar o volume da solução para 1 l adicionando água destilada. Conservar a solução a 4 °C (durante ~1 mês).
CUIDADO: O glutaraldeído e o paraformaldeído são tóxicos e devem ser manuseados dentro de uma capela de exaustão. Use luvas de nitrilo e óculos de proteção o tempo todo ao manusear esses reagentes. - Prepare o fixador de Karnovsky (modificado de Karnovsky)47. Para isso, adicione 10 mL de solução aquosa de glutaraldeído a 25% e 10 mL de solução aquosa de paraformaldeído a 20% a 25 mL de tampão de cacodilato de sódio 0,2 M e misture. Adicione 1 mL de cloreto de cálcio 0,1 M (CaCl2) e complete o volume para 100 mL adicionando água destilada. Conservar a solução a 4 °C (até 1 mês).
CUIDADO: O fixador de Karnovsky é prejudicial quando inalado e pode causar irritação na pele e nos olhos. Assim, evite respirar vapores, usando apenas ao ar livre ou em uma área bem ventilada. Use luvas de nitrilo e óculos de proteção o tempo todo ao manusear a solução. - Usando etanol absoluto (grau analítico), prepare soluções de etanol a 30%, 50%, 70% e 90% em água destilada; 100% de etanol também é necessário.
3. Fixação da amostra
NOTA: Os fixadores endurecem e preservam as amostras, mantendo as características morfológicas. Os aldeídos (como o paraformaldeído e o glutaraldeído) são fixadores não coagulantes do tipo reticulação, induzindo ligações cruzadas dentro e entre proteínas e ácidos nucléicos48.
- Remova operárias, ovos, pupas e larvas das amostras de jardim usando fórceps entomológicos (Figura 2B). Separe fragmentos de jardim não maiores que 5 mm3. Adicione os fragmentos a um tubo de 2 mL (Figura 2C).
- Nos tubos que contêm as amostras, use uma pipeta de vidro Pasteur para adicionar ~ 1 mL de solução fixadora de Karnovsky (certifique-se de que a amostra esteja completamente coberta). Misturar com agitação suave para ajudar a amostra a mergulhar e incubar a 4 °C durante pelo menos 24 h antes de continuar o processamento da amostra (figura 2C.1).
NOTA: Quando as seguintes etapas de desidratação não forem realizadas imediatamente após a fixação, o protocolo pode ser pausado nesta etapa e as amostras podem ser armazenadas por ~ 1 ano a 4 ° C.
Sugerimos o uso de uma pipeta de vidro Pasteur, pois é composta por um material inerte e é mais fácil de limpar para reutilização posterior. O material do jardim é altamente hidrofóbico, tendendo a flutuar sobre a superfície da solução fixadora. Geralmente, leva até 5 minutos para ficar completamente embebido no fixador. Certifique-se de que o fixador cubra os fragmentos do jardim, pois seu volume tende a ser reduzido à medida que entra nos poros da amostra. Verificamos empiricamente que, depois que a amostra foi encharcada e molhada, seus componentes (em particular, o delicado micélio fúngico) tornaram-se suscetíveis a quebrar quando misturados ainda mais. Assim, recomendamos evitar ao máximo agitar as amostras.
4. Desidratação da amostra
NOTA: A série de lavagem de etanol troca gradualmente a água em amostras por etanol. É importante começar com uma solução de etanol de baixa concentração (veja abaixo) para evitar danos excessivos ou colapso dessas amostras delicadas49.
- Remova completamente a solução fixadora de Karnovsky usando uma pipeta de vidro, tomando cuidado para não interromper a amostra (Figura 2C.1).
NOTA: Descarte o fixador de Karnovsky em um recipiente devidamente rotulado para gerenciamento de resíduos químicos tóxicos. - Imediatamente após a remoção do fixador, adicionar 1 mL de etanol a 30%, tomando cuidado para não romper a amostra, e incubar por 10 min em temperatura ambiente (Figura 2C.2).
NOTA: As amostras precisam sempre ser embebidas em solução. Certifique-se de substituir rapidamente as soluções durante a série de lavagem gradual do etanol. Como o jardim perde seu aspecto poroso e se agrega no fundo do tubo, 1 mL de etanol tende a ser suficiente para cobrir a amostra (quando a amostra não é maior que 5 mm3). No entanto, se as amostras não estiverem completamente cobertas, adicione etanol até que a amostra esteja completamente coberta. - Remova completamente o etanol a 30% com uma pipeta de vidro, tomando cuidado para não interromper a amostra. Descarte o etanol a 30% adequadamente.
NOTA: Para toda a série de lavagem gradual do etanol, descarte o etanol em um recipiente devidamente rotulado para gerenciamento de resíduos químicos tóxicos. - Adicione 1 mL de etanol a 50% e incube por 10 min em temperatura ambiente. Remova completamente o etanol a 50% com uma pipeta de vidro, tomando cuidado para não interromper a amostra. Descarte o etanol a 50% adequadamente.
- Adicione 1 mL de etanol a 70% e incube por 10 min em temperatura ambiente. Remova completamente o etanol a 70% com uma pipeta de vidro, tomando cuidado para não interromper a amostra. Descarte o etanol 70% corretamente.
NOTA: Na etapa de lavagem com etanol a 70%, o usuário pode pausar o protocolo, se necessário, pois os tubos de amostra podem ser armazenados durante a noite a 4 °C, quando o material não será processado imediatamente. - Adicione 1 mL de etanol a 90% e incube por 10 min em temperatura ambiente. Remova completamente o etanol a 90% com uma pipeta de vidro, tomando cuidado para não interromper a amostra. Descarte o etanol a 90% adequadamente.
- Adicione 1 mL de etanol 100% e incube por 10 min em temperatura ambiente. Remova completamente o etanol 100% com uma pipeta de vidro, tomando cuidado para não atrapalhar a amostra. Descarte o etanol 100% corretamente.
- Usando uma pinça e/ou uma espátula, transfira cuidadosamente as amostras para recipientes de amostra para o secador de ponto crítico (CPD), contendo etiquetas de identificação de amostra (feitas anteriormente em papel e lápis). Para evitar o ressecamento das amostras, essa transferência é realizada com o recipiente colocado em uma placa de Petri coberta com etanol 100%.
- Coloque as tampas nos recipientes e mergulhe-os em um copo de vidro graduado contendo 100% de etanol suficiente para submergir os recipientes. Cubra o copo de vidro e incube por 10 min em temperatura ambiente; Em seguida, descarte adequadamente o etanol 100%.
- Transfira os recipientes de amostra para outro copo de vidro graduado contendo etanol 100% suficiente para submergir os recipientes. Cubra o copo de vidro e incube por 10 min em temperatura ambiente; Em seguida, transfira os recipientes de amostras para o secador de ponto crítico.
NOTA: Após a série de lavagens, pipetas e copos de vidro devem ser abundantemente enxaguados com água destilada, e essa água residual deve ser descartada em um recipiente devidamente rotulado para gerenciamento de resíduos químicos tóxicos. Após o enxágue, os itens de vidro podem ser lavados com detergente neutro, enxaguados com água da torneira e secos ao ar.
5. Secagem do ponto crítico
NOTA: Um secador de ponto crítico troca o etanol nas amostras por dióxido de carbono líquido (CO2), que evapora da amostra em temperatura e pressão mais altas. Siga as instruções do fabricante para tais procedimentos.
- Ligue o equipamento.
- Abra a câmara, coloque os recipientes de amostra dentro e adicione etanol 100% até cobrir os recipientes. Feche a câmara.
- Ative a opção Cool e aguarde até que a temperatura atinja 10 °C.
- Abra a válvula do cilindro de CO2 e ative a opção Agitador .
- Ative a opção CO2 in , sempre verificando a câmara para verificar quanto CO2 já a encheu. Quando a câmara estiver quase cheia, desative a opção CO2 in e ative a opção Exchange , mantendo-a ativada até que haja CO2 suficiente apenas para cobrir os recipientes. Certifique-se de que os recipientes estejam sempre cobertos com CO2 (ou seja, desative a opção Troca antes que todo o CO2 saia da câmara). Repita a etapa 5.5 6x.
- Ative o CO2 uma última vez e encha a câmara até que os recipientes estejam cobertos.
- Ative a opção Aquecer e desative a opção Agitador . Feche a válvula do cilindro de CO2.
- Aguarde até que a temperatura suba para 35 °C; em seguida, ative a opção Gas out .
NOTA: A cerca de 30 °C, a câmara atinge pressões de 70-80 bar, atingindo o ponto crítico, onde o líquido desaparece. - Quando a pressão da câmara atinge 1 bar, todo o conteúdo de gás foi removido. Abra a câmara e remova os recipientes.
- Desligue o equipamento.
6. Montagem
- Prepare suportes de amostra SEM (ou seja, canhotos de alumínio; Figura 2C.4).
- Enrole os tocos com um pedaço de papel alumínio, cobrindo apenas a parte superior, para facilitar a limpeza do topo após a análise.
- Identifique os canhotos escrevendo o código/número da amostra na parte inferior do suporte, garantindo a identificação do que é colocado na parte superior.
- Cubra a parte superior com fita de carbono dupla face. Coloque os tocos em um porta-amostras.
- Abra a tampa do recipiente de amostra e transfira cuidadosamente a amostra seca para uma placa de Petri de vidro usando uma pinça e uma espátula.
NOTA: Jardins secos de pontos críticos tendem a se aglomerar formando amostras altamente compactadas que devem ser cuidadosamente separadas como fragmentos não maiores que 1 mm3de tamanho. - Coloque cuidadosamente os fragmentos na superfície pegajosa do toco coberto com fita. Uma vez que o fragmento do jardim toca a fita, é muito difícil (re)movê-lo, portanto, tome cuidado para não colocá-lo em locais ou posições indesejadas. Adicione até nove fragmentos por esboço.
- Repita as etapas 6.2 e 6.3 para cada amostra.
7. Revestimento por pulverização catódica com ouro
NOTA: O revestimento da amostra é necessário para evitar seu carregamento. Siga as instruções do fabricante para ajustar as configurações, como a pressão do gás de operação (0.5 × 10-1 mm Hg de pressão do gás neste protocolo), o tempo de pulverização catódica (220 s), a espessura da camada de ouro (~120 Å), a corrente (50 mA) e o voltage alimentação. A pulverização catódica tende a seguir um fluxo de trabalho comum, embora os equipamentos de diferentes fabricantes possam operar de maneira ligeiramente diferente.
- Abra o braço articulado e coloque os tocos na mesa de amostras.
- Feche o braço alvo articulado e verifique se a proteção contra estilhaços da câmara de vácuo de vidro está devidamente encaixada.
- Abra a válvula do cilindro de argônio e ligue o interruptor de alimentação principal.
- Siga o vácuo subindo no visor do equipamento até atingir a marca de 0.5 × 10-1 mm Hg no visor; em seguida, ative o Enxágue. Repita a operação 5x.
- Ligue o sistema de circulação de água, ative a opção HV On e abra a tampa da película dourada. Confirme se a cor do plasma é rosada. Para seguir este protocolo, configure 220 s de pulverização catódica com tensão de 50 mA, que depositará uma camada de ouro de ~ 120 Å (12 nm).
- HV On é desligado automaticamente. Desligue o sistema de circulação de água e feche a tampa da película de ouro.
- Desligue o interruptor de alimentação principal, permitindo que o ar entre na câmara de vácuo. Para amostras de jardim e resíduos, repita as etapas 7.1-7.7 3x.
NOTA: Se a imagem não ocorrer imediatamente após a preparação da amostra, armazene os stubs em um recipiente hermético cheio de uma camada de sílica para evitar a reidratação das amostras.
8. Exames por imagem
NOTA: Siga as instruções do fabricante para ajustar as configurações de SEM, como diâmetro da abertura objetiva, tensão operacional, alinhamento do sistema de feixe de elétrons, alinhamento axial e estigmatizadores.
- Coloque os tocos no porta-amostras, anotando a posição de cada amostra.
NOTA: Use luvas ao inserir ou remover o sample porta-luvas e mantenha-o o mais limpo possível. - Inicie o software de operação na área de trabalho.
- Selecione as configurações do instrumento. Visualize amostras de jardim com o diâmetro de abertura da objetiva de 30 μm (ou seja, no segundo estágio), operando em alto vácuo, detectando sinais de elétrons secundários (SED), acelerando a tensão de 20 kV, distância de trabalho de 15 - 20 mm, corrente da sonda de 40,0 (no modo de alta corrente) e variando a ampliação de acordo com a amostra.
- Seguindo as instruções de navegação, pressione o ícone Vent e aguarde a ventilação da câmara de amostra. Uma barra de progresso indica o status do vácuo.
- Quando a pressão atmosférica for atingida, abra a câmara de amostra e insira cuidadosamente o porta-amostras.
- Feche suavemente a porta da câmara e pressione o ícone Evac para evacuar o instrumento, seguindo o status do vácuo através da barra de progresso. O sistema de navegação mostrará a posição do estágio móvel e fornecerá um gráfico de suporte quando o movimento for concluído. Pressione o ícone Câmera para gravar uma foto do suporte para obter uma visão superior que ajudará a navegar entre as amostras durante a geração de imagens.
- Pressione o ícone Observação para ligar o canhão de elétrons e aguarde a formação da imagem. Use a interface manual do usuário (controle manual) para mover manualmente o eixo Z para a altura adequada (com base na altura da amostra e na distância de trabalho determinada).
NOTA: Para prolongar a vida útil do filamento de tungstênio, pause a função Observação sempre que não estiver criando imagens ativamente. - Use as opções mostradas na tela ou o controle manual para alterar a taxa de varredura e a posição da imagem. Mova o palco para obter uma visão completa da amostra, empregando a função RDC para se concentrar em áreas específicas. Ao observar uma estrutura alvo (ou interessante), ajuste a ampliação, o foco, o brilho, o contraste e a estigmatização de acordo. Corrija a estigmatização usando a interface manual do usuário para mover o palco para as direções X e Y.
- Ajuste a ampliação entre 100x e 700x para visualizar aspectos gerais do jardim (como densidade de hifas, substrato e padrões de colonização).
- Ajuste a ampliação entre 700x e 1.500x para visualizar os padrões espaciais da microbiota.
- Ajuste a ampliação entre 1.500x e 3.000x para observar as interações físicas da microbiota e do biofilme.
- Ajuste a ampliação entre 3.000x e 4.000x para focar em aglomerados microbianos específicos.
- Para salvar uma imagem, use a função Congelar , clique no ícone Foto e configure o caminho do arquivo. Como padrão, analise pelo menos três fragmentos de jardim, criando imagens de cada um em todas as faixas de ampliação mencionadas na etapa 8.8. Isso resulta em pelo menos 12 imagens por amostra, embora mais imagens possam suportar descrições mais detalhadas. Sugerimos que 15-25 imagens por amostra, variando todas as ampliações na etapa 8.8, tendem a fornecer detalhes finos para a descrição da amostra.
NOTA: Rastejar e salvar uma imagem leva alguns segundos durante os quais qualquer vibração na mesa flutuante SEM deve ser evitada. - Depois de terminar a imagem, pressione a opção Ventilar para ventilar a câmara, seguindo o status do vácuo através da barra de progresso. Remova cuidadosamente o suporte, feche suavemente a porta da câmara e pressione Evac para evacuar a câmara.
- Assim que a evacuação estiver concluída, saia do software de operação.
- Ajuste o brilho e o contraste para melhorar a visualização usando um editor de imagens.
Resultados
Aqui, apresentamos um protocolo simplificado para visualizar os componentes do jardim attine e amostras de resíduos, como hifas fúngicas, substrato, microbiota e biofilmes. O SEM melhorou nossa compreensão de como o jardim e os resíduos sustentam os padrões estruturais da microbiota (Figura 3). Em jardins attine, as hifas fúngicas são estruturas semelhantes a galhos que cobrem porções da superfície do substrato. Como as hifas fúngicas tendem a ser...
Discussão
O SEM usa um feixe de elétrons para escanear a amostra, gerando uma imagem ampliada dela de forma que se possa visualizar microestruturas tridimensionais em alta resolução. Como o SEM opera sob alto vácuo, é necessária a remoção de até/mais de 99% da água das amostras. Dentro da câmara de vácuo MEV, amostras parcialmente hidratadas podem desidratar e colapsar, além de espalhar elétrons. Para imagens de alta resolução em MEV, a preparação da amostra deve incluir procedim...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Os autores agradecem à Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) pelo apoio financeiro (Processo #2019/03746-0). O MOB agradece a bolsa de doutorado recebida da FAPESP (processo 2021/08013-0) e da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - Brasil (CAPES) - Código de Finanças 001. A AR também agradece ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) pela bolsa de pesquisa (#305269/2018). Os autores gostariam de agradecer a Marcia Regina de Moura Aouada e Antonio Teruyoshi Yabuki por ajudarem nos testes piloto para preparação de amostras, a Renato Barbosa Salaroli pela assistência técnica e a Enzo Sorrentino por ajudar na sessão de fotos. Este estudo foi realizado sob a autorização de acesso ao patrimônio genético # SISGen AA39A6D.
Materiais
| Name | Company | Catalog Number | Comments |
| 2 mL tube | Axygen | MCT-200-C-BRA | To fix and dehydrate samples |
| Calcium chloride anhydrous | Merck | C4901 | CaCl2 anhydrous to prepare Karnovsky’s fixative |
| Critical point dryer | Leica | EM CPD 300 | For critical point drying |
| Double Sided Carbon Conductive Tape, 12 mm (W) X 5 M (L) | Electron Microscopy Sciences | 77819-12 | For mounting samples |
| Entomological forceps | No specific supplier | To manipulate garden samples | |
| Ethyl alcohol (=ethanol), pure (≥99.5%) | Sigma-Aldrich | 459836 | For dehydration |
| Forceps | No specific supplier | To manipulate garden samples | |
| Glass beaker | No specific supplier | For dehydration | |
| Glass Petri dish | No specific supplier | To manipulate garden samples | |
| Glass pipette | No specific supplier | To fix and dehydrate samples | |
| Glutaraldehyde (Aqueous Glutaraldehyde EM Grade 25%) | Electron Microscopy Sciences | 16220 | To prepare Karnovsky’s fixative |
| Gold target | Ted Pella, Inc. | 8071 | To sputter coat with gold |
| Hydrochloric acid | Sigma-Aldrich | 320331 | For adjusting solutions pH |
| Image editor | Photoshop | any version | To adjust images |
| Paraformaldehyde (Paraformaldehyde 20% Aqueous Solution EM Grade) | Electron Microscopy Sciences | 15713 | To prepare Karnovsky’s fixative |
| Propilene recipient | No specific supplier | For maintaining alive ant colonies | |
| Scanning Electron Microscope | JEOL | IT300 SEM | For sample imaging |
| Sodium cacodylate trihydrate | Sigma-Aldrich | C0250 | For preparing sodium cacodylate buffer |
| Spatula | No specific supplier | To manipulate garden samples | |
| Specimen containers with 15 mm dia. x 10 mm high | Ted Pella, Inc. | 4591 | For critical point drying |
| Sputter coater | Baltec | SCD 050 | To coat with gold |
| Stub (Aluminium mount, flat end pin) 12.7 mm x 8 mm | Electron Microscopy Sciences | 75520 | For mounting samples |
Referências
- Turner, M. G. Landscape ecology: the effect of pattern on process. Annu Rev Ecol Evol Syst. 20 (1), 171-197 (1989).
- Rietkerk, M., Van de Koppel, J. Regular pattern formation in real ecosystems. Trends Ecol Evol. 23 (3), 169-175 (2008).
- Schmitz, O. J. Spatial dynamics and ecosystem functioning. PLOS Biol. 8 (5), e1000378 (2010).
- Pringle, R. M., Doak, D. F., Brody, A. K., Jocqué, R., Palmer, T. M. Spatial pattern enhances ecosystem functioning in an African savanna. PLOS Biol. 8 (5), e1000377 (2010).
- Wimpenny, J. W. Spatial order in microbial ecosystems. Biol Rev. 56 (3), 295-342 (1981).
- Martiny, J. B. H., et al. Microbial biogeography: putting microorganisms on the map. Nat Rev. Microbiol. 4 (2), 102-112 (2006).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nat Rev Microbiol. 22 (2), 105-118 (2024).
- Lamont, R. J., Hajishengallis, G., Koo, H. Social networking at the microbiome-host interface. Infec Immun. 91 (9), e00124-e00223 (2023).
- Welch, J. L. M., Rossetti, B. J., Rieken, C. W., Dewhirst, F. E., Borisy, G. G. Biogeography of a human oral microbiome at the micron scale. Proc Natl Acad Sci USA. 113 (6), E791-E800 (2016).
- Kim, D., et al. Spatial mapping of polymicrobial communities reveals a precise biogeography associated with human dental caries. Proc Natl Acad Sci USA. 117 (22), 12375-12386 (2020).
- Swidsinski, A., Weber, J., Loening-Baucke, V., Hale, L. P., Lochs, H. Spatial organization and composition of the mucosal flora in patients with inflammatory bowel disease. J Clin Microbiol. 43 (7), 3380-3389 (2005).
- Ibberson, C. B., Barraza, J. P., Holmes, A. L., Cao, P., Whiteley, M. Precise spatial structure impacts antimicrobial susceptibility of S. aureus in polymicrobial wound infections. Proc Natl Acad Sci USA. 119 (51), e2212340119 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Dejea, C. M., et al. Microbiota organization is a distinct feature of proximal colorectal cancers. Proc Natl Acad Sci USA. 111 (51), 18321-18326 (2014).
- Hanson, C. A., Fuhrman, J. A., Horner-Devine, M. C., Martiny, J. B. Beyond biogeographic patterns: processes shaping the microbial landscape. Nat Rev Microbiol. 10 (7), 497-506 (2012).
- Adade, E. E., Al Lakhen, K., Lemus, A. A., Valm, A. M. Recent progress in analyzing the spatial structure of the human microbiome: Distinguishing biogeography and architecture in the oral and gut communities. Curr Opin Endocr. 18, 275-283 (2021).
- Mony, C., Bohannan, B. J., Leibold, M. A., Peay, K., Vandenkoornhuyse, P. Microbial landscape ecology: Highlights on the invisible corridors. Front Ecol Evol. 9, 753213 (2021).
- Hölldobler, B., Wilson, E. O. . The Ants. , (1990).
- Chapela, I. H., Rehner, S. A., Schultz, T. R., Mueller, U. G. Evolutionary history of the symbiosis between fungus-growing ants and their fungi. Science. 266 (5191), 1691-1694 (1994).
- Mueller, U. G., Rehner, S. A., Schultz, T. R. The evolution of agriculture in ants. Science. 281 (5385), 2034-2038 (1998).
- Mueller, U. G., Gerardo, N. M., Aanen, D. K., Six, D. L., Schultz, T. R. The evolution of agriculture in insects. Annu Rev Ecol Evol Syst. 36, 563-595 (2005).
- Dentinger, B. T., Lodge, D. J., Munkacsi, A. B., Desjardin, D. E., McLaughlin, D. J. Phylogenetic placement of an unusual coral mushroom challenges the classic hypothesis of strict coevolution in the Apterostigma pilosum group ant-fungus mutualism. Evolution. 63 (8), 2172-2178 (2009).
- Schultz, T. R., Brady, S. G. Major evolutionary transitions in ant agriculture. Proc Natl Acad Sci USA. 105, 5435-5440 (2008).
- de Fine Licht, H. H., Boomsma, J. J. Forage collection, substrate preparation, and diet composition in fungus-growing ants. Ecol Entomol. 35 (3), 259-269 (2010).
- Martin, M. M. The biochemical basis of the fungus-attine ant symbiosis: A complex symbiosis is based upon integration of the carbon and nitrogen metabolisms of the two organisms. Science. 169 (3940), 16-20 (1970).
- Grell, M. N., et al. The fungal symbiont of Acromyrmex leaf-cutting ants expresses the full spectrum of genes to degrade cellulose and other plant cell wall polysaccharides. BMC Genomics. 14, 928 (2013).
- Lange, L., Grell, M. N. The prominent role of fungi and fungal enzymes in the ant-fungus biomass conversion symbiosis. Appl Microbiol Biotechnol. 98, 4839-4851 (2014).
- Huang, E. L., et al. The fungus gardens of leaf-cutter ants undergo a distinct physiological transition during biomass degradation. Environ Microbiol Rep. 6 (4), 389-395 (2014).
- Craven, S. E., Dix, M. W., Michaels, G. E. Attine fungus gardens contain yeasts. Science. 169 (3941), 184-186 (1970).
- De Fine Licht, H. H., Boomsma, J. J., Tunlid, A. Symbiotic adaptations in the fungal cultivar of leaf-cutting ants. Nat Commun. 5 (1), 5675 (2014).
- Aylward, F. O., et al. Enrichment and broad representation of plant biomass-degrading enzymes in the specialized hyphal swellings of Leucoagaricus gongylophorus, the fungal symbiont of leaf-cutter ants. PLoS One. 10 (8), e0134752 (2015).
- Moreira-Soto, R. D., Sanchez, E., Currie, C. R., Pinto-Tomás, A. A. Ultrastructural and microbial analyses of cellulose degradation in leaf-cutter ant colonies. Microbiology. 163 (11), 1578-1589 (2017).
- Erthal Jr, M., Silva, C. P., Cooper, R. M., Samuels, R. I. Hydrolytic enzymes of leaf-cutting ant fungi. Comp Biochem. 152 (1), 54-59 (2009).
- North, R. D., Jackson, C. W., Howse, P. E. Communication between the fungus garden and workers of the leaf-cutting ant, Atta sexdens rubropilosa, regarding choice of substrate for the fungus. Physiol Entomol. 24 (2), 127-133 (1999).
- Herz, H., Hölldobler, B., Roces, F. Delayed rejection in a leaf-cutting ant after foraging on plants unsuitable for the symbiotic fungus. Behav Ecol. 19 (3), 575-582 (2008).
- Schiøtt, M., De Fine Licht, H. H., Lange, L., Boomsma, J. J. Towards a molecular understanding of symbiont function: identification of a fungal gene for the degradation of xylan in the fungus gardens of leaf-cutting ants. BMC Microbiol. 8, 40 (2008).
- Bot, A. N., Currie, C. R., Hart, A. G., Boomsma, J. J. Waste management in leaf-cutting ants. Ethol Ecol Evol. 13 (3), 225-237 (2001).
- Scott, J. J., et al. Microbial community structure of leaf-cutter ant fungus gardens and refuse dumps. PLOS One. 5 (3), e9922 (2010).
- Lewin, G. R., et al. Cellulose-enriched microbial communities from leaf-cutter ant (Atta colombica) refuse dumps vary in taxonomic composition and degradation ability. PLOS One. 11 (3), e0151840 (2016).
- Suen, G., et al. An insect herbivore microbiome with high plant biomass-degrading capacity. PLOS Genet. 6 (9), e1001129 (2010).
- Aylward, F. O., et al. Convergent bacterial microbiotas in the fungal agricultural systems of insects. MBio. 5 (6), e02077 (2014).
- Barcoto, M. O., et al. Fungus-growing insects host a distinctive microbiota apparently adapted to the fungiculture environment. Sci Rep. 10 (1), 12384 (2020).
- Francoeur, C. B., et al. Bacteria contribute to plant secondary compound degradation in a generalist herbivore system. mBio. 11, e02146-e02220 (2020).
- Martiarena, M. J. S., Deveau, A., Montoya, Q. V., Flórez, L. V., Rodrigues, A. The hyphosphere of leaf-cutting ant cultivars is enriched with helper bacteria. Microb Ecol. 86 (3), 1773-1788 (2023).
- Leal-Dutra, C. A., et al. Evidence that the domesticated fungus Leucoagaricus gongylophorus recycles its cytoplasmic contents as nutritional rewards to feed its leafcutter ant farmers. IMA Fungus. 14 (1), 19 (2023).
- Sosa-Calvo, J., Jesovnik, A., Okonski, E., Schultz, T. R. Locating, collecting, and maintaining colonies of fungus-farming ants (Hymenoptera: Myrmicinae: Attini). Sociobiology. 62 (2), 300-320 (2015).
- Karnovsky, M. J. A formaldehyde glutaraldehyde fixative of high osmolality for use in electron microscopy. J Cell Biol. 27, 137-138 (1965).
- Eltoum, I., Fredenburgh, J., Myers, R. B., Grizzle, W. E. Introduction to the theory and practice of fixation of tissues. J Histotechnol. 24 (3), 173-190 (2001).
- Gusnard, D., Kirschner, R. H. Cell and organelle shrinkage during preparation for scanning electron microscopy: effects of fixation, dehydration and critical point drying. J Microsc. 110 (1), 51-57 (1977).
- Surman, S. B., et al. Comparison of microscope techniques for the examination of biofilms. J Microbiol Methods. 25 (1), 57-70 (1996).
- Augustin, J. O., et al. Yet more "weeds" in the garden: fungal novelties from nests of leaf-cutting ants. PLOS One. 8 (12), e82265 (2013).
- Montoya, Q. V., Martiarena, M. J. S., Polezel, D. A., Rodrigues, A. More pieces to a huge puzzle: Two new Escovopsis species from fungus gardens of attine ants. MycoKeys. 46, 97 (2019).
- Varanda-Haifig, S. S., et al. Nature of the interactions between hypocrealean fungi and the mutualistic fungus of leaf-cutter ants. Antonie van Leeuwenhoek. 110, 593-605 (2017).
- Custodio, B. C., Rodrigues, A. Escovopsis kreiselii specialization to its native hosts in the fungiculture of the lower attine ant Mycetophylax morschi. Antonie van Leeuwenhoek. 112, 305-317 (2019).
- Schröttner, H., Schmied, M., Scherer, S. Comparison of 3D surface reconstruction data from certified depth standards obtained by SEM and an Infinite Focus Measurement Machine (IFM). Microchim Acta. 155, 279-284 (2006).
- Zhou, W., Apkarian, R., Wang, Z. L., Joy, D., Zhou, W., Wang, Z. L. Fundamentals of scanning electron microscopy (SEM). Scanning microscopy for nanotechnology: Techniques and applications. , 1-40 (2007).
- Kannan, M., Raja, K., Subramanian, K. S., Kannan, M. Scanning electron microscopy: Principle, components and applications. A Textbook on Fundamentals and Applications of Nanotechnology. , 81-92 (2018).
- Ruffolo, J. J. Critical point drying of protozoan cells and other biological specimens for scanning electron microscopy: apparatus and methods of specimen preparation. Trans Am Microsc. 93 (1), 124-131 (1974).
- Echlin, P. Recent advances in specimen coating techniques for electron microscopy. Scanning Electron Microscopy 1981/1. , 79-90 (1981).
- Ris, H. The cytoplasmic filament system in critical point-dried whole mounts and plastic-embedded sections. J Cell Biol. 100 (5), 1474-1487 (1985).
- Bray, D. F., Bagu, J., Koegler, P. Comparison of hexamethyldisilazane (HMDS), Peldri II, and critical-point drying methods for scanning electron microscopy of biological specimens. Microsc Res Tech. 26 (6), 489-495 (1993).
- Bergmans, L., Moisiadis, P., Van Meerbeek, B., Quirynen, M., Lambrechts, P. Microscopic observation of bacteria: review highlighting the use of environmental SEM. Int Endod J. 38 (11), 775-788 (2005).
- Kirk, S. E., Skepper, J. N., Donald, A. M. Application of environmental scanning electron microscopy to determine biological surface structure. J Microsc. 233 (2), 205-224 (2009).
- Kemmenoe, B. H., Bullock, G. R. Structure analysis of sputter-coated and ion-beam sputter-coated films: a comparative study. J Microsc. 132 (2), 153-163 (1983).
- Kinden, D. A., Brown, M. F. Technique for scanning electron microscopy of fungal structures within plant cells. Phytopathology. 65, 74-76 (1975).
- Masaphy, S., Levanon, D., Tchelet, R., Henis, Y. Scanning electron microscope studies of interactions between Agaricus bisporus (Lang) Sing hyphae and bacteria in casing soil. Appl Environ Microbiol. 53 (5), 1132-1137 (1987).
- Massicotte, H. B., Melville, L. H., Peterson, R. L. Scanning electron microscopy of ectomycorrhizae potential and limitations. Scanning Microsc. 1 (3), 58 (1987).
- Visen, A., Singh, P. N., Chakraborty, B., Singh, A., Bisht, T. S. Scanning electron microscopy indicates Pseudomonad strains facilitate AMF mycorrhization in litchi (Litchi chinensis Sonn.) air-layers and improving survivability, growth and leaf nutrient status. Curr Res Microb Sci. 2, 100063 (2021).
- Fleeman, R. M., Mikesh, M., Davies, B. W. Investigating Klebsiella pneumoniae biofilm preservation for scanning electron microscopy. Access Microbiol. 5 (2), 000470-000473 (2023).
- Wells, M., Mikesh, M., Gordon, V. Structure-preserving fixation allows scanning electron microscopy to reveal biofilm microstructure and interactions with immune cells. J Microsc. 293 (1), 59-68 (2024).
- Nadell, C. D., Drescher, K., Foster, K. R. Spatial structure, cooperation and competition in biofilms. Nat Rev Microbiol. 14 (9), 589-600 (2016).
- Madsen, J. S., et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities. Environ Microbiol. 18 (8), 2565-2574 (2016).
- Madsen, J. S., Sørensen, S. J., Burmølle, M. Bacterial social interactions and the emergence of community-intrinsic properties. Curr Opin Microbiol. 42, 104-109 (2018).
- Flemming, H. C., Wuertz, S. Bacteria and archaea on Earth and their abundance in biofilms. Nat Rev Microbiol. 17 (4), 247-260 (2019).
- Bringhurst, B., Allert, M., Greenwold, M., Kellner, K., Seal, J. N. Environments and hosts structure the bacterial microbiomes of fungus-gardening ants and their symbiotic fungus gardens. Microb Ecol. 86 (2), 1374-1392 (2023).
- Bringhurst, B., Greenwold, M., Kellner, K., Seal, J. N. Symbiosis, dysbiosis and the impact of horizontal exchange on bacterial microbiomes in higher fungus-gardening ants. Sci Rep. 14 (1), 3231 (2024).
- Vargas, S., et al. Body-plan reorganization in a sponge correlates with microbiome change. Mol Biol Evol. 40 (6), msad138 (2023).
- Apprill, A., et al. Humpback whale populations share a core skin bacterial community: towards a health index for marine mammals. PLOS One. 9 (3), e90785 (2014).
- Fraune, S., et al. Bacteria-bacteria interactions within the microbiota of the ancestral metazoan Hydra contribute to fungal resistance. ISME J. 9 (7), 1543-1556 (2015).
- Gowen, R., Gamal, A., Di Martino, L., McCormick, T. S., Ghannoum, M. A. Modulating the microbiome for Crohn's disease treatment. Gastroenterology. 164 (5), 828-840 (2023).
- Moya, P., et al. Molecular phylogeny and ultrastructure of the lichen microalga Asterochloris mediterranea sp. nov. from Mediterranean and Canary Islands ecosystems. Int J Syst Evol Microbiol. 65 (6), 1838-1854 (2015).
- Zettler, E. R., Mincer, T. J., Amaral-Zettler, L. A. Life in the "plastisphere": microbial communities on plastic marine debris. Environ Sci Technol. 47 (13), 7137-7146 (2013).
- Porter, K. R., Kallman, F. The properties and effects of osmium tetroxide as a tissue fixative with special reference to its use for electron microscopy. Exp Cell Res. 4 (1), 127-141 (1953).
- Ligon, J. J., Abraham, J. L., Boyd, A. S. Traumatic osmium tetroxide inoculation. J Am Acad Dermatol. 45 (6), 949-952 (2001).
- Friedova, N., et al. Osmium absorption after osmium tetroxide skin and eye exposure. Basic Clin Pharmacol Toxicol. 127 (5), 429-433 (2020).
- Fratesi, S. E., Lynch, F. L., Kirkland, B. L., Brown, L. R. Effects of SEM preparation techniques on the appearance of bacteria and biofilms in the Carter Sandstone. J Sediment. 74 (6), 858-867 (2004).
- Dassanayake, R. P., et al. Identification of a reliable fixative solution to preserve the complex architecture of bacterial biofilms for scanning electron microscopy evaluation. PLOS One. 15 (5), e0233973 (2020).
- Desiro, A., et al. Detection of a novel intracellular microbiome hosted in arbuscular mycorrhizal fungi. ISME J. 8 (2), 257-270 (2014).
- Morales, D. P., et al. Advances and challenges in fluorescence in situ hybridization for visualizing fungal endobacteria. Front Microbiol. 13, 892227 (2022).
- Shi, H., et al. Highly multiplexed spatial mapping of microbial communities. Nature. 588 (7839), 676-681 (2020).
- Valm, A. M., Welch, J. L. M., Borisy, G. G. CLASI-FISH: principles of combinatorial labeling and spectral imaging. Syst Appl Microbiol. 35 (8), 496-502 (2012).
- Fernandez-Brime, S., Muggia, L., Maier, S., Grube, M., Wedin, M. Bacterial communities in an optional lichen symbiosis are determined by substrate, not algal photobionts. FEMS Microbiol Ecol. 95 (3), fiz012 (2019).
- Schluter, S., Eickhorst, T., Mueller, C. W. Correlative imaging reveals holistic view of soil microenvironments. Environ Sci Technol. 53 (2), 829-837 (2018).
- Barcoto, M. O., Rodrigues, A. Lessons from insect fungiculture: from microbial ecology to plastics degradation. Front Microbiol. 13, 812143 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados