Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Одной частицы электронной микроскопии Реконструкция комплекса Exosome Использование случайных Коническая Метод Tilt
В этой статье
Резюме
В этой статье описывается стандартный метод для получения трехмерных (3D) реконструкция биологических макромолекул с использованием отрицательных электронной микроскопии окрашивания (EM). В этом протоколе, мы объясняем, как получить 3D-структуры exosome комплекс CEREVISIAE Saccharomyces на среднего разрешения с использованием случайного конической метод реконструкции наклона (РКИ).
Аннотация
Single particle electron microscopy (EM) reconstruction has recently become a popular tool to get the three-dimensional (3D) structure of large macromolecular complexes. Compared to X-ray crystallography, it has some unique advantages. First, single particle EM reconstruction does not need to crystallize the protein sample, which is the bottleneck in X-ray crystallography, especially for large macromolecular complexes. Secondly, it does not need large amounts of protein samples. Compared with milligrams of proteins necessary for crystallization, single particle EM reconstruction only needs several micro-liters of protein solution at nano-molar concentrations, using the negative staining EM method. However, despite a few macromolecular assemblies with high symmetry, single particle EM is limited at relatively low resolution (lower than 1 nm resolution) for many specimens especially those without symmetry. This technique is also limited by the size of the molecules under study, i.e. 100 kDa for negatively stained specimens and 300 kDa for frozen-hydrated specimens in general.
For a new sample of unknown structure, we generally use a heavy metal solution to embed the molecules by negative staining. The specimen is then examined in a transmission electron microscope to take two-dimensional (2D) micrographs of the molecules. Ideally, the protein molecules have a homogeneous 3D structure but exhibit different orientations in the micrographs. These micrographs are digitized and processed in computers as "single particles". Using two-dimensional alignment and classification techniques, homogenous molecules in the same views are clustered into classes. Their averages enhance the signal of the molecule's 2D shapes. After we assign the particles with the proper relative orientation (Euler angles), we will be able to reconstruct the 2D particle images into a 3D virtual volume.
In single particle 3D reconstruction, an essential step is to correctly assign the proper orientation of each single particle. There are several methods to assign the view for each particle, including the angular reconstitution1 and random conical tilt (RCT) method2. In this protocol, we describe our practice in getting the 3D reconstruction of yeast exosome complex using negative staining EM and RCT. It should be noted that our protocol of electron microscopy and image processing follows the basic principle of RCT but is not the only way to perform the method. We first describe how to embed the protein sample into a layer of Uranyl-Formate with a thickness comparable to the protein size, using a holey carbon grid covered with a layer of continuous thin carbon film. Then the specimen is inserted into a transmission electron microscope to collect untilted (0-degree) and tilted (55-degree) pairs of micrographs that will be used later for processing and obtaining an initial 3D model of the yeast exosome. To this end, we perform RCT and then refine the initial 3D model by using the projection matching refinement method3.
протокол
1. Принцип метода случайных Коническая Tilt
- Принцип случайной конической метод наклона требует принятия пару микрофотографии же области образца внутри электронного микроскопа. Одна картина берется из образца в untilted положение (рис. 1А) и другие съемки образца под углом от 50 до 70 градусов (в нашем случае, мы используем 55 градусов). (Рис. 1В)
- Использование компьютера, оцифрованных пару микрофотография ставится рядом друг с другом и образы из тех же частиц будут выбраны. (Рис. 1C)
- В трехмерных координат, образы untilted частиц и их партнеров наклонены коррелируют друг с другом направлении оси наклона и угла наклона. (Рис. 1D)
- Выравнивание untilted изображения частиц приносит образы наклонена частиц с соответствующими азимутальным местах. (Рис. 1E)
- Использование нескольких изображений наклонных частиц, заполняющих пространство азимутальная, трехмерная структура молекулы может быть восстановлена с использованием резервного алгоритма проекции. (Рис. 1F)
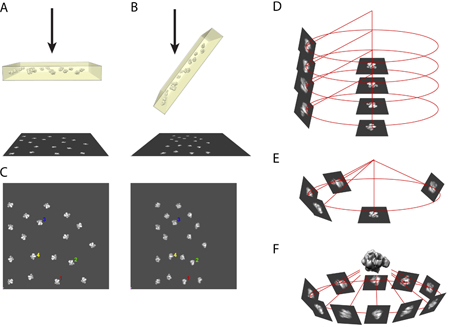
Рисунок 1. Иллюстрацией принципа РКИ реконструкции.
2. Подготовка Сетки Холей Carbon покрыты тонким Carbon
Обоснование: Мы используем отрицательные метод окрашивания исправить белка образец для случайных конической реконструкция наклона. В целях сохранения макромолекул без особых уплощение во время сушки, мы стараемся внедрить белковых молекул в глубоком пятно толщиной около измерении белков 4. В общем, непрерывное углерод используется в создании негативно окрашенных препаратов. Такой вид углерода, однако, трудно контролировать пятно толщиной около белковых частиц. Таким образом, использование самодельных дырявые углерода сетки покрыта тонким слоем пленки углерода (~ 5 нм толщиной), чтобы негативно окрашенных препаратов. Маленькая ямах, образованных отверстия позволяют сохранить решение белка и пятна раствора на сетку так гораздо проще встроить белка в оптимальное пятно толщиной. Кроме того, тонкий слой углерода над отверстием уменьшает фоновый шум значительно.
- Подготовка 0,5% формвар решение. В вытяжной шкаф, добавить 0,45 г поливинилового формальных смолы и 90 мл хлороформа в 100 мл стакан стекло. Использование алюминиевой фольги, чтобы покрыть стакан, и использовать небольшой бар движение, чтобы помочь растворения смолы на формальные магнитной мешалкой. Она занимает около 15 минут для растворения смолы.
- Во время роспуска формвар, чистые стекла микроскопа слайды в метаноле и вытереть насухо Kimwipes.
- После формвар смола полностью растворяется в хлороформе, добавляют 1 мл 50% глицерина на поверхности раствора. Регулировка громкости добавил глицерина влияет на плотность отверстий на углерод дырявые. Опустите кончик ultrasonicator в растворе около 1 дюйма глубиной и использования максимальной мощности, чтобы разрушать ультразвуком 1 мин, чтобы сделать эмульсию глицерина капель в растворе формвар. Решение становится молочно после этого шага. Более ультразвуком причинами меньших размеров отверстий на углерод дырявые.
- Сразу же после обработки ультразвуком, окуните очищать стеклянные пластинки вертикально в эмульсию в течение 1 секунды, вынуть их, и промокните днища слайдов с использованием фильтровальной бумаги для формирования тонкой пластиковой пленкой по поверхности горки. После испаряется choloroform, проверьте плотность и размер отверстия в фильме под световым микроскопом. Отрегулируйте подготовке условий в соответствии с потребностями. При условии, описанные здесь, мы в целом получить отверстия диаметром 3 ~ 4 микро-м и 10 ~ 20 отверстий в каждом квадрате 400 меш сетку.
- После стеклах сухие, вырезать края пластиковой пленки на поверхности слайда. Float фильм прочь на поверхности дистиллированной воды. Тонкая пленка на поверхности воды можно наблюдать на угол скольжения против отражения света. Место 400-сетка медных сеток на фильм один за другим, с сетками «гладкой поверхности, обращенной вниз.
- Возьмите пластиковую пленку с сетками на это с помощью листа бумаги. Переверните бумагу и дайте ему высохнуть в чашке Петри. Замочите бумагу в метаноле, чтобы удалить остаточную глицерина в отверстия и пусть бумаге высохнуть на воздухе.
- Пальто сетки с слоя углерода толщиной ~ 20 нанометров в углерод испарителя. Толщина может быть определено серый цвет углерода.
- Замочите сетки с углеродом в хлороформе в течение получаса, чтобы удалить формвар. После сетках сушат, мы получили самодельные сетки дырявые углерода.
- Evaporate тонкий слой углерода около 5 нанометров в толщину на свежем сколе поверхности слюды.
- Осторожно введите сетки дырявые углерода под дистиллированной воды. Float тонкие углерода с поверхности слюды на поверхности воды и поместить его на углерод дырявыесетками медленно. Сухой сеток в вытяжном шкафу.
3. Отрицательные Окрашивание Exosome комплекс
Обоснование: Есть немало тяжелых металлов пятно решений, которые могут быть использованы для отрицательных EM окрашивания, в том числе уранил ацетат, формиат уранила, фосфорно-вольфрамовой кислоты, молибдата аммония и других. Различные пятна решение имеет свои уникальные свойства. Например, уранилацетата обеспечивает высокую контрастность частицы, но может произойти сбой белковые комплексы, которые не любят кислую среду. Для тех образцов, phosphotungestic кислоты при нейтральном рН может быть хорошей окраски раствора. Мы выбираем насыщенные уранила Формиат (UF) решение из-за его тонкой детализации и высокой способностью проникновения в молекулах.
- Отварите дистиллированной воде в течение 1 мин. Охладить медленно до комнатной температуры. Этот шаг для удаления растворенного кислорода из воды.
- Сделать свежее 2% уранила Формиат (UF) решение. Смешайте 1 мл воды и 20 мг UF в 1,5 мл трубки. Vortex в течение 10 мин.
- Отрегулируйте рН до 5,0 путем добавления 2 мкл 10 М гидроксида калия. Смешайте немедленно. Цветовое решение должно быть более желтым. РН раствора не должна быть слишком высокой, иначе пятно осадков.
- Положите трубку на vortexer в течение еще 10 мин.
- Спиновые решение на настольных центрифуг на максимальной скорости в течение 10 мин.
- Фильтр решение через мембраны 0,2 микрометра PVDF. Это свежий раствор UF. Обложка решение трубки в кусок алюминиевой фольгой, чтобы свет. Решение должно быть использовано в тот же день.
- Тлеющий разряд один тонкий углерода по сравнению с аналогичным дырявые углерода сетке использовании тлеющего разряда аппарата в течение 30 секунд при 25 мА.
- Положите кусок чистой парафильмом на скамейке запасных. Поместите 3 капли на 50 мкл UF окраски раствора на вершине парафильмом.
- Развести exosome комплекса концентрации 50 ~ 100 нм, используя разбавления буфера (25 мМ Трис-HCl, рН 7,5, 100 мМ NaCl, 2 мМ DTT). Введите 4 мкл разбавленного белка на тлеющего выписан сетки. Пусть образец остаться на сетке в течение одной минуты. (Примечание:. Такая конечная концентрация молекул как правило, дает оптимальную плотность хорошо дисперсных частиц на отрицательно окрашенных сетки для формиата уранила или уранила acecate пятно раствор, фосфат или высокой концентрации соли (более 0,5 м) в целом не хорошо для получения хороших результатов окрашивания. Наш опыт показывает, что Hepes или трубы хорошо работать с уранил пятно решений.)
- Используйте кусок фильтровальной бумаги, чтобы уничтожить остаточные решение от края сетки и флип сетку сразу на вершине пятна капель и промыть сетку в течение приблизительно 10 секунд на каждую каплю.
- После последнего полоскания, не говоря пятна остаются на сетке еще 1 мин, а затем промокните пятно езды на кусочек фильтровальной бумаги. Держите тонким слоем пятна раствора на поверхности сетки, чтобы получить хороший глубокий пятно результаты. Пусть сетка быстро высыхают в вытяжном шкафу.
4. Электронная микроскопия Exosome комплекс
Обоснование: Любые просвечивающего электронного микроскопа с наклоном этапе может быть использована для сбора наклона пар образцов для РКИ реконструкции. В теории, высокий угол образца может быть наклонена для сбора данных, тем лучше. На практике, из-за дизайна держателя образца и геометрия сетки, максимальный угол рабочем ограничено с 50 до 70 градусов. В этом протоколе, мы только описать наши процедуры с использованием FEI Tecnai-12 электронного микроскопа. Для других моделей микроскопов, операции должны быть скорректированы в соответствии с требованиями проекта и имущества инструмента.
- Поместите образец сетки в держатель образца, а затем положить держатель в FEI Tecnai -12 электронного микроскопа. Микроскоп работает при 120 кВ. Мы используем Gatan Ultrascan4000 ПЗС-камера для съемки фотографий. Убедитесь, что 'Flip вокруг вертикальной оси "в диалоге" конфигурации камеры цифровой интерфейс Микрофотография снят для обеспечения правильного определения хиральности. (Примечание:. Это важно, особенно если читатель полагается на СПАЙДЕР РКИ процедуры реконструкции, чтобы получить 3D-модель)
- Проверьте образец сетки при малом увеличении, чтобы найти лучший окрашенных квадратов. Такого рода квадраты должны иметь десятков отверстий размером около 1 ~ 2 мкм и темное пятно областей в них. (Рис. 2)

Рисунок 2. Низкой микрофотография увеличением сетки показывает квадраты с хорошими и плохими пятен. - Включите режим пониженного дозу интерфейс пользователя FEI и выровнять поиска, фокус и выставить положение в режиме низкой дозе. Мы используем увеличениях 150000 для фокусировки, 52000 по выявлению и 1,5 метра длиной камеры при дифракции для поиска. Время экспозиции регулируется до 1 секунды. Установить фокус положение 2 микрометра от воздействия позition вдоль наклона оси.
- Найти отверстия с хорошим окрашивания в режиме поиска и сохранения мест. Хорошие отверстия, как правило, темное пятно градиент в них наблюдается в режиме поиска. Сфотографируйте ПЗС площади. Tilt образца до 55 градусов, принять другую картину. Сравните две фотографии, определить парные отверстия в две микрофотографии. (Рис. 3)
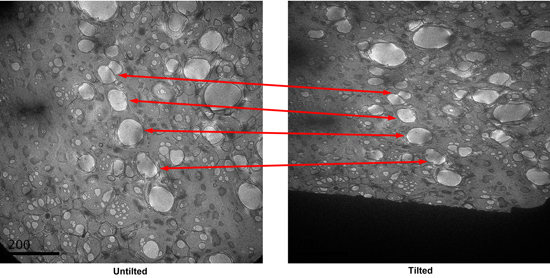
Рисунок 3. Наклона пару микрофотографии квадратный в режиме поиска. Корреспондент парные отверстия указаны. - Tilt этапе до 0 градусов. Использование малых доз комплект сфотографировать каждую лунку, определенных в режиме поиска при большом увеличении. Расфокусировать использовали около -0,7 микрометра.
- После того как все отверстия были взяты фотографии, наклон этапе до 55 градусов. Возьмите микрофотографии образца наклонена в то же увеличение, как те, с untilted расфокусировать около -1,2 микрометра. Определить соответствующий наклон пары микрофотографии на основе модели в низкий микрофотографии увеличения. Нерегулярные структуры самодельные дырявые сетки углерода помогает корреляции. Изучить наклона пар микрофотографии и удалить снимки с плохим пятна, такие как мелкие запятнанные области (частиц, похоже, ореол вокруг их под наклоном состоянии). (Рис. 4)
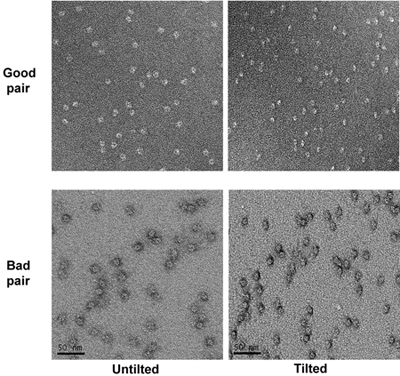
Рисунок 4. Два наклона пар микрофотографии образцов при большом увеличении. Хорошие и плохие микрофотографии выделены.
5. Обработка изображения из данных
Обоснование: Есть разные варианты и пакетов программного обеспечения для выполнения реконструкции РКИ в компьютере. Наиболее широко используемых СПАЙДЕР 5. Основной протокол для выполнения РКИ в СПАЙДЕР можно найти в веб-страницу http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Подробный протокол для выполнения РКИ в СПАЙДЕР описан в статье Шейх и др. 6. В нашем протокол, мы используем сочетание IMAGIC-5 7 и СПАЙДЕР в видео версию протокола. Мы также предоставляем альтернативную процедуру для использования исключительно СПАЙДЕР в текстовую версию протокола.
- Установка программ. Мы используем proc2d в EMAN 8 пакет изменить формат изображения с картинки Gatan Цифровой формат изображения для СПАЙДЕР. IMAGIC-5 используется для выполнения 2D выравнивания. СПАЙДЕР используется для выполнения 3D-реконструкции и утонченности.
Подраздел 1: Выбор наклона пар частиц.
- Преобразование *. дм3 Gatan цифровых изображений для СПАЙДЕР формат изображения использованием proc2d команду в EMAN. Наклонена и untilted пар названы в схеме, t.spi *** и *** u.spi, соответственно.
- Выберите пар частиц с помощью программы "паутина" распространяется в пакете программ СПАЙДЕР. Следуйте инструкциям в Координаты автоматически сохраняются в DCU ***. SPI и DCT ***. SPI для untilted и наклоняться частиц соответственно. Еще один документ, DCB ***. SPI содержит информацию наклона угол между наклонной и untilted частиц (Примечание:. Три угла во время примерки в WEB между наклона пар не гарантируют правильную направленность окончательной реконструкции, потому тета не имеет знак (положительное значение) и фи-и гамма полагаться на начальные значения, установленные для них. Рассматривая направления расфокусировки градиент на наклонном микрофотография поможет установить правильный начальные значения фи-и гамма до установки, чтобы получить правильную беспристрастности по реконструкции. Рисунок 5 иллюстрирует правильное конвенции.)
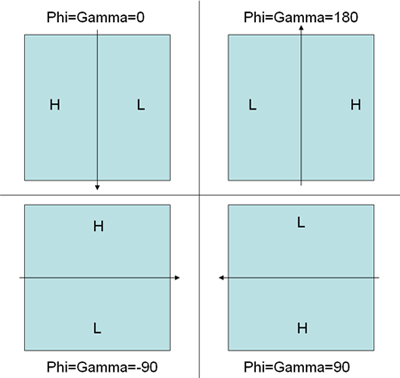
Рисунок 5. Иллюстрацией объяснить конвенции угла определение в WEB наклона пары частица выбора установки режима определяется градиент расфокусировать. H представляет высокого расфокусировать области в то время как L представляет низким расфокусировать области. Стрелка представляет наклон оси. Соответствующие правильные начальные углы PHI и GAMMA указаны для каждой схемы. - Коробка все взял частиц с использованием модифицированной версии сценария СПАЙДЕР, как показано на веб-странице http://www.wadsworth.org/spider_doc/spider/docs/partpick.html . Сценарий сохраняет untilted и наклоняться частиц стеки как u.spi и t.spi. Количество микрофотографии, где частицы от должны быть сохранены в particle_list.spi. (Примечание: Это очень важно для создания правильного угла Эйлера файлыRCT.)
Подраздел 2: Двумерная выравнивание и классификация untilted изображения частицы.
- Преобразование untilted частиц в IMAGIC-5 формат, используя em2em программы в Imagic-5 пакетов. Выравнивание и классификация частиц на однородные классы итеративно использованием IMAGIC-5 программ (Приложение). Использование MSA-имен в своем классе команду в IMAGIC-5 для создания классов таблицу поиска частиц, которые мы сохраняем в качестве imagic_classes.lis. (Примечание:. Классификации и выравнивания заключается в увеличении числа частиц с такой же формы, а также уменьшить изменением класса отклонений карте каждого класса могут предоставить информацию о качестве класса.)
- Создание участка файл (ali_50.plt в видео-демонстрации) для перемещения и вращения значения выравнивания каждой частицы использованием заголовка команды в IMAGIC-5.
- Преобразование классов по таблице в СПАЙДЕР файлы документов base_file ***. SPI использованием lis2spi.pl Перл распространяется в http://cryoem.berkeley.edu .
- Преобразование перемещения и поворота значения выравнивания каждой частицы от участка, созданного для перемещения и вращения значения в 5,6 шаг в СПАЙДЕР файла документа ali_50.spi помощью сценария plt2spi.pl распространяется в http://cryoem.berkeley.edu .
Мы используем IMAGIC-5 для 2D выравнивания и классификации, поскольку он дает лучшую производительность на эту работу в наших руках. Альтернативные стратегии в СПАЙДЕР на двумерной выравнивание и классификации можно найти на http://www.wadsworth.org/spider_doc/spider/docs/align.html . Мы также использовали СПАЙДЕР для выполнения 2D анализ untilted частиц exosome. Ниже приводится простая процедура.
Альтернативные 5.5) Используйте ссылки без выравнивания, как описано в выравнивания изображений. Два простых скриптов можно найти Сохранить вращение и сдвиг всех частиц в angular_file.spi файла документа.
Альтернативные 5,6) Классифицировать выровнены частиц в группы с тем же вид, как описано в Мы использовали К-средних метод классификации. Создание base_file ***. SPI на основе классификации.
Подраздел 3: Трехмерные реконструкции с использованием наклонной изображения частицы.
- Полосовой фильтр, маску и центр наклонена частиц так же, как для untilted частиц в IMAGIC-5. Создание нового набора данных для под названием частиц. (Примечание: Этот шаг является необязательным Это может быть сделано в СПАЙДЕР..)
- Создать anglular файлы документа из классов по таблице документы ali_50.spi, DCB ***. SPI файлы, созданные в шаге веб 5.3 и файл частиц список partile_list.spi с шагом 5,4 использованием СПАЙДЕР сценарий, как в приложении В.
- Использование скриптов в реконструкции СПАЙДЕР сделать реконструкцию каждого класса, как описано в http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html . Каждый класс частиц вносит свой вклад в одном томе реконструкции 3D. 3D модели могут быть рассмотрены в UCSF-Химера 9. Двумерные проекции 3D-модель в угол Эйлера (0,0,0), можно сравнить с соответствующей 2D средний класс от untilted частиц проверить качество реконструкции. Поиск похожих объемов, согласовать и объединить их для получения начального объемов в соответствии с процедурой http://www.wadsworth.org/spider_doc/spider/docs/techs/rancon/recn.html .
Подраздел 4: Уточнение 3D реконструкции с использованием untilted изображения частицы.
- Есть ли соответствующие проекции уточнение объединены первоначального объема против всех untilted частиц, чтобы получить более высокое разрешение Воламэлектронная, не пропуская конуса и уплощение артефакт использованием СПАЙДЕР скрипты, как описано в http://www.wadsworth.org/spider_doc/spider/docs/techs/recon/refine.html .
6. Представитель Результаты:
Используя метод РКИ, мы получили около 50 реконструкций exosome из общего числа 5000 пар наклона (рис. 6). С 50 3D-модели, мы можем видеть разные ориентации комплекса сидя на сетке в основном с двух ортогональных взглядов. Уплощение артефакт также обнаружены во многих томах в направлении, перпендикулярном к углеродной поверхности. Мы выполнили согласование и объединение 3D объемы для создания двух начальных томов на ортогональные виды. Использование 5000 untilted изображения частиц, мы получили окончательный же 3D-реконструкция exosome около 18 ангстрем разрешение от обоих первоначальных моделей (рис. 7). Структуры показали архитектуры дрожжей exosome и дают данные на вербовку РНК субстрат пути 10.

Рисунок 6. 50 3D модели exosome комплекса РКИ реконструкции.
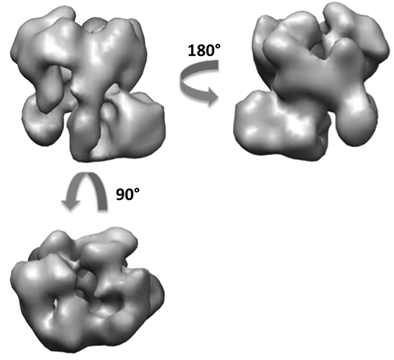
Рисунок 7. 3D-реконструкция exosome комплекса после доработки.
Приложение:
Приложение A. файл сценария для 2D выравнивания и классификации в IMAGIC-5.
Файл: auto_align_i.sh
Нажмите здесь для файла
Приложение B. Файл сценария для создания угловой файл для 3D реконструкции в СПАЙДЕР.
Файл: generate_angular_file.spi
Нажмите здесь для файла
Обсуждение
В этой статье мы представляем подробный протокол пробоподготовки и трехмерные реконструкции exosome комплекса с использованием отрицательных электронной микроскопии окрашивания. Используя этот метод, мы получили 3D-реконструкции с использованием метода случайной конической наклона бе...
Раскрытие информации
Благодарности
Авторы хотели бы поблагодарить членов Ногалес лаборатории в Калифорнийском университете в Беркли, помогая установить начальные протоколы и членам Ван лаборатории в Йельском университете в их помощью установить полный протоколов. Мы также признаем, штабов в крио-ЭМ объекта и высокопроизводительный вычислительный центр в Йельской школы медицины за их поддержку. HW является Смит Семья Лауреат премии.
Материалы
| Name | Company | Catalog Number | Comments |
| Polyvinyl Formal Resin | Electron Microscopy Sciences | 63450-15-7 | |
| Uranyl Formate | Electron Microscopy Sciences | 22451 | |
| Superfrost Microscope Slides | Thermo Fisher Scientific, Inc. | 4951F-001 | |
| 400 mesh grid regular | SPI Supplies | 3040C | |
| Carbon coater Auto 306 | Edwards Lifesciences | ||
| Tecnai-12 Electron Microscope | FEI | ||
| Glow Discharger | BAL-TEC | Sputter Coater SCD 005 |
Ссылки
- van Heel, M. Angular reconstitution: a posteriori assignment of projection directions for 3D reconstruction. Ultramicroscopy. 21, 111-123 (1987).
- Radermacher, M. Three-dimensional reconstruction of single particles from random and nonrandom tilt series. J Electron Microsc Tech. 9, 359-394 (1988).
- Penczek, P. A., Grassucci, R. A., Frank, J. The ribosome at improved resolution: new techniques for merging and orientation refinement in 3D cryo-electron microscopy of biological particles. Ultramicroscopy. 53, 251-270 (1994).
- Ohi, M., Li, Y., Cheng, Y., Walz, T. Negative staining and image classification - powerful tools in modern electron microscopy. Biol Proced Online. 6, 23-34 (2004).
- Frank, J., Radermacher, M., Penczek, P., Zhu, J., Li, Y., Ladjadj, M., Leith, A. SPIDER and WEB: processing and visualization of images in 3D electron microscopy and related fields. J Struct Biol. 116, 190-199 (1996).
- Shaikh, T. R., Gao, H., Baxter, W. T., Asturias, F. J., Boisset, N., Leith, A., Frank, J. SPIDER image processing for single particle reconstruction of biological macromolecules from electron micrographs. Nat Protoc. 3, 1941-1974 (2008).
- Heel, M. v. a. n., Harauz, G., Orlova, E. V., Schmidt, R., Schatz, M. A new generation of the IMAGIC image processing system. J Struct Biol. 116, 17-24 (1996).
- Ludtke, S. J., Baldwin, P. R., Chiu, W. EMAN: semiautomated software for high-resolution single-particle reconstructions. J Struct Biol. 128, 82-97 (1999).
- Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C., Ferrin, T. E. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25, 1605-1612 (2004).
- Wang, H. W., Wang, J., Ding, F., Callahan, K., Bratkowski, M. A., Buttler, J. S., Nogales, E., Ke, A. Architecture of the yeast Rrp44 exosome complex suggests routes of RNA recruitment for 3' end processing. Proc Natl Acad Sci USA. 104, 16844-16849 (2007).
- Scheres, S. H., Nunez-Ramirez, R., Sorzano, C. O., Carazo, J. M., Marabini, R. Image processing for electron microscopy single-particle analysis using Xmipp. Nat Protoc. 3, 977-990 (2008).
- Yoshioka, C., Pulokas, J., Fellmann, D., Potter, C. S., Milligan, R. A., Carragher, B. Automation of random conical tilt and orthogonal tilt data collection using feature-based correlation. J Struct Biol. 159, 335-346 (2007).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены