Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Высокопроизводительного скрининга грибной активность в эндоглюканазы Кишечной палочки
В этой статье
Резюме
Мы описываем низкая стоимость, высокая пропускная способность метода для выявления грибковых деятельности в эндоглюканазы Е. палочки. Метод основан на простой визуальный отсчет деградации субстрата, не требует очистки фермента, и является масштабируемой. Это дает возможность быстрого скрининга больших библиотек фермента вариантов.
Аннотация
Cellulase enzymes (endoglucanases, cellobiohydrolases, and β-glucosidases) hydrolyze cellulose into component sugars, which in turn can be converted into fuel alcohols1. The potential for enzymatic hydrolysis of cellulosic biomass to provide renewable energy has intensified efforts to engineer cellulases for economical fuel production2. Of particular interest are fungal cellulases3-8, which are already being used industrially for foods and textiles processing.
Identifying active variants among a library of mutant cellulases is critical to the engineering process; active mutants can be further tested for improved properties and/or subjected to additional mutagenesis. Efficient engineering of fungal cellulases has been hampered by a lack of genetic tools for native organisms and by difficulties in expressing the enzymes in heterologous hosts. Recently, Morikawa and coworkers developed a method for expressing in E. coli the catalytic domains of endoglucanases from H. jecorina3,9, an important industrial fungus with the capacity to secrete cellulases in large quantities. Functional E. coli expression has also been reported for cellulases from other fungi, including Macrophomina phaseolina10 and Phanerochaete chrysosporium11-12.
We present a method for high throughput screening of fungal endoglucanase activity in E. coli. (Fig 1) This method uses the common microbial dye Congo Red (CR) to visualize enzymatic degradation of carboxymethyl cellulose (CMC) by cells growing on solid medium. The activity assay requires inexpensive reagents, minimal manipulation, and gives unambiguous results as zones of degradation (“halos”) at the colony site. Although a quantitative measure of enzymatic activity cannot be determined by this method, we have found that halo size correlates with total enzymatic activity in the cell. Further characterization of individual positive clones will determine , relative protein fitness.
Traditional bacterial whole cell CMC/CR activity assays13 involve pouring agar containing CMC onto colonies, which is subject to cross-contamination, or incubating cultures in CMC agar wells, which is less amenable to large-scale experimentation. Here we report an improved protocol that modifies existing wash methods14 for cellulase activity: cells grown on CMC agar plates are removed prior to CR staining. Our protocol significantly reduces cross-contamination and is highly scalable, allowing the rapid screening of thousands of clones. In addition to H. jecorina enzymes, we have expressed and screened endoglucanase variants from the Thermoascus aurantiacus and Penicillium decumbens (shown in Figure 2), suggesting that this protocol is applicable to enzymes from a range of organisms.
протокол
1. Подготовка Скрининг пластины
- Добавить 25 г средний рост Л.Б., 15 г агара и 1,5 г карбоксиметилцеллюлозы (КМЦ) субстрата 1л дистиллированной воды, и автоклав для стерилизации.
- После автоклавирования среду позволяют охладить и добавить соответствующие антибиотики, и IPTG к конечной концентрации 100 мкм.
- Залить 200 мл каждая среда в 5 больших квадратных чашках Петри / биопроб лотки (240 х 240 х 20 мм и более). Разрешить пластины до полного высыхания.
2. Эндоглюканазы поколения библиотеки и экран
- Создание библиотеки эндоглюканазы каталитический домен (ы). Это может быть достигнуто разными способами (например, сайт-направленного мутагенеза или случайного, рекомбинации и т.д.) и определяется исследователем.
- Клон библиотеки в такой вектор, что они находятся под контролем промотора T7 с оператором лак, и содержат pelB сигнальной последовательности для ориентации в периплазме. Примером подходящего вектора pET22b (+) от Novagen.
- Преобразование эндоглюканазы библиотеки в E. палочки штамма BL21 (DE3) и пластины для отдельных колоний.
- Инкубируйте преобразования ночи при 37 ° C.
- Использование стерильных зубочистки, привить клонов в 96 и глубоких скважин чашки, содержащие 400μL средой LB добавлены все необходимые антибиотики. Инкубировать при 37 ° С в течение ночи при встряхивании при 250 оборотах в минуту.
- Добавить стерильным глицерином до 96 и ночью культур к конечной концентрации 10% для длительного хранения при температуре -80 ° C. Это будет мастер пластины.
- Использование 96-контактный штамп, повторить пластины ночь культур на скрининг чашки, содержащие IPTG и КМЦ. До 5 наборов ночь культур может нанести на каждый скрининга пластины.
- Этикетка скрининга пластин, так что личность и ориентация каждого 96-луночного планшета известно.
- Инкубируйте штамп библиотеки при комнатной температуре (17-25 ° С) в течение ночи или пока не видимые колонии образовали.
- Промыть пластины с дистиллированной водой, пока все следы колонии удаляются.
- Налейте конго красный (CR, 0,5% в воде) на мыть тарелку. Убедитесь в том, чтобы добавить ровно столько, чтобы покрыть CR пластины.
- Инкубировать 15 минут при комнатной температуре. Не превышать 15 минут для инкубации ЧР. Дольше время инкубации приведет к менее выраженным гало.
- Вылейте CR и добавить большое количество (более чем достаточно, чтобы покрыть пластины) 1 М NaCl.
- Инкубировать 15 минут при комнатной температуре.
- Вылейте NaCl выявить CMC ореолы деградации. Для лучшего разрешения ореолы, больше времени инкубации с NaCl или нескольких NaCl моет могут потребоваться.
- Использование стерильных зубочистки, выбрать активную клонов ферментов из мастер пластина для дальнейшей характеристики.
3. Представитель Результаты:
Пример считывания для этого экран высокого пропускной показано на рисунке 3. Клоны могут быть идентифицированы как неактивные, слабо активны, и очень активный в зависимости от размера деградации зон. Это особенно полезно, если фермент известных деятельность включена в библиотеку в качестве эталона. Как показано здесь, идентификация активных ферментов является надежной и воспроизводимой. Тем не менее, необходимо учитывать, что адекватное маркировки скрининга пластин является чрезвычайно важным. Исследователь должен быть в состоянии идентифицировать активные варианты после мытья колонии далеко, для того, чтобы отобрать их у мастера пластины хранят при -80 ° С для дальнейшей характеристики. Наконец, трудно оценить априорное отношение между деятельностью на искусственном против природных субстратов. Таким образом, исследователь должен проверить все положительные клоны на промышленно соответствующие материалы для определения белка фитнеса.
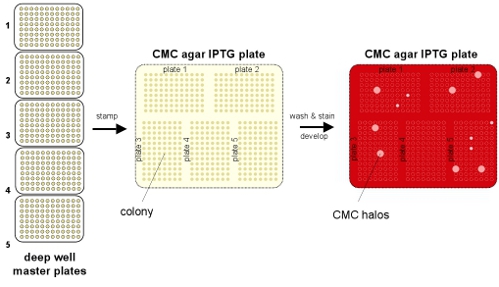
Рисунок 1. Схема контур процедура отбора. Клетки переходят с эндоглюканазы библиотеки и покрытием выборочно для отдельных колоний. Клоны собирают и выращивают в течение ночи на 96 и глубокий колодец пластин для (1) хранение при температуре -80 ° С и (2) реплики покрытие на пластинах содержащих КМЦ и IPTG, чтобы побудить эндоглюканазы выражения. Активная клоны выбрать из мастер пластины хранятся при температуре -80 ° C.

Рисунок 2. Клонирование и экспрессия грибковых каталитических доменов в E. палочки. (А) эндоглюканазы вектор экспрессии. Гены были клонированы в pET22b (+) (Novagen) под контролем промотора Т7. (B) правой панели: эндоглюканазы экспрессирующих BL21 (DE3) колоний. Левая панель: CMC ореолы деградации после мытья и развития с конго красный.
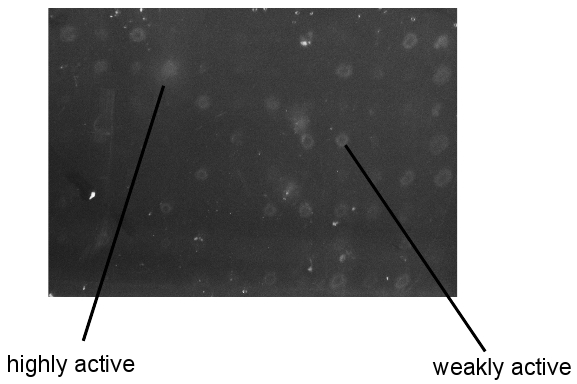
Рисунок 3. Пример результатов от CMC / Конго экране красный. Снимки, сделанные скрининга пластин после мытья и конго красный развития. Все колонии на Screening пластины такого же размера.
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Протокол, описанный здесь, позволяет быстро и высокую пропускную способность идентификации активных ферментов, с минимальной манипуляцией. Обнаружение активности является достаточно чувствительным, и качественно отражает количество активности внутри клетки. Его простота использов...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Эта работа финансировалась Гордона и Бетти Мур Фонда, а также UNCF / Merck Научная инициатива.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Реагент | Компания | Номер изделия | |
|---|---|---|---|
| IPTG | Сигма | I1284 | |
| Конго красный | Сигма | C6277 | |
| Карбоксиметилцеллюлоза | Сигма | 360384 | |
| NaCl | Сигма | S1679 | |
| Бакто-агар | BD Biosciences | 214030 | |
| Биопроб пластин | Thermo Scientific | 240845 |
Ссылки
- Atsumi, S., Hanai, T., Liao, J. C. Non-fermentative pathways for synthesis of branched-chain higher alcohols as biofuels. Nature. 451, 86-89 (2008).
- Wilson, D. B. Cellulases and biofuels. Curr Opin Biotechnol. 20, 295-299 (2009).
- Qin, Y. Engineering endoglucanase II from Trichoderma reesei to improve the catalytic efficiency at a higher pH optimum. J Biotechnol. 135, 190-195 (2008).
- Nakazawa, H. Directed evolution of endoglucanase III (Cel12A) from Trichoderma reesei. Appl Microbiol Biotechnol. 83, 649-657 (2009).
- Heinzelman, P. A family of thermostable fungal cellulases created by structure-guided recombination. Proc Natl Acad Sci. 106, 5610-5615 (2009).
- Heinzelman, P. SCHEMA recombination of a fungal cellulase uncovers a single mutation that contributes markedly to stability. J Biol Chem. 284, 26229-26233 (2009).
- Lantz, S. E. Hypocrea jecorina CEL6A protein engineering. Biotechnol Biofuels. 8, 3-20 (2010).
- Mahadevan, S. A. Site-directed mutagenesis and CBM engineering of Cel5A (Thermotoga maritima). FEMS Microbiol Lett. 287, 205-211 (2008).
- Nakazawa, H. Characterization of the catalytic domains of Trichoderma reesei endoglucanase I, II, and III, expressed in Escherichia coli. Appl Microbiol Biotechnol. 81, 681-689 (2008).
- Wang, H., Jones, R. W. Properties of the Macrophomina phaseolina endoglucanase (EGL 1) gene product in bacterial and yeast expression systems. Appl Biochem Biotechnol. 81, 153-160 (1999).
- Howard, R. L. Enzyme activity of a Phanerochaete chrysosporium cellobiohydrolase (CBHI.1) expressed as a heterologous protein from Escherichia coli. Afr J Biotechnol. 2, 296-300 (2003).
- Howard, Characterisation of a chimeric Phanerochaete chrysosporium cellobiohydrolase expressed from Escherichia coli. Afr J Biotechnol. 3, 349-352 (2004).
- Teather, R. M., Wood, P. J. Use of Congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl Env Microbiol. 43, 777-780 (1982).
- Gilkes, N. R. Mode of action and substrate specificities of cellulases from cloned bacterial genes. J Bio Chem. 259, 10455-10459 (1984).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены