Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Высокое разрешение Пространственно-временная Анализ рецептора Dynamics по одиночных молекул флуоресцентной микроскопии
В этой статье
Резюме
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Аннотация
Микроскопия одиночных молекул становится мощным подхода для анализа поведения сигнальных молекул, в частности в отношении тех аспектов (например,., Кинетика, сосуществование различных государств и групп населения, переходных взаимодействий), которые, как правило, скрыты в измерениях ансамбль, например, результатами, полученными с помощью стандартных биохимических или микроскопии методов. Таким образом, динамические события, такие как взаимодействия рецептор-рецепторов, можно проследить в режиме реального времени в живой клетке с высокой пространственно-временной резолюции. Этот протокол описывает метод, основанный на маркировке с небольшими светлыми и органических флуорофоров и полного внутреннего отражения флуоресценции (TIRF) микроскопия непосредственно визуализировать одиночные рецепторы на поверхности живых клеток. Такой подход позволяет точно локализовать рецепторы, измерить размер рецепторных комплексов, а также захватить динамические события, такие как переходных взаимодействий рецептор-рецепторов. Протокол содержит подробное описание того, как реrform эксперимент в одиночных молекул, в том числе подготовки проб, получения изображений и анализа изображений. В качестве примера, применение этого метода для анализа двух G-белком рецепторы, то есть., Β 2-адренорецепторов и γ-аминомасляной кислоты типа B (ГАМК B) рецептор, не сообщается. Протокол может быть адаптирована к других мембранных белков и различных моделей клеток, методы трансфекции и стратегии маркировки.
Введение
Рецепторы, расположенные на поверхности клеток ощутить внеклеточную среду и отвечать на различные стимулы, такие как отдушки, ионов малых нейротрансмиттеров и крупных белковых гормонов. Жидкость характер клеточных мембран позволяет движения рецепторов и других мембранных белков. Это необходимо для формирования белковых комплексов и возникновением переходных белок-белковых взаимодействий, таких как те, что используются рецепторов собрать в функциональных блоков и преобразовывать сигналы в внутрь клетки. Например, G-белком рецепторы (GPCR), которые составляют самый большой семейство рецепторов клеточной поверхности 1, были предложены для формирования di-/oligomers, которые, как представляется, участвуют в регуляции тонкой настройки передачи сигнала и могут иметь важные физиологические и фармакологические последствия, 2-5.
Микроскопия одиночных молекул имеет большой потенциал непосредственно визуализации с высоким spatiotempоральный разрешение динамическое поведение отдельных рецепторов, расположенных на поверхности живых клеток, в том числе их ассоциации с образованием димеров и высших порядков молекулярные комплексы 6-10. Это дает несколько преимуществ по сравнению со стандартными биохимическими и микроскопии методов, которые обычно сообщают среднее поведение тысяч или миллионы молекул.
Белок маркировка с достаточно яркой и фотостабильным флуорофором имеет важное значение для микроскопии одиночных молекул. Этот протокол использует недавно введенной SNAP тега 11 ковалентно присоединить небольшие и яркие органические флуорофоры чтобы рецепторами клеточной поверхности. SNAP является кДа белок тег 20 происходит от человека фермент репарации ДНК алкилтрансферазы O 6-алкилгуанин-ДНК, который может быть необратимо меченного флуорофора, конъюгированным бензилгуанином (флуорофор-BG) производные. CLIP, еще инженерии тег происходит от SNAP, можно вместо помечены флуорофора-сonjugated производные benzylcytosine 12.
Протокол сообщалось в этой рукописи объясняет, как трансфекции и этикетка SNAP-11 помечены рецепторы с малыми органическими флуорофоров и использовать полного внутреннего отражения флуоресценции (TIRF) микроскопия для визуализации одиночных рецепторы или рецепторных комплексов на поверхности живых клеток 10. Представленные результаты протокола на> 90% эффективности маркировки на внеклеточно SNAP-меченых клеток поверхностного белка 10. Более подробную информацию о том, как использовать данные одной молекулы анализировать размер и подвижность рецепторных комплексов, а также на захват переходных взаимодействий рецептор-рецептор, предоставляется. Схематическое рабочий процесс всей протокола дается на рисунке 1. Например, трансфекция яичника китайского хомячка (СНО) с SNAP-меченых G-белком рецепторы (GPCR) следуют маркировка с производным флуорофором BG как а также его применение в quantifу и di-/oligomerization рецепторов монитор описаны. Этот протокол может быть распространен и на другие поверхности клеток белков и флуоресцентных меток (например., Клип), а также на другие трансфекции и маркировки методов.
протокол
1. Подготовка образца
- Покровные очистки
ПРИМЕЧАНИЕ: Работа под вытяжкой.- Используйте чистые пинцет, чтобы разместить покровные стекла (диаметром 24 мм) в держатель покровное, которая отделяет отдельные покровные.
- Установите держатель с покровные в стакан и добавить хлороформ до покровные не покрыты. Стакан накрывают алюминиевой фольгой, чтобы уменьшить испарение и обрабатывают ультразвуком в ванне для обработки ультразвуком в течение 1 часа при комнатной температуре. Возьмите держатель покровное из стакана и пусть покровные сухой.
- Повторите шаг 1.1.2 с 5M NaOH решения вместо хлороформа.
- Возьмем держатель покровное в новый стакан и мыть три раза дистиллированной водой. Поместите очищенные покровные стекла в клеточной культуральной пластины, наполненной 100% этанола.
- Подготовка образцов калибровочных
- Растворите флуоресцентный краситель в соответствующем растворителе.
- Приготовьте 1:10 серийное разведение флуоресцентного красителя в диапазонес 1 пМ до 1 нМ фильтр стерилизуют воду (0,22 мкм).
- Возьмем очищенные покровные, сохраненные в 100% этаноле и промывают стерилизованным фильтрацией воды. Пятно 20 мкл каждого разбавления флуоресцентным красителем на отдельном очищенной покровное. Пусть покровные высохнуть в стерильной крышкой. Защитите покровные от света и пыли до использования. Используйте эти образцы для оценки интенсивности отдельных флуоресцентных молекул (см. шаг 3).
- Трансфекция
- Культура клеток СНО в 1:01 модификации Дульбекко / питательной смеси Дульбекко F-12 (DMEM/F12) дополненной 10% фетальной бычьей сыворотки (FBS), 100 Ед / мл пенициллина и 100 мкг / мл стрептомицина при 37 ° С, в . 5% СО 2 ПРИМЕЧАНИЕ: Используйте фенол-красный свободные средства массовой информации в течение всего эксперимента, чтобы минимизировать аутофлюоресценция.
- Возьмем очищенные покровные из 100%-ным раствором этанола, промойте стерильной фосфатно-солевом буфере (PBS), и разместить один покровное в каждую лунку 6 мкл клеточной культуре хорошоели.
- Trypsinize, считать и семян клетки СНО при плотности 3 х 10 5 клеток / лунку в клеточной культуры пластины 6-а, содержащего покровные. Пусть клетки растут в инкубаторе (37 ° C, 5% CO 2) в течение 24 часов, чтобы достичь прибл. 80% слияния, который является оптимальным плотность клеток для трансфекции.
- Для каждой лунки, разбавленных 2 мкг целевого плазмидной ДНК (например., SNAP-меченый β 2-адренергических рецепторов) и 6 мкл липофектамина 2000 в двух отдельных пробирки, содержащие 500 мкл среды OptiMEM. Инкубируют при комнатной температуре в течение 5 мин.
- Объедините решения, начиная с шага 1.3.4 в одну пробирку и смешать, чтобы получить трансфекции смесь. Инкубируйте трансфекции смесь при комнатной температуре в течение 20 мин.
- Во время инкубации (1.3.5), взять клетки СНО и мыть дважды подогретого (37 ° С) PBS. Заменить PBS с 1 мл / лунку фенолового красного среде DMEM/F12, дополненной 10% FBS, но без антибиотиков.
- Добавить весь трансфекцииионный смесь (1 мл) с этапа 1.3.5 каплям в каждую лунку и осторожно раскачивать пластину назад и вперед для обеспечения полного перемешивания.
- Выдержите в течение 2 до 4 часов при температуре 37 ° С, в 5% CO 2 и приступить сразу же после этого к следующему шагу. ПРИМЕЧАНИЕ: Эти условия трансфекции были оптимизированы для достижения плотности рецепторов <0,45 частиц / мкм 2, которые подходят для одно- томография молекула. Корректировки могут потребоваться при использовании различных клеток, конструкции или реагентов.
- Белок маркировки
- Развести 1 мкл флуорофор-BG маточного раствора в 1 мл среды DMEM/F12, дополненной 10% FBS, чтобы получить конечную концентрацию 1 мкМ. Возьмем трансфицированных клеток из инкубатора и дважды промывали нагретого (37 ° C) PBS. Заменить PBS с 1 мл 1 мкМ раствора флуорофором BG и инкубировать в течение 20 мин при 37 ° C 5% CO 2 в инкубаторе.
- После инкубации промыть клетки трираза с среде DMEM/F12, дополненной 10% FBS, каждый раз с последующей 5-минутной инкубации при 37 ° С Возьмите покровное (с меченых клеток) с помощью пинцета и поместить его в камеру изображения.
- Мыть два раза с 300 буфера изображений мкл. Добавить 300 мкл свежего буфера изображений и немедленно приступить к визуализации (Часть 2).
2. Приобретение изображения
ПРИМЕЧАНИЕ: Используйте полного внутреннего отражения флуоресценции (TIRF) микроскоп, оснащенный масло-погружения высокой числовой апертурой объектива (например, 100x magnification/1.46 числовой апертурой.), Подходящих лазеров (например, 405 нм, 488 нм, 561 нм и 645 нм диодные лазеры), электрон умножения прибор с зарядовой связью (EMCCD) камера, инкубатор и контроль температуры визуализировать одиночные флуоресцентных молекул.
- Установите желаемые параметры микроскопа, т.е.., Лазерная линия, угол TIRF (этот параметр управляет проникновенияглубины проникновения от затухающих области), время экспозиции, частота кадров и количество кадров в фильме 10. Держите контроль нагреватель / инкубатор и температуры всегда, чтобы избежать температурных заносы и конденсации влаги.
- Поместите каплю иммерсионного масла на 100X объектив микроскопа. Поместите изображения камеры с меченых клеток на держатель образца микроскопа, и принести клетки в фокусе использованием светлое освещение.
- Перейти к TIRF освещения. Хранить мощность лазера как можно ниже, чтобы поиск требуемой клетке, но в то же время сводя к минимуму фотообесцвечивания.
- Выберите нужную ячейку и тонкой регулировки фокуса. Установите мощность лазера до уровня, который позволяет визуализировать отдельных флуорофоров. Приобретать последовательность изображений и сохранить файл необработанного последовательность изображений, как. Размолвки.
3. Калибровка (Одноместный Флуорофоры на стекла и мономерных / димерных рецепторов управления)
- Соберите каждый калибровки SAMPLе полученного, как описано в п. 1.2 в камере изображений. Поместите каждый образец на микроскопа и выбрать образец, содержащий хорошо разделенных дифракционной пятна, которые отбеливатель в одну стадию. Примечание: Эти пятна представляют отдельные молекулы флуоресцентного красителя.
- Приобретать последовательности изображений TIRF, как описано в шаге 2. Важно: те же параметры визуализации должны использоваться для всех экспериментов, в том числе тех, для калибровки.
- Выполните обнаружение и сопровождение анализ как подробно изложено в 4,1 - 4,2. Выписка интенсивность каждой частицы, как описано в 4.2.6. Исходя из этих данных, вычислить среднее (μ) и стандартное отклонение (σ) интенсивности отдельных флуорофоров.
- Дополнительно: выполнить тот же анализ на клетках, трансфицированных мономерного рецептора на поверхности клетки (. Например, CD86), N-терминально отмеченных либо одного или двух копий SNAP 10 и помечены с производной флуорофором BG. ФоллоW методике, описанной выше для одного флуорофора на стекле. Оценить эффективность маркировки, как описано в Calebiro, D. и соавт. 10
4. Анализ изображений
- Подготовка последовательность изображения
- Используйте программное обеспечение для обработки изображений (например,., ImageJ), чтобы обрезать изображения.
- Сохраняет отдельные кадры как отдельный. TIFF изображения в новой папке, указывая на каждом изображении номер кадра.
- Измерьте площадь поверхности клетки, рисуя область интереса (ROI) по контуру клетки и с помощью инструмента измерения в ImageJ или аналогичного инструмента в другое программное обеспечение. Это значение можно использовать для расчета плотности частиц путем деления общего количества частиц в начале фильма на площадь поверхности клетки.
- Обнаружение частиц и отслеживания
ПРИМЕЧАНИЕ: Используйте некоммерческой программы, такие как U-трек 13, работая в Matlabсреда, для автоматического обнаружения и отслеживания частиц одиночных рецепторов.
Примечание: алгоритм U-трек основан на подходе отслеживания нескольких гипотез. Этот подход связывает частицы между кадрами путем создания стоимости матриц, где отдельные вероятности того, что данная частица в одном кадре соответствует данной частицы в следующем кадре, оказывается, исчезает или слияний / разбивает с / от других частиц назначены. Решение, которое во всем мире минимизирует затраты, то есть., Тот, с наибольшей вероятностью, будет, наконец, выбрали. Это также позволяет отслеживания временно исчезают частицы, типичный феномен, вызванный флуорофора миганием. В последней версии U-трек (2.1.0) имеет графические пользовательские интерфейсы, которые облегчают выполнение этих анализов.- В командной строке Matlab, типа "movieSelectorGUI", чтобы открыть интерфейс выбора фильма. Следуйте инструкциям, чтобы создатьНовая база данных фильмов, начиная с ранее сохраненным отдельных изображений (см. 4.1.2).
- Обеспечить размер пикселя в нм, интервал времени в секундах, числовую апертуру, глубину камеры битов и длины волны излучения флуорофора, необходимое для регистрации частиц и отслеживания. Сохраните базу данных фильмов.
- Из интерфейса выбора кино, запустить анализ, выбирая "одиночные частицы" как тип объектов. Появится новое окно, где параметры, используемые для обнаружения частиц и отслеживания может быть определена. Начните с параметрами по умолчанию. Позже, настроить эти параметры, если качество обнаружения и / или отслеживания не является удовлетворительным (например, некоторые частицы не обнаружены или дорожек фрагментированы) Дополнительно:. В настройках слежения, проверить "отслеживания результатов Экспорт в формат матрицы", чтобы сохранить координаты и амплитуды всех частиц в одной матрице (поле под названием "trackedFeaturedInfo»). Для получения подробного описания этих параметров, обратитесь к документации у-трека.
- Запустите алгоритм обнаружения. Этот алгоритм автоматически определить местоположение и интенсивность выше фона каждой дифракционной месте (то есть., Одиночные рецепторы / рецепторных комплексов) путем установки двумерный гауссовой функции со стандартным отклонением, равным функции рассеяния точки микроскопа около локальных максимумов интенсивности . Затем запустите алгоритм отслеживания. Храните результаты анализов в. Файл мат.
- Используйте "movieViewer" рутины, содержащийся в пакете у-трека или аналогичные новые пользовательские режимы визуализировать треки и проверить качество обнаружения и сопровождения.
- Откройте файл. Мат, чтобы увидеть положение и амплитуду (т.е.., Интенсивность) отслеживаемых частиц в каждом кадре. Данные, полученные в шаге 4.2.4 содержатся в области tracksCoordAmpCG АР tracksFinalлуч и / или в trackedFeaturedInfo. Из общего числа частиц обнаружено рассчитать плотность частиц Разделив эту величину на площадь поверхности клеток, измеренной при 4.1.3.
- Дополнительно: Используйте координаты частиц с течением времени (см. 4.2.6), чтобы проанализировать движение частиц рецепторов. Рассчитать средние квадратных смещения (MSD) и коэффициентов диффузии (D), используя Matlab или подобное программное обеспечение. Для каждой частицы и каждого интервала времени (Δ т) считается, вычислить средний квадрат смещения (MSD) по следующей формуле:
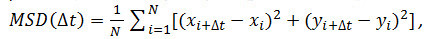
где Δ т это интервал времени в кадрах, N-число шагов проанализированных х и у х частицы и у координаты в кадре, показанном индекса. Используйте MSD течением времени участков оценить тип движения данной частицы: линейные соотношенияобозначения свободного распространения (т.е.., броуновское движение), на положительную кривизну (IE., кривая выглядит как параболы) предполагает направленное движение, отрицательной кривизны свидетельствует о замкнутом движения 14. В случае свободно диффундирующих частиц, вычисления коэффициента диффузии (D) каждой частицы путем подгонки полученные данные MSD с помощью следующего уравнения: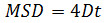
- Расчет размера частиц с гауссовой фитинга методом
ПРИМЕЧАНИЕ: После того, как распределение интенсивности калибровочных образцов (Single флуорофоры на стекле и / или флуоресцентной меченых мономерных рецепторов), как известно, выполняют смешанную Gaussian посадку на распределение интенсивности частиц в начале последовательности изображений, чтобы определить размер рецепторных комплексов (т.е.., число рецепторов на одну частицу) 10. Выполните эти анализы с использованием Matlab или секimilar программного обеспечения.- Рассчитать интенсивность каждой частицы путем усреднения интенсивности частицы от первого кадра к кадру до первого изменения интенсивности произошло (в большинстве случаев снижение из-за фотообесцвечивания).
- Выполнение смешанную гауссову присоединение по следующей формуле:
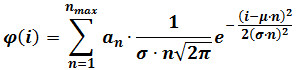
где φ (I) является частота частицы, имеющие интенсивность I, N есть число компонент, α н это параметр, который вносит свой вклад в высоту компонента п, μ и σ представляют собой среднее и стандартное отклонение интенсивности опорных отдельных флуорофоров . (рассчитывается как описано в шаге 3) ПРИМЕЧАНИЕ: Определите тон Максимальное число компонент (N Максимум) для каждого последовательности изображений, постепенно увеличивая N Макс до добавления компонента уже больше не производят статистически лучше фитинг, если судить по F-теста. - Дополнительно: (. Например, последние 60 кадров из 400 последовательности кадра) Выполните смешанную Gaussian посадку на распределение интенсивности, полученные на последних кадрах фильма. Заменить μ и σ со значениями, полученными после этого фитинг, которые обеспечивают изысканные оценки этих параметров, и повторите шаг 4.3.2.
- Вычислить площадь под кривой (AUC) для каждого компонента смешанного гауссова подгонки. Вычислить относительное содержание частиц рецепторов разного размера (т.е. мономер, димер, тример и т.д.) путем деления значения AUC каждого компонента по AUC всей распределения.
- Дополнительно: Использовать данные из разных клеток и соответствующих плотностей частиц (рассчитывается как описано в 4.2.6) для создания участков, где распределение частиц с различными размерами коррелирует с плотностью частиц 10.
- Расчет размера частиц с шагом установки методом
ПРИМЕЧАНИЕ: Используйте шаг облегающие анализ в качестве альтернативного метода определения размера рецепторов комплексов 10. Основой для этого анализа является то, что светоиндуцированная уничтожения (фотообесцвечивания) одного флуорофорных приводит к ее мгновенной исчезновения - таким образом частицы, содержащие п флуорофоры, как ожидается, постепенно отбеливатель производя профиль интенсивности с п шагов.- Выписка профили интенсивности каждой частицы из файла мат., Порожденной U-трека или аналогичного программного обеспечения обнаружения / слежения (см. 4.2.6).
- Используйте шаг облегающие алгоритм, например, той, которую представилв работе. 10, для подсчета количества шагов отбеливания для каждой частицы.
- Дополнительно: использовать результаты для создания дистрибутивов, показывающие относительное обилие частиц рецепторов разного размера и соотнести их с плотностью частиц, как описано выше за результаты смешанной Гаусса фитинга (см. п. 4.3).
Результаты
Описанный протокол может быть применен к различных мембранных белков. Например, представительные результаты, полученные с β 2-адренергических и GABA B-рецепторов приведены 10. Поскольку люминесцентные сигналы от одиночных молекул слабы, минимизация фоновой флуоресценц?...
Обсуждение
Описанный протокол позволяет анализ пространственного расположения, подвижности и размера рецепторных комплексов на поверхности клеток на уровне отдельных молекул. По сравнению с использованием флуоресцирующих белков, маркировка с малых органических флуорофоров, которые светлее и...
Раскрытие информации
The authors declare that they have no competing financial interests.
Благодарности
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
Материалы
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
Ссылки
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены