Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Последовательность конкретных Маркировка нуклеиновых кислот и белков с метилтрансферазы и кофактора аналогов
В этой статье
Резюме
ДНК и белки-последовательность специально помечены близости или люминесцентных репортер групп с использованием ДНК или белка-метилтрансферазы и синтетические аналоги кофактора. В зависимости от кофактора специфики ферментов, азиридина или двойных активированных аналогов кофактора используются для одно- или двухступенчатой маркировки.
Аннотация
S -Adenosyl-L-метионин (AdoMet или SAM) зависимых метилтрансферазы (МТАазе) катализируют перенос активированного метильной группы от AdoMet к отдельным позициям в ДНК, РНК, белков и малых биомолекул. Это естественная реакция метилирования может быть расширена до широкого спектра реакций алкилирования с использованием синтетических аналогов кофактора. Замена реактивной сульфониевой центре AdoMet с Азиридиновое кольцо приводит к кофакторов, которые могут быть в сочетании с различными ДНК MTases ДНК. Эти кофакторы азиридиновые может быть оснащен репортерных групп в различных положениях аденина и используется для S equence конкретных М ethyltransferase- Я nduced л Abel Ing ДНК (ДНК улыбается). В качестве типичного примера приведем протокол для биотинилирования pBR322 плазмиды ДНК в последовательности 5'-ATCG Т-3 "с ДНК МТАазе M.BseCI и азиридинового кофактора 6BAz водин шаг. Расширение активированного метильной группы с ненасыщенных алкильных групп приводит к другому классу AdoMet аналогов, которые используются для м ethyltransferase-направленный Т ransfer Л ctivated G roups (MTAG). Поскольку расширенные боковые цепи активируются сульфониевой центра и ненасыщенной связи, эти кофакторы называются дважды активированные аналоги AdoMet. Эти аналоги не только в качестве кофакторов ДНК MTases, как азиридиновых кофакторов, но и для РНК, белков и малых MTases молекулы. Они обычно используются для модификации ферментативной МТАазы субстратов с уникальными функциональными группами, которые меченных репортерных групп во втором химическом этапе. Это видно на примере протокола для флуоресценции маркировки гистона H3 белка. Небольшой пропаргил группы передается от аналогового кофактором SeAdoYn с белком в гистона Н3 лизина 4 (H3K4) МТАазы Set7 / 9 с последующим щелчком маркировкиalkynylated гистонов H3 с TAMRA азида. МТАазе-опосредованной маркировки с кофактора аналогов технологией для многих интересных приложений, включая идентификацию и функциональный изучения МТАазе субстратов, а также генотипирования ДНК и обнаружения метилирования.
Введение
Удельный маркировка нуклеиновых кислот 1,2 и белков 3,4 представляет большой интерес для функциональных характеристик, медицинской диагностики и (нано) биотехнологии. Здесь мы представляем ферментативного метода маркировки для этих биополимеров, который основан на S -adenosyl-L-метионина (AdoMet или SAM) -зависимых метилтрансфераз (MTases). Этот класс ферментов (EC 2.1.1.) Цели отдельных нуклеофильные позиции (азот, кислород, сера и атомами углерода) в пределах конкретных остатков нуклеиновых кислот и белков, и, естественно, передает активированного метильной группы кофактора AdoMet (1А) 5. Кроме того, MTases могут использовать синтетические аналоги кофактором для конкретного маркировки со сродством теги, флуорофорами или других меток (рис 1б) 6. Два класса AdoMet аналогов были разработаны: Азиридин кофакторов для S equence специфической M ethyltransferase- I Nduced L Абель Инг (улыбается) 7 и двухместные активированные аналоги AdoMet для м ethyltransferase-направленного T ransfer Л ctivated G roups (Мечел Трейдинг АГ) 8.

Рисунок 1: реакций, катализируемых метилтрансфераз (MTases) Групповой трансфер А. Метил из природного кофактора AdoMet (SAM) на различных подложках, включая ДНК, РНК, белков и малых биомолекул Б. Маркировка / функционализации нуклеиновых кислот и белков (NNNNN =.. пар оснований для ДНК, нуклеотиды РНК и аминокислот для белков; XXXXX = последовательность признание МТАазе с целевой остатка в зеленый) с синтетическими аналогами кофактора. Азиридин кофакторов содержащие репортер группу (синий шар)прикреплен к аденин кольце последовательность специально в сочетании с целевой остаток (слева) и дважды активируется аналогов AdoMet привести к передаче расширенных алкильных цепей, несущих Chemical Reporter Y (справа), которые могут быть помечены bioorthogonal реакции мыши на втором этапе. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
Азиридин алгебраические лучше всего работают с ДНК MTases. Они содержат трехчленное кольцо с атомом азота 9 (или N -mustard 10,11) вместо сульфониевого центра, реактивная группа. Протонирование этого атома азота активирует Азиридиновое кольцо для нуклеофильной атаки по целевой нуклеотид, что приводит к ковалентного связывания всей кофактора с ДНК. Присоединив репортерных групп в кольце аденин азиридиновые кофакторы могут быть использованы в сочетании с ДНК MTases маркировать ДНК в одну стадию ( г> Рисунок 1B, слева) 7,12. Это показано более подробно на биотинилирования ДНК с 6BAz 13 - 15 (азиридинового кофактора с биотином, прикрепленной к 6 положении кольца аденина) и аденин-специфической ДНК МТАазы из Bacillus stearothermophilus (M.BseCI) 16 (фиг.2, смотрите раздел протокола 2: один шаг маркировки ДНК с помощью азиридиновых кофакторов). В дополнение к M.BseCI ('Sequence признание, ДНК MTases из Thermus адиайсиз (M.TaqI, 5'-TCG -3 5'-ATCG Т-3)'), из Haemophilus heamolyticus (M.HhaI, 5 '-g C GC-3') и от Spiroplasma (M.SssI, 5'-C G-3 ') были успешно использованы для biotinylate ДНК с 6BAz 17. Кроме того, азиридиновые кофакторы могут быть использованы для одностадийного маркировки флуоресценции ДНК 18,19.
ontent "FO: Keep-together.within-страницу =" всегда ">
Рисунок 2:. Последовательность, специфичную один шаг биотинилирование ДНК с M.BseCI и 6BAz ДНК МТАазы M.BseCI распознает последовательность двухцепочечной ДНК 5'-ATCG Т-3 'и, естественно, метилирует аминогруппы второй аденина Остаток (зеленый) с помощью AdoMet. С азиридинового кофактора 6BAz ходом реакции изменяется и M.BseCI приводит к последовательностью, специфичной биотинилирование ДНК путем связывания всю кофактор в том числе биотин (синий) с целевой аденина. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
Двойные активированные аналоги AdoMet содержат расширенные ненасыщенные боковые цепи вместо метильной группы в сульфониевого центра (рисунок 1b , справа) 20. Ненасыщенные двойную или тройную связь в β-положении к сульфониевых центра компенсирует неблагоприятные стерических эффектов в переходном состоянии от конъюгативного стабилизации. Так как сульфоний центр и ненасыщенные связи активировать боковую цепь для ферментативного переноса, эти алгебраические были названы дважды активированные аналоги AdoMet. Как правило, они используются для передачи боковые цепи с уникальными химических групп (химические репортеров), как и аминокислоты, алкинов и азид групп, для химико-селективный маркировки на втором этапе 8,21. В общем, двойные активированные аналоги AdoMet не может работать только в качестве кофакторов ДНК MTases 8,20,21, но и для РНК MTases 22,23 и белковых MTases 24 - 28 позволяет дополнительно маркировки РНК и белков. Тем не менее, расширенные боковые цепи являются стерически более жесткими, чем метильной группой и расширение МТАазы активные участки от белка инженерии частоан требуется для получения эффективных скорости передачи. Другое решение этой проблемы состоит в использовании аналога AdoMet с небольшим пропаргил группы (три атомов углерода), где терминал алкина выполняет две функции: 1. стабилизации переходного состояния во время передачи ферментативной и реактивной 2. ручкой для следующей химической модификации от медью катализируемой азид-алкин циклоприсоединения (CuAAC) нажмите химию. Выяснилось, что в результате пропаргиловой AdoMet аналог 29 является довольно нестабильны в нейтральной или слегка щелочной среде, и только ограниченное применение. Этот недостаток можно исправить, заменив атом серы с селеном. Полученный кофактором 5 '- [(Se) [(3S) -3-амино-3-карбоксипропил] проп-2-ynylselenonio] -5'-дезоксиаденозин (SeAdoYn, рисунок 3) принимается дикого типа ДНК, РНК, и белковые MTases 30 - 32, которые отменяют необходимость белковой инженерии во многих случаях. Это подтверждается флуоресценции про белок маркировка с гистона H3 лизина 4 (H3K4) МТАазе Set7 / 9 33 (рис 3, смотрите раздел протокола 3: маркировка двухступенчатой белка, с помощью двойного активированных кофакторов).
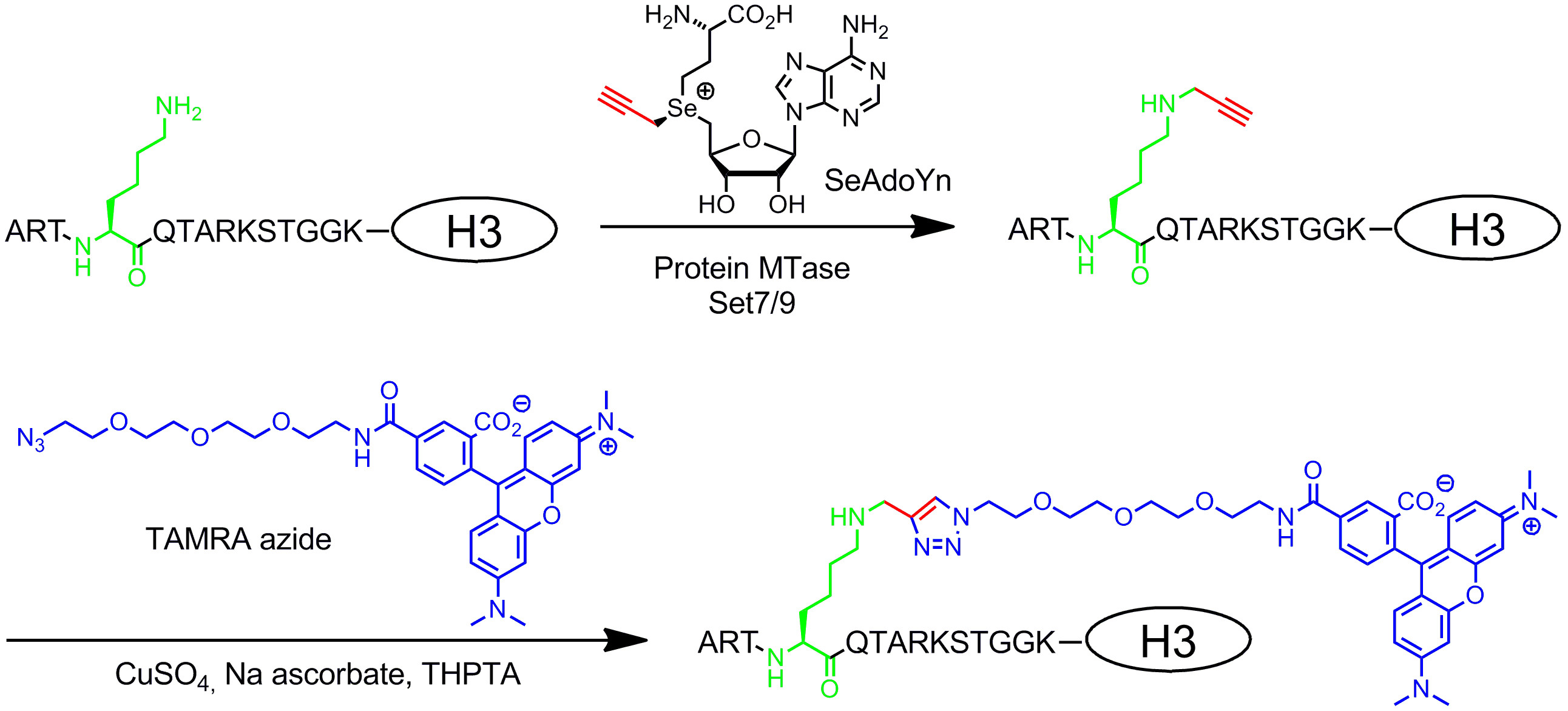
Рисунок 3:. Последовательность конкретных двухступенчатый флуоресценции маркировки гистона Н3 с Set7 / 9, SeAdoYn и TAMRA азид белок МТАазы Set7 / 9, естественно метилирует аминогруппы лизина 4 в гистона Н3 (H3K4, зеленый), используя AdoMet. С двойной активирована кофактора SeAdoYn МТАазы передает небольшой пропаргил группу (красный) до остатка лизина. Прилагается терминал тройная связь выборочно модифицировать в bioorthogonal реакции мыши (медно-катализируемой азид-алкин циклоприсоединения, CuAAC) азидной-производные TAMRA (тетраметилродамин, синий) флюорофора.нагрузка / 52014 / 52014fig3highres.jpg "TARGET =" _ пустое "> Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этой цифры.
протокол
1. Общие инструкции
- Магазин азиридин кофактором 6BAz (в ДМСО), и белок МТАазы Set7 / 9 при -80 ° С, и все другие реагенты, включая двойной активирована кофактора SeAdoYn и ДНК МТАазы M.BseCI (в 50% глицерина) при -20 ° С.
- Определите концентрацию 6BAz и SeAdoYn с помощью UV / VIS спектроскопии с использованием коэффициентов экстинкции ε 269nm (6BAz) = 16000 см -1 М -1 и ε 260 нм (SeAdoYn) = 15400 см -1 М -1 в деионизированной воде. Определить концентрацию MTases с помощью анализа Брэдфорда или, если коэффициент экстинкции доступен, с помощью прямого поглощения при 280 нм.
- Старайтесь избегать образования пузырьков от интенсивного пипетки или вортексе для предотвращения потери ферментативной активности. Вместо этого, перемешать, осторожно пипеткой вверх и вниз.
- При добавлении кофакторов азиридиновые из маточных растворов в ДМСО убедиться, что конечная концентрация ДМСО в анализе составляет менеечем на 5%. Всегда включать 10 мМ ионы магния в буфере для анализа, чтобы предотвратить неспецифические реакции с ДНК.
- При добавлении двойные активированные кофакторов из кислых маточных растворов используют небольшие объемы (в высоких концентрациях растворы), чтобы избежать изменения рН и убедитесь, что рН раствора анализа существенно не изменится. Избегайте тиолы, например, β-меркаптоэтанол или дитиотреитол (DTT), в буфере для анализа, поскольку они могут помешать реакции щелчок комплексообразования необходимых ионов меди.
2. Один шаг Маркировка ДНК с помощью Азиридин кофакторами
- Последовательность конкретных метилтрансферазы-индуцированной Этикетка ING (улыбается) плазмидной ДНК с M.BseCI ДНК МТАазе и азиридин кофактора 6BAz.
- Оттепель кофактора раствора при 20 ° С и подготовить реакционные смеси на льду.
- В дополнение к анализа выполнить контроль "кофактора", чтобы визуализировать любые неспецифические изменения, а & #8220; фермент "контроль, чтобы убедиться, что подготовка МТАазе свободен от природного кофактора AdoMet.
- Для анализа смеси 2 мкл 10х буфера (модификации, содержащей 100 мМ Трис-HCl, 100 мМ MgCl 2, 20 мМ β-меркаптоэтанола, рН 7,4), 2 мкл pBR322 (0,5 мкг / мкл), 10 экв. M.BseCI в последовательности узнавания на ДНК последовательности (1 распознавания в pBR322) и азиридинового кофактора 6BAz до конечной концентрации 60 мкМ в общем объеме 20 мкл. Добавить кофактором и ДНК МТАазе в прошлом.
ПРИМЕЧАНИЕ: β-меркаптоэтанол является токсичным, коррозии и вредным для окружающей среды. - Для контроля »кофактора" Добавить деионизированной воды вместо M.BseCI и для "фермент" контроля добавить деионизированной воды вместо 6BAz.
- Смешайте решения осторожно пипеткой вверх и вниз.
- Инкубировать пробирки при 55 ° С в течение 1 часа.
- Центрифуга кратко, чтобы собрать все жидкости в нижней части трубы.
- Рестрикции-модификации анализ, чтобы проверить модификации ДНК.
- Готовят раствор путем смешивания 10 мкл 10х буфера (R.TaqI, содержащий 100 мМ Трис-HCl, 50 мМ MgCl 2, 1 М NaCl, 1 мг / мл бычьего сывороточного альбумина, рН 8,0), 80 мкл деионизированной воды и 3,3 мкл рестрикции (REase) из Thermus адиайсиз (R.TaqI, 10 U / мкл). Убедитесь в том, чтобы добавить REase на последней стадии.
- В каждую пробирку с 2.1.7 добавить 2 мкл 10х Р. TaqI буфер и 28 мкл раствора сверху (2.2.1).
- Смешайте решения осторожно пипеткой вверх и вниз.
- Инкубировать пробирки при 65 ° С в течение 30 мин.
- Центрифуга кратко, чтобы собрать все жидкости в нижней части трубы.
- Электромобилей сдвиг (тест EMSA) стрептавидином проверить функциональную модификацию.
- Удалить 25 мкл из каждой пробирки (2.2.5) и добавить 2,4 мкл стрептавидина решения (1 мМ по отношению к стрептавидин- мonomer в стрептавидин буфера, содержащего 100 мМ Na 2 HPO 4, 100 мМ NaCl, рН 7,5; 4 эквивалентов общей биотин). Добавить 2,4 мкл стрептавидина буфера для остальных труб.
- Инкубируйте все пробирки при 37 ° С в течение 1 часа.
- Анализ с помощью электрофореза в агарозном геле.
- Добавить 5 мкл 6x буфера для нанесения (0,25% бромфенола синего, 30% глицерина) в каждую пробирку.
- Смешайте решения осторожно.
- Добавить 10 мкл каждого образца в лунки агарозном геле (1% агарозы в 0.5x КЭ буфера, содержащего 1x GelRed из 10,000x маточного раствора).
- Запустите гель в 0,5 × КЭ буфера с 80 V прим. 1 час.
- Визуализация полос ДНК на УФ таблицы (312 нм) с ПЗС-камерой, снабженной фильтром (540 ± 50 нм).
ПРИМЕЧАНИЕ: УФ-излучение повреждает глаза и кожу.
3. Двухступенчатая белка маркировки с помощью двойного Активированные кофакторы
- Methyltransfстереть-направленного переноса активированных групп (Мечел Трейдинг АГ) с Set7 / 9 и дважды активируется кофактора SeAdoYn для гистона H3 лизина 4 маркировка (стадии модификации).
- Оттепель компонентов и подготовки реакционных смесей на льду. ПРИМЕЧАНИЕ: Всегда держите SeAdoYn охлаждением, чтобы избежать деградации.
- В дополнение к анализа выполнить контроль "кофактора", чтобы визуализировать любые неспецифические изменения, и "фермент" контроль, чтобы исключить неспецифические реакции флуоресцентного зонда.
- Подготовка Анализируемый раствор (20 мкл), содержащий модификации буфера (50 мМ Трис-HCl, 5% глицерина, рН 8,5), 10 мкМ гистон H3, 10 мкМ Set7 / 9 и 600 мкМ SeAdoYn (смесь обоих эпимеров в селена). В последних шагов добавить кофактор, а затем МТАазе.
- Для контроля »кофактора" подготовить решение в тестах в 3.1.3 и добавить 60 мм AdoMet конкурировать с синтетическим кофактора. Для контроля "фермент" добавить деионизированной воды вместо SeAdoYn.
- Смешайте решения медленно пипетки вверх и вниз. Проверка рН путем добавления 1 мкл каждого раствора в верхней области в полосе рН (диапазон рН 5 - 10).
- Инкубируют при 37 ° С в течение 2 ч.
- В то же время подготовить 12% SDS полиакриламидном геле (гель работает: 357 мМ бис-Трис, рН 6.5-6.8, 0.1% (вес / объем) APS, 0,04% (объем / объем) TEMED и 12% акриламида / бис-акриламида 37,5: 1 ; загрузка гель: 357 мМ бис-Трис рН 6.5-6.8, 0.1% (вес / объем) APS, 0,04% (объем / объем) тетраметилендиамин и 5% акриламида / бис-акриламида 37,5: 1).
ПРИМЕЧАНИЕ: акриламид / бис-акриламида является токсичным и здоровье опасными. Надевайте перчатки во время этой процедуры.
- Химическая маркировка alkinylated лизина 4 в гистона H3 по медному катализируемой азид-алкин циклоприсоединения (CuAAC) (маркировки шаг).
- Перед конце реакции модификации подготовить 5x щелчок смеси, содержащей 3 мМ CuSO 4, 3 мМ трис (3-гидроксипропил-triazolylmethyl) амин (THPTA), 250 мМ аскорбата натрия и 6 мМ азида с TAMRAОбщий объем 20 мкл.
- Добавить 5 мкл свежеприготовленного 5x мыши смеси в каждую пробирку, чтобы начать CuAAC и гашения реакции модификации.
- Осторожно перемешать с помощью пипетки вверх и вниз.
- Защитите все трубы с алюминиевой фольгой от света, чтобы избежать фото-отбеливание флуорофора.
- Инкубируют при 20 ° С в течение 1 часа.
- Белок осадков удалить избыток свободного TAMRA флюорофора.
- Чтобы избежать затмевая флуоресцентного помечены гистона H3 по интенсивной в геле флуоресценции свободного TAMRA флюорофора, удалить излишки флуорофора путем осаждения белков (3.3.2 - 3.3.4) 34.
- Добавить 75 мкл метанола, 18,8 мкл хлороформа и 50 мкл деионизованной воды в каждую пробирку и вихря на короткое после каждого добавления. Центрифуга при 16000 х г в течение 5 мин. Удалить верхнюю фазу, не нарушая интерфейс слой, который содержит белок.
- Добавить 56,3 мкл метанола в оставшейся фазе яп каждая трубка, вихревые и центрифуге при 16000 х г в течение 5 мин для осаждения белка. Удалить супернатант. Повторите этот шаг, чтобы смыть таблетку.
- Обложка открытые трубы с безворсовой ткани и дайте им высохнуть в течение 15 - 30 мин.
- Анализ с помощью SDS-PAGE.
- Растворить осажденных белков из 3.3.4 в 20 мкл SDS загрузки буфера (50 мМ Трис-HCl, 2,5% (вес / объем) SDS, 10% (об / об) глицерина, 320 мМ β-меркаптоэтанола и 0,05% (вес / V) бромфенол синий, рН 6,8). Убедитесь в том, чтобы полностью растворить таблетку путем промывки стенок труб с помощью пипетки.
- Инкубируйте образцы при 95 ° С в течение 10 мин и позволить им остыть до 20 ° С.
- Центрифуга кратко, чтобы собрать все жидкости в нижней части трубы.
- Загрузите всю сумму каждого образца в лунки в полиакриламидном геле SDS (3.1.7). Использование 50 мМ MOPS, 50 мМ Трис-X (Трис-основание), 5 мМ ЭДТА, 0,1% (вес / объем) SDS, как работает буфер для электрофореза.
- Запустите гель с 120 V в течение ок. 90 мин.
- Визуализация флуоресценцию в геле на УФ таблицы (312 нм) с ПЗС-камерой, снабженной фильтром (540 нм ± 50 нм).
ПРИМЕЧАНИЕ: УФ-излучение повреждает глаза и кожу.
Результаты
Один шаг Маркировка ДНК с помощью Азиридин кофакторами
В этом примере реакцию проводят с ДНК МТАазы M.BseCI, который изменяет второй аденин остаток в 5'-ATCG Т-3 'последовательности двунитевой и имеет один сайт узнавания на плазмиде pBR322 (фиг.4А). Чтобы проверит...
Обсуждение
Один шаг маркировки ДНК с MTases ДНК и азиридиновых кофакторов (улыбается ДНК) является надежным методом, но некоторые аспекты следует учитывать при планировании эксперимента.
Азиридин кофактором: концентрация 6BAz для маркировки ДНК с M.BseCI был 60 мкм. При использовании ?...
Раскрытие информации
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Благодарности
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
Материалы
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Ссылки
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены