Method Article
Объективное Deep секвенирование РНК вирусов из клинических образцов
В этой статье
Резюме
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Аннотация
Здесь мы опишем протокол секвенирования РНК следующего поколения , которая позволяет De Novo узлов и внутри хоста вариантные вызовы вирусных геномов , собранных из клинических и биологических источников. Метод объективной и универсальной; он использует случайные праймеры для синтеза кДНК и не требует предварительных знаний о вирусного контента последовательности. До создания библиотеки, избирательное переваривания РНКазы Н на основе используется для истощить нежелательные РНК - в том числе поли (р.а.) и носитель рибосомной РНК - от вирусного образца РНК. Селективный истощение улучшает как качество данных и количество уникальных просмотров в вирусных РНК библиотек секвенирования. Кроме того, стадия а транспозазу на основе 'tagmentation' используется в протоколе, поскольку это сокращает общее время строительства библиотеки. Протокол позволил быстрое глубокое секвенирование более 600 вируса Ласса и Эбола образцы коллекций, в том числе с обеих крови и ткани изолирует-и широко применима к другим микробные геномики исследований.
Введение
Следующее поколение Секвенирование вирусов из клинических источников может сообщить передачу и эпидемиологии инфекций, а также помочь поддержать роман диагностики, вакцины и терапевтическое развитие. Синтез кДНК с использованием случайных праймеров позволило обнаружить и сборку геномов из расходящегося, сострахования инфицировании или даже новых вирусов 1,2. Как и с другими объективными методами, нежелательные примеси занимают много секвенирование чтения и негативно повлиять на результаты секвенирования. Хост и поли (РЗ) РНК-носитель являются примеси, присутствующие во многих существующих вирусных коллекций образцов.
Протокол описывает эффективный и экономичный способ глубоких вирусных геномов секвенирование РНК на основе объективной общей Секвенирование РНК. Метод использует РНКазы Н селективного истощения шаг 3 для удаления нежелательных рибосом хозяина и РНК - носитель. Селективный истощение обогащает для вирусного контента (рисунок 1) и улучшает общее качество данных секвенирования(Рисунок 2) из клинических образцов. Кроме того, tagmentation применяется к протоколу, поскольку это значительно сокращает сроки строительства библиотеки. Эти методы были использованы для быстрого генерирования больших наборов данных Эбола и Ласса геномов вируса 2,4,5 и может быть использована для изучения широкого спектра РНК -содержащих вирусов. И, наконец, этот подход не ограничивается образцов человека; Полезность селективного разрушения была продемонстрирована на образцах ткани , собранных из Ласса-инфицированных грызунов и приматов модели заболеваний , отличных от человека 5,6.

Рисунок 1. Общая РНК Содержание Отражает Обогащение Ласса вируса содержимого с помощью Selective Истощение. Запуск общего содержания (вход РНК) и обогащение уникальных вируса Ласса (LASV) читает (содержание библиотеки) при истощении рРНК из девяти различных клинических изолятов. Эта цифра была изменена с 6 . Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
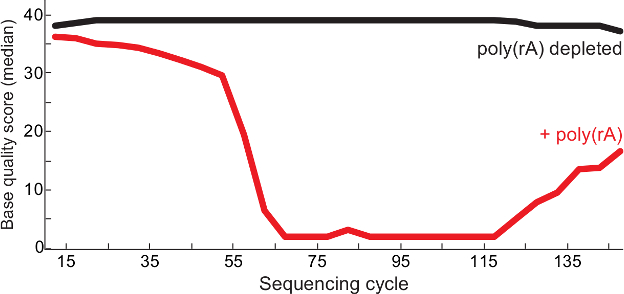
Рисунок 2. Высшее качество Секвенирование После того, как РНК Carrier Истощение. Срединные базовые качества на секвенирования цикла поли (РЗ) -contaminated вируса Ласса библиотеки (красный) и контроль (без носителя наблюдается в библиотеке, черный) из доклада QC 13. Как прочитать 1 и читать 2 парных концевых чтений объединяются в BAM файл библиотеки и оценки качества представлены на каждой базе. Эта цифра была изменена с 6. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Вирусная РНК-сл детали протокола строительство библиотек непосредственно из извлеченныйРНК собирали из клинических и биологических образцов. Для обеспечения личной безопасности, все вирусные сыворотки, плазмы и ткани образцы должны быть инактивируется в соответствующих буферов до экстракции РНК. В некоторых инактивация и извлечения комплектов, несущей поли (гА) РНК входит; это будет удалена во время начального этапа РНКазы Н селективного истощения. На основе полного выздоровления, ожидаемая концентрация РНК-носителя составляет 100 нг / мкл. В протоколе, 110 нг / мкл олиго дТ РНК (1.1x концентрация носителей) используется для истощения. Если поли (гА) носитель не присутствует в образце, то олиго (дТ) не должны быть добавлены до истощения.
Следующий протокол предназначен для 24 реакций в формате ПЦР пластины (до 250 мкл объем). Более ранняя версия этого протокола было сообщено в Matranga, и др. 6.
протокол
Заявление по этике: пациенты лихорадки Ласса были набраны для данного исследования с использованием протоколов, утвержденных комитетами человеческих субъектов в Университете Тулейн, Гарвардский университет, Broad Institute, Irrua специалист по Teaching Hospital (ISTH), больницы Кенема правительства (KGH), Ойо министерства здравоохранения штата, Ибадан , Нигерии и Сьерра-Леоне Министерство здравоохранения. Все пациенты получали аналогичным стандартом медицинской помощи и предложили препарат рибавирин, решили ли они принять участие в исследовании. Для лихорадки Ласса пациентов (LF), лечение рибавирином следовали рекомендуемые в настоящее время руководящие принципы и в целом предложен как только был сильно подозревал LF.
В связи с тяжелой вспышки Эбола вируса болезни (EVD), пациенты не могли быть согласие через наши стандартные протоколы. Вместо использования клинических избыточных образцов от больных EVD была оценена и одобрена институциональными советами в Сьерра-Леоне и в Гарвардском университете. OffiСЕ по вопросам этики и научной экспертизы Комитета по Сьерра-Леоне, Леоне Министерства здравоохранения и санитарии Сьерра и Гарвардской Комитета по использованию прав субъектов предоставили отказ от согласия на последовательность и сделать общедоступный вирусные последовательности, полученные из образцов пациентов и контактных собранные во время вспышки лихорадки Эбола в Сьерра-Леоне. Эти органы также предоставляется использование клинических и эпидемиологических данных для обезличенной образцов, взятых из всех пациентов с подозрением на EVD, получающих помощь в ходе реагирования на вспышки. Сьерра-Леоне Министерство здравоохранения и санитарии также одобрила поставки неинфекционным, небиологических образцов из Сьерра-Леоне в Институте Брода и Гарвардского университета для геномных исследований образцов вспышек.
1. ДНКазы обработка образца РНК (до 55 мкл экстрагированной суммарной РНК в ~ 4 ч)
- Настройка реакции ДНКазы в пластинке 96-луночного ПЦР на льду в шкафу биологической безопасности , как описано в таблице 1 , Шаг 1.1 (общий объем, 70 мкл / лунку). Примечание: мастер микс может быть подготовлен.
- Вихревой аккуратно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
- Инкубируют при 37 ° С в течение 30 мин.
- Очистка с помощью твердой фазе Реверсивный иммобилизация (SPRI) бусинки РНК.
- Теплые РНК бусин до комнатной температуры в течение 30 мин.
- Слегка встряхните РНК шарики бутылку ресуспендирования любые магнитные частицы, которые могут быть урегулированы. Добавить объем 1.8x (126 мкл) РНК бус ДНКазы обработанной РНК (70 мкл), перемешать с помощью пипетки 10 раз и инкубировать в течение 5 мин при комнатной температуре (общий объем в колодезной, 196 мкл).
- Поместите смесь на магнитной станции. Дождитесь решения, чтобы очистить (5 - 10 мин).
- Удалить очищенную решение в то время как на станции пипеткой и выбросьте. В то время как на станции, мыть шарики, покрывая гранулы с 70% этанола и инкубировать в течение 1 мин. Удалить этанола с пипеткой и выбросьте. Повторить в общей сложности двух стирок.
Примечание: Используя именно 70% свежеприготовленный егhanol имеет решающее значение, так как более высокий процент приведет к неэффективному омывания меньшего размера молекул, в то время как <70% -ного этилового спирта может привести к потере образца 7. - Держите тарелку на станции и оставить открытым высохнуть на воздухе. Примечание: Убедитесь, чтобы гранулы полностью высохнуть, пока шарики не начнут трескаться.
- Добавьте 55 мкл нуклеазы без воды к пластине для элюирования РНК. Удалить пластину от станции, чтобы смешать шарики и воду пипеткой тщательно. Примечание: В качестве альтернативы, использовать меньшее количество воды (≤ 10 мкл) для того, чтобы концентрировать тотальную РНК.
- Поместите пластину обратно на станцию. Подождите, пока раствор не станет прозрачным для передачи с помощью пипетки к новым завинчивающейся крышкой трубки для длительного хранения (-80 ° C). Поместите 5 мкл РНК в новой 96-луночного ПЦР планшет для обеднения (этап. 2.4).
- Дополнительно:. Сохранить и разбавить 1 мкл в 19 мкл воды (1:20) для QRT-PCR рРНК (например, 18S, 28S рРНК) (таблица 2) и вирусные маркеры 5
2. Избирательные Истощение рибосомного и несущей РНК из вирусной РНК образца (~ 4 ч)
- Сделать 5x гибридизацию и 10х буфера реакции РНКазы Н и нуклеазы без воды с линейным носителем акриламид , как описано в таблице 1.
- Настройка реакции гибридизации путем объединения РНК с истощением рРНК олигонуклеотидами (таблица 3) и олиго (дТ) на льду в 96-луночный планшет для ПЦР , как описано в таблице 1.
Примечание: мастер микс может быть подготовлен. 50 femtograms (FG) уникального синтетической РНК (ERCCs 8) могут быть добавлены для отслеживания как вирусный процесс секвенирования и потенциального индекса чтения перекрестного загрязнения.- Вихревой аккуратно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
Инкубируют при 95 ° С в течение 2 мин, медленно наращивают до 45 ° C при -0.1 ° С в секунду. Приостановка Термоциклер при 45 ° С.
- Вихревой аккуратно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
- Настройка РНКазы H реакционной смеси на льду, как описаноd в таблице 1, а затем предварительно нагреть при 45 ° С в течение 2 мин. Примечание: мастер микс может быть подготовлен.
- Добавляют РНКазы Н смесь предварительно нагревают до реакции гибридизации в пластине при сохранении пластины в амплификатор при 45 ° С.
- Хорошо перемешать, осторожно пипеткой 6 - 8 раз. Инкубируют при 45 ° С в течение еще 30 мин. Место на льду.
- Настройка реакционной смеси ДНКазы на льду , как описано в Таблице 1 Примечание. Мастер - микс может быть получен.
- Добавить в реакции РНКазы Н в пластине, вихревой нежно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин. Инкубируют при 37 ° С в течение 30 мин.
- Остановка реакции ДНКазы добавлением 5 мкл 0,5 М ЭДТА. Вихревой аккуратно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
- Очистка с помощью РНК-бусинки (этап 1.3), используя объем 1.8x (144 мкл) бусин. Элюции в 11 мкл нуклеазы без воды. Примечание: Для безопасного хранения в холодильнике, хранить обедненный RNА образцы при -80 ° C O / N.
3. Синтез кДНК (~ 6 ч)
- Смешайте рРНК / носитель обедненного РНК со случайными праймерами на льду в 96-луночный планшет для ПЦР , как описано в таблице 1, вихревое осторожно и тщательно, а затем центрифугировать при 280 х г при комнатной температуре в течение 1 мин.
- Нагревают смесь до 70 ° С в течение 10 мин в амплификатор. Сразу после тепловой денатурации, поместите РНК на льду в течение 1 - 5 мин. Не позволяйте РНК стоять (даже на льду) в течение более 5 мин до первой нити реакции.
- Установка для синтеза первой цепи реакции синтеза смесь на льду , как описано в таблице 1.
Примечание: Мастер-микс может быть подготовлен.- Добавить в РНК / случайного праймера в смеси пластины, вихревая осторожно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин. Инкубируют при 22 - 25 ° С в течение 10 мин.
- Инкубируют при 55 ° С в воздушном инкубаторе в течение 60 мин. Поместите пластину на лед, чтобы закончить реакцию. Нее: использование воздушного инкубатора рекомендуется создать постепенное потепление первой нити реакции, в течение которого праймеры отжигают и первая нитка начинает удлиняться.
- Настройка второго прядей реакции синтеза смеси на льду , как описано в таблице 1.
Примечание: Мастер-микс может быть подготовлен.- Добавить к первой цепи реакции синтеза на пластине, вихревое осторожно и тщательно, а затем центрифугировать при 280 х г при комнатной температуре в течение 1 мин. Инкубировать в течение 2 ч при 16 ° С (держать крышку при 25 ° C). Не позволяйте температуре подниматься выше 16 ° C.
- Поместите пластину на льду, а затем инактивируют реакцию добавлением 5 мкл 0,5 М ЭДТА, осторожно и тщательно перемешать, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
- Очистка с бисером ДНК (см шаг 1.3 для протокола) с использованием 1.8x объема (153 мкл) из бисера. Элюции в 9 мкл буфера для элюции (EB). Сохранить 1 мкл для количественного определения. Используйте 1 нг кДНК для subsequeнт шаги. Если концентрация кДНК слишком мала, чтобы обнаружить, использовать 4 мкл кДНК tagmentation (см шаг 4.1).
- Для безопасного хранения в холодильнике, хранить двухцепочечной кДНК при 4 ° CO / N или -20 ° С для длительного хранения.
4. Библиотека Подготовка - ДНК Библиотека Строительство (~ 4 часа)
- Передача 4 мкл кДНК в 96-луночный планшет и сохранить оставшуюся кДНК для второй попытки, если это необходимо.
- Настройка реакции tagmentation на льду , как описано в таблице 1.
Примечание: Мастер-микс может быть подготовлен. Для уменьшения фона и общую стоимость, общий объем реакционной смеси tagmentation уменьшается от 20 до 10 мкл. Поскольку кДНК является ограничивающим фактором, количество банкоматов (т.е. transposome) , используемый в реакции, также уменьшается , чтобы уменьшить число интеграции сайтов.- Добавить tagmentation смесь в кДНК в плите, вихрем мягко и тщательно и центрифуге при 280 мкг (при комнатной температуре) в течение 1 мин.Инкубируют при 55 ° С в течение 5 мин, выдержка при 10 ° С.
- После того, как при 10 ° С, немедленно добавляют 2,5 мкл обезвредить Tagment буфера (NT), чтобы закончить реакцию. Смешайте с помощью пипетки вверх и вниз, а затем центрифуге при 280 мкг (при комнатной температуре) в течение 1 мин.
- Инкубируют при комнатной температуре в течение 5 мин.
- Настройка ПЦР реакции амплификации на льду , как описано в таблице 1.
- Вихревой аккуратно и тщательно, а затем центрифуге при 280 мкг при комнатной температуре в течение 1 мин.
- Выполните ПЦР на амплификатор с использованием условий , описанных в таблице 1.
Примечание: 12 циклов ПЦР Предложены 1 нг кДНК tagmented; Однако, вирусные клинические образцы часто имеют неопределяемый количество кДНК. Для малых количеств кДНК (<1 нг), использовать до 18 циклов ПЦР, чтобы создать достаточно библиотеку для секвенирования.
- Библиотека подготовка - очистка и аккумулирования для секвенирования
- Довести образец до 50 мкл с БЭ.
- Очистка сбусинки ДНК (см шаг 1.3 для протокола) с использованием 0.6x объема (30 мкл) бусин. Элюции в 15 мкл EB.
- Определить концентрацию библиотеки (рисунок 3) путем проведения анализа региона ( от 150 до 1000 пар оснований ) с помощью программного обеспечения Bioanalyzer 9, за исключением димеров праймеров (~ 120 б.п.) из анализа области. Примечание: В качестве альтернативы, КПЦР может быть использован для количественного определения библиотек 10.
- библиотеки бассейн по самой низкой молярной концентрации 1 нМ или больше. Если библиотека находится ниже 1 нанометра, добавить небольшой объем библиотеки до пула (~ объем 1x других библиотек) для сбора информации о последовательности из этих библиотек.
- Cleanup бассейн с бисером ДНК 0.7x, как описано выше (см шаг 2). Элюции в 15 мкл EB. Примечание: Объем бисера будет зависеть от конечного объема бассейна.
- Анализировать бассейн 9. Определить молярную концентрацию путем проведения анализа региона ( от 150 до 1000 пар оснований ) 9. Примечание: В качестве альтернативы, КПЦР может быть использован для количественного определения библиотеки пул 10 </ SUP>.
- Нагрузка секвенсор при концентрации библиотеки 10 пМ для генерации 101 пар оснований , парноконцевое чтение с двойной штрих - код считывает 11.
Рисунок 3. Библиотеки Изготовленные из Эбола вируса клинических образцов. Гель изображение 4 представителя вируса Эбола (EBOV) библиотек. Регионы библиотеки и праймеров димеров показаны. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Результаты
Описанный протокол позволяет поколение высококачественных секвенирования считывает из малозатратных вирусных образцов РНК, обогащая для уникального вирусного контента. Как показано на рисунке 1, протокол , обогащенный уникальный контент вируса Ласса , по крайней мере в пять раз во всех образцах ( по сравнению с не обедненного контроля), по меньшей мере , один миллион копий 18S рРНК (~ 100 пг общей РНК). Точно так же, секвенирование успех также коррелирует с количеством вируса в данном образце. Использование QRT-PCR в качестве суррогата вирусного количества, образцы, которые содержали ~ 1000 или более вирусных копий генома наиболее часто создаются полные сборки (данные не показаны). Кроме того, истощение поли (р.а.) носителя уменьшает гомополимеров последовательности А и Т в библиотеках, что приводит к более чистых препаратов и обеспечивает более высокое качество последовательности операций чтения (рисунок 2). Конечные библиотеки от низкого уровня входных вирусных клинических образцов, часто имеют широкий длины фрагмента от 150 до 1000 пар оснований(Рисунок 3).
После секвенирования для уменьшения выборки и неправильной идентификации перекрестных помех между библиотеками в пуле 12, только индекс считывает с базового показателя качества 25 (Q25) и обеспечить нулевые несовпадений сохраняются в процессе демультиплексирования. Вирусные геномы собираются с помощью биоинформатики трубопровода , специфичную для расходящихся вирусов 2,4-6. Эти инструменты доступны в https://github.com/broadinstitute/viral-ngs или через коммерческие облачных платформ 4.
| Шаг 1.1: ДНКазная реакция | |
| реактив | Объем на реакцию (мкл) |
| 10X буфера ДНКазы | 7 |
| Нуклеазы без воды | 6 |
| Извлеченные вирусной РНК | 55 |
| ДНКазы (2 U / & #181; л) | 2 |
| Общий объем | 70 |
| Шаг 2.1: 5x буфера гибридизации | |
| реактив | Объем по 1 мл (мкл) |
| 5 М NaCl | 200 |
| 1 М Трис-HCl (рН 7,4) | 500 |
| Нуклеазы без воды | 300 |
| Общий объем | 1000 |
| Шаг 2.1: 10x РНКазы H реакционного буфера | |
| реактив | Объем по 1 мл (мкл) |
| 5 М NaCl | 200 |
| 1 М Трис-HCl (рН 7,5) | 500 |
| 1 М MgCl 2 | 200 |
| Нуклеазы без воды | 500 |
| Общий объем | 1000 |
| Шаг 2.1: Вода жIth линейной акриламид | |
| реактив | Объем 1 мл буфера (мкл) |
| Нуклеазы без воды | +992 |
| Линейный акриламид (5 мг / мл) | 8 |
| Общий объем | 1000 |
| Шаг 2.2: реакция гибридизация для селективного разрушения | |
| реактив | Объем на реакцию (мкл) |
| 5x Гибридизация буфера | 2 |
| рРНК-Истощение олиго смесь (100 мкМ) | 1,22 |
| Олиго (d) T (550 нг / мкл) | 1 |
| ДНКазы лечение тотальной РНК | до 5 |
| Spike-РНК (это необязательно) | 0,5 |
| Вода (с линейной акриламид) | довести до 10 всего |
| Итого Voluменя | 10 |
| Шаг 2.3: РНКазы Н реакции для селективного разрушения | |
| реактив | Объем на реакцию (мкл) |
| Буфер реакции 10x РНКазы Н | 2 |
| Вода (с линейной акриламид) | 5 |
| Термостабильная РНКазы Н (5 ед / мкл) | 3 |
| Общий объем | 10 |
| Шаг 2.4: ДНКазная реакция пост селективное истощение | |
| реактив | Объем на реакцию (мкл) |
| 10x ДНКазы буфера | 7.5 |
| Вода (с линейной акриламид) | 44,5 |
| ингибитор РНКазы (20 ед / мкл) | 1 |
| РНКазы ДНКазы I (2,72 ед / мкл) | 2 |
| Общий объем (с реакцией РНКазы Н) | 75 |
| Шаг 3.1: Синтез кДНК, случайным образом праймер гибридизация | |
| реактив | Объем на реакцию (мкл) |
| рРНК / носитель обедненного РНК | 10 |
| 3 мкг случайных праймеров | 1 |
| Общий объем | 11 |
| Шаг 3.2: первой цепи реакции синтеза кДНК | |
| реактив | Объем (мкл) |
| 5x первой цепи буфера реакции | 4 |
| 0,1 М DTT | 2 |
| 10 мМ дНТФ смесь | 1 |
| ингибитор РНКазы (20 ед / мкл) | 1 |
| Обратной транскриптазы (добавить последний) | 1 |
| Общий объем (с РНК выше) | 20 |
| Шаг 3.3: Вторая цепь реакции синтеза кДНК | |
| реактив | Объем (мкл) |
| РНКазы вода | 43 |
| 10x второго Strand буфера реакции | 8 |
| 10 мМ дНТФ смесь | 3 |
| Кишечной палочки ДНК - лигазы (10 ед / мкл) | 1 |
| Кишечной палочки ДНК - полимеразы I (10 ед / мкл) | 4 |
| Кишечная палочка РНКазы H (2 ед / мкл) | 1 |
| Общий объем (с 1 - й реакционную цепь) | 80 |
| Шаг 4.2: реакция Tagmentation | |
| реактив | Объем (мкл) |
| Amplicon Tagment Mix (ATM) | 1 |
| Tagment ДНК Буфер (TD) | 5 |
| Общий объем (кДНК) | 10 |
| Шаг 4.3: Библиотека реакции ПЦР | |
| реактив | Объем (мкл) |
| PCR Master Mix (НПМ) | 7.5 |
| Индекс 1 праймер (i7) | 2.5 |
| Индекс 2 праймера (i5) | 2.5 |
| Общий объем (с tagmented кДНК) | 25 |
| Шаг 4.3.2: Библиотека Условия ПЦР | |
| 72 ° С, 3 мин | |
| 95 ° С, 30 сек | |
| до 18 циклов-10 с при 95 ° С, 30 сек при 55 ° С, 30 сек при 72 ° C | |
| 72 ° С, 5 мин | |
| 10 ° C, навсегда | |
Таблица 1:. Реакция настройки и буферы Шаг за шагом таблицы с содержимым всех буферов и реакционных смесей.
Таблица 2: QRT-ПЦР - праймеры Последовательности праймеров , используемых для измерения хозяина (18S рРНК) и вирусный контент (Эбола и Ласса).. 'KGH' Госпиталь Кенема правительство в Сьерра - Леоне, где праймеры Эбола были испытаны 2. 'Кулеш' является исследователь , который разработал набор праймеров 14.
Таблица 3: рибосомальной РНК (рРНК) Истощение олигонуклеотиды 195 50-нуклеотидные длинные последовательности , комплементарные к человеческому рРНК для селективного истощения шага 6.. Пожалуйста , нажмите здесь , чтобы загрузить этот файл.
| Oligo Имя | Последовательность (5 'к 3') |
| Эбола KGH FW | GTCGTTCCAACAATCGAGCG |
| Эбола KGH Р.В. | CGTCCCGTAGCTTTRGCCAT |
| Эбола КУЛЕШ FW | TCTGACATGGATTACCACAAGATC |
| Эбола КУЛЕШ Р.В. | GGATGACTCTTTGCCGAACAATC |
| Ласса SL FW | GTA AGC CCA НОД GYA AAB CC |
| Ласса SL Р.В. | AAG CCA CAG AAA RCT GGS AGC |
| 18S рРНК FW | TCCTTTAACGAGGATCCATTGG |
| 18S рРНК Р.В. | CGAGCTTTTTAACTGCAGCAACT |
Обсуждение
Изложенная подход позволяет надежный, универсальный, быстрое секвенирование и был использован для секвенирования вируса Эбола во время вспышки 2014 2,4. Объединяя селективное истощение и синтез кДНК со строительством библиотеки tagmentation, общее время процесса был уменьшен на ~ 2 дней с предыдущими методами лигирования адаптеров. Совсем недавно, этот протокол был использован международными сотрудниками и другими с большим успехом 15,16 и будут развернуты в лаборатории в Западной Африке для поддержки местных геномики на основе научных исследований и диагностики 17.
Протокол, описанный здесь, использует случайные праймеров для подготовки кДНК вирусных РНК-сл библиотек. В отличие предыдущих вирусных РНК-сл подходов, она не требует априорного знания последовательности данных или сложного и трудоемкого дизайна праймеров для конкретного вируса или клады. Этот метод может быть применен к любой вирусной РНК образца. Например, он был использован для создания вирусного контента из обоих лихорадку Эболаи образцы Ласса 6. Протокол также может быть использован для хоста транскриптомных, метагеномных и обнаружения патогена секвенирования проектов 1.
Важным шагом протокола направлена РНКазы Н пищеварение, высокую пропускную способность, низкая стоимость метода для удаления нежелательного носителя и хозяина РНК из вирусных образцов. Избирательное стадия истощения протокола использует множество компонентов и требует мастерства и точности. Дополнительное время и следует соблюдать осторожность во время начальной установки.
Поскольку большинство клинических сыворотки и плазмы образцы часто имеют очень мало материала нуклеиновых кислот, загрязнение и потеря образца являются общими. Чтобы избежать этих проблем, особое внимание следует соблюдать осторожность при использовании этого протокола. Во-первых, РНК очень восприимчивы к деградации; поэтому все районы должны быть чистыми и свободными от нуклеаз. Во- вторых, чтобы идентифицировать образцы , пригодные для использования в данном протоколе, QRT-ПЦР для обеих РНК - хозяина и вируса следует использовать для количественной оценки 5,6 . При сравнении ввода суммы с результатами секвенирования из протокола, секвенирование успеха (т.е. генерация достаточных данных для полной вирусной сборки) коррелируют с образцами , которые содержали по меньшей мере , 100 мкг общей РНК и 1000 копий вируса. В-третьих, воздействие окружающей среды источники нуклеиновых кислот, их следует избегать. Протокол указано здесь делается в кабинете биологической безопасности для техники безопасности и для ограничения загрязнителей окружающей среды. Кроме того, наша группа и другие заметили , что коммерческие ферменты могут быть еще одним источником загрязнения бактериальных нуклеиновых кислот в низких входных выборок 6,18. Использование чистого рабочего пространства (например, ПЦР капот, биозащитой) и отрицательный контроль (например, вода или буфер) поможет облегчить и отслеживать загрязнения, соответственно. Для образцов с <100 пг общей РНК, только поли (гА) РНК-носитель, не рРНК, должны быть истощены, чтобы гарантировать результаты секвенирования высокого качества при одновременном ограничении потери материала. Для оченьнизкие входные выборки, методы амплификации кДНК-могут быть более подходящими 19, хотя и поли (гА) носитель должен быть удален до синтеза кДНК.
Истощение хозяина рРНК обогащают для вирусного контента в библиотеках секвенирования и применима к различным коллекциям образцов , включая сыворотки или плазмы, а также несколько типов тканей от грызунов и приматов , отличных от человека 5,6. В нечеловеческих организмов, совместив чтений к 28S рРНК оставались после истощения, предлагая 28S рРНК менее консервативными между людьми и другими видами 6,20. При использовании этого метода с нечеловеческими изолятов, может быть необходимо дополнить с олигонуклеотидами ДНК , комплементарных расходящихся последовательностей рРНК конкретного хоста 3,21.
Поскольку протокол является несмещенной, вирусный читает может представлять лишь малую часть от общего содержания библиотеки. Хотя рРНК является наиболее распространенным видов РНК-хозяина и лишь небольшой процент рРНК (читает0; 1%) обнаружены после селективного истощения, все другие хост - РНК (например, мРНК) остается после того, как истощение и может объяснить многие секвенирование считывает из образца. Поэтому "передискретизации" (т.е. oversequencing) отдельные библиотеки требуется для того , чтобы иметь достаточное освещение для сборки и вирусных вариантов вызовов. Для наших исследований, мы пытаемся последовательность ~ 20 миллионов просмотров на пробу , чтобы иметь достаточную глубину для анализа вирусного генома и связанного вариантов, а также содержание метагеномной 2,5. Для метагеномных и возбудителем исследований обнаружения, важно отметить, что загрязняющие ДНК хозяина удаляют с помощью ДНКазы пищеварения. Поэтому вирусы и другие болезнетворные микроорганизмы, которые содержат геномы ДНК, могут быть потеряны в ходе процесса, однако промежуточные продукты РНК могут все еще быть секвенированы.
Раскрытие информации
The authors have no competing financial interests.
Благодарности
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Материалы
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
Ссылки
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены