Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Микроскопия основе Анализы для высокопроизводительного скрининга на хост факторов, участвующих в
* Эти авторы внесли равный вклад
В этой статье
Резюме
Two assays for microscopy-based high-throughput screening of host factors involved in Brucella infection are described. The entry assay detects host factors required for Brucella entry and the endpoint assay those required for intracellular replication. While applicable for alternative approaches, siRNA screening in HeLa cells is used to illustrate the protocols.
Аннотация
Бруцелл являются факультативными внутриклеточными патогенами , которые инфицируют животных в их естественных хозяев. Передача инфекции человеку чаще всего вызывается при непосредственном контакте с инфицированными животными или при употреблении зараженной пищи и может привести к серьезным хроническим инфекциям.
Brucella могут вторгнуться профессиональные и непрофессиональные клетки фагоцитоза и размножается в пределах эндоплазматической сети (ER) -derived вакуолей. Факторы , принимающие необходимые для ввода Brucella в клетки - хозяева, предотвращение деградации лизосомной и репликации в ER-подобные отделения остаются неизвестными. Здесь мы опишем два анализов для выявления факторов хозяина , участвующих в записи Brucella и репликации в клетках HeLa. Протоколы описывают использование РНК-интерференции, в то время как альтернативные методы скрининга могут быть применены. Анализы основаны на обнаружении флуоресцентно меченных бактерий в флуоресцентно меченных клетках-хозяевах с использованием автоматизированныхширокого поля микроскопии. Флуоресцентные изображения анализируются с использованием стандартизированного трубопровода для анализа изображений в CellProfiler, который позволяет одноклеточной на основе инфекции скоринг.
В анализе конечных точек, внутриклеточный репликации измеряется через два дня после инфицирования. Это позволяет бактериям трафика на их репликативной нишу , где распространение инициируется около 12 часов после того, как бактериальная запись. Бруцелл , которые успешно создали внутриклеточный нишу, таким образом , сильно пролиферируют внутри клеток - хозяев. Так как внутриклеточных бактерий значительно превышает количество индивидуальных внеклеточные или внутриклеточные нерепликативная бактерии, штамм конститутивно экспрессирующих GFP может быть использован. Сильный сигнал GFP, затем используется для идентификации зараженных клеток.
В отличие от этого, для анализа начального необходимо проводить различие между внутриклеточным и внеклеточным бактерий. Здесь используется кодирующий штамм для тетрациклина индуцируемого GFP. индукционныйиз GFP с одновременной инактивацией внеклеточных бактерий гентамицин позволяет дифференцировать внутриклеточным и внеклеточным бактерий на основе сигнала GFP, с только внутриклеточных бактерий в состоянии выразить GFP. Это позволяет надежное детектирование единичных внутриклеточных бактерий, прежде чем внутриклеточный пролиферации инициируется.
Введение
Бруцелл являются грамотрицательные, факультативные внутриклеточные возбудители , принадлежащие к классу альфа-протеобактерий. Они вызывают выкидыши и бесплодие в их естественных хозяев, таких как крупный рогатый скот, коз, овец или приводит к серьезным экономическим потерям в эндемичных районах. Бруцеллез является одним из самых важных зоонозных заболеваний во всем мире , вызывая более полумиллиона новых человеческих инфекций ежегодно 1. Передача к человеку чаще всего вызывается при непосредственном контакте с инфицированными животными или при употреблении зараженной пищи, такие как непастеризованного молока. Симптомы лихорадочного заболевания неспецифичны, что вызывает трудности в диагностике бруцеллеза. При отсутствии лечения у пациентов может развиться хроническая инфекция с более тяжелыми симптомами , такими как артрит, эндокардит и невропатии 2.
На клеточном уровне бруцелл способен к вторжению фагоцитоза и не фагоцитарной клетки и реплицируется в внутриклеточнымкупе известна как Brucella катио- вакуоли (BCV). Интернализации бактерий требует цитоскелета перестроек с помощью Rac, Rho и прямой активации Cdc42 3. Внутри эукариотической клетки - хозяина, то BCV трафиков вдоль эндоцитотического пути и , несмотря на взаимодействие с лизосом, бактерии удается избежать деградации 4. Подкисление BCV по везикулярного АТФазы требуется , чтобы индуцировать экспрессию системы секреции IV типа (бактериальный T4SS) 5. Считается , что бактериальные эффекторы , секретируемые T4SS имеют важное значение для бруцелл , чтобы установить свою нишу репликативной, так как делеции T4SS 6 или ингибирования везикулярного АТФазы приводят к дефектам в создании внутриклеточного ниши 7. Бактерии не размножаются во время фазы торговли, пока они достичь ЭР-производных вакуолярной отсек 8. После того, как происходит внутриклеточное пролиферации, то BCV находится в слОСЭ ассоциации с маркерами ER , таких как калнексину и глюкозо-6-фосфатазы 6.
Молекулярные механизмы , с помощью которых Brucella проникает в клетку, позволяет избежать деградации лизосомами, и , наконец , размножается в ER-подобные отделения остаются неизвестными. Факторы хозяина , участвующие в различных стадиях инфекции главным образом были определены целевые подходы или малых интерферирующих РНК экрана малого масштаба (миРНК) в исполнении Drosophila клеток 9. Они проливают свет на вклад отдельных факторов хозяина во время Brucella инфекции , но мы все еще далеки от полного понимания всего процесса.
Здесь, протоколы, позволяющие идентифицировать факторов хозяина человека с использованием крупномасштабной РНК-интерференция (RNAi) скрининг в сочетании с автоматизированной широким полем флуоресцентной микроскопии и анализа изображения в автоматическом режиме представлены. Обратный миРНК трансфекции клеток HeLa проводили как describред ранее 10,11 с незначительными изменениями. Анализ конечных точек покрывает большую часть внутриклеточного жизненного цикла Brucella , за исключением выхода и инфекции соседних клеток. Для дополнительной характеристики хитов, выявленные при анализе конечных точек, модифицированный протокол с целью выявления факторов, участвующих в ранних стадиях инфекции используется.
Выкидыш штамм бруцелл , который конститутивно экспрессирует GFP используется для анализа конечных точек , где бактерии пoлучают инфицировать клетки в течение двух дней. За это время, бактерии проникают в клетки, трафик в ЭР-производных репликативной нишу, и реплицировать в пери-ядерном пространстве. Высокие уровни сигнала GFP может быть использован, чтобы надежно обнаружить отдельные клетки, которые содержат реплицирующиеся бактерии.
Для включения в исследование бруцелл в тесте с высокой пропускной способностью , важно , чтобы иметь возможность различать внутриклеточной и внеклеточной бактерий. Изложенный здесь метод circumvЭНЦ дифференциальное окрашивание антитела внутриклеточной и внеклеточной бактерий. Он основан на штамма Brucella , выражающей Тетрациклин-индуцируемых GFP в сочетании с конститутивной экспрессии DsRed. Присутствие конститутивным DsRed маркера позволяет идентифицировать все бактерии, которые присутствуют в образце. выражение GFP индуцируется добавлением нетоксичного тетрациклина аналогового anhydrotetracycline (АТЦ) одновременно с инактивацией внеклеточных бактерий гентамицина (Gm). В то время как клетка-непроницаемой антибиотик Gm убивает внеклеточные бактерии, АТГ могут проникать в клетку-хозяина и индуцирует экспрессию GFP избирательно в внутриклеточных бактерий. Этот двойной репортер позволяет надежное разделение отдельных внутриклеточных бактерий (GFP и DsRed сигналов) от внеклеточных бактерий (только DsRed сигналов) с использованием широкого поля микроскопии. Для того , чтобы достичь обнаруживаемого экспрессии GFP с помощью внутриклеточной Brucella мы обнаружили , что 4 ч индукции AtC приводит к RELIсостоянии сигнала. Подобные схемы индукции выборочно выражающие GFP в внутриклеточных бактерий был использован ранее для изучения внутриклеточных Shigella 12.
протокол
Примечание: Все работы с живыми Аборт штаммов бруцелл должны быть выполнены в лаборатории уровня биологической безопасности 3 (BSL3) с учетом всех необходимых предписаний и инструкций по технике безопасности.
1. Приготовление экранирующих пластин и Культивирование бактерий и клеток
- Приготовление Brucella выкидыш заквасок
- Серия Brucella выкидыш 2308 (Б. Аборт) от -80 ° C молока на складе триптический соевый агар (TSA) пластину , содержащую 50 мкг / мл канамицина (АСП / км). Инкубируйте пластины в течение 3-4 дней при 37 ° С. Использование штамма Brucella выкидыш 2308 pJC43 (apHT :: GFP) 13 для анализа конечных точек и штамма Brucella выкидыш pAC042.08 (apht :: DsRed, Teto :: тетр-GFP) для анализа ввода.
Примечание: Гладкая липополисахарида (LPS) бруцелл Аборт является важным фактором , определяющим вирулентность , но грубые мутанты могут происходить при относительно высоких частотах 14. Wе, таким образом, рекомендуется протестировать статус штамма культуры перед началом с этого эксперимента. - Restreak бактерии на четырех АСП / км пластин покрывают полную тарелку и инкубировать в течение 2-3 дней при 37 ° С.
- Использование одноразовой пластиковой петли, передачи и ресуспендирования бактерий из пластин TSA / км в 10 мл 10% автоклавного обезжиренного молока. Убедитесь в том, что бактерии хорошо ресуспендировали для обеспечения последовательности бактериальных концентраций во всех заквасок.
- Аликвотные 250 мкл бактериальной суспензии в 2 мл пробирки с завинчивающейся пробкой и замораживают при -80 ° С.
- Оттепель аликвоту закваски и Перенесите 25 мкл, 50 мкл и 100 мкл бактериальной культуры стартера, чтобы отделить 250 мл с завинчивающейся пробкой бутылки с 50 мл трипсиновый соевый бульон (TSB), содержащий 50 мкг / мл канамицина (ТСБ / км) , Печать бутылки с парафильмом.
- Выдержите бутылки на ночь на орбитальном шейкере при 100 оборотах в минуту и температуре 37 ° С.
- Измерьте оптическую плотность при 600 Нм (OD 600) , чтобы определить объем закваски , необходимой для достижения OD 600 = 0,8-1,1 после культивирования в течение ночи.
- Серия Brucella выкидыш 2308 (Б. Аборт) от -80 ° C молока на складе триптический соевый агар (TSA) пластину , содержащую 50 мкг / мл канамицина (АСП / км). Инкубируйте пластины в течение 3-4 дней при 37 ° С. Использование штамма Brucella выкидыш 2308 pJC43 (apHT :: GFP) 13 для анализа конечных точек и штамма Brucella выкидыш pAC042.08 (apht :: DsRed, Teto :: тетр-GFP) для анализа ввода.
- Получение клеток HeLa
- Рост клеток HeLa в Дульбекко модификации Дульбекко (DMEM) , дополненной 10% фетальной телячьей сыворотки (FCS) (DMEM / 10% FCS) во влажном инкубаторе при 37 ° C с 5% CO 2. Рост клеток до 80% сплошности.
- Подготовьте экранирующих пластин с миРНК.
- Развести киРНК в РНКазу воде до конечной концентрации 0,32 мкМ. Передача 5 мкл / лунку в черные четко и плоским дном 384-луночные планшеты.
- Используйте столбцы 1, 2, 23 и 24 для стандартных элементов управления. Для отрицательных контролей, используйте ненацеливании миРНК (закодированная) и имитировали скважин без миРНК (трансфекция только реагента). Для управления трансфекции используют Серна , что вызывает гибель клеток (KIF11), а также в качестве положительных контролей известных факторов хозяина , участвующих в Brucella инфекции (например, ARPC3,компонент Arp2 / 3 комплекса, участвующего в полимеризации актина). Передача коммерчески доступные библиотеки миРНК или пользовательские миРНК в остальные лунки.
- Уплотнительные пластины с отслаиваемыми алюминиевой фольги. Хранить планшеты при -20 ° C.
Примечание: Плиты можно хранить при температуре -20 ° С в течение не менее 6 месяцев и до года, в зависимости от рекомендаций производителя.
- Развести киРНК в РНКазу воде до конечной концентрации 0,32 мкМ. Передача 5 мкл / лунку в черные четко и плоским дном 384-луночные планшеты.
2. Обратный миРНК Трансфекция
- Растаяйте черный 384-луночный планшет центрифугированием при 300 х г в течение 20 мин при комнатной температуре.
Примечание: Это приведет к снижению всю жидкость, которая может прилипать к крышке или боковой лунках. - Подготовьте трансфекция среду путем разбавления реагента для трансфекции 1: 200 в среде DMEM без FCS при комнатной температуре. Приготовить трансфекции среду не более чем за 20 мин до использования. Тщательно перемешать раствор перед использованием.
- Добавьте 25 мкл среды для трансфекции в каждую лунку с помощью дозатора реагентаи смешивать решения, перемещая пластину назад и вперед.
- Инкубируйте планшет в течение 1 ч при комнатной температуре, чтобы позволить миРНК / трансфекция реагента комплексообразования.
- В то же время, подготовить клетки HeLa путем промывки суб-сливающийся клетки в 75 см 2 колбу один раз с 2,5 мл 0,05% трипсин-ЭДТА в PBS (трипсин).
- Добавить 1,5 мл свежего Трипсин и передать колбу до 37 ° С в течение 2-3 мин до тех пор, пока клетки не окружать.
- Ресуспендируют клеток в 10 мл подогретого DMEM / 16% FCS.
- Граф клеток с использованием автоматического счетчика клеток и подготовить клеточную суспензию 10000 клеток / мл в DMEM / 16% FCS.
- Добавить приостановку 50 мкл клеток в каждую лунку с помощью дозатора реагента, что приводит к 500 клеток / лунку.
- Переместить пластину назад и вперед, чтобы достичь равномерного распределения клеток. Оставьте пластину при комнатной температуре в течение 5-10 мин, чтобы позволить клеткам осесть.
- Уплотнение пластины с парафильмом и инкубировать ее в течение 72 ч на подогретого алюминияпластины в 37 ° C влажной инкубаторе с 5% CO 2.
Примечание: предварительно нагретом алюминиевая пластина позволяет равномерное распределение температуры по всей 384-луночного планшета.
3. Заражение и фиксация
- За один день до инфицирования, прививают суммы , определенной в 1.1.7 В. выкидыш стартер культуры в 50 мл среды TSB / км в 250 мл завинчивающейся крышкой бутылки. Уплотнение бутылку с парафильмом и расти бактерии в течение ночи при 37 ° С и 100 оборотов в минуту на орбитальном шейкере до OD 600 = 0,8-1,1.
- Мера OD 600 бактериальной культуры и подготовить инфекции среду путем разведения бактериальной культуры в среде DMEM / 10% FCS для достижения желаемой кратности инфекции (MOI).
Примечание: Для того, чтобы получить уровень инфекции 5-10% в клетках HeLa, А.Н. MOI 10000 используется. Кривая титрования MOI должна быть выполнена для каждого типа клеток, чтобы получить оптимальные уровни инфекции. - Заменить трансфекции среду 384-луночного планшетас 50 мкл среды инфекции с использованием автоматизированной промывки планшетов.
- Центрифуга 384-луночный планшет при 400 х г и 4 ° С в течение 20 мин.
Примечание: Это помогает синхронизировать процесс заражения. - Уплотнение пластины с Parafilm и инкубировать ее на подогретого алюминиевой пластины во влажном инкубаторе при 37 ° C с 5% CO 2 в течение 4 часов.
- Промывают клетки DMEM / 10% FCS, содержащей 100 мкг / мл Gm инактивировать внеклеточных бактерий с использованием автоматизированной промывки планшетов (используют 200 мкл / лунку перелива промывку).
Примечание: Во время стирки перелива, автоматизированная машина одновременно пластины дозирует и всасывает среду. - Для анализа на вход, промыть клетки второй раз с DMEM / 10% FCS, содержащей 100 мкг / мл Gm и 100 нг / мл AtC с использованием автоматизированной промывки планшетов (используют 200 мкл / лунку перелива промывку).
- Уплотнение плита с парафильмом и вернуть его в предварительно нагретой алюминиевой пластины во влажном инкубаторе при 37 ° C с 5% CO 2 для другого40 ч или 4 ч, для анализа конечных точек и анализа ввода, соответственно.
- Для фиксации, промыть лунки PBS с использованием автоматизированной промывки планшетов (используют 200 мкл / лунку перелива промывку).
- Обмен PBS с 50 мкл 3,7% параформальдегида в 0,2 М HEPES при рН 7,4 (PFA) с использованием автоматизированной промывки планшетов.
Примечание: Внимание: PFA токсичен при вдыхании, при контакте с кожей и при проглатывании. - Инкубировать в течение 20 мин при комнатной температуре.
- Заменить фиксации среду с 50 мкл PBS с использованием автоматизированной промывки планшетов. Примечание: Образцы теперь готовы быть вывезены из BSL3, если это необходимо.
Внимание: Удаление любого образца из BSL3 объекта подлежит оценке риска и проверки процедуры и зависит от применяемых правил биологической безопасности.
4. Окрашивание
- Промыть клетки дважды с 50 мкл PBS.
- Проницаемыми клеток в 50 мкл 0,1% Тритон-Х-100 в PBS в течение 10 мин.
- Wзольные клетки три раза PBS. Добавьте 20 мкл ФБР, содержащем 1 мкг / мл DAPI (4 ', 6-диамидино-2-фенилиндол) и инкубируют в течение 30 мин при комнатной температуре.
- Мыть клетки три раза в PBS и защищают от света, окрашивание с алюминиевой фольгой.
5. обработки изображений
- Настройка автоматизированной широкого поля микроскоп для получения изображения.
Примечание: Установки для молекулярного ImageXpress Устройство микроскопа с использованием программного обеспечения MetaXpress описаны ниже. Альтернативные 2D-микроскопические устройства широким полем с соответствующими спецификациями аппаратных и программных средств обработки изображений можно использовать (см Таблицу материалов / оборудования для деталей).- Выберите цель 10X, камеры биннинга = 1, коэффициент усиления = 1.
- Выберите формат пластины, соответствующую 384-луночного планшета.
- Приобретать 9 сайтов на лунку.
- Используйте "Включить лазер на основе фокус" и "Фокус на тарелку и хорошо дно".
- Набор "Первый хорошо" в качестве исходной скважины длянайти образец и "Все сайты" для сайта автофокусировкой.
- Используйте канал DAPI вручную установить Z-Offset для фокусировки.
- Вручную выбрать Z-Offset от DAPI для всех остальных каналов.
- Вручную исправить время экспозиции для всех каналов, чтобы обеспечить широкий динамический диапазон с низкой передержки.
- Приобретать изображение репрезентативной сайта с помощью функции автоэкспозиции. Используйте это время экспозиции изображения 3-5 местах по всей пластине и регулировать время экспозиции вручную, пока самые яркие пиксели не достигают ~ 80% от максимальной яркости по всем узлам.
- Изображение полной пластины, используя каналы DAPI и GFP для анализа конечной точки. Для анализа ввода выберите RFP канал в дополнение.
6. Автоматический анализ изображений
Примечание: CellProfiler 2 15 используется в качестве программного обеспечения для анализа изображений в сегменте сотовой связи и бактериальных объектов и выполнять автоматизированные измерения дЗЛЫ в пределах определенных объектов. Программное обеспечение предоставляет алгоритмы анализа изображений в отдельных модулях, которые могут быть объединены в трубопровод, который будет выполнять модули последовательно на всех изображениях, для автоматического выполнения конкретной задачи анализа изображений. Для того, чтобы следовать протоколу, установите CellProfiler 2.1.1 или более новую версию. Затем загрузите прилагаемый трубопровод и следуйте инструкциям ниже, чтобы настроить необходимые параметры в модулях. Описание отдельных модулей всех трубопроводов можно найти в дополнительных файлов.
Примечание: Две отдельные трубопроводы используются для каждого анализа. Первый трубопровод вычисляет модель затенения, которая используется второй трубопровод для коррекции изображений до анализа. коррекция затенения применяется к изображениям, чтобы уменьшить влияние неоднородного светового пути от микроскопа. Вычислительная модель затенения в первом трубопроводе представляет собой длительный процесс, но результаты будут более высокой точности.
- Анализ конечных точек
- Вычислить модель затенения
- Перейдите в меню "Файл" выберите "Импорт" → "Трубопровод из файла ..." и выберите предоставленный трубопровод "BrucellaShadingCorrectionPart1-Ver007". На вопрос о том, чтобы преобразовать устаревший трубопровод, ответ "Не преобразовывать".
- Перейти к "Выход" → "Настройки" Просмотр вывода и выберите входную папку с изображениями и выходную папку для полученных моделей затенения.
- В модуле (1) "LoadImages", отрегулируйте название "Текст, что эти образы имеют в общем", чтобы соответствовать имена изображений.
- Убедитесь, что ни один из модулей не показывают их отображение перед запуском полный анализ отключив все символы глаз рядом с модулями. Примечание: Это значительно снижает необходимые вычислительные ресурсы при обработке трубопровода.
- Нажмите кнопку "Анализ изображений", чтобы начать вычисление модели затенения.
- Выполнить анализ изображений
- Перейдите в меню "Файл" выберите "Импорт" → "Трубопровод из файла ..." и выберите предоставленный трубопровод "BrucellaEndpointWithShadingCorrectionPart2-Ver007". На вопрос о том, чтобы преобразовать устаревший трубопровод, ответ "Не преобразовывать".
- Перейти к "Выход" → "Настройки" Просмотр вывода и выберите входную папку с изображениями и выходную папку для результирующего таблицы.
- В модуле (1) "LoadImages", настроить имя "Текст, что эти образы имеют в общем", чтобы соответствовать имена изображений.
- В модуле (2) "LoadSingleImage", загрузите модели затенения, рассчитанные в соответствии с 6.1.1, выбрав расположение и имена файлов двух моделей затенения.
- В модулях (4) - (5) "ImageMath", установите параметр "Умножим первое изображение путем" согласования битовой глубины камеры микроскопа. Для 8 и 16 битизображения установите параметр "1.0", для 12 битных изображений установите параметр "16,0".
- В модуле (9) "ImageMath" настроить параметр "Умножить второе изображение с помощью" до значения (начать с 0.25) , который будет подавлять сигнал Brucella в окрашивании DAPI , не подавляя ядер.
- Нажмите кнопку "Mode Start Test", затем активировать символ паузы и функцию отображения для модуля (9), нажав на соответствующие иконки модуля.
Примечание: Символ паузы будет появляться в желтый цвет, в то время как символ глаз покажет открытый глаз, если активирован. - Нажмите кнопку "Выполнить", чтобы запустить анализ до модуля (9) с символом активной паузы. Чтобы проверить значения для модуля, нажмите "Step".
- Щелкните правой кнопкой мыши на недавно открывшийся изображения и установить "контраст изображения", чтобы "Log нормализуется". Многократно шаг назад к модулю (9), установите параметр "Умножить второе изображение с помощью", и перешагивать тон модуль еще раз, до тех пор пока сигнал бруцелл не будет полностью подавлен в то время как в то же самое время черные области , которые указывают на чрезмерно вычитании сведены к минимуму в результате изображения.
- Нажмите кнопку "Mode Start Test", затем активировать символ паузы и функцию отображения для модуля (9), нажав на соответствующие иконки модуля.
- В модуле (10) "IdentifyPrimaryObjects", установите параметр "Нижняя граница порога" достаточно высоко (начать с нуля), так что ядра и сегментированный фон игнорируется.
- Для того, чтобы определить хорошее значение для модуля (10), шаг за модуль и отобразить входное изображение, как описано в шагах 6.1.2.6.1 и 6.1.2.6.2.
- Щелкните правой кнопкой мыши на изображение ввода и отображения гистограммы.
Примечание: Пик гистограммы, как правило, указывает на то фон. - Начиная с интенсивности фона увеличить параметр до хорошей сегментации ядер не будет достигнуто, как показано в выходном изображении.
Примечание: Установка порога выше, чем интенсивность фона особенно важно для пустых сайтов.
- В модуле (15)4; FilterObjects ", установите параметр" Минимальное значение " , так что клетки с четко видимой бруцелл в ядре сохраняются, а все остальные отфильтровываются На этом этапе, игнорировать Brucella вне ядра..
- Для того, чтобы определить хорошее значение, шаг за модуль и отображения выходного изображения, как описано в шагах 6.1.2.6.1 и 6.1.2.6.2. Начать с нуля, который идентифицирует все клетки как инфицированные, и увеличивать значение малыми шагами , пока только клетки с четко видимой бруцелл на ядре сохраняются.
- В модуле (16) "FilterObjects", установите параметр "Минимальное значение" , так что клетки с четко видимой бруцелл в то perinucleus сохраняются, а все остальные клетки отфильтровывают. С помощью такой же подход, что и в предыдущем модуле.
- В модуле (17) "FilterObjects", установите параметр "Минимальное значение" , так что клетки с четко видимой бруцелл на Ворonoi тела клетки сохраняются, а все остальные клетки отфильтровывают. С помощью такой же подход, что и в предыдущем модуле.
Примечание: Тело клетки Вороного является радиальная протяженность ядра на 25 пикселей, не перекрывается с соседними телами Вороного клеток. - Выход из режима тестирования и убедитесь, что ни один из модулей не показывают их отображение перед запуском полный анализ отключив все символы глаз рядом с модулями.
Примечание: Это значительно снижает необходимые вычислительные ресурсы при обработке трубопровода. - Нажмите кнопку "Анализ изображений", чтобы начать анализ.
- Когда анализ закончен, инспектировать полученные изображения в формате PNG, а также лист CSV, чтобы гарантировать, что анализ надежно работал по всей пластине.
- Вычислить модель затенения
- Вступление Анализ
- Вычислить модель затенения
- Перейдите в меню "Файл" выберите "Импорт" → "Трубопровод из файла ..." и выберите обеспечитьd трубопровода "BrucellaShadingCorrectionPart1-Ver007". На вопрос о том, чтобы преобразовать устаревший трубопровод, ответ "Не преобразовывать".
- Перейти к "Выход" → "Настройки" Просмотр вывода и выберите входную папку с изображениями и выходную папку для полученных моделей затенения.
- В модуле (1) "LoadImages", настроить имя "Текст, что эти образы имеют в общем", чтобы соответствовать имена изображений.
- Убедитесь, что ни один из модулей не показывают их отображение перед запуском полный анализ отключив все символы глаз рядом с модулями.
Примечание: Это значительно снижает необходимые вычислительные ресурсы при обработке трубопровода. - Нажмите кнопку "Анализ изображений", чтобы начать вычисление модели затенения.
- Выполнить анализ изображений
- Перейдите в меню "Файл" выберите "Импорт" → "Трубопровод из файла ..." и выберите Providред трубопровода "BrucellaEntryWithShadingCorrectionPart2-Ver007". На вопрос о том, чтобы преобразовать устаревший трубопровод, ответ "Не преобразовывать".
- Перейти к "Выход" → "Настройки" Просмотр вывода и выберите входную папку с изображениями и выходную папку для результирующего таблицы.
- В модуле (1) "LoadImages", настроить имя "Текст, что эти образы имеют в общем", чтобы соответствовать имена изображений.
- В модуле (2) "LoadSingleImage", загрузите модели затенения, рассчитанные в соответствии с 6.2.1, выбрав расположение и имена файлов двух моделей затенения.
- В модулях (4) - (5) "ImageMath", установите параметр "Умножим первое изображение путем" согласования битовой глубины камеры микроскопа. Для 8 и 16-битные изображения установите параметр "1.0", 12 битных изображений установите параметр "16,0".
- В модуле (6) "IdentifyPrimaryObjects", установите параметр "LoWER ограничение на порог "достаточно высока, что только ядра на сегменты и фон игнорируется.
- Для того, чтобы определить хорошее значение для модуля (6), шаг над модулем и отобразить входное изображение, как описано в шагах 6.1.2.6.1 и 6.1.2.6.2.
- Щелкните правой кнопкой мыши на изображение ввода и отображения гистограммы.
Примечание: Пик гистограммы, как правило, указывает на то фон. - Начиная с интенсивности фона увеличить параметр до хорошей сегментации ядер не будет достигнуто, как показано в выходном изображении.
Примечание: Установка порога выше фона имеет важное значение для пустых сайтов.
- В модуле (8) "IdentifyPrimaryObjects", установите параметр "Нижняя граница порога" достаточно высоко , что только Brucella на сегменты и фон игнорируется.
- Для того, чтобы определить хорошее значение для модуля (8), шаг над модулем и отобразить входное изображение, как описано в шагах 6.1.2.6.1 и 6.1.2.6.2.
- Щелкните правой кнопкой мыши на изображение ввода и отображения гистограммы.
Примечание: Пик гистограммы, как правило, указывает на то фон. - Начиная от интенсивности фона увеличьте значение параметра до хорошо сегментации бруцелл , как показано на выходном изображении не будет достигнута.
Примечание: Установка порога выше фона имеет важное значение для пустых сайтов.
- В модуле (11) "FilterObjects", установите параметр "Минимальное значение" , так что фон и артефакты отфильтровывают, и только бруцеллы колонии сохраняются.
- Для того, чтобы определить хорошее значение, шаг за модуль и отображения выходного изображения, как описано в шагах 6.1.2.6.1 и 6.1.2.6.2. Начните с выбранного значения в 6.2.2.7 и ступенчато увеличивать его , пока только объекты бруцеллы не сохраняются.
- Выход из режима тестирования и убедитесь, что ни один из модулей не показывают их отображение перед запуском полный анализ с помощью uncheCKING все символы глаз рядом с модулями.
Примечание: Это значительно снижает необходимые вычислительные ресурсы при обработке трубопровода. - Нажмите кнопку "Анализ изображений", чтобы начать анализ.
- Когда анализ закончен, инспектировать полученные изображения в формате PNG, а также лист CSV, чтобы гарантировать, что анализ хорошо работал по всей пластине.
- Вычислить модель затенения
7. Инфекция Scoring
Примечание: миРНК, которые оказывают существенное влияние на жизнеспособность клеток должны рассматриваться с осторожностью, так как это может способствовать ложноположительные открытий. Измененная число клеток влияет на фактическую MOI и адресности важных генов могут иметь плейотропных воздействие на возбудителя инфекции. В то время как неполное истощение путем киРНК позволяет для изучения важных генов, такие цели должны быть подтверждены альтернативными методами (например, фармацевтические помехи) , чтобы подтвердить свою роль в качестве факторов хозяинаво время инфекции.
- Анализ конечных точек
- Откройте лист CSV сгенерированный на этапе 6.1.2.12 и рассчитать на лунку уровень заражения путем деления числа инфицированных клеток (CellProfiler счётчика: "Count_InfectedCells") от общего количества клеток (CellProfiler отсчета: "CountNuclei"). Если несколько сайтов на лунку были отображены, сначала суммировать все сайты для каждой скважины, построить за скважине количество зараженных клеток и за а общее количество клеток.
- Выполните Z балльной нормализации для учета пластины к пластине вариаций, если пластины содержат достаточное количество не пострадавших миРНК. Предположим, достаточное количество не хит миРНК на пластину, если полные библиотеки экранированы.
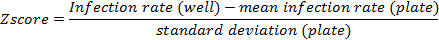
- Вступление Анализ
- Откройте лист CSV сгенерированный на этапе 6.2.2.10 и рассчитать на лунку уровень заражения путем деления числа инфицированных клеток (CellProfiler отсчет: "Count_InfectedCeЛЛС ") от общего числа клеток (CellProfiler отсчет:". CountNuclei ") Если несколько сайтов на лунку были отображены, сначала суммировать все сайты для каждой скважины, построить за-а количество инфицированных клеток и за хорошо общее количество ячеек.
- Для количественной оценки бактериальной нагрузки инфицированных клеток, оценить среднюю площадь, занимаемую внутриклеточных бактерий в инфицированной клетке. С этой целью разделить объединенную площадь всех внутриклеточных бактерий перекрывающихся с клетками (CellProfiler считывания: "AreaOccupied_AreaOccupied_TrueIntPathogenInCells") по количеству инфицированных клеток в этом месте. Для учета артефактов, использовать медиану всех участков этого отсчета как считывании скважины.
Примечание: Если этот анализ используется в качестве последующих мер анализа , содержащего большое количество генов , участвующих в Brucella инфекции, не применяются Z оценка нормализации. Вместо этого, выполнить нормализацию с использованием фиктивных скважин в качестве эталона для учета изменения пластины к пластине.
Результаты
На фиг.1А показан пример анализа изображения , используемого для автоматического определения инфицированных клеток в анализе конечных точек. были идентифицированы Ядра клеток HeLa, окрашенных DAPI, были рассчитаны околопубертатном ядро 8 пикселей в ширину, о?...
Обсуждение
Bacterial pathogens have evolved numerous strategies to manipulate eukaryotic host cells to their benefit. Pathogens causing acute infections often show rapid proliferation which is accompanied by significant alarming of the immune system and loss of viability of infected cells. In contrast, Brucella and other pathogens that cause chronic infections manage to establish long-lasting interactions within host cells. Therefore, bacteria need to fine tune host cell functions to their benefit without disrupting cellul...
Раскрытие информации
The authors declare no conflicts of interest.
Благодарности
This work was supported by grants 51RT 0_126008 and 51RTP0_151029 for the Research and Technology Development (RTD) project InfectX and TargetInfectX, respectively, in the frame of SystemsX.ch, the Swiss Initiative for Systems Biology. We acknowledge grant 310030B_149886 from the Swiss National Science Foundation (SNSF). Work of S.H.L and A.C. was supported by the International PhD Program "Fellowships for Excellence" of the Biozentrum. Simone Muntwiler is acknowledged for technical assistance. We would like to thank Dirk Bumann for providing pNF106 and Jean Celli for pJC43 and pJC44.
Материалы
| Name | Company | Catalog Number | Comments |
| Tryptic Soy Agar (TSA) | BD | 236950 | |
| Tryptic Soy Broth (TSB) | Fluka | 22092 | |
| Kanamycin sulfate | Sigma-Aldrich | 60615 | |
| Skim milk | |||
| 250 ml screw cap bottle | Corning | 8396 | |
| DMEM | Sigma-Aldrich | D5796 | |
| Fetal Calf Serum (FCS) | Gibco | 10270 | Heat-inactivated |
| Trypsin-EDTA (0.5%) | Gibco | 15400-054 | 10x stock solution, dilute 1:10 in PBS |
| Scepte 2.0 Cell Counter | Merck Milipore | PHCC20060 | Alternative cell counting devices can be used |
| Greiner CELLSTAR 384-well plate | Sigma-Aldrich | M2062 | |
| Peelable aluminum foil | Costar | 6570 | |
| Reagent dispenser: "Multidrop 384 Reagent Dispenser" | Thermo Scientific | 5840150 | Alternative reagent dispenser can be used. |
| Transfection reagent: "Lipofectamine RNAiMAX | Invitrogen | 13778-150 | |
| Automated plate washer: "Plate washer ELx50-16" | BioTek | ELX5016 | This plate washer contains a 16-channel manifold suitable for 384-well plates. It fits into a biosafety cabinet and has a lid covering the plate during washing which reduces the risk of aerosol production. Alternative plate washers with similar features could be used. |
| Gentamicin | Sigma-Aldrich | G1397 | |
| Anhydrotetracycline hydrochloride | Sigma-Aldrich | 37919 | 100 μg/ml solution in 100% ethanol is kept at -20 °C protected from light in aluminum-foil |
| PBS | Gibco | 20012 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Dissolve in 0.2 M HEPES buffer, pH 7.4. Store at -20 °C and thaw freshly the day before use. Caution, PFA is toxic by inhalation, in contact with skin and if swallowed. |
| HEPES | Sigma-Aldrich | H3375 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| DAPI | Roche | 10236276001 | |
| Scrambled siRNA | Dharmacon | D-001810-10 | |
| Kif11 siRNA | Dharmacon | L-003317-00 | |
| ARPC3 siRNA | Dharmacon | L-005284-00 | |
| Brucella abortus 2308 | |||
| pJC43 (apHT::GFP) | Celli et al.12 | ||
| pAC042.08 (apht::dsRed, tetO::tetR-GFP) | Construction: pJC44 4 was digested with EcoRI followed by generation of blunt ends with Klenov enzyme and subsequent digestion with SalI. TetR-GFP was amplified from pNF106 using primer prAC090 and prAC092. Following digestion with SalI, the TetR-GFP product was ligated to the digested pJC44 vector. | ||
| Primer prAC090 | Sigma-Aldrich | TTT TTG AAT TCT GGC AAT TCC GAC GTC TAA GAA ACC | |
| Primer prAC092 | Sigma-Aldrich | TTT TTG TCG ACT TTG TCC TAC TCA GGA GAG CGT TC | |
| HeLa CCL-2 | ATCC | CCL-2 | |
| ImageXpress Micro | Molecular Devices | IXM IMAGING MSCOPE | Automated cellular imaging microscope equipped with a precision motorized Z-stage. Alternative systems for automated microscopy and alternative components for hard- and software specified below can be employed. |
| High-Speed Laser Auto-Focus | Molecular Devices | 1-2300-1037 | |
| CFI Super Fluor 10X objective | Nikon | MRF00100 | N.A 0.50, W.D 1.20 mm, DIC Prism: 10X, Spring loaded |
| Photometrics CoolSNAP HQ Monochrome CCD Camera | Molecular Devices | 1-2300-1060 | 1,392 x 1,040 imaging pixels, 6.45 x 6.45 µm pixels, 12 bits digitization |
| MetaXpress software | Molecular Devices | 9500-0100 | |
| LUI-Spectra-X-7 | Lumencor | SPECTRA X V-XXX-YZ | Light engine. The following light sources are used: violet (DAPI), cyan (GFP), green/yellow (RFP) |
| Single Band Exciter for DAPI | Semrock | FF01-377/50-25 | |
| Single Band Emitter for DAPI | Semrock | FF02-447/60-25 | |
| Single Band Dichroic for DAPI | Semrock | FF409-Di03-25x36 | |
| Single Band Exciter for GFP | Semrock | FF02-472/30-25 | |
| Single Band Emitter for GFP | Semrock | FF01-520/35-25 | |
| Single Band Dichroic for GFP | Semrock | FF495-Di03-25x36 | |
| Single Band Exciter for RFP | Semrock | FF01-562/40-25 | |
| Single Band Emitter for RFP | Semrock | FF01-624/40-25 | |
| Single Band Dichroic for RFP | Semrock | FF593-Di03-25x36 |
Ссылки
- Pappas, G., Papadimitriou, P., Akritidis, N., Christou, L., Tsianos, E. V. The new global map of human brucellosis. Lancet Infect Dis. 6, 91-99 (2006).
- Atluri, V. L., Xavier, M. N., de Jong, M. F., den Hartigh, A. B., Tsolis, R. E. Interactions of the human pathogenic Brucella species with their hosts. Annu Rev Microbiol. 65, 523-541 (2011).
- Guzman-Verri, C., et al. GTPases of the Rho subfamily are required for Brucella abortus internalization in nonprofessional phagocytes: direct activation of Cdc42. J Biol Chem. 276, 44435-44443 (2001).
- Starr, T., Ng, T. W., Wehrly, T. D., Knodler, L. A., Celli, J. Brucella intracellular replication requires trafficking through the late endosomal/lysosomal compartment. Traffic. 9, 678-694 (2008).
- Boschiroli, M. L., et al. The Brucella suis virB operon is induced intracellularly in macrophages. Proc Natl Acad Sci U S A. 99, 1544-1549 (2002).
- Celli, J., et al. Brucella evades macrophage killing via VirB-dependent sustained interactions with the endoplasmic reticulum. J Exp Med. 198, 545-556 (2003).
- Porte, F., Liautard, J. P., Kohler, S. Early acidification of phagosomes containing Brucella suis is essential for intracellular survival in murine macrophages. Infect Immun. 67, 4041-4047 (1999).
- Anderson, T. D., Cheville, N. F. Ultrastructural morphometric analysis of Brucella abortus-infected trophoblasts in experimental placentitis. Bacterial replication occurs in rough endoplasmic reticulum. Am J Pathol. 124, 226-237 (1986).
- Qin, Q. M., et al. RNAi screen of endoplasmic reticulum-associated host factors reveals a role for IRE1alpha in supporting Brucella replication. PLoS Pathog. 4, e1000110 (2008).
- Ramo, P., et al. Simultaneous analysis of large-scale RNAi screens for pathogen entry. BMC genomics. 15, 1162 (2014).
- Kuhbacher, A., Gouin, E., Cossart, P., Pizarro-Cerda, J. Imaging InlC secretion to investigate cellular infection by the bacterial pathogen Listeria monocytogenes. J Vis Exp. , e51043 (2013).
- Kentner, D., et al. Shigella reroutes host cell central metabolism to obtain high-flux nutrient supply for vigorous intracellular growth. Proc Natl Acad Sci U S A. 111, 9929-9934 (2014).
- Celli, J., Salcedo, S. P., Gorvel, J. P. Brucella coopts the small GTPase Sar1 for intracellular replication. Proc Natl Acad Sci U S A. 102, 1673-1678 (2005).
- von Bargen, K., Gorvel, J. P., Salcedo, S. P. Internal affairs: investigating the Brucella intracellular lifestyle. FEMS microbiology reviews. 36, 533-562 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27, 1179-1180 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены