Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Выбор нескольких подмножеств биомаркеров с аналогичным образом эффективное двоичной классификации выступления
В этой статье
Резюме
Существующие алгоритмы генерации одно решение для обнаружения набора биомаркеров. Этот протокол демонстрирует существование нескольких аналогично эффективных решений и представляет удобного программного обеспечения для биомедицинских исследователи расследовать их наборы данных для предлагаемого вызов. Программисты могут также предоставлять эту функцию в их биомаркер алгоритмы обнаружения.
Аннотация
Биомаркер обнаружения является одним из более важных вопросов биомедицинских для высокой пропускной способности «омику» исследователей, и почти все существующие алгоритмы обнаружения биомаркер генерировать один биомаркер подмножество с оптимизированной производительности измерений для заданного набора данных . Однако недавнее исследование продемонстрировал существование нескольких подмножеств биомаркеров с аналогичным образом эффективным или даже идентичные классификации спектаклей. Этот протокол предоставляет простой и понятной методологии для обнаружения подмножеств биомаркеров с двоичной классификации спектакли, лучше, чем отсечки, определяемой пользователем. Протокол состоит из подготовки данных и загрузки, базовой информации уплотнения, настройки параметров, биомаркер скрининг, результат визуализации и интерпретации, биомаркер гена аннотации и результат и визуализация экспорт в качество издания. Предлагаемая биомаркеров, скрининг стратегия является интуитивно понятным и демонстрирует общее правило для разработки алгоритмов обнаружения биомаркеров. Дружественный графический интерфейс пользователя (GUI) был разработан с использованием языка программирования Python, позволяя биомедицинских исследователей, чтобы иметь прямой доступ к их результатам. Исходный код и руководство kSolutionVis можно загрузить с http://www.healthinformaticslab.org/supp/resources.php.
Введение
Двоичной классификации, один из наиболее часто расследование и сложной интеллектуального анализа проблем в области биомедицинских данных используется для построения модели классификации обучение на две группы образцов с наиболее точные дискриминации мощность1, 2 , 3 , 4 , 5 , 6 , 7. Однако, большие данные, полученные в области биомедицины имеет присущие «большой p малых n» парадигма, с числом функций, обычно намного больше, чем количество образцов6,8,9. Таким образом биомедицинских исследователи должны снизить возможность измерения перед использованием классификации алгоритмов, чтобы избежать overfitting проблема8,9. Диагноз биомаркеров определяются как часть обнаруженных функций отделения больных данной болезни от здорового управления образцы10,11. Пациенты обычно определяются как положительные образцы, и здоровые элементы управления определяются как негативные примеры12.
Недавние исследования показали, что существует более чем одно решение с идентичными или аналогичным образом эффективной классификации спектакли для биомедицинских данных5. Почти все алгоритмы выбора функции являются детерминированными алгоритмы, производить только одно решение для одного набора данных. Генетические алгоритмы могут одновременно создавать несколько решений с аналогичными характеристиками, но они все еще пытаются выбрать одно решение с лучших фитнес-функции в качестве выходных данных для заданного набора данных13,14.
Функция выбора алгоритмов можно подразделить примерно как фильтры или оболочки12. Фильтр алгоритм выбирает Топ -k особенности ранжированных по их значительные индивидуальные ассоциации с двоичной класса этикетки, основывается на предположении, что функции являются независимыми друг от друга15,16,17 . Хотя это предположение не справедливы для почти всех реальных наборов данных, эвристический фильтр выполняет хорошо, во многих случаях, например, mRMR (избыточность минимальной и максимальной релевантности) алгоритм, Вилкоксон тест на функцию фильтрации (WRank) алгоритм и сюжет ROC (приемник эксплуатационные характеристики) на основе алгоритма фильтрации (ROCRank). mRMR, представляет собой эффективный фильтр алгоритм, потому что он приближает комбинаторной оценки проблемы с серией гораздо меньше проблем, сравнивая алгоритм выбора функций максимум зависимость, каждая из которых включает только две переменные, и Таким образом использует попарно совместных вероятностей, которые являются более надежные18,19. Однако mRMR может недооценивать полезность некоторых функций, как это не мера взаимодействия между функциями, которые можно увеличить релевантность и таким образом пропускает некоторые функции комбинаций, которые индивидуально бесполезны, но полезны только при сочетании. WRank алгоритм вычисляет-параметрическая оценка как дискриминационный функцию между двумя классами образцов, и известен за его надежность для останцы20,21. Кроме того ROCRank алгоритм оценивает, насколько важно для исследуемых двоичной классификации производительности22,23площадь под ROC кривой (AUC) конкретного компонента.
С другой стороны оболочка оценивает предварительно определенные классификатора производительность данной функции подмножества, многократно порожденных эвристические правила и создает подмножество функций с лучшими измерения производительности24. Оболочка обычно превосходит фильтра в классификации производительности, но работает медленнее25. Например упорядочить случайный лес (СБР)26,27 алгоритм использует жадные правило, путем оценки функции на подмножества обучающих данных на каждом узле случайных лесных, чьи оценки важности функция оцениваются индекс Джини . Выбор новой функции будут наказаны, если его прироста информации не улучшить, что выбранной функции. Кроме того анализ прогнозирования для Microarrays (PAM)28,29 алгоритм, также оболочку алгоритм, вычисляет средневзвешенные для каждого класса знаков, а затем выбирает возможности уменьшить центроиды гена к общей центроид класса. Пэм надежной для удаленных компонентов.
Несколько решений с производительностью лучших классификации могут быть необходимы для любого заданного набора данных. Во-первых цель оптимизации детерминированного алгоритма определяется математическую формулу, например, минимальная ошибка ставка30, который не обязательно подходит для биологических образцов. Во-вторых набор данных может иметь несколько, значительно отличаются, решения с аналогичными эффективным или даже одинаковых спектаклей. Почти все существующие алгоритмы выбора функция будет случайным образом выбирать одну из этих решений как вывода31.
Это исследование представит аналитический протокол информатики для генерации несколько функция выбора решений с аналогичными спектаклей для любой данной двоичной классификации набора данных. Учитывая, что наиболее биомедицинских исследователи не знакомы с информатики методы или компьютер кодирование, дружественный графический интерфейс пользователя (GUI) была разработана для облегчения быстрого анализа биомедицинских двоичной классификации наборов данных. Аналитически протокол состоит из загрузки данных и подведение итогов, настройки параметров, конвейер выполнения и интерпретации результатов. С помощью простого клика исследователь способен генерировать биомаркер подмножеств и публикации качество визуализации участков. Протокол был протестирован с помощью transcriptomes двух наборов двоичной классификации острый лимфобластный лейкоз (все), то есть, ALL1 и ALL212. Наборы данных ALL1 и ALL2 были загружены из широкой институт генома данных анализа центра, доступны на http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. ALL1 содержит 128 образцы с 12,625 функциями. Из этих образцов, 95 являются B-клетка все и 33 являются Т-клеток всех. ALL2 включает в себя 100 образцов с 12,625 функций, а также. Из этих образцов есть 65 пациентов, которые пострадали рецидива и 35 пациентов, которые не. ALL1 был простой двоичной классификации dataset с минимальной точностью четырех фильтров и четыре обертки 96,7%, и 6 8 функция выбора алгоритмов достижения 100%12. В то время как ALL2 был набор более сложным, с выше 8 функция выбора алгоритмов достижения не лучше, чем 83,7% точность12. Это лучший точность была достигнута с 56 функций, обнаруженных алгоритмом оболочки, на основе корреляции функция выбора (CFS).
протокол
Примечание: Следующий протокол описывает детали аналитические процедуры информатики и псевдо-коды основных модулей. Система автоматического анализа была разработана с использованием Python версия 3.6.0 и панды модулей Python, abc, numpy, scipy, sklearn, sys, PyQt5, sys, mRMR, математика и matplotlib. Материалы, используемые в данном исследовании, перечислены в Таблице материалов.
1. Подготовка данных матрицы и класса этикетки
- Подготовьте файл матрицы данных как файл матрица табуляции или запятыми, как показано на рисунке 1A.
Примечание: Каждая строка содержит все значения функции, и первым пунктом является имя компонента. Probeset ID для набора данных на основе microarray транскриптом или функция может быть другое значение ID как остатков цистеина с ее метилирование значения в наборе данных methylomic. Каждый столбец дает возможность значения данного образца, с первым элементом является имя образца. Строки разделяются на столбцы табуляции (рис. 1B) или запятая (рис. 1 c). Файл с РАЗДЕЛИТЕЛЯМИ табуляциями матрица признается .tsv Расширение файла, и файл с разделителями запятыми матрица имеет расширение .csv. Этот файл может быть порождена сохранения матрицы либо как .tsv или CSV формат от программного обеспечения, таких как Microsoft Excel. Матрица данных также может быть порождена компьютер кодирования. - Подготовьте файл меток класса в формате табуляции или запятыми матрицы (рис. 1 d), аналогично файлу данных матрицы.
Примечание: В первой колонке приводится образец имена, и класс этикетке каждого образца дается в столбце под названием класса. Максимальная совместимость рассматривается в процессе кодирования, так что могут быть добавлены дополнительные столбцы. Ярлык файла класса может быть отформатирован как .tsv или CSV-файл. Имена в столбце класс может быть каких-либо условий, и может быть более двух классов образцов. Пользователь может выбрать любые два из классов для следующих анализа.
2. загрузить матрицы данных и класса этикетки
- Загрузка матрицы и класса Метки данных в программное обеспечение. Нажмите на кнопку загрузки данных матрицы для выбора файла пользователем данных матрицы. Нажмите на кнопку Load класса этикетки выбрать соответствующий файл класса label.
Примечание: После того, как оба файлы загружаются, kSolutionVis будет проводить обычные экран совместимости между двумя файлами. - Резюме особенности и образцы из файла данных матрицы. Оцените размер файла данных матрицы.
- Суммировать образцы и классов из файла класса label. Оцените размер файла класса label.
- Проверьте, имеет ли каждый образец из матрицы данных класса label. Суммируйте количество образцов с метками класса.
3. обобщить и отображения статистики базового набора данных
- Нажмите на кнопку суммировать, без ввода указанного ключевого слова, и программное обеспечение будет отображать 20 индексированных функции и соответствующие имена функций.
Примечание: Пользователи должны указать имя компонента, которые они хотели найти, чтобы увидеть ее базовой статистики и соответствующего значения распределения среди всех входных образцов. - Предоставляют ключевое слово, например «1000_at», в текстовом поле функция найти конкретную возможность резюмировать. Нажмите на кнопку конспект для получения базовой статистики для этой заданной функции.
Примечание: Ключевое слово может появиться в любом месте в целевых имен компонентов, облегчить процесс поиска для пользователей. - Нажмите на кнопку, подвести итог найти более чем одну функцию с заданным ключевым словом, а затем укажите идентификатор уникальной особенностью приступить к шаг выше подведения итогов одну особенность.
4. определение класса этикетки и количества функций, топ рейтинг
- Выберите имена положительных («P (33)») и отрицательным («N (95)») классы в раскрывающихся списках Класс положительных и Отрицательных класса, как показано на рисунке 2 (в центре).
Примечание: Рекомендуется выбрать что сбалансированного бинарные классификации набора данных, то есть, разница между количеством положительных и отрицательных примеров является минимальным. Количество выборок, также приводится в скобках после имени каждого класса Метки в двух раскрывающихся списках. - Выберите 10 как количество лучших возможностей (параметр pTopX) в раскрывающемся списке Top_X (?) для комплексного экрана особенность-подмножества.
Примечание: Программное обеспечение автоматически занимает все функции по P-значение , вычисленное t тест по каждой функции сравнения положительных и отрицательных классов. Функция с меньше P-значение имеет более требовательных власти между двумя классами образцов. Модуль комплексного скрининга интенсивных вычислений. Параметр pTopX -по умолчанию 10. Пользователи могут изменить этот параметр в диапазоне от 10 до 50, до тех пор, пока они находят удовлетворение располагают подмножества с хорошим классификации спектаклей.
5. Настройте параметры системы для различных исполнений
- Выбор показателей (pMeasurement) точность (АКК) в раскрывающемся списке АКК/bAcc (?) для выбранного классификатора крайняя машина обучения (вяз). Еще один вариант этого параметра является измерение сбалансированный точность (bAcc).
Примечание: Пусть TP, FN, TN, и FP количество истинных положительных результатов, ложные негативов, правда негативов и ложных срабатываний, соответственно. Измерения АКК определяется как (TP+TN)/(TP+FN+TN+FP), который лучше всего работает на сбалансированный набор6. Но классификатора, оптимизированный для АКК стремится присвоить негативные классу все образцы, если количество отрицательных образцов гораздо больше, чем положительных. BAcc определяется как (Sn + Sp) / 2, где Sn = TP/(TP+FN) и Sp = TN/(TN+FP) правильно предсказал ставки за положительные и отрицательные примеры, соответственно. Таким образом bAcc нормализует предсказания выступления над двумя классами и может привести к сбалансированной предсказание производительности свыше двух классов несбалансированным. АКК является выбором по умолчанию pMeasurement. Программное обеспечение использует классификатор ELM по умолчанию для вычисления классификации спектаклей. Пользователь может также выбрать классификатора из SVM (поддержка векторных машина), KNN (k ближайшего соседа), дерево принятия решений или Байеса. - Выберите пороговое значение 0,70 (параметр pCutoff) для измерения производительности указанного в поле ввода pCutoff:.
Примечание: АКК и bAcc в диапазоне между 0 и 1, и пользователь может указать значение pCutoff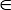 [0, 1] как отсечки для отображения соответствующего решения. Программное обеспечение выполняет всеобъемлющую функцию подмножества скрининг, и подходящим выбором pCutoff сделают 3D визуализации, более интуитивным и явной. Значение по умолчанию для pCutoff- 0,70.
[0, 1] как отсечки для отображения соответствующего решения. Программное обеспечение выполняет всеобъемлющую функцию подмножества скрининг, и подходящим выбором pCutoff сделают 3D визуализации, более интуитивным и явной. Значение по умолчанию для pCutoff- 0,70.
6. Запуск конвейера и интерактивная визуализация результатов
- Нажмите кнопку Analyze для запуска конвейера и создавать участки визуализации, как показано на рисунке 2 (внизу).
Примечание: Левая таблица дает все подмножества компонентов и их pMeasurement рассчитывается путем десятикратного стратегии перекрестной проверки классификатора вяз, как описано ранее5. Для процедуры скрининга особенность подмножество с текущими настройками параметра создаются два 3D точечные участки и участки две линии. - Выберите 0,70 как значение по умолчанию pMeasurement среза (параметр piCutoff, поле ввода значение) и 10 по умолчанию число подмножеств Лучшая особенность (параметр piFSNum).
Примечание: Конвейера выполняется с использованием параметров pTopX, pMeasurement и pCutoff. Функцию обнаруженных подмножества может быть далее экранированный, с помощью отсечки piCutoff, однако piCutoff не может быть меньше, чем pCutoff. Таким образом piCutoff инициализируется как pCutoff и будут визуализированы только подмножества функция с ≥ измерения производительности piCutoff . Значение по умолчанию piCutoff pCutoff. Иногда kSolutionVis обнаруживает множество решений и только лучшие piFSNum (по умолчанию: 10) будет визуализирована функция подмножеств. Если число подмножеств особенность, обнаруженных программой меньше чем piFSNum, все подмножества функция будет визуализирована. - Собирать и интерпретировать особенности обнаружены программного обеспечения, как показано на рисунке 3.
Примечание: В таблице в левом окне показывает обнаруженные функции подмножества и измерения их производительности. Имена первых трех столбцов являются «F1», «F2» и «F3». Три функции в каждой функции подмножества приводятся в порядке их рейтинга в одной строке (F1 < F2 < F3). Последний столбец дает измерение производительности (АКК или bAcc) подмножества каждого компонента, и его имя столбца (АКК или bAcc) является значение pMeasurement.
7. интерпретировать 3D точечные участки визуализации и интерпретации функции подмножества с аналогичным образом эффективное двоичной классификации выступления с использованием 3D точечные участки
- Нажмите кнопку анализ для создания 3D точечная подмножеств Топ 10 функция с лучших спектаклей классификации (АКК или bAcc) обнаружены программного обеспечения, как показано на рисунке 3 (средний ящик). Сортировать три функции в подмножестве особенность в возрастающем порядке их ряды и использовать ряды трех функций как F1/F2/F3 осей, т.е., F1 < F2 < F3.
Примечание: Цвет точка представляет двоичной классификации производительность соответствующего подмножества компонентов. Набор данных может иметь несколько подмножеств функция с аналогичным образом эффективность измерений. Таким образом интерактивный и упрощенной точечной является необходимым. - Измените значение на 0,70 в поле ввода pCutoff: и нажмите кнопку анализ для создания 3D Точечная функция подмножеств с производительности измерений ≥ piCutoff, как показано на рисунке 3 (в правом окне). Нажмите кнопку 3D тюнинг открыть новое окно вручную настроить углы 3D точечную диаграмму.
Примечание: Каждое подмножество функций представляет точку в так же, как выше. 3D точечная сгенерирована угла по умолчанию. Для облегчения 3D визуализации и тюнинг, отдельном окне будет открыта, нажав на кнопку 3D тюнинг. - Нажмите кнопку уменьшить уменьшить избыточность обнаруженных функция подмножеств.
Примечание: Если пользователи хотят далее выберите функцию тройни и свести к минимуму избыточность функция подмножеств, программное обеспечение также предоставляет эту функцию, используя алгоритм выбора функций mRMR. После нажатия на кнопку уменьшить , kSolutionVis удаления этих избыточных функций в функцию тройни и восстановить таблицу и два разброс участков, упомянутых выше. Удалены функции функция тройни будет заменен ключевым словом в таблице. Значения None в оси F1/F2/F3 будет обозначать как значение piFSNum (диапазон нормальной стоимости F1/F2/F3 — [1, top_x]). Таким образом точки, которые включают значение None может показаться «выброс» точек в 3D участков. Вручную перестраиваемый 3D участки могут найти в «Ручная настройка 3D точка участков» в дополнительном материале.
8. найти ген аннотации и их ассоциации с заболеваний человека
Примечание: Шаги 8 – 10 будет показано, как для аннотирования ген от уровня последовательности ДНК и белка. Во-первых ген символ каждого биомаркер ID из выше шаги будут извлечены из базы данных Дэвид32, а затем два представителя веб-сервера будет использоваться для анализа этот ген символ от уровней ДНК и белка, соответственно. Сервер GeneCard обеспечивает всеобъемлющие функциональные аннотации данного гена символа, и Менделевское наследование у человека базы данных (Маккусика) обеспечивает наиболее полное курирование болезни Джин ассоциаций. Сервер UniProtKB является одним из наиболее всеобъемлющей базы данных белков, и сервера на основе группы прогнозирования системы (GPS) предсказывает сигнализации фосфорилирование в очень большой список киназы.
- Копировать и вставить веб-ссылку базы данных Дэвид в веб-браузер и откройте веб-страницу этой базы данных. Нажмите на ссылку Гена ID преобразования в рисунке 4A и ввода функцию идентификаторы 38319_at/38147_at/33238_at первого биомаркер подмножества набора данных ALL1 (Рисунок 4B). Нажмите на ссылку Генов списка и нажмите кнопку Отправить список , как показано на рисунке 4В. Извлечь заметки интерес и нажмите кнопку Показать список гена (рис. 4 c). Получить список символов гена (рис. 4 d).
Примечание: Символы гена, полученные здесь будет использоваться для дальнейшего функциональных аннотации в следующих шагах. - Копировать и вставить веб-ссылку базы данных ген карты в веб-браузер и откройте веб-страницу этой базы данных. Поиск имя гена CD3D в поле ввода запроса базы данных и найдите аннотации этого гена Джин карт33,34, как показано в таблице 1 и Рисунок 5A.
Примечание: Джин карт является геном всеобъемлющей базы знаний, обеспечивая номенклатуры, геномики, протеомики, субцеллюлярные локализации и участие пути и других функциональных модулей. Он также предоставляет внешние ссылки на различных других биомедицинских базах данных как PDB/PDB_REDO35, Джин Entrez36, Маккусика37и38UniProtKB. Если имя компонента не является символом стандартного гена, используйте базу данных ENSEMBL преобразовать39. CD3D — это имя ген Т-клеточный рецептор T3 Дельта цепи. - Копировать и вставить веб-ссылку базы данных Маккусика в веб-браузер и откройте веб-страницу этой базы данных. Поиск имени гена CD3D и найти заметки этого гена из базы данных Маккусика37, как показано в таблице 1 и на рисунке 5B.
Примечание: Маккусика служит теперь одним из наиболее всеобъемлющим и авторитетным источников человеческого гена соединения с наследуемыми заболеваний. Маккусика был инициирован д-р Виктор а. McKusick каталог связанные заболевания генетические мутации40. Маккусика в настоящее время охватывает более 15 000 генов человека и более 8500 фенотипов, по состоянию на Декабрь 1st 2017.
9. Аннотирование закодированные белков и столб-поступательные изменения
- Копировать и вставить веб-ссылку базы данных UniProtKB в веб-браузер и откройте веб-страницу этой базы данных. Поиск имени гена CD3D в поле ввода запроса UniProtKB и найти заметки этого гена из базы данных-38, как показано в таблице 1 и на рисунке 5 c.
Примечание: UniProtKB собирает богатым источником аннотации для белков, в том числе номенклатуры и функциональной информации. Эта база данных также содержит внешние ссылки на другие широко используемых баз данных, включая PDB/PDB_REDO35, Маккусика37и41Pfam. - Копировать и вставить веб-ссылку на веб-сервер GPS в веб-браузер и откройте веб-страницу этого веб-сервера. Извлечение последовательности белка, кодируемых биомаркер гена CD3D от UniProtKB базы данных38 и предсказать белка столб-поступательные изменения (ПТМ) остатков с помощью онлайн-инструмент GPS, как показано в таблице 1 и на рисунке 5 d.
Примечание: Биологические системы является динамичным и сложным, и существующих баз данных собирать только известной информации. Таким образом биомедицинских предсказания онлайн-инструментов, а также автономные программы может предоставить полезные доказательств для дополнения механизма гипотетической. GPS был развитых и улучшение на протяжении 12 лет7,42 и могут быть использованы для прогнозирования белка PTM остатков в43,данной пептида в последовательности44. Инструменты также доступны для различных темы исследований, включая прогнозирование белка субцеллюлярные расположение45 и транскрипционным фактором привязки мотивы 46 среди других.
10. Аннотирование белок белковых взаимодействий и их обогащенных функциональных модулей
- Копировать и вставить веб-ссылку на веб-сервер строку в веб-браузер и откройте веб-страницу этого веб-сервера. Поиск в списке для генов CD3D и P53 и найти их организовали свойств, с помощью базы данных строка47. Такая же процедура может осуществляться с помощью другой веб-сервер, Дэвид32.
Примечание: Помимо вышеупомянутых аннотации для отдельных генов, есть много крупномасштабных информатики инструментов, доступных для изучения свойств группы генов. Недавнее исследование показало, что индивидуально плохо маркерных генов может представлять набор значительно улучшилась гена5. Таким образом стоит вычислительная стоимость экран для более сложных биомаркеров. Базы данных строка может визуализировать подключения известных или прогнозируемое взаимодействие, и Дэвид сервера может обнаружить функциональные модули с значительным фенотип ассоциаций в запрашиваемый гены47,32. Также доступны различные другие инструменты анализа крупномасштабных информатики.
11. экспорт подмножеств сгенерированный биомаркеров и участки визуализация
- Экспорт обнаруженные биомаркер подмножеств .tsv или CSV текстовый файл для дальнейшего анализа. Нажмите кнопку экспортировать таблицу под столом всех подмножеств обнаруженных биомаркеров и выбрать какой формат текста, чтобы сохранить как.
- Экспортируйте участки визуализация файла изображения. Нажмите на кнопку сохранить под каждого участка и выбрать какой формат изображения, чтобы сохранить как.
Примечание: Программное обеспечение поддерживает .png формат пикселей и векторный формат .svg. Пиксель изображения являются хорошими для отображения на экране компьютера, в то время как векторных изображений могут быть преобразованы в любой резолюции, необходимые для целей публикации журнала.
Результаты
Цель этого рабочего процесса (рис. 6) является выявление нескольких подмножеств биомаркеров с аналогичными эффективности для двоичной классификации набора данных. Весь процесс иллюстрируется двумя пример наборов данных ALL1 и ALL2, извлеченные из недавно...
Обсуждение
Это исследование представляет протокол обнаружения и характеристика easy-to последующие мульти решение биомаркер для пользовательской двоичной классификации набора данных. Программное обеспечение ставит акцент на удобство и гибкий импорт/экспорт интерфейсы для различных форматов фай...
Раскрытие информации
У нас нет конфликтов интересов, связанных к настоящему докладу.
Благодарности
Эта работа была поддержана стратегических приоритетных исследований программы Китайской академии наук (XDB13040400) и запуска грант от университета Цзилинь. Анонимные Отзывы и биомедицинских испытаний пользователей были оценены за их конструктивные замечания по улучшению юзабилити и функциональность kSolutionVis.
Материалы
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Ссылки
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены