Method Article
ТРИФОСФАТЫ руководство РНК клонирования млекопитающих систем
* Эти авторы внесли равный вклад
В этой статье
Резюме
Здесь приводится простой, эффективной и экономически эффективным методом sgRNA клонирования.
Аннотация
Изложил протокол описывает упрощенные методы для эффективного и экономичного поколения Cas9-связанные руководство РНК. Две альтернативные стратегии для клонирования руководство РНК (gRNA) изложены, основанные на использовании энзима ограничения типа IIS BsmBI в сочетании с набором совместимых векторов. За пределами доступ к Сэнгер последовательности услуг для проверки созданного векторов помимо тех, которые являются стандартными в современной молекулярной биологии лаборатории требуются никакого специального оборудования или реагентов. Изложил метод предназначен прежде всего для клонирования одного единого gRNA или одной паре gRNA выражая вектора одновременно. Эта процедура не масштабируется для создания библиотек, содержащих тысячи оформления. Для этих целей рекомендуется использовать альтернативные источники синтеза олигонуклеотида как синтез oligo чип. Наконец в то время как этот протокол фокусируется на набор векторов, млекопитающих, Общая стратегия пластика и применима к любой организм, при наличии соответствующей gRNA вектор.
Введение
ТРИФОСФАТЫ связанные белком 9 (Cas9) является РНК руководствуясь эндонуклеазы, которая направлена на желаемому геномной когда complexed с надлежащим образом оформленные небольшое руководство РНК (gRNA)1,2. Оформления составляют 20-нуклеотидной последовательности (protospacer), который дополняет геномные последовательности. Рядом с геномной целевой последовательности является 3' protospacer связанные мотив (PAM), которая требуется для привязки Cas9. В случае Streptococcus Pyogenes Cas9 (SpCas9), это имеет последовательность NGG. После привязки целевого ДНК, Cas9 отщепляет обе нити ДНК, тем самым стимулируя ремонт механизмов, которые могут быть использованы для изменения Локус интерес. Активации ошибкам non гомологичных прихода в конце пути (NHEJ) может привести к срыву целевого гена. Кроме того ремонт сделал через гомологичная рекомбинация может стимулировать целенаправленной интеграции желаемой последовательности ДНК если шаблон доноров предоставляется6. Нуклеаза Мертвого Cas9 варианты (dCas9) также может быть сгенерированный3 , мутирует остатков в Cas9, которые необходимы для endonucleolytic деятельности. dCas9 остается компетентным для связывания ДНК, но не режет его связанного целевого объекта. Фьюзинг различных эффекторных домены dCas9 и направляя его вблизи гена transcriptional стартовый сайт, dCas9 может использоваться для выборочного гена индукции или частичной гена, глушителей4,5.
В то время, как это невероятно мощный, возможность использования Cas9 целевой геномные последовательности интерес требует, что пользователь сначала генерирует gRNA уникальный для целевого сайта. Различные методы для создания gRNA векторы выражения ранее были описаны многие из различной эффективности и стоимость6,,78. Автономные ампликонов ПЦР может использоваться в качестве оформления для быстрого скрининга, идя от олигонуклеотида доставки трансфекции в течение нескольких часов; Однако, это требует Ultramer (более 100 bp) oligo синтеза, который является дорогим9. Это также можно купить gBlocks, которые являются более рентабельными, чем Ultramers. Как Cas9 быстро стал неотъемлемой частью современной молекулярной биологии инструментарий, простой, недорогой и очень эффективный метод для клонирования gRNA бы благом для поля. В нашей группе и сотрудничающих лабораторий за последние несколько лет для создания более 2000 уникальных оформления4использовался метод, описанный здесь.
Указывается метод фокусируется на методах для клонирования, лентивирусные совместимый векторов, содержащий один gRNA или самое большее два оформления. Для поколения плазмид, содержащие более двух оформления, или клонировать библиотеку оформления,9,10,,1112рекомендуются альтернативные подходы.
протокол
Примечание: Следующий протокол описывается, как выполнять gRNA дизайн с помощью онлайн-инструментов (шаги 1.1-1.4), которые являются общими для всех методов строительства gRNA плазмида, подробно изложены в этой рукописи. После того, как желаемый оформления определены, наряду с несколько различных методов для введения олигонуклеотиды в векторы выражения (например, pSB700) описаны шаги для заказа необходимых олигонуклеотиды. Представлены 2 методы для клонирования единичных векторов выражения руководство на основе либо перевязка в predigested выражение вектор (шаги 2-7.3) или Золотые ворота клонирования (шаги 8-8.4). Далее приводится стратегия для клонирования парных Путеводитель выражая векторов с помощью полимеразной цепной реакции (ПЦР) следуют Золотые ворота клонирования (шаги 11-16). Наконец также изложены общие методы для выполнения химической трансформации E. coli (шаги 9-9,6) и вектора выражения последовательности проверки (шаги 10 до 10.2.3).
- Дизайн gRNA олигонуклеотиды с использованием онлайновых инструментов следующим образом.
- Дизайн оформления с помощью онлайн инструменты, такие как sgRNA бомбардиром 2.0 онлайн-инструмент (https://crispr.med.harvard.edu/sgRNAScorerV2/)13 или альтернативы, такие как средство проектирования широкого sgRNA (http://portals.broadinstitute.org/gpp/public/analysis-tools/ sgrna дизайн)14 и несколько других15.
- Используйте для создания потенциальных gRNA последовательностей гена имя, символ или сырой последовательности ДНК.
Примечание: Создается файл электронной таблицы (.csv/.xls) со всех потенциальных оформления против ген или предоставленной последовательности. Качество оформления потенциальных будет сообщено как Оценка , если используются sgRNA бомбардиром 2.0 онлайн-инструмент, или как % целевой эффективности если используется средство проектирования широкого sgRNA. Обе эти меры являются производными на основе деятельности на цели резки. - Рассмотрим деятельность - целевой в выборе gRNA при ранжировании оформления.
Примечание: Оптимального руководства являются те, с наибольшей эффективностью на цель и наименее-целевого действия. Для активации (CRISPRa) оформления предназначены для целевой регион от 1-200 bp, вверх по течению transcriptional начало сайта (TSS)4,15. Для ингибирования (CRISPRi), оформления предназначены для целевой регион от 50 bp вверх по течению ТВУ до 300 bp по течению ТП5. Чтобы отключить гена с помощью Cas9 резки, оформления предназначены для целевой первый 10-50% кодирования последовательности по течению инициирующей кодон (как оформления ориентации 3' конца гена показали менее эффективными)13. - Обеспечение не внутренние сайты BsmBI присутствуют если олигонуклеотиды используется для клонирования, Золотые ворота, поскольку это приведет к отожженная олигонуклеотиды, переваривается13.
- Выберите геномные последовательности и удалить 3' NGG PAM (оставляя только последовательность protospacer) для изменения последовательности олигонуклеотида одного gRNA (например, начиная последовательность: CGCGTGCTCTCCCTCATCCATGG).
Примечание: При использовании таблицы, следующая формула будет удалить 3' NGG: =LEFT(A2,LEN(A2)-3), где ячейка A2 содержит последовательности целевых объектов и Пэм.- Добавьте CACCG к 5' концу oligo.
Примечание: После перевязки pSB700 магистрали, эта последовательность будет состоять из 3' конца промотор U6, вождение транскрипции gRNA. Последовательность «CACC» гарантирует, что oligo совместим с свесы переваривается BsmBI pSB700 вектора. «G» является требованием полимеразы III промоутеров и обеспечивает эффективное инициации транскрипции gRNA. Используйте следующую формулу в электронной таблице для выполнения этой задачи: =CONCATENATE("CACCG",(B2)), где B2 — это ячейка, содержащая последовательность protospacer [например, добавив 5' CACCG последовательности для protospacer: CACCGCGCGTGCTCTCCCTCATCCA (это Это окончательный вперед oligo, которое заказывается)]. - Создайте обратный комплемента (rc) protospacer последовательности.
Примечание: например, обратная дополняют вышеупомянутые protospacer-TGGATGAGGGAGAGCACGCG. - Добавьте AAAC к 5' концу rc protospacer. Добавьте дополнительные C 3' концу rc protospacer (обратный oligo).
Примечание: Последовательности «AAAC» гарантирует, что oligo совместим для клонирования в BsmBI усваивается pSB700 вектор. Дополнительные «C» на конце 3' необходим для отжига последовательности для инициирующей «G», добавил к вперед oligo. Используйте следующую формулу в электронную таблицу для выполнения этой задачи =CONCATENATE("AAAC",(C2),"C"), где C2 — это ячейка, содержащий последовательность обратная дополнения [например, AAACTGGATGAGGGAGAGCACGCGC (это заключительный обратный oligo, приказал)].
- Добавьте CACCG к 5' концу oligo.
- Закажите олигонуклеотиды, без дополнительных изменений на олигонуклеотиды.
- Порядок минимальное количество oligo требуется, как только очень небольших количествах необходимы для клонирования реакций.
- Разбавьте лиофилизированные грунтовки до конечной концентрации 100 мкм в 1 x TE буфера (10 мм трис-Cl, pH 7.5 с 1 мм ЭДТА).
- Аликвота 1:1 прямого и обратного олигонуклеотиды (например, 10 мкл каждого) в PCR трубки газа Кап.
Примечание: Это позволит быстрого клонирования многих оформления одновременно с помощью многоканальных дозаторов.- Вортекс и спин вниз oligo смеси (100 x g 15 s). Инкубируйте реакции при комнатной температуре за 5 мин до создания перевязки.
Примечание: Нет необходимости для тепла и затем прохладной oligo смеси для облегчения их отжиг. Стоит отметить, что 3-4 пары gRNA олигонуклеотиды могут объединить и отжигом в одном реакции (например, смесь 3 мкл вперед и 3 мкл обратная олигонуклеотиды вместе с до 4 пар oligo gRNA, давая общий объем 24 мкл). Объем олигонуклеотиды, используемые для отжига может корректироваться с учетом потребностей пользователей.
Примечание: Шаги 6-7.3 описывают predigestion pSB700. Для альтернативного метода см. шаг 8, который описывает одноступенчатых BsmBI ограничение перевязки.
- Вортекс и спин вниз oligo смеси (100 x g 15 s). Инкубируйте реакции при комнатной температуре за 5 мин до создания перевязки.
- Дайджест 1-5 мкг вектора выражения руководство выбранного pSB700 с BsmBI (0,5 мкл на 1 мкг) за 1 ч при температуре 55 ° C15. Проводить пищеварение вектора выражения gRNA pSB700 в общем объеме 40 мкл (Таблица 1).
- Запустите пищеварение продуктов на 1,5% (wt/vol) легкоплавких геля агарозы.
- Вырезать переваривается вектор костяк группы, которая соответствует фрагмент ~ 9 kb в размер и место среза геля в 1,5 мл microcentrifuge трубку (рис. 1).
- Используйте комплект очистки коммерческих гель для извлечения ДНК из геля агарозы согласно инструкциям производителя.
- Элюировать ДНК в 10-15 мкл буфера TE (10 мм трис, рН 7,5; 10 мм трис, pH 8.0) для получения концентрированных элюата.
- Для клонирования gRNA олигонуклеотиды в предварительно усваивается pSB700 выражение вектор перевязать отожженная gRNA олигонуклеотиды из шага 4 в BsmBI усваивается pSB700 руководство выражение вектор в 20 мкл реакции (Таблица 2).
- Во-первых, добавить воды, буфер и ДНК, а затем вихревой смесь и добавить фермент, вихревые 1 окончательное время после добавления фермента и спин вниз решение (100 x g 15 s). Инкубируйте реакции при комнатной температуре на ночь.
- Включать без Вставка отрицательный контроль реакции, что одни, без вставки oligo отожженная gRNA переваривается BsmBI вектор. 1 мкл воды может использоваться вместо 1 мкл gRNA олигонуклеотиды для без Вставка отрицательного контроля.
- Перейти к трансформации (шаг 9).
Примечание: Шаг 8 это альтернативный метод для predigestion pSB700, как описано в шагах 6-7.3.
- Ввести gRNA олигонуклеотиды (описанные в шагах 1-5.1) в pSB700 вектор с помощью Золотые ворота клонирования для единый этапа лигирование BsmBI ограничение.
- Соберите главный микс (1 x) — Если несколько оформления время клонируются, подготовить мастер смесь без вставки олигонуклеотиды добавил (Таблица 3). Аликвота мастер смешать в PCR трубы и использовать многоканальные пипетки для добавления gRNA олигонуклеотиды из шага 4.2. Включите элемент управления без вставка с помощью 1 мкл воды.
- Включая BsmBI фермента и T4 ДНК лигаза в такую же реакцию, достигается одновременное ограничение пищеварение и перевязки (т.е., клонирование Золотые ворота). Используйте следующие простые ворота протокол дайджест вектор и перевязать вставок в 1 реакции: Держите реакции при 37 ° C на 2 ч, при 50 ° C за 10 мин, при 65 ° C для 15 мин, при 80 ° C в течение 15 мин и наконец , держите его на 4 ° C.
- Следует отметить убедитесь, что олигонуклеотиды не содержат BsmBI сайтов перед использованием этого метода. Если оформления, которые копируются в настоящее время содержат места ограничения BsmBI, они будут переварены после добавления BsmBI. Это позволит предотвратить получение любых трансформантов пользователя.
- После первоначальной реакции простого ворот добавить дополнительные 0,5 мкл BsmBI фермента в каждой реакции и поместите их обратно в 55 ° C за 1 ч. После переваривания удерживайте реакции на 4 ° C, или заморозить его до готовности для преобразования. Этот второй раунд энзима ограничения инкубации удаляет любые непереваренных или religated одичал тип pSB700 векторные позвоночника от реакционной смеси, так что только векторов, которые получили oligo вставки остаются нетронутыми и даст трансформантов.
- Перейти к трансформации (шаг 9).
- Трансформировать 0.5 мкл реакционной смеси в 8 мкл сведущее Escherichia coli [например, нос DH5a (1-3 х 109 cfu/мкг ДНК pUC19) или НЭБ стабильной 1 - 3 х 109 cfu/мкг ДНК pUC19). Для лентивирусные плазмид стабильной НЭБ клетки обеспечит более последовательной плазмида урожайности.
- Удаление кишечной палочки от хранения при температуре-80 ° C и оттепели на льду для 5-10 мин.
- Добавить 0,5 мкл реакция смеси 8 мкл сведущее Escherichia coli и держать смесь на льду за 30 мин.
- Тепловой шок смесь при 42 ° C для 45 s.
- Остальные его на льду на 2 мин.
- Восстановите культуру на роторный шейкер в 250 мкл SOC СМИ за 1 ч при 37 ° C для NEB DH5a и при 30 ° C для NEB конюшни.
- Пластины 80 мкл культуры на соответствующие антибиотикорезистентности Lysogeny бульон (LB) плита и проинкубируйте его на ночь при 37 ° C для NEB DH5a и при 30 ° C для NEB конюшни [например, pSB700 является покрытием на фунтов с пластинами ампициллин (100 мкг/мл)] (рис. 3).
- Проверить последовательность gRNA выражение плазмида: проверить последовательность каждой колонии Сэнгером виртуализации, используя праймер 5'-GAGGGCCTATTTCCCATGATTCC-3' который воспламеняет на U6 промоутер вверх по течению gRNA oligo insert.
- Выберите 2-3 колоний на реакции для виртуализации (Таблица 4).
Примечание: Несколько провайдеров виртуализации теперь предлагают услуги, которые могут выполнять Сэнгер секвенирования непосредственно от бактериальных колоний, которые значительно сокращает время и расходы, устраняя необходимость очищения плазмиды до последовательности. Кроме того плазмиды изоляции комплект может использоваться для восстановления плазмида ДНК от индивидуальных бактериальных клонов. Очищенные плазмид затем могут быть представлены для виртуализации. Если выполняется реакции перешнуровки пула, используйте Таблица 4 в качестве руководства для количество колоний, которые должны быть выбраны и представлен для виртуализации. - После подтверждения последовательности изолируйте gRNA выражение плазмид.
- Используйте кончик стерильные пипетки для инокуляции желаемого колонии в 5 мл культуру LB носитель, содержащий 100 мкг/мл ампициллина для векторов выражения pSB700.
- Рост клеток в Ротари инкубатора при 100 об/мин при 37 ° C ночь (30 ° C, если используется НЭБ конюшни).
- Изолируйте плазмида ДНК от культур с использованием подготовка комплекта плазмида, после производителя протокол.
Примечание: Хотя выше методы позволяют пользователям создавать единичных векторов gRNA содержащих, этот протокол позволит пользователям создавать векторов, которые содержат 2 оформления, каждый обусловлен их собственных РНК полимеразой III промоутер.
- Выберите 2-3 колоний на реакции для виртуализации (Таблица 4).
- Для парных gRNA дизайн использовать выходной файл из инструментов дизайна sgRNA, описанные выше (шаги 1.1-1.4) и выберите 2 направляющие интерес.
- Для активации или репрессий, выбрать пар гидов, которые по крайней мере 30 nt друг от друга.
- Для оформления используются для резки выберите руководства, ориентированные на 2 разных экзонов внутри ген.
- Инструкции ниже назначить первый gRNA в массиве gRNA-A и второй gRNA в массиве gRNA-B, для создания изменения последовательности олигонуклеотида парных gRNA.
Примечание: Oligo, используемый для создания gRNA-A будет называться Англии, и обратный oligo используется для введения gRNA-B будет именоваться РБ. Метод клонирования ниже автоматически добавляет инициирующей G к 5' концу каждого из оформления.- Создайте соответствующие фа oligo следующим образом.
Примечание: 20 нуклеотидов, включающий gRNA ориентации последовательности для gRNA-A будет представлять последовательность первоначальный ОС, на которой следующие шаги будут строить (например, protospacer - aggggctacaccactcattg).- Добавьте TTTTCGTCTCTCACCG к 5' концу ОС (например, последовательности TTTTCGTCTCTCACCGaggggctacaccactcattg).
- Добавьте GTTTTAGAGCTATGCTGAAAAGCA в 3' конца ОС (например, последовательности TTTTCGTCTCTCACCGaggggctacaccactcattgGTTTTAGAGCTATGCTGAAAAGCA). Это окончательная версия oligo Англии, которые будут заказаны.
- Создайте необходимые oligo РБ следующим образом.
Примечание: 20 нуклеотидов, включающий gRNA таргетинга последовательности для gRNA-B будет являться исходной последовательности РБ, на котором следующие шаги будут строить (например, о начальной последовательности gtcccctccaccccacagtg).- Обратный дополнение РБ = (Ревком) РБ (например, cactgtggggtggaggggac).
- Добавить TTTTCGTCTCTAAAC к 5' концу (Ревком) РБ (например., TTTTCGTCTCTAAACcactgtggggtggaggggac).
- Добавить CGGTGACCCAGGCGGCGCACAAG к 3' концу (Ревком) РБ (например, TTTTCGTCTCTAAACcactgtggggtggaggggacCGGTGACCCAGGCGGCGCACAAG). Это окончательная версия oligo РБ, которые будут заказаны.
Примечание: следующие формулы могут использоваться с общей таблицы программное обеспечение автоматически редактировать oligo последовательности: = CONCATENATE("TTTTCGTCTCTCACCG",(A2), «GTTTTAGAGCTATGCTGAAAAGCA»), ячейка, содержащая последовательность Англии и , где A2 = Concatenate("TTTTCGTCTCTAAAC",(B2), «CGGTGACCCAGGCGGCGCACAAG»), где B2 ячейку, содержащую последовательность РБ (Ревком).
- Создайте соответствующие фа oligo следующим образом.
- Заказывайте олигонуклеотиды от поставщика синтез выбора. Дополнительные изменения не требуются.
- Для выполнения ПЦР с фа и РБ грунтовки используйте подготовленные плазмидной ДНК. Это будет придавать как вперед и обратного оформления для ПЦР фрагментов генерируется эта плазмида и будет позволить каждому выразил от ее собственных промоутер (U6 и 7SK промоутеров — для предотвращения вирусных рекомбинации в этой конструкции используются различные промоутеры).
- Используйте специальный протокол постановки ПЦР Phusion GC для создания необходимого фрагмента: Держите смеси на 98 ° С в течение 1 мин (1 цикл), в 98 ° C 15 s, в 53 ° C 15 s, 65 ° с 2 мин, а в 72 ° C для 4 мин (30 циклов) , а затем, удерживая его на 4 ° C (Таблица 5).
- Запустить продукт PCR на геле агарозы 1% и проверить, что 1 группа рассматривается в ~ 490 bp (рис. 2).
- Вырезать и извлечь этот продукт PCR, используя набор извлечения геля выбора. Элюировать ДНК в 15 мкл буфера TE (10 мм трис, рН 7,5; 10 мм трис, рН 8,0) или дистиллированную воду.
- Измерьте концентрацию извлечения продукта наряду с pSB700 вектора с спектрофотометр для расчета Молярность ДНК. Нормализовать продукт PCR и вектор к концентрации 40 femtomoles/мкл.
Примечание: Некоторые сайты готовы помочь с расчетом Молярность данного решения ДНК, на основе длине ДНК и его концентрация в растворе (например, Promega: http://www.promega.com/a/apps/biomath/index.html?calc=ugpmols). Калькулятор использует формулу: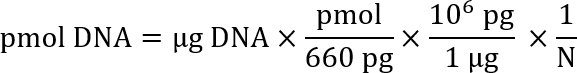
Здесь N — длина последовательности нуклеотидов.
- Настройка одновременного ограничения пищеварение и перевязка реакции с использованием протокола, изложенные в шаги 8-8.4 для клонирования PCR фрагмент в pSB700 вектор.
- Вместо добавления 1 мкл раствора грунт на реакцию, добавьте 1 мкл 40 fmole/мкл продукта PCR. Кроме того используйте 1 мкл pSB700 вектора в концентрации 40 fmole/мкл.
Примечание: Одинаковый молярный соотношения приводят к последовательным результатам, хотя даже в тех случаях, когда не используются точные коэффициенты, могут быть получены колоний.
- Вместо добавления 1 мкл раствора грунт на реакцию, добавьте 1 мкл 40 fmole/мкл продукта PCR. Кроме того используйте 1 мкл pSB700 вектора в концентрации 40 fmole/мкл.
- После завершения процесса реакции Золотые ворота (шаги 8-8.4), переходите к трансформации и последовательности проверки (шаги 9 - 10.2.3).
Результаты
Методы, изложенные в настоящем Протоколе, для создания векторов выражения либо единого или парные gRNA. Одноместный gRNA, выражая вектора могут быть созданы либо predigesting вектор позвоночника (рис. 1) следуют безлигатурные в серии коротких олигонуклеотиды или с помощью клонирования одновременно дайджест вектор позвоночника и перевязать его в Золотые ворота Серия коротких олигонуклеотиды в одном реакции. Также это метод производства векторов, которые содержат 2 оформления, каждый свой собственный независимый промоутер, движимый клонирование фрагмента пользовательский ПЦР (рис. 2).
Успешное клонирование для любого из намеченных протоколов приведет к внешний вид значительно больше колоний для преобразований с соответствующим Вставка ДНК чем на пластину без вставить элемент управления (рис. 3).
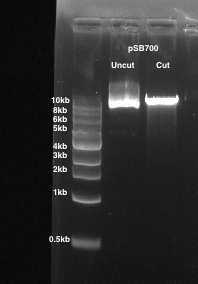
Рисунок 1: пищеварение вектора выражения gRNA pSB700. гель агарозы 1% используется. Переулок 1: режиссерский pSB700. Участок 2: pSB700 вырезать с BsmBI.
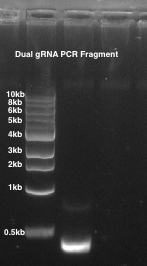
Рисунок 2: Парные gRNA ПЦР. гель агарозы 1% используется. Переулок 1: ПЦР фрагмент ~ 490 парных руководство bp.
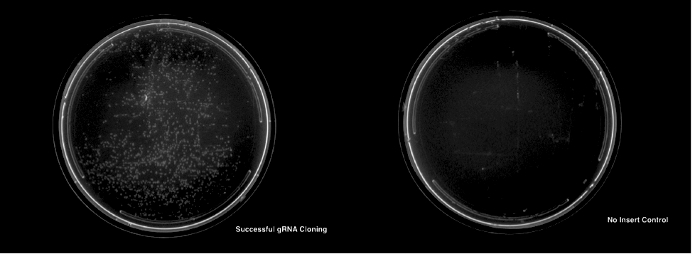
Рисунок 3: успешное клонирование и трансформация gRNA плазмида. Левая панель показывает LB и ампициллин пластины с успешно преобразован колоний. Правая панель показывает без Вставка управления пластины, показаны не колоний. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
| pSB700 руководство выражение вектор | 1-5 мкг |
| НЭБ буфер 3.1 | 4 МКЛ |
| BsmBI | 1 МКЛ |
| Дистиллированная вода | до 40 мкл |
| Общий объем | 40 МКЛ |
Таблица 1: Пищеварение ограничения вектора выражения gRNA pSB700 с BsmBI.
| Отожженная gRNA oligos (одинарные или пуле реакции) | 1 МКЛ |
| BsmBI усваивается pSB700 руководстве выражение вектор | 1 мкл (~ 100-250 нг) |
| 10 x T4 ДНК лигаза реакции буфера | 2 мкл (1 x конечная концентрация) |
| T4 ДНК лигаза | 1 мкл (120 шт.) |
| Дистиллированная вода | 15 МКЛ |
| Общий объем | 20 мкл |
Таблица 2: Перевязка реакция gRNA олигонуклеотиды в вектор выражения gRNA predigested pSB700.
| Реагент | Объем (1 x) |
| H2O | 6 МКЛ |
| Лигаза T4 | 0.5 МКЛ |
| ATP | 1 МКЛ |
| BsmBI | 0.5 МКЛ |
| НЭБ буфер 3.1 | 1 МКЛ |
| Вектор (например, pSB700) (40 фмоль) | 1 μlL |
| Вставка - вперед и обратить вспять oligos или ПЦР-фрагмент для двойной руководство (40fmol) | 1 мкл (0.5 мкл вперед и обратного 0.5 мкл) |
Таблица 3: Один шаг BsmBI ограничения и реакции перешнуровки gRNA mix.
| Для 1 гид | последовательность 3 колоний |
| 2 направляющих | последовательность 5-10 колоний |
| Для 3 руководства | последовательность колоний 10-15 |
| Для 4 направляющих | последовательность колоний 15-20 |
Таблица 4: Рекомендуемое количество колоний в последовательность для объединения gRNA клонирования реакций.
| ПЦР компонент | Реакция 50 мкл |
| 10 мкм вперед грунт | 2.5 МКЛ |
| Обратный Primer 10 мкм | 2.5 МКЛ |
| 5 x Phusion ВЧ или GC буфера | 10 МКЛ |
| дНТФ 10 мм | 1 МКЛ |
| Шаблон ДНК | 100 нг |
| ДНК-полимераза Phusion | 0.5 МКЛ |
| Нуклеаза свободной воды | До 50 мкл |
Таблица 5: Парные gRNA ПЦР смесь.
Обсуждение
Конструкции вектора выражения gRNA были внесены ряд изменений времени и экономии. Единый этапа ограничения и перевязка протокол изложен метод парных gRNA выражение вектор строительства. Парные gRNA выражение достигается за счет амплификации PCR используя олигонуклеотиды, содержащих последовательности целевого объекта вперед gRNA (№20) и обратный дополнения другой цели (№20). Это затем вводит промоутера вторичного РНК Pol III (7SK) а также sgRNA хвост в условно выражением единого gRNA выражение плазмид.
Тщательного олигонуклеотида дизайн обеспечит успешного PCR для парных gRNA строительства, а также липкой конце перевязки. После ограничения одноступенчатых и реакции перешнуровки, помимо дальнейшего BsmBI обеспечит что вырезать все неизмененные выражение векторов в реакции. Это значительно снизит фон после преобразования. С помощью НЭБ-Stable компетентным E. coli и роста на 30 ° C увеличит доходность успешных преобразований.
Преимущества, которые этот метод имеет над общей практики включают в себя упрощение процесса отжига, ограничение одноступенчатых gRNA вектора выражения и перевязка олигонуклеотиды, и способность использовать обычные олигонуклеотида векторы выражения одного gRNA для выражения парных гидов. В то время как протокол описанные здесь сосредоточена на наборе млекопитающих векторов, Общая стратегия пластика и применима к любой организм, условии доступен соответствующий gRNA вектор. В целом протокол, в описанный экономит время и затраты.
Следует, однако, отметить, что изложенные процедура приобретения отдельных олигонуклеотиды не масштабируется хорошо для создания библиотек, содержащих тысячи оформления. Для этих целей рекомендуется использовать альтернативные источники синтеза олигонуклеотида как oligo чип синтеза.
Это выгодно включить элемент управления без Вставка при клонировании оформления. Эта реакция может быть легко настроена параллельно с реакции интерес и может использоваться для определения, является ли неполное переваривание начальный вектор способствовала колоний, наблюдаемое в конце процедуры. Кроме того если один из вышеуказанных протоколов клонирования не удалось из-за неполностью переваренной вектор позвоночника или плохой перевязки эффективности, мы предлагаем попробовать альтернативный метод, который мы наметили клонирования.
При использовании клонирования одновременно дайджест вектор и перевязать в небольшой олигонуклеотиды в пределах одной реакции Золотые ворота, важно проверить, что gRNA, что клонируются в вектор не BsmBI сайт внутри него, как это приведет к t Он gRNA сокращаются и отсутствие колоний после преобразования.
Клонирование стратегия, которую мы наметили gRNA позволяет быстрое и экономически эффективным поколения оформления на ~ 10 долларов США за руководство, с основной объем расходов, исходя из синтеза олигонуклеотида и последовательность проверки. Хотя указывается метод предназначен чтобы позволить пользователям создавать оформления для использования с SpCas9, протокол может быть легко адаптирована для использования с Cas9 orthologues или других РНК руководствуясь эндонуклеазами, например Cpf1 или C2C2, с небольшими изменениями для векторных позвоночника и свес олигонуклеотида последовательности16,17.
Изложенные выше протокол будет обеспечивать проверку последовательности gRNA выражение плазмид в 3 d (начиная с надлежащим образом оформленные олигонуклеотидов), который значительно быстрее, чем нынешние методы. Это включает в себя дизайн gRNA 1 h (шаги 1-2.3), разбавление и Алиготе олигонуклеотиды 10 мин (шаги 3-5.1), пищеварения и очищения вектора выражения gRNA pSB700 2 h (шаги 6-6.4), клонирование gRNA олигонуклеотиды в предикаты предложенную pSB700 gRNA выражение вектора на ночь при комнатной температуре (шаги 7-7.3), лигирование ограничение BsmBI сингл шаг 3 ч. и 40 мин (шаги 8-8.4), преобразование 16 h (шаги 9-9,6) и проверки последовательности gRNA выражение плазмиды 24 ч (при продолжительности в зависимости от доступности последовательности Сэнгер и скорость после представления бактериальные колонии; шаг 10). Парные gRNA дизайн и изменения последовательности занимает 1 час (шаги 11-13). Парные gRNA ПЦР занимает 3.5 h (шаги 14-14.4). Клонирование PCR фрагмента в вектор pSB700 занимает 3 ч. и 40 мин (шаг 15-16).
Скорее всего проблемы, которые могут столкнуться с настоящим Протоколом относятся к неэффективности в Ассамблее Золотые ворота и трансформации. Включать элемент управления без вставка во время Ассамблеи Золотые ворота для визуализации эффективности Ассамблеи. Во время преобразования puc19 положительный контроль обеспечит контроль эффективности преобразования. НЭБ-Stable E. coli должен использоваться для лентивирусные совместимый плазмидов и часто требуют до > 16 h 30 ° c для получения видимых колоний.
Раскрытие информации
Джордж м. Церковь имеет справедливости в Editas и карибу Biosciences, которые стремятся коммерциализировать ТРИФОСФАТЫ-Cas9 генома редактирования технологии (полное раскрытие списка, см. http://arep.med.harvard.edu/gmc/tech.html).
Благодарности
Sathiji Nageshwaran поддерживается Наследственная атаксия Фридрейха исследования альянса (07340305 - 01) и национальных атаксии фонд стипендий (7355538-01). Алехандро Чавес финансировался в Национальный институт рака Грант 5T32CA009216-34 и Берроуз Уэллком фонд карьеры награды для ученых-медиков. Джордж м. Церковь поддерживается, США национальных институтов здравоохранения (НИЗ) национального человеческого генома научно-исследовательский институт Грант RM1 HG008525 и Институт Wyss биологически вдохновил инженерных. Джеймс Коллинз Дж. признает поддержку агентством по уменьшению угрозы обороне Грант HDTRA1-14-1-0006 и G. Пол Аллен границы группы. Алехандро Чавес разработал метод клонирования двойной гид. Sathiji Nageshwaran, Алехандро Чавес и Nan Шер ЕО написал рукопись с вводом от всех авторов.
Материалы
| Name | Company | Catalog Number | Comments |
| BsmBI | New England Biolabs | R0580L | |
| T4 DNA ligase | Enzymatic/New England Biolabs | L6030-LC-L/M0202S | |
| Buffer 3.1 | New England Biolabs | B7203S | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| QIAprep spin miniprep kit | Qiagen | 27104 | |
| Chemically competent E. coli | New England Biolabs | C3019l, C2987l, or C3040H | |
| Standard microcentrifuge tubes, 1.5 mL | Eppendorf | 0030 125.150 | |
| Axygen 8-Strip PCR tubes | Fischer Scientific | 14-222-250 | |
| Thermocycler with programmable temperature-stepping control | BioRad, | 1851148 | |
| UV spectrophotometer (NanoDrop 2000c) | Thermo Scientific | ||
| pSB700 plasmid | Addgene | #64046 | |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | c3040 | |
| Lysogeny broth (LB) | |||
| Ampicillin | |||
| TE buffer (1 mM EDTA, 10 mM Tris-Cl, pH 7.5) | |||
| pSN007 plasmid | Addgene | 102440 |
Ссылки
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America. 109 (39), E2579-E2586 (2012).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J., Charpentier, E. A Programmable Dual-RNA-Guided DNA Endonuclease in Adaptive Bacterial Immunity. Science. 337 (6096), 816-821 (2012).
- Shalem, O., Sanjana, N., Zhang, F. High-throughput functional genomics using CRISPR-Cas9. Nature Reviews Genetics. 16 (5), 299-311 (2015).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Gilbert, L., et al. CRISPR-Mediated Modular RNA-Guided Regulation of Transcription in Eukaryotes. Cell. 154 (2), 442-451 (2013).
- Yang, L., Yang, J., Byrne, S., Pan, J., Church, G. CRISPR/Cas9-Directed Genome Editing of Cultured Cells. Current Protocols in Molecular Biology. , 31.1.1-31.1.17 (2014).
- Yang, L., Mali, P., Kim-Kiselak, C., Church, G. CRISPR-Cas-Mediated Targeted Genome Editing in Human Cells. Methods in Molecular Biology. 1114, 245-267 (2014).
- Vidigal, J., Ventura, A. Rapid and efficient one-step generation of paired gRNA CRISPR-Cas9 libraries. Nature Communications. 6, 8083 (2015).
- Joung, J., et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nature Protocols. 12 (4), 828-863 (2017).
- Kabadi, A., Ousterout, D., Hilton, I., Gersbach, C. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Research. 42 (19), e147 (2014).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Scientific Reports. 4 (1), (2014).
- Chari, R., Yeo, N., Chavez, A., Church, G. sgRNA Scorer 2.0: A Species-Independent Model To Predict CRISPR/Cas9 Activity. ACS Synthetic Biology. 6 (5), 902-904 (2017).
- Doench, J., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nature Biotechnology. 32 (12), 1262-1267 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2014).
- Engler, C., Marillonnet, S., Valla, S., Lale, R. Golden Gate Cloning. DNA Cloning and Assembly Methods. , 119-131 (2013).
- Zetsche, B., et al. Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System. Cell. 163 (3), 759-771 (2015).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены