Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Дизайн и синтез аккордеон Шкаф реконфигурируемых ДНК
* Эти авторы внесли равный вклад
В этой статье
Резюме
Мы описываем подробный протокол для проектирования, моделирования, мокрый лабораторных экспериментов и анализа для реконфигурируемых ДНК аккордеон Шкаф 6 на 6 сеток.
Аннотация
ДНК, на основе наноструктурированных механических систем или ДНК наномашины, который производят сложные наноразмерных движения в 2D и 3D в нанометровом Ангстрем резолюции, показывает большой потенциал в различных областях нанотехнологий как молекулярные реакторов, Доставка лекарств, и nanoplasmonic систем. Реконфигурируемые ДНК Аккордеонный стойку, который коллективно могут манипулировать 2D или 3D наноразмерных сетевых элементов, в несколько этапов в ответ на ввод ДНК, описан. Платформа имеет потенциал для увеличения количество элементов, которые наномашин ДНК может контролировать от нескольких элементов в масштабе сети с несколькими этапов реорганизации.
В настоящем протоколе мы описывают весь экспериментальный процесс реконфигурируемых аккордеонных стойки ДНК 6 на 6 сеток. Протокол включает в себя дизайн правила и моделирования процедура структур и мокрой лаборатории эксперимента для синтеза и реконфигурации. Кроме того анализ структуры с использованием ТЕА (просвечивающей электронной микроскопии) и лад (передачи энергии резонансной флюоресценции) включен в протокол. Роман методы проектирования и моделирования, охватываемых в настоящем протоколе будет оказывать помощь исследователям использовать ДНК аккордеонных стойку для дальнейшего применения.
Введение
Механические системы, основанные на ДНК наноструктур или ДНК наномашин1,2,3,,45 являются уникальными, поскольку они производят сложные наноразмерных движения в 2D и 3D в нанометровом для Ангстрем резолюция, согласно различным биомолекулярных раздражители2,3,6. Подключая функциональных материалов на этих структур и контролируя их позиции, эти структуры могут применяться в различных областях. Например ДНК наномашин были предложены для молекулярной реактора7, наркотиков доставки8и nanoplasmonic систем в9,10.
Ранее мы внедрили перестраиваемой ДНК Аккордеонный стойку, которой можно манипулировать в 2D или 3D сети наноразмерных элементов11 (рис. 1A). В отличие от других ДНК наномашины, что контролировать только несколько элементов платформы можно коллективно управлять периодически выстроились 2D или 3D элементы в различных стадиях. Мы ожидаем, что программируемый химические и биологические реакции сети или молекулярные вычислительной системы может быть построен из нашей системы, увеличивая количество управляемых элементов. ДНК аккордеонных стойку является структурой, в котором сеть нескольких пучков ДНК подключен к суставов, состоящий из одноцепочечной ДНК (рис. 1B). Аккордеонный стойку, порожденных ДНК балки перенастраивается ДНК замками, которые гибридизируйте важная часть балки и изменить угол между балками согласно длина моста части замков (состояние блокировки). Кроме того многоступенчатый реконфигурации проявляется путем добавления новых замков после формирования свободного государства путем отсоединения замки ДНК через нити на основе зацепки перемещения12,13.
В этом протоколе мы описывают весь процесс проектирования и синтез реконфигурируемых ДНК аккордеонных стойки. Протокол включает в себя проектирование, моделирование, мокрый лабораторных экспериментов и анализ для синтеза ДНК аккордеонных стойки 6 на 6 сеток и реорганизации этих. Структура, охваченных Протоколом является базовой модели предыдущих исследований11 и 65 нм по 65 нм в размер, состоящий из 14 балки. С точки зрения проектирования и моделирования конструкции аккордеона стойки отличается от обычных ДНК оригами14,15 (т.е., плотно упакованы). Таким образом от традиционных методов были изменены правила, касающиеся конструкции и молекулярного моделирования. Для демонстрации, мы показываем методика проектирования, используя модифицированный подход caDNAno14 и моделирование аккордеонных стойки, с помощью oxDNA16,17 с дополнительных скриптов. Наконец описаны оба протокола ТЕА и лад для анализа структур настроенных аккордеонных стойку.
протокол
1. дизайн 6 по 6 ДНК аккордеон стойку с caDNAno14
- Скачать и установить caDNAno 2.0 программного обеспечения14 дизайн аккордеона стойку ДНК (caDNAno 2.5 также доступна на https://github.com/cadnano/cadnano2.5). Откройте caDNAno14 и нажмите Квадрат инструмент , чтобы добавить новую часть с квадратную решетку.
- Номер каждый луч аккордеонных стойки и опираться на панели слева решетки caDNAno14 (рис. 2).
- Выберите инструмент карандаш и рисовать каждый луч на панели справа редактирования caDNAno14. Балки перерыв каждые 32 bp, которая является для швов между смежными балки. Место основных кроссоверы в той же позиции, что суставы. Используйте Insert инструмент и Инструмент карандаш в caDNAno14 чтобы суставы имеют дополнительные одноцепочечной кроссоверы.
- Щелкните Инструмент карандаш и подключить суставов. Каждый луч имеет семь швов.
- Генерировать эшафот кроссоверы объединить подмости в единый цикл с помощью маршрутизации алгоритм11сообщалось ранее эшафот. Не позволяйте минимальный связывающий домен между леску и штапель нитка быть менее 8 bp (рис. 3).
- Место строительные леса, которые не используются в Ассамблее на вершины, расположенные на противоположных сторонах аккордеонных стойки, как показано на рисунке 3.
- Нажмите кнопку разорвать инструмент. Разорвать нитей, где основной нити круговой или длиннее 60 bp.
- Дизайн блокировки нитей ДНК.
- Нажмите кнопку разорвать инструмент. Bp перерыв 8 основных ДНК региона сделать липкое части и удалить 8 bp штапельные региона ДНК. Есть 18 липкие частей (рис. 1) в 6 по 6 аккордеонных стойку.
- Место последовательностей, которые обратный дополнение к липкой частей на обоих концах пряди блокировки и соединить их преодоление регион, который состоит из нитей поли T желаемой длины (рис. 1B).
- Для перенастройки, добавить 8 bp зацепки последовательностей в конце ДНК замки для перемещения стренги. Зацепки последовательность, используемая в таблице 2.
- Подготовьте поли A нити, которые являются вспять дополняет преодоление региона.
- Дизайн нитей, которые являются обратный дополняют ДНК замки для перенастройки эксперимент.
- Нажмите Последовательность инструмент и нажмите эшафот ДНК. Выберите эшафот, как стандартный M13mp18. Нажмите кнопку Экспорт инструмент и сохранить последовательность в формате csv (Таблица 1).
2. Имитация структуры с oxDNA
- Скачать и установить16,oxDNA17. Последние исходный код доступен на https://sourceforge.net/projects/oxdna/files/.
- Сделайте начальной конфигурации файлов из файла14 caDNAno, с помощью скрипта python «cadnano_interface.py», который находится в пакете17 oxDNA16,. Использование является следующим: «python cadnano_interface.py cadnano_file.json sq». В настоящее время создаются топологии файла и файла конфигурации.
Примечание: Файл топология включает в себя сколько прядей и нуклеотидов в структуре и информацию о позвоночник основа связей между нуклеотидов. Файл конфигурации содержит общие сведения, такие как скорость, энергию и размер коробки. Ориентация информацию, такую как вектор, позвоночник база вектор, вектор нормали, скорости и угловой скорости нуклеотидов является также включены (рис. 4). - Измените информацию в файле топологии и конфигурации caDNAno14 , чтобы они отражали реальные структурной информации аккордеонных стойку. Все лучи расположены параллельно, когда топологии и конфигурации файлы из caDNAno14 визуализируются. Однако Аккордеонный стойку является структура решетки поэтому расстояние между кабального нуклеотиды являются далеко для моделирования (рис. 5).
- Повернуть и двигаться каждый луч к структуре требуемый решетке. Девять столбцы слева в файле конфигурации являются вектор, позвоночник база вектор и вектор нормали (рис. 4). Чтобы повернуть луч, поверните все позиции, позвоночник база и нормальной векторов с использованием вращения преобразования. Затем переместите луч, изменив радиус-вектором найти его, как показано на рисунке 5.
- Расслабьтесь структуры, используя сценарий, предоставленный в пакете oxDNA (см. пример в $oxDNA/примеры/RELAX_INITIAL_CONFIGURATION для получения дополнительной информации).
- Запуск моделирования молекулярной динамики для десяти миллионов шагов, с помощью файла конфигурации расслабленной. Использование является следующим: '. / oxDNA < input >' сохранить данные каждые 5000 или 10000 шагов.
- Визуализация
Примечание: Структуры были визуализируется с помощью cogli.- Загрузите и установите последнюю версию cogli (https://sourceforge.net/projects/cogli1/).
- Запустите cogli с файлами, топологии и конфигурации из oxDNA моделирования. Использование является следующим: '. / cogli1 -t < топологии файл >< файл конфигурации >'.
- Скройте поле, нажав b.
3. Синтез структуры
Примечание: Метод синтеза адаптирована от предыдущего протокол15,18.
- Приобрести разработан ДНК скобы от поставщика олигонуклеотида.
- Отрегулируйте концентрацию этих ДНК скоб до 100 мкм с использованием воды, свободной от нуклеиназы.
- Бассейн каждой стренге дна, который представляет собой структуру «свободный состояние» в одну трубу и регулировка концентрации до 2 мкм для каждой стренги.
- Пул блокировки ДНК пряди, Длина и количество блокировки сайтов в пробирки и регулировка концентрации до 2 мкм для каждой стренги. 18, 9 и 4 Блокировка сайтов используются. Добавьте поли A пряди, которые являются взаимодополняющими в преодоление регион на такой же концентрации.
- Пула нитей, которые являются вспять дополняет ДНК блокировки нити длиной в пробирки и регулировка концентрации до 2 мкм для каждой стренги.
- Приготовляют раствор2 MgCl 300 Нм, смешивая 70 мкл нуклеиназы свободной воды и 30 мкл раствора2 MgCl 1 мкм. Подготовьте 5 x трис ЭДТА решение путем смешивания 95 мкл нуклеиназы свободной воды и 5 мкл раствора ЭДТА трис 100 x.
- Добавить 2 мкл штапель ДНК, 1.1 мкл раствора MgCl2 , 2 мкл раствора ЭДТА трис, 7.6 мкл нуклеиназы свободной воды и 7.3 мкл эшафот ДНК из которых концентрация составляет 110 Нм сделать 20 мкл смешанных запасов. Установите конечная концентрация эшафот ДНК до 40 Нм, штапельное ДНК 200 Нм,2 MgCl до 16 мм и трис ЭДТА решение 0.5 x.
- Быстро тепло смешанных Стоковый раствор в тепловой циклователь до 80 ° C и охладить до 60 ° C со скоростью 4 мин на ° C и прохладный от 60 ° С до 4 ° C со скоростью 40 мин в ° C.
4. Очистка структуры
Примечание: Образцы всех структур были очищены перед анализом. В этом разделе мы описываем протокол PEG очистки, который заимствован из предыдущих литература19. Образец также может быть очищен электрофорезом геля, как описано в предыдущих литературы15,18.
- Подготовьте 5 М NaCl и 100 x трис ЭДТА.
- Подготовка осадков буфера путем смешивания 150 мкл PEG 8000, 500 мкл, 100 x трис ЭДТА и 101 мкл 5 М NaCl и 249 мкл воды, свободной от нуклеиназы.
- Подготовка целевого буфера путем смешивания 5.5 мкл раствора 300 Нм MgCl2 от раздел 3.3, 10 мкл раствора ЭДТА трис 5 x из раздела 3.3 и 84,5 мкл нуклеиназы свободной воды.
- Микс 20 мкл синтезированные структуры из раздела 3 и 20 мкл осадков буфера раздел 4.2. То спина смешанных запасов на 16000 x g при 4 ° C. Удалить супернатант и растворяют гранулы в целевой буфер из раздел 4.3.
5. Перенастройка аккордеонных стойки от «Свободное государство» заблокирован государства
- Синтезировать структуры без блокировки ДНК для настройки эксперимента.
- Подготовка нитей ДНК блокировки из раздела 3.
- Добавьте 2 мкл блокировки нитей ДНК желаемой длины в 20 мкл синтезированные структуры. Концентрация ДНК пряди блокировки пять раз выше, чем структура.
- Проинкубируйте образцы для 0, 10, 25, 50 или 100 минут, чтобы увидеть как быстро перенастройка происходит.
- Для 100 минут инкубации инкубации образца при температуре 50 ° C за 30 минут и медленно остыть до 25 ° C в размере 0,33 ° C в минуту.
- За 50 минут инкубации инкубации образца при температуре 50 ° C на 15 минут и медленно остыть до 25 ° C со скоростью 0.66 ° C в минуту.
- За 25 минут инкубации инкубации образца при температуре 50 ° C для 7,5 минут и медленно остыть до 25 ° C со скоростью до 1,32 ° C в минуту.
- За 10 минут инкубации инкубации образца при температуре 50 ° C за 3 минуты и медленно остыть до 25 ° C со скоростью 3,3 ° C в минуту.
- Для 0-минутный инкубации добавляются образец магазина на 4 ° C сразу после блокирует ДНК пряди.
- Сразу после крепления шаг быстро охладить вниз образца до 4 ° C для предотвращения нежелательных денатурации.
6. Перенастройка аккордеонных стойки от «Заблокирован государство» к «Свободное государство»
- Синтезировать структуры ДНК пряди нужной длины для настройки эксперимента.
- Подготовьте обратный дополнительных прядей от раздела 3.
- Добавьте 2 мкл нитей, которые являются вспять дополняет стренги блокировки требуемой длины в 20 мкл синтезированные структуры. Концентрация ДНК пряди блокировки пять раз выше, чем структура.
- Проинкубируйте образцы для 0, 12, 60, 120, 240 минут, чтобы увидеть как быстро перенастройка происходит.
- Для 12, 60, 120, 240 минут инкубации, быстро нагреть образца до 40 ° C и медленно остыть до 20 ° C для времени, соответствующие каждому. Сразу после отсоединение шаг быстро охладить вниз образца до 4 ° C для предотвращения нежелательных денатурации.
- Для 0-минутный инкубации Храните образец справа 4 ° C, после добавления обратного дополнительные пряди.
7. ТЕА изображений
Примечание: ТЕА изображений протокол был адаптирован от предыдущих литература18,20.
- Готовят раствор NaOH 1,25 М путем смешивания 87.5 мкл нуклеиназы свободной воды и 12,5 мкл раствора NaOH 10 М.
- Добавьте 50 мкл 2% раствор уранилнитрата формате 1 мкл раствора NaOH 1,25 М.
- Вихрь решение для 3 минуты и центрифуги на Макс скорость на 3 минуты. Депозит 3 мкл очищенный образца на сетке ТЕА свечения выписан 3 минуты и быстро промойте фильтр-бумаги.
- Депонирование 7 мкл подготовленных уранила Формиат решения для 30 секунд и быстро промойте фильтр-бумаги.
- Измерьте длину и угол аккордеонных структуры, отображаемого на ТЕА.
8. ладу анализ
- Используйте Атто 550 и Atto 647N краска, для которого Фёрстер расстояние составляет 6,5 Нм. Замените дневно помечены пряди штапель 58 и 117 штапель в таблице 1 . Затем, синтезировать структуры с дневно помечены нити методом, описанным в разделе 3.
- Измерьте концентрацию очищенный образца.
- Нормализовать образца до 10 Нм и нагрузки 50 мкл до 384 планшет хорошо.
- Возбуждают образца с доноров и акцепторов красители на 550 Нм и мера спектр флуоресценции от 570 Нм до 800 нм с флуориметр.
- Измерьте спектр флуоресценции доноров только образца таким же образом.
- Возбуждают красители образца на 650 нм и спектр флуоресценции и измерьте расстояние от 670 нм до 800 Нм. Это измерить концентрацию акцептор.
- Получение стандартных отклонений, повторяя же эксперимент с трех образцов, которые синтезируются и очищается отдельно.
- Рассчитайте эффективность ладу с соотношением метод как описано уравнением ниже21.
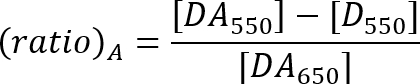
Да550: акцептор пика интенсивности флуоресценции образца с доноров и акцепторов в 550 Нм возбуждения.
550D: интенсивности флуоресценции в диапазон выбросов акцептора доноров только образца на 550 Нм возбуждения.
Да650: акцептор пика интенсивности флуоресценции образца с доноров и акцепторов в 650 Нм возбуждения.
Результаты
Аккордеонный стойку разработан 6 по 6 ДНК моделируется от16,oxDNA17 и результаты показаны на рисунке 6. В результате моделирования было подтверждено, что предполагаемой структуры формируется без искажения структуры.
Обсуждение
Этот протокол вводит весь процесс от проектирования, моделирования, синтеза и анализа основных 2D ДНК аккордеонных стойки. Модифицированный дизайн и моделирование правила были описаны потому что правило дизайн отличается от стандартных ДНК-оригами, в том, что ДНК аккордеон Шкаф имеет д...
Раскрытие информации
Авторы не имеют ничего сообщать
Благодарности
Это исследование было частично поддерживается глобальной программы центр исследований развития через Национальный исследовательский фонд Korea(NRF) финансируется министерством науки и ИКТ (MSIT) (2015K1A4A3047345) и Nano· Программа развития технологии материал через национальных исследований фонда из Кореи (NRF) финансируется министерством науки и ИКТ (MSIT) (2012M3A7A9671610). Институт инженерных исследований в Сеульском национальном университете представила научно-исследовательских учреждений для этой работы. Авторы признают благодарность Tae-Янг Юн (биологических наук, Сеульский Национальный университет) относительно флуоресцентной спектроскопии для анализа ладу.
Материалы
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Ссылки
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -. G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127 (2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102 (2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101 (2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272 (2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486 (2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены