Method Article
Изотропная световая микроскопия и автоматизированный анализ линии клеток для каталога Caenorhabditis elegans Embryogenesis с подклеточным разрешением
В этой статье
Резюме
Здесь мы представляем комбинаторный подход с использованием микроскопии высокого разрешения, вычислительных инструментов и одноклеточной маркировки в живых эмбрионах C. elegans для понимания динамики одной клетки во время нейроразвития.
Аннотация
Канорхабдит элеганы (C. elegans) выделяется как единственный организм, в котором проблема понимания клеточного происхождения всей нервной системы можно наблюдать, с разрешением одной клетки, in vivo. Здесь мы представляем комплексный протокол для обследования нейроразвития эмбрионов C. elegans. Наш протокол сочетает в себе визуализацию, линейный и нейроанатомический отслеживание одиночных клеток в развивающихся эмбрионах. Мы достигаем долгосрочной, четырехмерной (4D) визуализации живых эмбрионов C. elegans с почти изотропным пространственным разрешением с помощью перевернутой селективной микроскопии плоскости (diSPIM). Ядра и нейрональные структуры в эмбрионах нематод изображены и изотрополово слиты, чтобы дать изображения с разрешением 330 нм во всех трех измерениях. Эти поминутные наборы данных с высоким разрешением 4D затем анализируются для корреляции окончательных клеточных лидентий с экспрессией генов и морфологической динамикой на одноклеточных и субклеточных уровнях детализации. Наш протокол структурирован таким образом, чтобы обеспечить модульную реализацию каждого из описанных шагов и усилить исследования по эмбриогенезу, экспрессии генов или нейроразвитии.
Введение
C. elegans выделяется как единственный организм, в котором каждая клетка эмбриона может наблюдаться на протяжении всего развития нервной системы. При всей клеточной линииизвестны и инвариант1 , а также с развитием новых инструментов, которые позволяют маркировки и непрерывной визуализации одиночных клеток в эмбрионах, биологи теперь могут начать изучение различных шагов в развитии нематод нервной система со всех сторон - рождение клеток; миграция и дифференциация; невритовое образование, целенаправленный рост и фасцикуляция; синапсовое образование; и настройка функциональных схем. Захват динамики роста нейронов в эмбрионе C. elegans, сочетая заведомые репортеры и флуоресценцию микроскопии, ценен для научного сообщества.
Исследования в области развития в C. elegans часто используют инвариантные карты клеточной линии и клеточной судьбыэтого вида для расширения контекстуального понимания на одноклеточном уровне в нетронутом организме 1. Автолинейный анализ - с использованием StarryNite2,3,4 и AceTree5,6,7,8 программного обеспечения - выгоды от высокой контрастации, высокого разрешения изображения флуоресцентных ядер. Для оптимальной работы StarryNite/AceTree также зависит от предсказуемой ограниченной ориентации изображенных эмбрионов во время развития. Конфокальная микроскопия, сделанная в эмбрионах C. elegans, сжатых между двумя крышками, является стандартным методом автоматической линии микроскопии уже более десяти лет, поскольку она обеспечивает как высокий контрастный/высокий, так и предсказуемый ограниченный ориентация эмбриона7,8. Ранее мы описали строительство и использование нового светлиста на основе двойного вида перевернутый селективный микроскоп освещения плоскости (diSPIM) для живой образец изображения, такие как C. elegans эмбриогенез9,10 , 11 Год , 12 Лет , 13. Светолистная микроскопия, в общем, обеспечивает низкую фототоксичность, высокую скорость и долгосрочную визуализацию живых 3D-образцов14,15. Метод diSPIM, в частности, производит четырехмерные (4D) изображения с почти изотропным пространственным разрешением примерно 330 нм9.
По сравнению с конфокальной микроскопией, diSPIM предлагает более высокую скорость сигнала к шуму и скорости, более изотропное пространственное разрешение, и больше подходит для долгосрочного in vivo изображений16. Поэтому мы работали над адаптацией данных diSPIM для ввода в StarryNite/AceTree и исследовали, будет ли это способствовать расширению линейных анализов. Основным препятствием является то, что diSPIM образцы не могут быть легко ограничены яичной скорлупы сжатия принять ожидаемые ориентации для StarryNite / AceTree. Случайная ориентация клеточных позиций в анализируемом объеме ухудшает точность ауто-линейного анализа.
Поэтому мы использовали CytoSHOW, пользовательский интерфейс, управляемый зрителем, который позволяет пользователям выбирать точную 3D-ориентацию эмбрионов во время предварительной обработки изображений diSPIM, принося данные изображений, которые являются одновременно оптимизированными и контексто-осведомленными для ввода в StarryNite /AceTree. При выборе пользователями изображений эмбрионов, CytoSHOW организует автоматизированный конвейер обработки данных. Обрезанные и фоновые изображения эмбрионов сохраняются в файлах стека TIFF для каждой позиции, временной точки и представления. CytoSHOW затем итеративно вызывает программу SpimFusion совместно зарегистрироваться и совместно deconvolve два предварительно обработанных просмотров, используя Ричардсон-Люси17,18 алгоритм для получения изотропных высокой разрешения объемных изображений. Для StarryNite был оптимизирован набор параметров diSPIM для управления своим поведением во время сегментации изображений и отслеживания ядер на изотропически сброшенных изображениях. Сброшенные изображения и результаты lineaging затем редактируются с помощью AceTree, что позволяет пользователям выявлять и исправлять любые ошибки в авто-линии трассировки, генерируемых StarryNite. AceTree также может представить линию дерева и 3D моделируемых визуализаций гусеничных ядер в эмбрионе. Мы находим, что скорость и точность автоматической линии заметно повышаются с помощью изотропически сброшенных изображений, по сравнению с необработанными изображениями с камеры SPIM. Наш протокол, хотя и оптимизирован для описанного здесь приложения C. elegans, в целом может быть адаптирован для автоматического оформления данных diSPIM, полученных для других видов или образцов. Если это предполагаемое использование протокола, обратите внимание, что дополнительная настройка параметров StarryNite, вероятно, потребуется для новых образцов, как описано3,4.
Успешная реализация этого протокола приводит к изображениям с 4D-изотропным разрешением и позволяет биологам отслеживать линии клеток, одновременно выявляя и анализируя нейроны в развивающемся эмбрионе C. elegans. Более того, объединив несколько алгоритмов пост-обработки - с аппаратным ускорением, который является наиболее трудоемким из них - теперь мы можем анализировать как мелкие субклеточные детали, так и клеточные линии и клеточные судьбы живых эмбрионов практически в режиме реального времени. Этот новый протокол позволяет точные, обоснованные манипуляции и наблюдения поведения клеток во время доказательных исследований дифференциации и морфогенеза in vivo. В этой рукописи мы представляем подробное объяснение усовершенствованных протоколов, разработанных нами для отслеживания линий и клеток при разработке эмбрионов C. elegans, для повышения уровня исследований эмбриогенеза, экспрессии генов или нейроразвития.
протокол
1. Сборка камеры визуализации diSPIM стальной с поли-L-лизин покрытием coverslip
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМ. Несколько вариантов (указанных как таковые) могут быть опущены для экспериментов, которые требуют отслеживания линий клеток C. elegans.
- Нарисуйте небольшой прямоугольник (2 мм х 5 мм) в центре чистого прямоугольного обкрытия (No 1.5, 24 мм x 50 мм) с помощью остроты (или аналогичной ручки).
- Переверните крышку на немаркированную сторону и поместите 10 зл поли-L-лизина (кошка. нет. P1524) над отмеченный прямоугольник.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ОДНОЗАМЕЧАНИе, сделать рабочую концентрацию 1 мг/мл поли-л-лизин растворяется в фильтрофильтротой воде (или эквивалент). Для длительного хранения, подготовить 5-10 мл aliquots и хранить на 20 градусов по Цельсию. После размораживания, aliquots могут храниться при комнатной температуре (23 кВ) в течение 3-4 недель. - Разрешить поли-L-лизин, чтобы покрыть крышку в течение 5 минут(рисунок 1A). Поли-L-лизин используется для покрытия стеклянной крышкой скользит, где эмбрионы будут установлены, что позволяет эмбриона яичной скорлупы твердо придерживаться coverslip, даже когда цели погружены в изображении среды.
- Поместите поли-L-лизин покрытием coverslip в нижней половине камеры изображения стали.
- Поместите верхнюю половину стальной камеры изображения на нижнюю половину с крышкой и затяните с четырьмя винтами, связанными с камерой. Проверьте визуально со стороны, чтобы убедиться, что верхняя половина равномерно сидит в нижней половине(рисунок 1B).
- Заполните камеру 7-8 мл буфера M919, изотонический буфер, который помогает предотвратить ранние эмбрионы стадии от поддавшись аномальное осмотическое давление. 1-клеточные, и даже 2-клеточные эмбрионы осмотически чувствительны и могут развиваться аномально, если не в этом изотоновом буфере. Эмбрионы также, как правило, арест на 3-кратном этапе, если M9 заменяется водой.
2. Подготовка образцов эмбрионов C. elegans для монтажа
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ примечание мною, приблизительно 18 часов до изображения, 5 молодых (1 день с окончательного молочницы) взрослые и 10 личинок этап 4 (L4) C. elegans выбраны к среде роста нематода (NGM) агарплита ссеянная с штаммом OP50 кишечной палочки. Платиновые провода выбирает используются для перемещения личинок и молодых взрослых C. elegans без ущерба для животного19.
- Приготовьте 1% метилцеллюлозы (кошка. нет. Решение H7509-25G в буфере M9.
ПРИМЕЧАНИЕ Примечание Примечание Примечание, метиловая целлюлоза должна быть перемешивают в теплой M9, пока он не растворяется. После подготовки, это решение может храниться при комнатной температуре. - Добавьте 500 зл 1% метиловой целлюлозы в M9 растворвиться в депрессии вогнутого слайда микроскопа. Этот вязкий буфер будет использоваться в двух случаях, 1) при сборе ранних эмбрионов путем вскрытия взрослых червей и 2) при мытье эмбрионов поздней стадии, собранных непосредственно из пластины NGM.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, метилцеллюлоза используется для предотвращения эмбрионов от прилипания к микроскопу слайд. - Для визуализации эмбрионов поздней стадии, выбрать заложены C. elegans эмбрионов (желательно из NGM пластины с молодыми взрослыми настоящее время) с помощью ресниц забрать, и переместить эмбрионы на 1% метиловой целлюлозы на вогнутый микроскоп слайд. Выбор ресниц помогает уменьшить силу и тем самым свести к минимуму стресс или повреждение эмбрионов во время обработки. Процедура, чтобы сделать ресницы забрать покрывается Харт20.
- При втором выборе ресниц (в противоположной руке) аккуратно коснитесь обе ресницы вместе, чтобы приостановить эмбрионы в метиловой целлюлозе.
- Дополнительно ещё при планировании линии эмбриональных клеток с помощью StarryNite необходимо смонтировать 1-клеточные до 4-клеточных эмбрионов. Для этого сначала выберите молодых людей из пластины NGM и переместите их в раствор целлюлозы M9-метил овой на вогнутом микроскопии слайд с помощью платинового провода забрать.
- Дополнительно ели к заточенным кончикам подкожных игл (No 18G x 1 1/2), нарежьте животное поперечным на среднем теле, чтобы выпустить 1-клеточные до 4-клеточных эмбрионов.
3. Рот Пипетта.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, Мы используем трубку аспиратора с рукой вытащил микрокапиллярной пипетки вставляется в резиновой прокладкой трубки. Это позволяет нам перенести эмбрионы из скольжения на поверхность с покрытием Споли-Л-лизин омицы в заполненной буфером камере изображений.
- Вручную потяните микрокапиллярную пипетку над открытым пламенем, чтобы создать две половинки с растянутыми кончиками.
- Возьмите половину микрокапиллярной пипетки и вставьте тупой конец в резиновую прокладку трубки аспиратора(рисунок 1C). Установите другую половину микрокапиллярной пипетки в сторону для использования позже (при необходимости).
- С собранной трубкой аспиратора оснащены микрокапиллярной пипеткой, аккуратно разорвать кончик микрокапиллярной пипетки и создать отверстие, которое будет соответствовать около 1-2 двух эмбрионов (отсюда на этом инструменте называется "рот пипетка").
4. Монтаж C. elegans эмбрионов на Поли-L-лизин покрытием coverslip
- С аспиратором мундштук провел мягко между зубами, предварительно заполнить микрокапиллярный пипетка с 10-15 Зл Л L M9 Buffer, а затем осторожно сосать несколько эмбрионов из вогнутой слайд в капилляр.
- Перенесите эмбрионы в стальную камеру визуализации, наполненную M9 Buffer, позиционируя кончик капилляра так, чтобы эмбрионы попали в центральный прямоугольник крышки.
- Избегая травмы эмбрионов, аккуратно перемещать их с ресницазабратьи забрать или использовать пипетку рот, чтобы положение эмбрионов вертикально, чтобы сориентировать эмбрионы так, что длинная ось эмбриона перпендикулярно длинной оси крышки (Рисунок 1B вставляя , нижняя панель).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ отображение эмбриона в этой ориентации сводит к минимуму количество срезов к изображению, тем самым уменьшая дозировку света и время обработки данных при одновременном повышении скорости приобретения. - Поместите стальную камеру изображения в держатель образца на стадии микроскопа(Рисунок 1D).
5. Сборка, настройка программного обеспечения и лазерная оптимизация для эмбриональной визуализации с использованием diSPIM
- Смотрите пошаговые инструкции о том, как собрать весь волоконно-связанный diSPIM из коммерчески доступных частей в Кумар и др.10,11 и в http://www.dispim.org. Видеопротокол о том, как собрать diSPIM также доступен на веб-сайте ASI (http://www.asiimaging.com).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ОДНОЗАМЕЧАНИЕ, установка инструмента для этого протокола идентична Кумар у и др.10,11 diSPIM, который использует 40x 0.8NA линзы погружения воды для визуализации. Единственное различие между установкой в этом протоколе и Kumar et al.10,11 является добавлением дихромного зеркала (расщепление на 560 нм) и красных и зеленых фильтров bandpass внутри устройства расщепления изображений (модель A12801-01), установленного на как diSPIM изображений оружия. Добавление расщепления изображения позволяет одновременно захватизображения с двух различных фторфоров - возбужденных 561 нм и 488 нм лазеров - путем разделения полосы выбросов на две половины одного чипа камеры. - После сборки прибора проверьте оптическое выравнивание diSPIM перед визуализацией.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, чтобы правильно выровнять diSPIM видеть https://youtu.be/qnOrg30NNuE, и для аппаратной информации, http://dispim.org/hardware/objectives и http://www.asiimaging.com. - Используйте платформу с открытым исходным кодом Micro-Manager (https://micro-manager.org/)21, которая была оптимизирована для работы световых листов микроскопов для высокой пропускной связи22. Мы рекомендуем использовать плагин ASI diSPIM для многопозиционного приобретения, который позволяет одновременно с изображением до 30 эмбрионов, как описано23.
- С открытым микро-менеджером, установите интенсивность лазера до 179 зВт (0,5) для 488 нм и 79 мВт (0,25) за 561 нм(рисунок 2A, красный прямоугольник).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ- Рекомендуемые настройки для долгосрочной визуализации эмбрионов C. elegans с использованием 1-минутных интервалов. Во время долгосрочного двухцветного изображения лазер 561 нм используется для изображения ядер(mCherry.ru),пока эмбрионы не находятся на стадии фаны, после чего лазер 488 нм затем включится, чтобы также сделать изображение нейронов с маркировкой GFP. Эти условия визуализации оптимизированы для минимизации фототоксичности и обеспечения выживания и вылупления эмбрионов, позволяя при этом длительное (12-14 часов) непрерывное получение данных нейроразвития и линии. - В Micro-Менеджер, выберите меню Плагины (gt; Управление устройством) Выберите вкладку приобретения. В разделе Параметры сохранения данных этой вкладки (зеленый прямоугольник), раздел «Настройки объема» (синий квадрат) и раздел «Параметры фрагмента» (оранжевый квадрат) убедитесь, что каждый параметр настроен так, как показано на рисунке 2B.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ОДНОКРАТНАЯ обработка данных CytoSHOW адаптирована для работы с другими дополнительными форматами выходных данных, такими как серии файлов OME-TIFF и серия файлов TIFF-stack, созданная после приобретения за счет использования экспортной функции, встроенной в Micro-Manager. Как правило, используется одновременный стек, собранный формат файловых данных OME-TIFF, поскольку он позволяет в режиме реального времени просматривать и обрабатывать объем изображения сразу же после приобретения исходных данных.
6. Оптимизированные параметры автофокуса для долгосрочной визуализации эмбрионов C. elegans
- Установите параметры автофокуса Micro-Manager в настройки, оптимизированные для долгосрочного линейного качества diSPIM-изображения эмбрионов C. elegans. В окне ASI diSPIM щелкните вкладку Автофокус (рисунок 2C). В разделе General Autofocus Options (черный квадрат) указаны параметры точно так, как показано на рисунке. Обратите внимание, что канал Автофокуса (красный квадрат) должен указать ваш канал флуоресценции ядерного канала в линейных экспериментах.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, Если максимальная смещение больше, чем 5 мкм, изображения, как правило, дрейфовать из фокуса. - Нажмите, Плагины (ru) и инструменты приобретения ( и шаблон наложения.
- В окне накладки шаблона щелкните Show Grid.
- В окне ASI diSPIM щелкните вкладку «Навигация».
- Нажмите флажки для пучка и листа Пути A или B, а затем нажмите Live. Начинается приобретение изображения. Открывается окно Live view. Выберите область анализа автофокусировки эмбриона, нарисовав коробку вокруг эмбриона на выбранном канале от 6.1.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, мы обычно захватываем 420 точек времени для 10 эмбрионов за сеанс изображения. Необработанные данные на сеанс изображения обычно составляют 1,7 ТБ, в то время как данные deconvolved и StarryNite — 1,4 ТБ (см. Шаги 9 и 10). Мы рекомендуем использовать HDD большой емкости (18 ТБ в нашей текущей системе) для получения изображений и облачные платформы для хранения изображений. - Нажмите Начало Приобретение в "Приобретение вкладка, чтобы начать долгосрочный многомерный захват изображения (Рисунок 2B).
7. Открытие необработанных изображений микро-менеджера в CytoSHOW
- Скачать пакет программного обеспечения из http://dispimlineage.wormguides.org.
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ примечание программирования будет downloaded как архив .zip и будет необходимо извлечь к «C'» каталог перед использованием. Более подробная информация для установки приведена в http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm . - Дважды щелкните файл C.'CytoSHOWextrasForC-CytoSHOW-APP.jnlp, чтобы начать работу CytoSHOW.
- Выберите меню Файл (new) и диспим монитор (Микро-менеджер). Найдите папку набора корневых данных, в которой были сохранены папки времени приобретения. Выберите папку временной точки и нажмите Open. Многомерные навигационные окна (так называемые окна монитора diSPIM) открываются автоматически как для SPIMA, так и для SPIMB(рисунок 3A).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ- Эти окна будут контролировать папку исходных данных для недавно сохраненных необработанных стеков тайм-пойнтов (в случае, если образец все еще записывается). После того, как каждый новый тайм-пойнт приобретается, каждое из окон, отслеживающих различные позиции SPIM и образец, освежается для отображения всего многоканального 4D-набора данных для каждого эмбриона.
8. Создание Макс проекционные изображения с CytoSHOW
Еще до деконволюации необработанные данные могут быть быстро обработаны для оценки глобальных особенностей образца.
- Нажмите кнопку «К-MIP» на левой боковой панели окна изображения(рисунок 3A, красный прямоугольник),чтобы сделать максимальные проекции интенсивности через полную глубину и полный курс данной позиции или руки SPIM. Появится окно гиперстога с проекцией.
- В окне гиперстога проекции, установить тип проекции к интенсивности Max. Укажите, какие каналы, срезы и кадры временных точек для обработки на основе предпочтений пользователя.
- Нажмите OK при завершении.
- Выберите местоположение папки, чтобы сохранить выводы интенсивности Max из окна диалогов файлов, а затем нажмите OK. Разрешить некоторое время (15-20 минут, в зависимости от размера набора данных и вычислительной мощности компьютера) для CytoSHOW для создания проекционных изображений.
9. Анализ клеточных линий в изотропных объемных данных с высоким разрешением
- Дополнительное средство для открытия необработанных данных с помощью монитора diSPIM в CytoSHOW выберите инструмент выбора Полигона (рисунок3A,черные стрелки) и нажмите только за пределами передней, задней, спинной и вентральной краев (в том, что точный порядок) создать "боути" над эмбрионом. Делайте для обоих представлений (SPIM-A и SPIM-B, рисунок 3A).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ, этот выбор определяет эллиптической области интереса (ROI), в котором эмбрион в центре и записи передней задней оси эмбриона. Шаблон bowtie сигналии CytoSHOW, что пользователь планирует дополнительно указать точное вращение окончательного изотропно слитого тома в ориентацию, которая является оптимальной для линейных анализов StarryNite / AceTree. В тех случаях, когда lineaging StarryNite не является частью экспериментального плана, другие инструменты и формы выбора могут быть выбраны для установки рентабельности инвестиций для обработки изображений. - Если несколько эмбрионов были одновременно изображены с помощью многопозиционного варианта приобретения, откройте и выполните Шаг 9.1 для всех эмбрионов. Это позволит параллельно вывести будущие шаги для всех эмбрионов за один сеанс. Закройте окна SIMA и SPIMB для любых эмбрионов, которые вы не хотите обрабатывать.
- Нажмите кнопку diSPIM на левой боковой панели окна монитора diSPIM(рисунок 3A, выделенжелтый желтым цветом). Это показывает подпанель элементов управления, специфичных для обработки diSPIM.
- Выровняйте зеленые и красные каналы для каждой руки SPIM. Поскольку сплиттеры каналов выбросов используются для захвата различных красных и зеленых изображений одновременно на одной камере, важно визуально выровнять точную регистрацию пикселей этих двух физически смежных полей изображения, когда они наложены. Повторное использование одних и тех же корректировок выравнивания, как правило, возможно во многих последовательных сеансах визуализации, но должно быть проверено (как в шагах 9.4.1-9.4.5).
- Начиная с панели SPIMA, выберите красный канал, переместив бар прокрутки CM влево (рисунок3A,оранжевая стрелка, левая панель).
- Используя x-, y-и-регуляторы(рисунок 3A,оранжевый квадрат), сдвините красный канал, чтобы соответствовать зеленому цвету.
- Нажмите кнопку diSPIM (рисунок 3A, выделенный желтым цветом), чтобы закрыть подпанель и вызвать распространение тех же сдвигов на все другие окна позиции.
- Подтвердите, что правильное выравнивание распространяется на другие кадры и временные точки, перемещая бар прокрутки "z" (рисунок3A, синяя стрелка, левая панель) и/или "t" прокрутка бар (рисунок3A, зеленая стрелка, левая панель). Если было выполнено многопозиционное приобретение и было изображено несколько эмбрионов (Шаг 5.3), выравнивание должно было также распространяться на эти эмбрионы. Подтвердите, также изучая номера для x-, y-, и z-регуляторов(рисунок 3A, оранжевый квадрат, который должен быть одинаковым для панели SPIMA всех эмбрионов).
- Повторите шаги 9.4.1-9.4.4 для окна монитора SPIMB diSPIM(рисунок 3A,правая панель).
- Нажмите кнопку"diSPIM", а затем кнопку "Предохранитель" (рисунок3A, синий прямоугольник), чтобы открыть диалоговое окно под названием "Deconvolve/Fuse diSPIM Raw Data Volumes" (Рисунок 3B). Установите параметры, как показано на рисунке 3B. Эти параметры кратко рассматриваются в следующих подшагах.
- Установите регистрацию ключей на Первом канале (488 нм лазер) или 2 (561 нм лазер). Выберите канал с более плотным или более вездесущим сигналом. Для линейных экспериментов всегда выбирайте канал, используемый для изображения вездесущей ядерной флуоресценции гистона.
- Установите объемы ввода относительной ориентации на 1 или -1. Правильный индекс ориентации зависит от размещения камеры конкретного diSPIM(рисунок 4).
ПРИМЕЧАНИЕ ПРИМЕЧАНИЕ- Если неопределенным, проверить каждый вариант, дублируя один тайм-пойнт из обоих SPIM A и B diSPIM монитор окно, следующие шаги 9.1-9.12 и произвольно выбирая входной объемы относительной ориентации для тестирования. Неправильные ориентации дадут размытые изображения с артефактами, в то время как правильные ориентации дадут четкие изображения. Значение входная ввода относительной ориентации, которое дает четкое изображение, может быть повторно использовано для всех будущих данных из данного инструмента diSPIM. - Выберите, следует ли ориентироваться на объем сверкаемых данных так же, как и объем ввода A или B (на основе предпочтений пользователя).
- Выберите "Свежая регистрация для каждого тома". Эта пара контролирует, как SpimFusion вычисляет регистрационные матрицы для каждой пары громкости в каждой временной точке. Опция "Свежий" позволяет алгоритму оптимизировать регистрацию адаптивно в каждый момент времени.
- Установите число итераций деконволюции до 10. Это число, как правило, надежно дает желаемое высокое разрешение в экономичном с точки зрения времени порядке.
- Дополнительно еле- Если автолиния пожелана (настоятельно рекомендуется), проверьте автозапуск StarryNite линии слитых томов. Эта опция автоматически запустит StarryNite для сегментации и отслеживания клеток в изображениях объемов, производимых SpimFusion.
- Дополнительно еле- Для получения максимальной точности в автоматизированном lineaging, лучше всего перепозиционировать изотропическисброшенные объемы эмбриона в каноническую ориентацию "ADL" (Nterior x-west), Dorsal'y-north, Left'z-near). Выберите ориентацию вывода объема Define в опции предварительного просмотра, чтобы указать этот выбор. CytoSHOW ответит на обработку начальной пары изотропически сброшенных томов, что позволит пользователю внимательно наблюдать и указывать вращения, необходимые для достижения регистрации ADL.
- Нажмите «Да», как только будут выбраны все параметры.
- Укажите каталог вывода, в котором можно сохранить обработанные файлы. Нажмите OK.
- Необязательное определение ориентации вывода объема в опции предварительного просмотра было выбрано, установите бар прокрутки t (рисунок3A,зеленая стрелка, левая панель) в окне SPIM-A до ранней временной точки, на которой ячейки ABa и ABp достигли метафазы. Установите панель прокрутки t в окне SPIM-B на более позднюю стадию запятой разработки. Это поможет в определении ориентации ADL.
- Дополнительное время, когда они готовы, нажмите OK. Если вариант предварительного просмотра в 9.5.7 выше был выбран, только два тома предварительного просмотра будут изотропически слиты для временных точек, указанных t-ползунами из окон изображений SPIM-A и SPIM-B. Эти две точки предварительного просмотра могут быть использованы для определения точной переориентации объемов вывода эмбриона на ориентацию ADL, как показано ниже.
- Найдите недавно отображаемый 3DProjY-Decon-Fuse.... окно. Переместите панель прокрутки t в точку 2 времени этого окна предварительного просмотра. Переместите ползунок до тех пор, пока не будет показано представление прямо вниз по длинной оси эмбриона.
- Переместите t прокрутки бар обратно в точку времени 1 из 3DProjY-Decon-Fuse.... окно. Выберите инструмент выбора линии и нарисуйте выделение линии из ячейки EMS (вентральное самое круглое ядро) через плоскость метафазных пластин AB-клетки.
- Нажмите на кнопку оранжевый diSPIM Предварительный просмотр на 3DProjY-Decon-Fuse.... окно. Тонкие корректировки ориентации предварительноого изображения громкости будут сохранены для использования при обработке полного набора данных.
- Дополнительно ежемое диалог о объемах данных Deconvolve/Fuse diSPIM сырье появится, так же, как и в шаге 9.5 выше. Нажмите "Да", не выбрав ориентацию на выход объема Define volume в опции предварительного просмотра. Укажите систему вывода для полного выполнения обработки данных.
- Установите t прокрутки баров (рисунок3A, зеленая стрелка, левая панель) из diSPIM монитор окна к точке начала (SPIMA) и окончание тайм-пойнт (SPIMB) полный промежуток изображений для обработки. Затем нажмите Ok.
- По мере прогрессирования SpimFusion CytoSHOW открывает и обновляет многомерное окно, показывающее нарезанный 4D изотропный объем для каждого эмбриона, а также два окна с вращающимися-4D максимальная интенсивность проекций изотропного объема. В течение этого времени не нарушайте и не закрывайте окно CytoSHOW до тех пор, пока не будет завершено изотропное слияние и отслеживание линии.
- Дополнительно ею, обратите внимание, что после появления экрана StarryNite, а затем исчезнувшего, полный конвейер обработки данных был завершен. Это окно не должно быть закрыто во время обработки или StarryNite будет прервано.
10. Открытие StarryNite линии след серии в AceTree (по желанию)
- Откройте настраиваемую версию "AceTree-16BitCompat.jar" при условии.
- Выберите меню файла (Open configurefile). Найдите свой каталог вывода, ранее указанный в CytoSHOW. Откройте подфюм Decon-Fuse... Выберите aaa'edited.xml и Открыть.
- Используйте меню AceTree Отек и эрит инструменты, чтобы открыть Edit Track и настроить или удалить окна клеток.
- Нажмите на полузатенный круг Рисунок 5B, красный квадрат, чтобы настроить красный и зеленый интенсивности.
- Продолжить с визуализацией и редактированием линии, как ранее описано5,6,8 (рукописи также включены в наш пакет загрузки).
Результаты
Сначала мы подтвердили жизнеспособность эмбрионов, изображенных с помощью параметров протокола для приобретения diSPIM (разделы 1-6). Десять эмбрионов были одновременно изображены при 20 градусах Цельсия, одном томе/эмбрионе/минуте, от 2-клеточной стадии до 2-кратной стадии (7,5 часов, 451 том/эмбрион). Для мониторинга деления клеток во всем эмбриогенезе, мы использовали штамм BV514, который повсеместно выражает mCherry(-Histone reporter constructs from the integrated transgene array ujIs11324. На рисунке 6 показана хронология первой половины эмбрионального развития одного из изображенных эмбрионов. Каждое изображение представляет собой проекцию максимальной интенсивности с одним видом (произведенную шагами 7-8) изображенного эмбриона. Мы обнаружили, что оптимизированные протоколы не вызывают каких-либо обнаруженных фототоксичности эмбрионов, как оценивается по срокам деления клеток (не показано), время вылупления, и сроки, связанные с развитием вехи (Рисунок 6 и ссылки1 , 25 , 26).
Затем мы применили протокол для анализа динамики роста одиночных нейронов в развивающихся эмбрионах. Мы изображения DCR7692 (olaex4655), трансгенный штамм нематод, который выражает GFP от нейропептида flp-19 промоутер в подмножестве неопознанных клеток (DACR2819, Pflp-19 (3.6kb) (3.6kb)(Syn21/GFP)(CAAX/:p10 3'UTR) . Следуя этапам протокола, изложенному здесь, мы определили, что неопознанные клетки соответствуют моторным нейронам RMDDL и RMDDR, к клетке выделительного канала и двум мышечным клеткам(рисунок 7). Затем мы изучили и количественно динамику роста RMDDL и RMDDR нейронов. Мы заметили, что RMDDL и RMDDR нейроны косо формы уже в 360 минут после оплодотворения, с большей клеточной оси, представляющих последующую ось для невритовых наростов(Рисунок 7 и фильм S1). Используя плагин «простой нейрит» в FIJI и применяя его к 3D-реконструкциям изотропически сросшиеся тома, мы затем количественно оценили стереотипный рост RMDDL и rmDDR neurites для шести эмбрионов. Мы определили, что динамика роста была стереотипной для RMDDL и RMDDR через эмбрионы (здесь называется RMDDs). С 385-410 минут после оплодотворения, RMDDs нейритов продлен 6,0 '0,5 м (средний SEM; n - 12 невритов) передних тел клеток(рисунок 7B,C,I). От 415-445 минут после оплодотворения, оба неврита распространяются дорсально в и вокруг предполагаемого нервного кольца (звездочка на рисунке 7D). В среднем, каждый rmDD нейрит продлен 11,0 и 0,6 мкм (средний SEM; n 12 невритов) из клеточного тела перед синхронно встречающ свой контралатеральный двойник на вершине кольца(рисунок 7I). Важно отметить, что наши репрезентативные результаты показывают, что мы можем изучить, сравнить и количественно нейронных особенностей развития для отдельных идентифицируемых клеток с помощью нашего интегрированного протокола(Рисунок 7 и Рисунок 8).
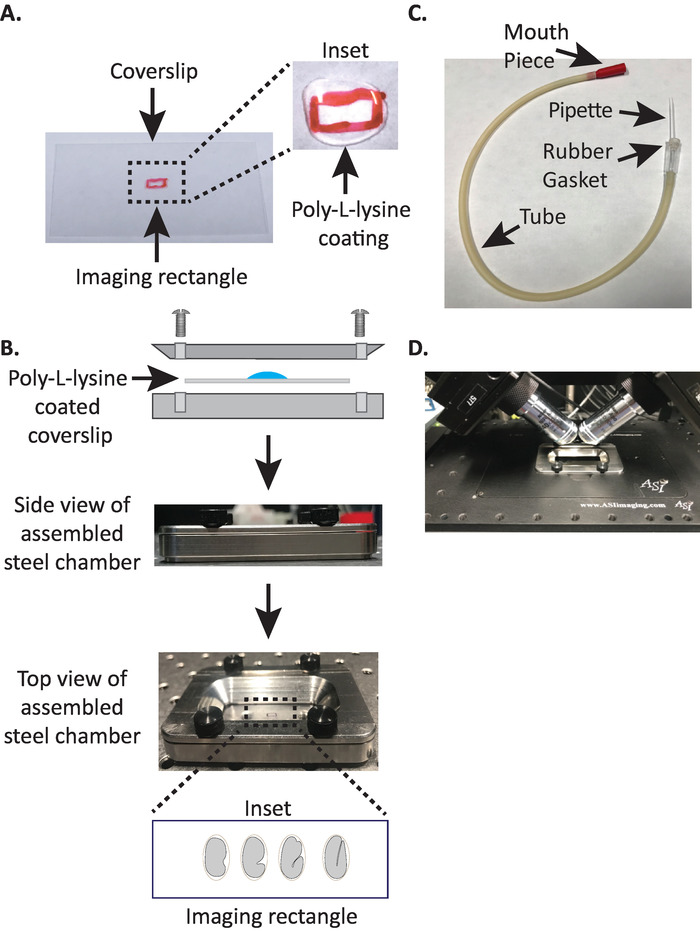
Рисунок 1. установка установки установки образца diSPIM. (A) Подготовка облика с поли-L-лизин. В входе, 10 Л из лизина Поли-Л был использован для покрытия крышкой в течение 5 минут. Поли-L-лизин позволяет эмбриона яичной скорлупы твердо придерживаться крышки в прямоугольнике. (B) Схема стальной камеры изображения и собранной камеры. В вставке репрезентативные эмбрионы ориентируются с передней задней оси перпендикулярно длинной оси на крышке. (C) Собранная трубка аспиратора с микрокапиллярным пипеткой. (D) Стальная камера визуализации установлена в выборке держателя под diSPIM 40x целей. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
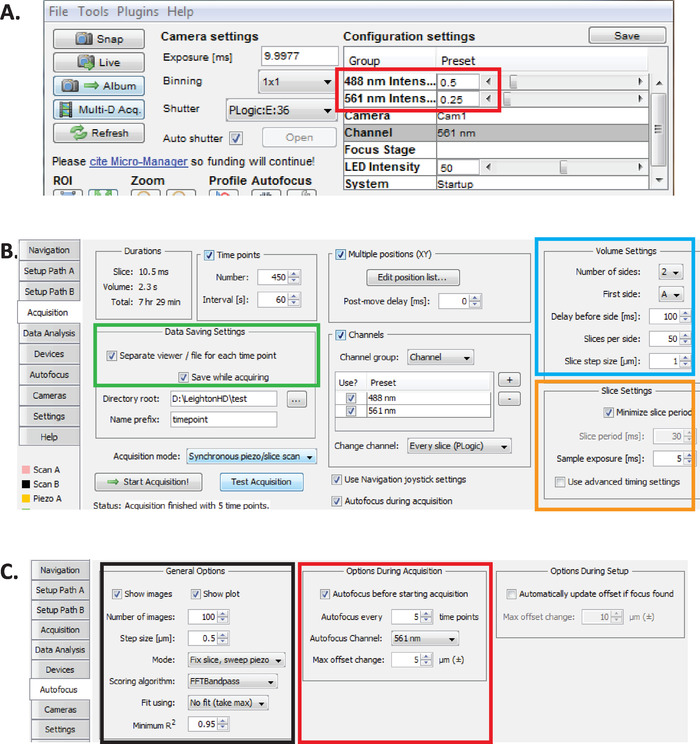
Рисунок 2. Долгосрочная настройка визуализации diSPIM в Micro-Manager. (A) Рекомендуемые параметры лазерной мощности diSPIM (красный прямоугольник), оптимизированные для длительной визуализации при одновременном снижении фототоксичности (по более высокой скорости вылупления эмбрионов C. elegans). Установите лазер 561 нм до 79 МВт (0,25) и 488 нм лазера до 179 МВт (0,5). Обратите внимание, что точная калибровка параметров программного обеспечения для лазерной мощности варьируется между diSPIM установок. Рекомендуется, чтобы пользователи измеряли и калибровали мощность лазера, чтобы достичь 79 ЗВт (561 нм) и 179 МВт (488 нм) лазерной мощности. (B) параметры diSPIM для сохранения данных (зеленый прямоугольник), настройки громкости (синий квадрат) и настройки срезов (оранжевый квадрат). (C) параметры автофокуса diSPIM для долгосрочной визуализации эмбриогенеза C. elegans (см. шаги 6.1-6.6). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.

Рисунок 3. Визуализация изображения и настройка обработки данных с помощью CytoSHOW. (A) Сырье diSPIM изображения, открытые CytoSHOW. CytoSHOW может открывать изображения, снятые обеими траекториями камеры (SPIM A и B). Эти необработанные изображения открываются в многомерных окнах под названием монитор diSPIM. В мониторе diSPIMсоздается «шаблон bowtie» для выбора передней, задней, спинной и брюшной краев эмбриона (см. шаг 9.1). Лук-связь выбор указывает на ориентацию эмбриона для деконволуации и StarryNite при содействии lineaging трассировки. (B) Оптимизированные параметры, используемые для создания изотропных изображений. В Деконволве при приобретении окна устанавливаем параметры, указанные в шагах 9.5.1-9.5.8. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
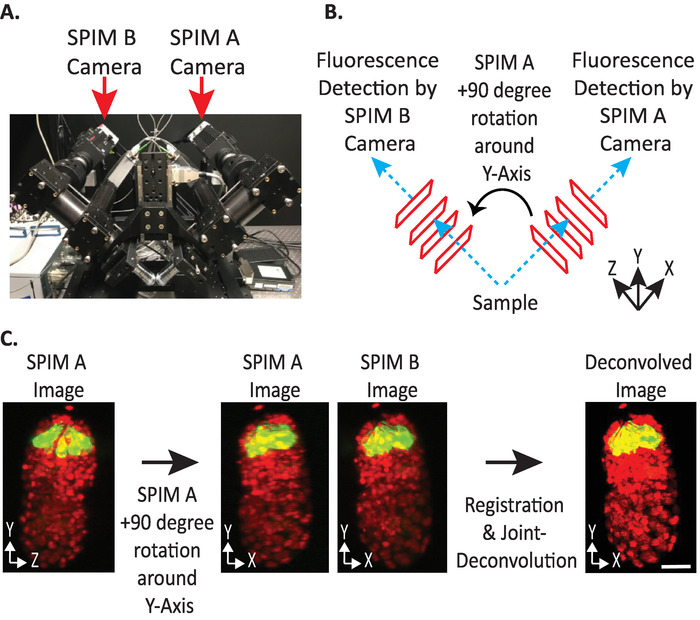
Рисунок 4. конфигурация камеры diSPIM. (A) Фотография diSPIM размещения камеры и ориентаций. (B) Изображение 90-градусных вращений SPIM A в соответствии с собранными изображениями SPIM B. (C) Объемы ввода по отношению к индексу ориентации No1 на основе конфигурации камеры нашего diSPIM (см. шаг 9.5.2). Мы поворачиваем изображение SPIM A (ы) на 90 градусов вокруг Y-Axis перед регистрацией в соответствии с изображением SPIM B (ы). Шкала баров - 10 мкм. Изображения являются репрезентативными одноэтажными, максимально интенсивными проекциями и деконволюционными изображениями 1,5-кратного эмбриона с маркированными ядрами (561 нм, красным) и нейронами (488-нм, зеленый). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
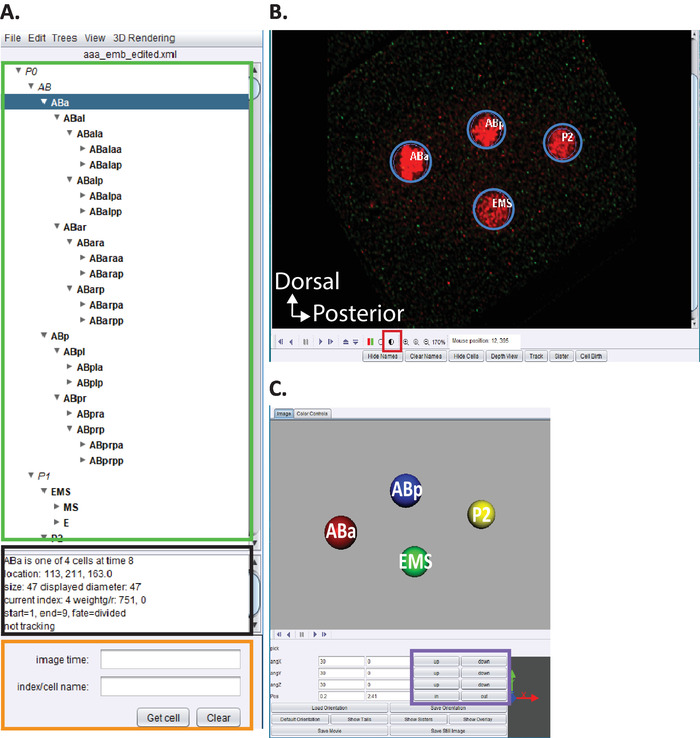
Рисунок 5. Курирование и редактирование C. elegans эмбриональной линии в AceTree. (A) Мы используем AceTree для отсеивания следов линии StarryNite (см. ссылки5,6,8;рукописи также включены в наш пакет загрузки). AceTree будет отображать C. elegans систематические имена для каждого ядра (зеленый прямоугольник) по завершении шагов 10.1-10.2. Это окно(A ) предоставляет информацию (черный прямоугольник) о каждой ячейке в следе линии (ABa, выделенный синим цветом), которые помогают пользователям при отслеживании и редактировании следов линии. Рекомендуется, чтобы пользователи проверяли и сравнивали линейные клетки и их положение с эмбриональной линией клеток C. elegans, о которых ранее сообщал Sulston et al.1 Кроме того, если пользователи заинтересованы в поиске конкретных клеток в серии данных deconvolved (см. ниже, B) введите C. elegans систематическое имя в панели поиска (оранжевый прямоугольник). (B) Серия деконволвированных данных пользователя также открывается автоматически после завершения шагов 10.1-10.2. Здесь показано изотропное слитое изображение четырехклеточного эмбриона с ядрами, помеченными красным цветом. Во время отслеживания ядер, пользователи должны изменить интенсивность изображения (красный квадрат) и перемещаться во времени и z с помощью клавиш и стрелки на клавиатуре (время-лево/право, z-up/down). (C) 3D мультфильм временной точки в (B) с определенными функциональными возможностями (фиолетовый прямоугольник), что позволяет вращаться 3D-визуализации. Для обзора AceTree и его 3D функциональности,см ссылки 5,6,8. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
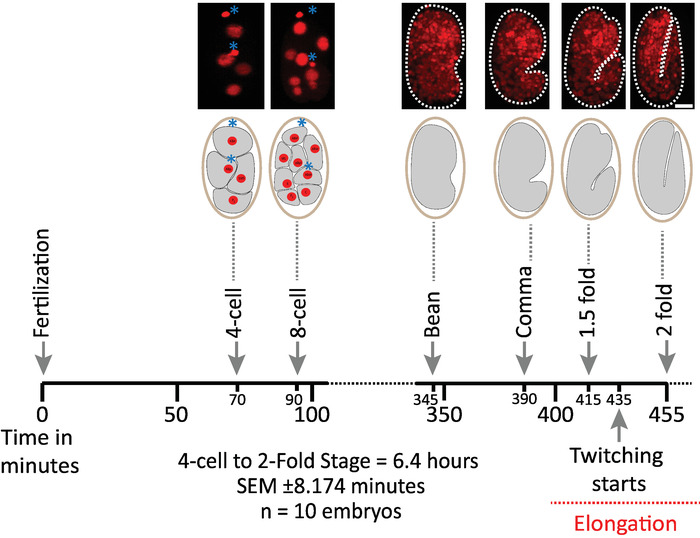
Рисунок 6. Приуроченная динамика развития эмбрионов C. elegans на diSPIM. Верхняя панель, diSPIM изображения, показывающие первую половину эмбрионального развития для одного из изображенных эмбрионов (Strain BV514 ujIs11324). Эмбрионы были изображены непрерывно, каждую минуту в течение 7,5 часов (при 20 градусах Цельсия). Первые два изображения верхней панели представляют 4- и 8-клеточные эмбрионы с ядрами (красными) и положениями полярных тел (плотные красные сферы, отмеченные синими звездочками). Каждое изображение представляет собой одновидовую максимально интенсивную проекцию изображенного эмбриона. Шкала баров 10 мкм. Хронология (горизонтальная планка) представляет собой минутное послеоплодное (m.p.f.) развития эмбрионов C. elegans. Мы подтвердили, что параметры нашего протокола для приобретения diSPIM не вызывали какой-либо обнаруживаемой фототоксичности для изображенных эмбрионов, оцениваемых по жизнеспособности, срокам деления клеток, срокам вылупления и срокам разработки вех (см. справки 1 , 25 , 26). Мы отмечаем, что время развития вех было воспроизводимо по эмбрионам с нашими параметрами изображения (SEM - 8,174 минуты для 6,4-часовой сессии визуализации; n No 10). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
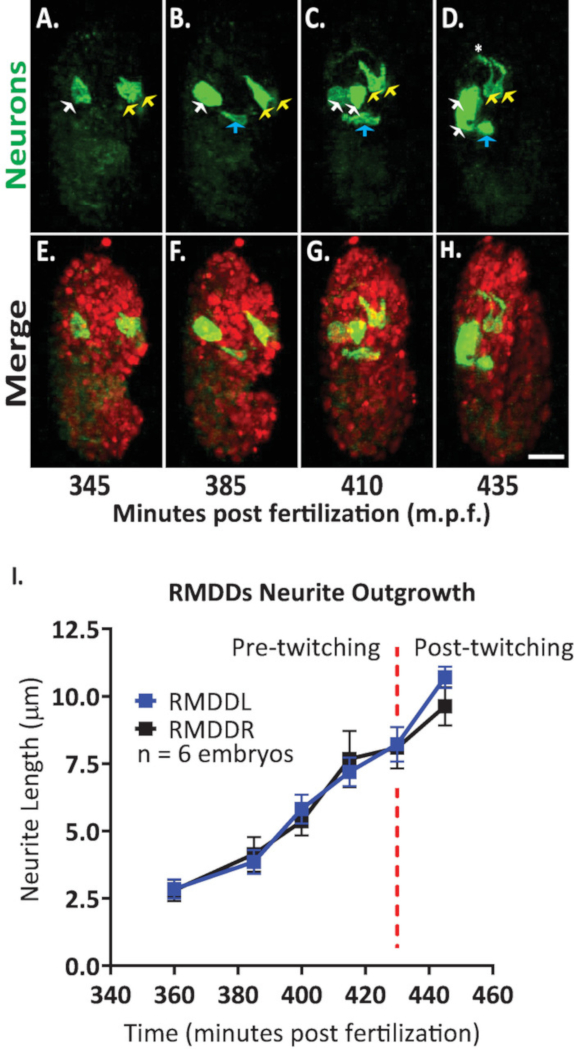
Рисунок 7. Клеточная идентификация и одноклеточная характеристика динамики неврита в развивающихся эмбрионах C. elegans. Двухцветные изображения штамма, сделанного путем пересечения BV514 ujIs11324 (для lineaging) и DCR7692(olaex4655), трансгенный штамм нематод, который выражает GFP от нейропептид флп-19 промоутер в подмножество неопознанных клеток. (A-H) Следуя описанным здесь этапам протокола, мы определили, что неопознанные клетки соответствуют моторным нейронам RMDDL и RMDDR (желтые стрелки), клетке выделительного канала (синие стрелки) и двум мышечным клеткам (белым стрелкам). (I) Количественная оценка динамики роста нейронов RMDDL и RMDDR с помощью плагина FIJI "простой неврит трассировки" и применения его к 3D-реконструкциям изотрополоилобъемных томов. Обратите внимание, как RMDDL и RMDDR показывают стереотипную динамику роста, каждая из которых синхронно расширяется на общую длину 11,0 х 0,6 м (средняя - SEM; n 12 невритов) и встреча на вершине дорасталора нерва (см. также Фильм S1). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
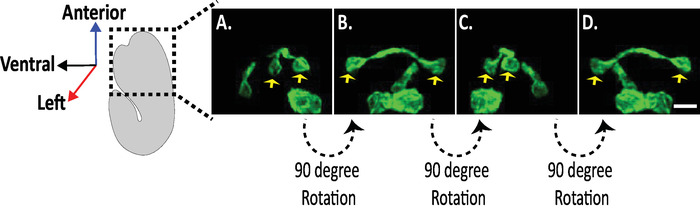
Рисунок 8. Изучение изотропных изображений нейронных морфологий в эмбрионах C. elegans. Изотропная визуализация нейронов AVHL и AVHR (желтые стрелки). Используя diSPIM, нейронные морфологии могут быть захвачены уступая четырехмерных (4D) изображений с изотропным пространственным разрешением около 330 нм. DiSPIM позволяет пользователям практически вращать объемы изображений с одинаковым разрешением во всех направлениях. Изображения в A-D являются максимально интенсивными проекциями одного и того же изотропно слитого объема изображения diSPIM от различных вращений вокруг длинной оси эмбриона. Шкала баров 5 мкм. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Дополнительный фильм S1. C. elegans эмбрион развивается от 280 до 434 минут после оплодотворения. Изотропный фильм штамма DCR7692(olaex4655) выражая ujIs113 повсеместно с DACR2819 редко маркировки RMDD нейритов (Рисунок7A-D, желтые стрелки). DACR2819 также маркирует две мышечные клетки(рисунок 7A-D,белые стрелки) и выделяемые клетки канала (рисунок7A-D,синяя стрелка) во время эмбрионального развития (рисунок7A-D). Шкала баров 10 мкм. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
C. elegans выделяется как единственный организм с окончательными позициями и связь каждого взрослого нейрона известны27. Однако динамика развития, приводящая к организации рабочих схем и сетей, вкоторыехавших коннектом C. elegans, остается неизвестной. Основываясь на возможностях, возникающих в результате достижений в области световой микроскопии, мы можем теперь фиксировать и анализировать клеточные позиции, морфогенез и нейрогенез на протяжении всего эмбрионального развития C. elegans.
Процедура, которую мы описали и которую мы регулярно используем в лаборатории, дает 4D-изотропные изображения помеченных нейронов и ядер для клеточной линии в эмбрионах C. elegans. Что еще более важно, мы оптимизировали долгосрочные условия визуализации с diSPIM и в сочетании с полуавтоматическими линейными возможностями с изображениями с высоким разрешением для повышения скорости и точности анализа эмбриогенеза C. elegans. Этот интегрированный протокол позволит пользователям визуализировать и идентифицировать клетки и количественно трехмерных объектов, таких как нейритная миграция и фасцикуляция аксона через начало раннего подергивания. Эта процедура может быть легко адаптирована в любой объект с системой ASI diSPIM, и мы рекомендуем эту систему специально для этого протокола. Другие формулировки SPIM, предлагаемые коммерчески, могут отличаться от конфигурации АСИ в образцовых камерах и оптических свойствах. Тем не менее, данные, экспортируемые с других платформ, также могут быть помещены через наш конвейер данных. Таким образом, оценка их стоимости в lineaging, требовательный тест качества изображения и стабильности приборов, является возможным. Несмотря на то, что мы активно используем diSPIM для регулярного изображения других образцов (таких как дрозофилы и эмбрионы зебры), описанный и всеобъемлющий линейный анализ эмбрионов по-прежнему в настоящее время ограничивается нематодными видами. Для больших или толстых образцов мы предпочитаем использовать подходы к сканированию стадии, которые сканируют образцы через стационарный световой лист. Kumar et al. ранее продемонстрировали это улучшенное diSPIM секционирование для получения высококачественных изображений из толстых образцов без дополнительных изменений в diSPIM10.
Критические шаги в протоколе включают монтаж эмбрионов C. elegans на поли-L-лизин покрытием coverslip, сбор данных, и обработки данных. Сбор урожая и монтаж C. elegans эмбрионов на стеклянном крышке может быть сложной задачей для неопытных пользователей, но здесь мы предоставляем подробный протокол ключевых шагов для облегчения обучения. Если долгосрочная визуализация желательна, мы получаем наилучшие результаты по сбору четырехклеточных или более ранних эмбрионов у 8-10 молодых людей28. Обратите внимание, что пожилые люди менее желательны для сбора эмбрионов ранней стадии, потому что они, как правило, содержат старые эмбрионы в матке и неоплодотворенные яйца. Что касается монтажа эмбрионов, такие проблемы, как блокировка в собранном аспираторе (ротпипетная пипетка) или слишком большое отверстие в микрокапиллярной пипетке, могут предотвратить правильное монтаж и ориентацию эмбрионов. Чтобы подготовиться к оптимальной визуализации, мы проводим предварительное тестирование на ранних и поздних предварительно подергивания эмбрионов, чтобы проверить производительность световых листов, камер, целей и автофокусировки. Мы получаем лучшие результаты, когда все эти операции проверены и дают высококачественные изображения во время нашего предварительного тестирования приобретения. Это особенно актуально для создания изображений с изотропным пространственным разрешением, для которых необработанные изображения, полученные из обоих представлений (целей), должны быть высококачественными. После приобретения объемы, приобретенные для каждого представления, обрабатываются для получения изотропных изображений. Важно использовать соответствующую графическую обработку (GPU) карты, как описано в этом протоколе (см. ниже). Это улучшает скорость обработки, с которой генерируются изотропически сброшенные изображения, сокращая время анализа данных. Кроме того, крайне важно, чтобы пользователи запускали последнюю версию CytoSHOW и используют параметры, предоставляемые с нашей загрузки расслоение для StarryNite автоматической линии. Если пользователи заинтересованы в использовании автоматической линии для других образцов (например, зебрафиш, дрозофила и т.д.), то потребуетсядополнительная оптимизация параметров, используемых в StarryNite (см. справки 3,4).
Хотя наш интегрированный протокол обеспечивает изображения и линейные результаты в предварительно подергиваем эмбрионе, пользователи должны знать, что автоматизированное lineaging в послепотливом эмбрионе в настоящее время не представляется возможным? после подергивания эмбриона, слишком быстро, чтобы позволить линии отслеживания. Тем не менее, diSPIM действительно продемонстрировали многообещающую способность захватить нейроразвития событий и отслеживать некоторые позиции клеток в после подергивания этапов эмбриогенеза23,29. Если пользователи заинтересованы в изучении после подергивания эмбриона, diSPIM обеспечивает скорость получения объемных снимков и отслеживать тонкие события нейроразвития, такие как неврит овый рост, в быстро движущихся эмбрионов.
Этот протокол будет основополагающим для завершения клеток за ячейкой Атлас WormGUIDES30, так как он обеспечит комплексный подход с высоким разрешением изотропных изображений для выявления и захвата 3D морфологии помеченных нейронов во время первые 430 минут эмбриогенеза. В его нынешнем виде прототип атласа WormGUIDES обеспечивает ядерное положение клеток в развивающемся эмбрионе и направлен на захват динамики развития подмножества эмбриональных нейронов. Этот протокол будет ключом к интеграции дополнительных развивающихся нейронов в атлас WormGUIDES30.
Наш интегрированный протокол также упростит изучение новых профилей экспрессии генов в эмбрионе C. elegans. В трансгенных C. elegans, многие клетки конкретных промоутеров пространственно и временно контролировать трансгенное выражение. В то время как модели экспрессии большинства генов были широко охарактеризованы у взрослого животного31,32,33,34, почти все еще не охарактеризованы в развивающихся (особенно поздней стадии) эмбриона. C. elegans promoterome был полезным ресурсом для сообщества червей для привода клеточного экспрессии трансгенов, а также определить, является ли генная функция клеточной или неавтономной. Захват изотропных моделей высокого разрешения и динамического выражения генов, а также точное определение выражающих сярлица клеток с помощью линии будут полезны для многих в научном сообществе.
Эмэмегенез включает в себя два взаимосвязанных основных процесса, клеточной дифференциации и морфогенеза тканей. Многое известно о механизмах и молекулах, которые определяют различные типы клеток во время развития C. elegans. Тем не менее, мало что известно о механизмах, важных для миграции клеток, клеточной адгезии и формы клеток в эмбрионе C. elegans. С C. elegans инвариантной линии клеток известны, наш протокол позволяет нам легко различить каталогизировал 3D-микроанатомии эмбриона во время морфогенеза на новых уровнях детализации, например, аксон фасцикуляции, синаптогенеза, и нейронной активности. Ardiel et al. ранее продемонстрировали силу diSPIM для улавливания переходных кальция на уровне одного нейрона в эмбрионах C. elegans 23. Многие другие аспекты физиологии развития созрели для исследования этими методами.
Наконец, этот протокол в значительной степени автоматизирован и систематически сокращает время, необходимое для генерации изображений деконволуции и выполнения клеточной линии через StarryNite и Acetree. Стратегии программного обеспечения, используемые в этом протоколе, могут быть применены ко многим вопросам биологии, далеким от очень специфических областей, в которых мы их продемонстрировали здесь.
Подробная информация о совместимости программного обеспечения и доступе к загрузке
Информация о Micro-Manager и плагинах для визуализации diSPIM доступна в http://dispim.org/software/micro-manager и https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
В настоящее время для конвейера обработки данных требуется операционная система Windows. Мы упаковали единый архивный файл для упрощения установки всех необходимых программ обработки данных и файлов поддержки. Он доступен для скачивания в http://dispimlineage.wormguides.org.
CytoSHOW (http://run.cytoshow.org/) основан на широко используемой платформе анализа изображений с открытым исходным кодом ImageJ (v1). Java должна быть установлена и обновлена на компьютере для использования CytoSHOW, а обновления CytoSHOW автоматически развертываются через Java Web Start. Многие функции CytoSHOW на основе ImageJ описаны и проиллюстрированы на https://imagej.nih.gov/ij/docs/examples/index.html. CytoSHOW был настроен для отображения многомерных необработанных данных из ASI diSPIM, а также другого программного обеспечения для визуализации, которое создает выход TIFF. В принципе, другие многовидовые системы визуализации SPIM могут быть подкреплены незначительными модификациями CytoSHOW, позволяющими осуществлять этот протокол на различных системах микроскопов.
SpimFusion был написан в CUDA/C е с помощью Visual Studio 2013 с помощью инструментария CUDA v7.5. Запуск SpimFusion требует специального компьютерного оборудования, NVIDIA графический процессор (GPU) карты с CUDA вычислительной возможности 1.0 или выше и минимум 2 ГБ памяти видеокарты. На момент публикации нашего протокола SpimFusion не публикуется (Мин Го и Шрофф), но доступен в вышеупомянутом архиве пакета программного обеспечения.
Специально построенная командная версия StarryNite требует установки свободно доступного компилятеля MATLAB Runtime, но не требует лицензии на коммерческое программное обеспечение MATLAB. MatLAB Compiler Runtime включен в упомянутый выше архив пакета программного обеспечения. Код для StarryNite, используемый в этом протоколе, по существу не изменился по сравнению с тем, который используется для конфокальных изображений6. Тем не менее, несколько оперативных вопросов в создании входных изображений для обработки StarryNite и обработки результатов StarryNite были рассмотрены здесь методами в CytoSHOW, которые позволяют непрерывной обработки данных трубопровода для слитого изотропного diSPIM объемов. Эти изменения автоматизированы кодом CytoSHOW, который обрабатывает эти пред- и пост-обработки шагов. CytoSHOW также редотирует предварительно оптимизированный шаблон StarryNite, установленный для автоматической настройки алгоритма сегментации на интенсивность флуоресценции ядер в изображении данных. Уникальные параметры, используемые StarryNite на каждом наборе данных diSPIM, затем сохраняются в файле вместе с выходным изображением и линейными данными.
Пользовательская версия AceTree, которая работает с 16-битными изображениями и поддерживает совместимость с визуализацией Java3D, лучше всего подходит для этого протокола. Он также включен в архив пакета программного обеспечения, упомянутый выше.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы благодарим Джона Мюррея за интегрированный штамм, ujIs113, для генерации линейных штамм BV514; Брэндон Харви (NIBIB) за помощь в тестировании протокола; Джон Дэниелс и Гэри Рондо (Прикладное научное оборудование) за помощь с Micro-Manager и diSPIM инструмент; и Эндрю йорк и Хэнк Иден за их критические отзывы о системе diSPIM. Мы также благодарим Исследовательский центр для меньшинств учреждений программы и Института нейробиологии Хосе дель Кастильо (Университет Пуэрто-Рико) для обеспечения встречи и мозгового штурма платформы. Большая часть этой работы была проведена в Морской биологической лаборатории в Вудс-Хоул в рамках программы Уитмена. Эта работа была поддержана интрамуральных научно-исследовательских программ НИЗ Национальный институт биомедицинской визуализации и биоинженерии и НИЗ грант No. U01-HD075602 и No. R24-OD016474. Марк У. Мойл был поддержан F32-NS098616 и Лейтон Х. Дункан был поддержан разнообразие дополнение к R24-OD016474.
Материалы
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
Ссылки
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. . WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- . WormBase Available from: https://www.wormbase.org (2019)
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены