Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Высокой пропускной способ в Ситу Метод оценки гепатоцитов ядерной плоиди у мышей
В этой статье
Резюме
Мы представляем надежный, экономичный и гибкий метод измерения изменений в количестве гепатоцитов и ядерной флоиди в фиксированных/криоконсервированных образцах тканей, которые не требуют цитометрии потока. Наш подход обеспечивает мощную выборку всей подписи цитологии печени идеально подходит для отслеживания прогрессирования травмы печени и болезни.
Аннотация
Когда печень повреждена, число гепатоцитов уменьшается, в то время как размер клеток, ядерный размер и ploidy увеличиваются. Расширение непаренхимальных клеток, таких как холангиоциты, миофибробласты, прародители и воспалительные клетки, также указывает на хроническое повреждение печени, ремоделирование тканей и прогрессирование болезни. В этом протоколе мы описываем простой высокопроизводительный подход к расчету изменений в клеточеском составе печени, связанных с травмами, хроническими заболеваниями и раком. Мы показываем, как информация, извлеченная из двухмерных (2D) разделов ткани может быть использована для количественной оценки и калибровки гепатоцитов ядерной ploidy в образце и позволяют пользователю найти конкретные подмножества ploidy в печени на месте. Наш метод требует доступа к фиксированной/ замороженной печени материал, основные иммуноцитохимии реагентов и любой стандартной высокой содержаниеизображения платформы. Он служит мощной альтернативой стандартным методам цитометрии потока, которые требуют нарушения свежесобранных тканей, потери пространственной информации и потенциальной предвзятости дезагрегирования.
Введение
Гепатоциты в печени млекопитающих могут пройти застопорился цитокинез для производства двухядерных клеток, и ДНК эндорепликации для производства полиплоидных ядер, содержащих до 16N содержание ДНК. Общее количество клеточных и ядерных ploidy увеличивается во время послеродового развития, старения и в ответ на различные клеточные стрессы1. Процесс полиплоидизации динамичный иобратимый 2,хотя его точная биологическая функция остаетсянеясной 3. Повышенная ploidy связана с уменьшенной пролиферативной емкостью4,генетическим разнообразием2,адаптацией к хронической травме5 и защитой от рака6. Гепатоцит ploidy изменения происходят в результате изменения циркадного ритма7, и отляги8. Наиболее примечательно, ploidy профиль печени изменяется травмы и болезни9, и убедительные доказательства свидетельствуют о том, что конкретные изменения ploidy, такие как увеличение 8N ядер или потери 2N гепатоцитов, обеспечивают полезные подписи для отслеживания безалкогольных жировых заболеваний печени (NAFLD) прогрессии3,10, или дифференциального воздействия вирусных инфекций11.
В общих чертах, повреждение печени и регенерации связаны с увеличением размера клеток гепатоцитов и ядерной области12, вместе с сокращением общего числа гепатоцитов, особенно те, с содержанием ДНК 2N10,11. Паранхимальная травма печени также часто сопровождается расширением непаранхимальных клеток (НПК), включая стромальные миофибробласты, воспалительные клетки и бипотентные клетки-прародители печени. Высокопроизводительные методы, которые обеспечивают количественный цитологический профиль parenchymal номер клетки и ядерной ploidy, в то время как также учета изменений в NPCs, поэтому имеют значительный потенциал в качестве научных исследований и клинических инструментов для отслеживания реакции печени во время травмы и болезни. Принуждение последние на месте анализ ploidy спектра в человеческих образцах гепатоцеллюлярной карциномы также показывают, что ядерная плоида резко увеличивается в опухолях и специально усиливается в более агрессивных подтипов опухоли с уменьшенной дифференциацией и потерей TP5313. Таким образом, существует высокая вероятность того, что методологические достижения в количественной оценке ядерной плоиди помогут в будущем прогностическом профилировании рака печени.
В этом протоколе описана гибкая методика высокой пропускной связи для сравнительного анализа секций тканей печени мыши, которая обеспечивает детальное цитометрическое профилирование числа гепатоцитов, ответ NPC и внутренне откалиброванный метод оценки ядерной флоидии(рисунок 1). Гепатоциты отличаются от НПК иммуномаркировкой гепатоцитов 4 альфа (HNF4) до характеристики ядерного размера и ядерной морфометрии. "Минимальное содержание ДНК" оценивается для всех круговых ядерных масок путем интеграции средней интенсивности Hoechst 33342 (прокси для плотности ДНК) с интерполированным трехмерным (3D) ядерным объемом. Гепатоцитминимальное содержание ДНК затем откалибровано с помощью NPC для создания ядерного профиля ploidy.
Приобретение изображений, ядерная сегментация и анализ изображений выполняются с использованием изображений с высоким содержанием, что позволяет обсуливать большие участки двумерных (2D) секций печени, содержащих десятки тысяч клеток. Предусмотрена специально написанная программа для автоматической пост-обработки данных анализа изображений высокого содержания для получения профиля флоиди для всех круговых ядер гепатоцитов. Это выполняется с помощью бесплатного скачать программное обеспечение для расчета ядерной ploidy на основе стерилологического анализа изображений (SIA)10,11,14,15. Методология SIA была ранее подтверждена цитометрией потока как точный, хотя и трудоемкий, метод для оценки гепатоцитов ядерной флоиди и в печени14, предполагая круговую ядерную морфологию и монотоническую связь между ядерным размером и содержанием ДНК. В этом протоколе оба ядерных параметра измеряются путем оценки ядерной морфометрии и маркировки Hoechst 33342. Расчет «минимального содержания ДНК» для каждой ядерной маски сопровождается калибровкой гепатоцитной ядерной флоиды с использованием NPC, которые имеют известное содержание ДНК 2х4N и поэтому служат полезным внутренним контролем.
По сравнению с обычными методами цитометрии потока16 описанный подход позволяет оценивать ядерную флуиду гепатоцитов на месте и не требует доступа к свежим тканям или методам дезагрегирования, которые могут предусмотреть результаты и быть трудно стандартизированными. Как и во всех подходах, основанных на SIA, подклассы ядерных ploidy sgt;2N недопредставлены 2D-выборкой из-за секции больших ядер за пределами экваториальной плоскости. Профиль флоиди по всей ткани также описывает минимальное содержание ДНК для всех круглых гепатоцитов ядерных масок и не проводит прямой различать моноядерные гепатоциты и двухядерные клетки, которые имеют два дискретных ("не прикосновения") ядра одной и той же флоиди. Однако простота этого протокола позволяет адаптировать его к дополнительным параметрам, таким как межядерное расстояние или анализ периметра ячейки, что облегчит идентификацию двухядерных ячеек, обеспечивающих более детальную оценку клеточной плоиды.
Access restricted. Please log in or start a trial to view this content.
протокол
Все эксперименты на животных были ранее одобрены комитетом по этике CIPF. Мыши были размещены в центре, свободном от патогенов, в Центре по борьбе с патогенами Пренсипе Фелипе (Валенсия, Испания), зарегистрированных в качестве экспериментального животновода, пользователя и центра снабжения (reg. No. ES 46 250 0001 002) в соответствии с действующими европейскими и испанскими правилами защиты животных (RD 53/2013).
1. Сбор тканей и подготовка образцов
ПРИМЕЧАНИЕ: Этот протокол описывает, как заморозить ткани без предварительной фиксации или криоконсервации. Для ранее фиксированных/криоконсервированных образцов переходят в раздел 2 и опускают шаг 3.1. Все анализы проводились с использованием взрослых самок C57BL/6 мышей в возрасте 12–16 недель.
- Пожертвование животных фентанил / пентобарбитальной интраперитонеальной инъекции с последующим вывихом шейки матки. С мышью, обращенной к вентральной стороне вверх, откройте брюшную полость и разоблачить печень, схватив кожу пинцетом и выполняя вертикальный разрез от основания нижней части живота до основания грудины с помощью хирургических ножниц.
- Аккуратно удалите желчный пузырь с помощью тонкого пинцета, вскрыть печень и промыть выбранную печень lobule в 10 см Петри блюдо пластины заполнены фосфат-буфера соливого (PBS).
ПРИМЕЧАНИЕ: Рекомендуется сравнить одну и ту же доли печени для каждого животного, в этом случае была использована средняя доли. - Заполните помеченный криомольд с оптимальной температурой резки (OCT) среды при комнатной температуре (RT). Избегайте пузырьков OCT. Если они появляются, нажмите их на край формы с помощью иглы или пипетки отзыв.
- Вложить печень lobule в заполненные OCT криомолд и сразу же поместите его на сухой лед, чтобы обеспечить быстрое замораживание. Храните криомольды при -80 градусов по Цельсию до криосекции.
2. Криосекция
- Транспорт криомолды на сухом льду, чтобы избежать деградации тканей. Перед криосекцией уравновешивать внутри криостата установить до -20 градусов по Цельсию в течение 20 мин.
- Извивайте образец, применяя давление на основание пластикового криомолда. Нанесите жидкий OCT на теплый образец диска на RT, положение в криостате и приложите OCT встроенный образец печени. Применить мягкое давление и ждать 3 мин для OCT заморозить обеспечения образца прилипает к диску.
ПРИМЕЧАНИЕ: Избегайте обработки образца пальцами как можно больше, чтобы избежать деградации тканей. - Заблокируйте образец в руку криостата и отрегулируйте ориентацию так, чтобы край образца был параллельным лезвию криостата. Вырезать в образец, пока ткань не будет достигнута.
- Разделите образец толщиной 6 мкм. Поместите помеченные полиамид покрытием слайд над образцом в течение 5 с, чтобы образец придерживаться на слайде. Поместите слайд на RT в течение 3-5 минут, затем, для достижения наилучших результатов, перейдите непосредственно к разделу 3.
ПРИМЕЧАНИЕ: Для обработки нескольких свежезамороженных образцов воспроизводимые результаты были получены путем временного хранения слайдов в слайд-бокснах на сухом льду до тех пор, пока все образцы не будут обработаны. При использовании этого подхода позволяют всем слайдам уравновесить RT, прежде чем перейти к разделу 3. Формалин фиксированной парафина встроенных (FFPE) образцы могут быть использованы, хотя фон овефлуоресценции увеличивается с помощью этого метода. Чтобы исходить из образцов FFPE, раздел на 4 мкм. Гора, ловя разделы от 40 градусов по Цельсию водяной ванны на полиамид-обработанных горках. Тепло слайдов для 1 ч при температуре 60 градусов по Цельсию, затем deparaffinize серийным RT washes (5 мин) в Коплин банки, содержащие ксилен (x2), этанол 100% (x2), 96% (x2), 70% (x1) и dH2O (x1). Чтобы разоблачить антигены место слайды в цитрат буфера в течение 20 минут при температуре 90 градусов по Цельсию до закалки слайдов в PBS на RT. Приступить к шагу 3.2.
3. Иммуномаркировка флуоресценции
- Исправить секции тканей в дым капот, применяя 1 мл 4% параформальдегида (PFA) в PBS в течение 10 минут на RT. Передача слайдов на PBS заполнены Coplin банку и мыть в течение 3 минут с использованием нежного возбуждения (повторить 3x).
ПРИМЕЧАНИЕ: Отныне и до конца процесса иммуностоинга, избегайте высыхания образца. - Высушите область вокруг каждой секции ткани и окружите с помощью гидрофобной ручки. Пермяки с 0,5% неионический сурфактант (т.е. Тритон X-100) в PBS в течение 15 минут на RT. Затем промыть в PBS заполнены Коплин банку в течение 3 минут с помощью нежного возбуждения (повторить 2x).
- Блок с помощью фильтрованного раствора 1% бычьего сывороточного альбумина (BSA), 5% лошадиной сыворотки, 0,2% неионического сурфактанта в PBS (не менее 1 ч на RT).
- Инкубировать с первичными HNF4 'антитела разбавленной в блокирующем буфере в течение ночи при 4 градусах по Цельсию в темной влажной камере окрашивания (см. Таблица материалов для антител и специфических разбавлений).
- Место слайды в PBS заполнены Coplin банку и мыть в течение 3 минут с помощью нежного возбуждения (повторить 4x).
- Инкубировать с Alexa-488 конъюгированных вторичных антител и Hoechst разбавленной в фильтрованных 1% BSA и 0,2% нонионических сурфактант ов 2 ч на RT в темной влажной камере окрашивания (см. Таблица материалов для антител и конкретных разбавлений).
- Место слайды в PBS заполнены Coplin банку и мыть в течение 3 минут с помощью нежного возбуждения (повторить 4x). Вымойте в ddH2O в течение 3 мин с помощью нежного возбуждения (повторить 2x).
- Маунт слайды, поместив две капли флуоресцентных монтажных носителей на крышку (24 х 60 мм) и прокладка слайдов над ним, устраняя пузырьки, применяя мягкое давление. Для длительного хранения, печать coverslip по краям с четким лаком для ногтей и хранить в темноте при 4 градусах Цельсия.
- Перед началом работы проверьте слайды с помощью обычного флуоресцентного микроскопа, чтобы обеспечить хорошую фиксацию и иммуномаркировку.
ПРИМЕЧАНИЕ: См Рисунок 2A,B для ожидаемых результатов.
4. Приобретение изображения флуоресценции
ПРИМЕЧАНИЕ: Для этого шага требуется платформа высококонтентных изображений(Таблица материалов),которая поддерживает автоматическое приобретение изображения флуоресценции.
- Включите систему визуализации и откройте новый протокол приобретения.
- Выберите 10-разную цель, обратите внимание на область поля зрения (в данном случае 0,6 мм2).
- Установить параметры для получения флуоресценции изображения с использованием соответствующих фильтров возбуждения и выбросов (в соответствии с шагом 3.6). Для Hoechst и Alexa-488 выберите каналы "DAPI" и "GFP" с 390/18 и 438/24 нм и 432,5/48 и 475/24 нм соответственно.
- Сосредоточьте образец и убедитесь, что интенсивность сигнала не насыщена. Убедитесь, что захват изображения осуществляется с одинаковым временем экспозиции для всех изображений или использовать систему, где интенсивность флуоресценции корректируется для времени экспозиции.
- Сканирование образца и получить достаточно евких изображений для получения полного покрытия ткани разделе (приблизительно 20-50 полей зрения, в зависимости от размера выборки).
- Просмотрите базу данных изображений, вручную устраняя (i) плохо сфокусированные поля, (ii) те, на границах каждого раздела ткани (чтобы избежать смещения расчетов плотности клеток), и (iii) те, которые содержат сложенные/физически поврежденные участки ткани, если они присутствуют.
5. Автоматизированный анализ флуоресценции изображения
ПРИМЕЧАНИЕ: Этот шаг требует соответствующего программного обеспечения для анализа изображений(Таблица материалов),способных: (1) автоматически идентифицировать ядра Hoechst в пределах изображений на уровне 405 нм (ядерная сегментация), (2) оценка средней ядерной интенсивности и морфометрии, и (3) анализ порога для определения состояния ядерной флуоресценции на уровне 488 нм (HNF4). Некоторые основные подготовки оператора / экспертизы требуется для визуальной оценки и корректировки сегментации и пороговых параметров в рамках программы, чтобы обеспечить оптимальное состояние ядер и HNF4 "/- статус оптимально закрытой(рисунок 2).
- В программном обеспечении для анализа изображений откройте файл приобретения, содержащий изображения Hoechst (405 нм) и HNF4 (488 нм) со ступени 4.5, и создайте новый протокол анализа.
- Определите длины волн, которые будут использоваться для ядерной сегментации (Hoechst, 405 нм) и для анализа пороговых значений гепатоцитов/NPC (HNF4, 488 нм).
- Отрегулируйте параметры ядерной сегментации программного обеспечения (такие как "минимальная ядерная область" и "чувствительность к обнаружению ядерного оружия"), чтобы обеспечить оптимальную сегрегацию ядер.
ПРИМЕЧАНИЕ: Хорошая сегментация гепатоцитов должна быть приоритетной по сравнению с NPC. Ядра гепатоцитов характерно округлены (диапазон размеров межкварти: 40-64 мкм2). NPC ядра, такие, как синусоидальные эндетели, сплющены / эллиптические или нерегулярные формы и, как правило, меньше и более тесно упакованы, чем у гепатоцитов (диапазон размеров межквартилы: 30-43 мкм2). Для печени мыши использовалась минимальная ядерная область 23 мкм2 и «чувствительность» обнаружения 65% (см. рисунок 2C,D для ожидаемых результатов). Чувствительность определяет, как пиксельные кластеры распознаются как отдельные ядра на основе их интенсивности и должны быть эмпирически протестированы для каждого образца, установленного пользователем, прежде чем приступить к автоматическому анализу изображений. - Измените пороговую интенсивность на уровне 488 нм, чтобы обеспечить оптимальное gating гепатоцитов (HNF4) и непархимальных клеток (HNF4'-).
ПРИМЕЧАНИЕ: См Рисунок 2C,D для ожидаемых результатов. Значение интенсивности порога является относительным и будет зависеть от эффективности окрашивания и параметров приобретения, таких как интенсивность лазера. Поэтому он должен быть стандартизирован пользователем. Используйте известные HNF4 -клетки, такие как эндотелиальные клетки и перипортальные NpCs в качестве внутреннего отрицательного контроля и двухядерных ядер гепатоцитов в качестве положительной ссылки на окрашивание. Проверьте параметры анализа с использованием небольшого количества изображений, чтобы обеспечить хорошую ядерную сегментацию и сегрегацию порога интенсивности перед применением параметров анализа ко всему набору данных. - Выберите следующие ядерные параметры, которые будут количественно: (1) ядерная область, основанная на окрашивании Hoechst (мкм2),(2) означает ядерная интенсивность Hoechst (RU), (3) ядерный коэффициент удлинения (среднее отношение короткой оси ядра к длинной оси ядра, в тех случаях, когда центросимметричный «неудлиненный» объект имеет значение 1, (4) Nuc 1/(форм-фактор), означает ядерный индекс «округлие», рассчитанный по периметру 2/(4'x области). Значения варьируются от 1 до бесконечности, где 1 является идеальным кругом, (5) статус HNF4 "(положительный-1 или отрицательный-0), и (6) ядерных x/y координаты на основе "центр тяжести" (cg), метод для определения центра объекта из серого масштаба изображения с субпиксельной точностью.
- Выполнить анализ для всех наборов выборок и экспортных численных данных от шага 5.5 до программного обеспечения для электронных таблиц.
6. Анализ данных
ПРИМЕЧАНИЕ: Шаг анализа данных может быть выполнен с помощью любого стандартного программного обеспечения электронной таблицы.
- Рассчитайте гепатоциты и номера клеток, не являевась гепатоцитами.
- Рассчитайте общую площадь раздела печени, проанализированное для каждого образца, умножая количество полей зрения по области поля зрения (шаг 4.2).

- Работая с файлами электронной таблицы, созданными для каждого раздела печени, отфильтруйте данные, выбрав только ядра HNF4. Рассчитайте общее количество проанализированных ядер HNF4 и разделите это по общей площади, проанализированному для получения средней плотности гепатоцитов для каждого образца(рисунок 2F).

- Выполните тот же расчет для не-паранхимальных клеток, фильтруя электронную таблицу для клеток HNF4 " (Рисунок 2E).
- Рассчитайте общую площадь раздела печени, проанализированное для каждого образца, умножая количество полей зрения по области поля зрения (шаг 4.2).
- Рассчитайте распределение ядерных размеров гепатоцитов.
- Используя программное обеспечение для электронных таблиц, фильтруйте данные, чтобы выбрать только ядра HNF4.
- Участок значения ядерной области в гистограмме(Рисунок 2G). Установите ширину бендов до 5 мкм2.
ПРИМЕЧАНИЕ: Значения частоты могут быть скорректированы для области (ядра/мм2)в каждом шаге 6.1.1.
- Выполните гепатоцитный ядерный анализ ploidy.
ПРИМЕЧАНИЕ: Данные электронной таблицы с шага 5.6 используются для создания профиля ядерной ploidy для каждого образца. Этот процесс был автоматизирован и может быть выполнен с помощью пользовательского письменного программного обеспечения, которое свободно доступно для загрузки с вспомогательной информацией и демонстрационными наборами данных вhttps://github.com/lukeynoon(см.Дополнительные файлы). Исходный код предоставляется пользователям, желающим адаптировать методологию. Ниже описано описание алгоритма, а также инструкции по установке и использованию. Программа использует данные электронных таблиц для автоматического разделения ядер гепатоцитов на две группы; (1) те, с "простыми" круговыми ядрами и (2) "сложными" некруглыми ядрами, репрезентативными двухядерных клеток с плоейдию 2с. Минимальное содержание ядерной ДНК (функция ядерной области и плотность ДНК) затем рассчитывается для всех "простых" ядер. Последующий шаг затем автоматически калибрует HNF4 "гепатоцитов ядерной ploidy с помощью HNF4 "-ядра, как известный 2'4N внутреннего контроля.- Скачать и установить программное обеспечение.
- Скачать упакованное приложение от: https://github.com/lukeynoon
- Запуск MATLAB. Перейдите на вкладку APP toolstrip, нажмите Установить приложение и открыть загруженное приложение под"Ploidy_Application.mlappinstall". Появится сообщение, подтверждая успешную установку.
ПРИМЕЧАНИЕ: Приложение теперь готово к использованию и останется во вкладке APP toolstrip.
- Данные ввода формата.
ПРИМЕЧАНИЕ: До автоматизированного анализа ядерной ploidy, все файлы электронной таблицы, содержащие данные высокой визуализации контента (шаг 5.6) должны храниться и отформатироваться в соответствии со следующими инструкциями.- В каждом экспортируемом файле данных (. XLS 97-2004 трудовая книжка) от шага 5.6, включите лист термин «измерения клетки» содержа все данные необходимые для анализа ploidy установленных в столбцах(рисунок 3A). Убедитесь, что макет таблицы, включая имена заголовков столбцов, остается неизменным по сравнению с рисунком 3A,поскольку метод анализа находит правильные данные столбца, ища эти имена (см. наборы демонстрационных данных в дополнительных файлах для справки). Если, например, программное обеспечение для анализа изображений с высоким содержанием контента не производит столбец "Light flux"(рисунок 3A),вручную вставьте столбец "Light flux" в том же месте, т.е. столбец K и заполните его нулями.
- Для каждого экспериментального состояния (например, "Injured-d14") предусмотрен контрольный набор данных, который будет использоваться для расчета внутреннего контроля для калибровки ядерной флоидной 2'4N (шаг 6.3.4.3). Здесь выберите образцы печени из необработанных взрослых littermates ("Контроль-d0"; Рисунок 3БКД).
- Для биологических репликаций (в каждом состоянии) храните каждую электронную таблицу в своей папке (как на рисунке 3B). Назовите префиксы папки постепенно, например, "Sample1, Sample2, Sample3... SampleN", согласно именам файлов, содержащимся внутри. Таким образом, каждая папка набора данных (например, "Control-d0") должна содержать ряд субфолок ("Sample1", "Sample2" и т.д.), каждый из которых содержит файл электронной таблицы с одинаковым соответствующим именем.
- Запустите приложение.
- В MATLAB, запустить "Ploidy_Application", нажав на значок в вкладке MY APPS toolstrip (Рисунок 3C). Появится Ploidy_Application графический пользовательский интерфейс (GUI)(рисунок 3C).
- Нажмите кнопку «Путь» для управления данными, чтобы перейти к папке, в которой находятся репатриды данных управления (например, «Control-d0»). Этот путь данных будет отображаться в интерфейсе (например, /Пользователи/Настольная/Контрольная группа).
- Далее, в типе "папка префикса" имя, которое будет дано выходным файлам (например, "Образец").
ПРИМЕЧАНИЕ: Эта приставка может быть изменена на любой текст, при условии, что папки и имена файлов остаются постепенно названными. - Нажмите кнопку «Путь» и перейдите к папке, в которой находятся сравнительные репликации данных (например, «Поврежденный-d14»). Этот путь данных будет отображаться в интерфейсе (например, /Пользователи/Настольная группа/Поврежденный-d14).
- Нажмите Run!. Когда анализ будет завершен, в баре статуса будет написано "Анализ завершен!..".
ПРИМЕЧАНИЕ: Приложение будет сообщать, для каждого образца, стратификации "простых" ядер в 2n, 2n-4n, 4n'8n и 8n " с точки зрения абсолютных подсчетов и в процентах от общего числа (Рисунок 3D). Эти файлы будут автоматически сохранены в каждой папке образца:"Count_2n.txt","Count_2n_to_4n.txt", "Count_4n_to_8n.txt", "Count_8n_and_higher.txt", "Percentage_2n.txt", "Percentage_2nto4n.txt", "Percentage_4nto8n.txt", "Percentage_8n_and_higher.txt". Ploidy_Application автоматически сохранит список для каждого образца, из всех индивидуальных оценок ploidy для "простых" гепатоцитов и негепатоцитов ядер в "Ploidy_All_Hepatocytes.txt" и "Ploidy_NonHepatocytes.txt". Для набора данных управления метод также сохраняет минимальные пороги содержания ДНК, рассчитанные для расслоения ploidy (см. шаг 6.3.4.3.7) в файле под названием "Normalised_Thresholds_Control". Наконец, приложение будет производить папку как для управления, так и для выбранных данных сравнительного состояния под «Summary». Эта папка содержит два субфалеры, "Ploidy" и "Стратификация", которые содержат средние показатели всех предоставленных образцов(рисунок 3D).
- Описание методологии.
ПРИМЕЧАНИЕ: В следующем разделе подробно описывается методология, используемая программным обеспечением для анализа ядерной плоиди. Если пользователь решит не использовать приложение, эти шаги могут быть продолжены с помощью программного обеспечения электронной таблицы для расчета профиля ядерной ploidy вручную.- Отделите ядра на «простые» или «сложные» в соответствии с ядерной морфометрией.
- Рассчитайте "индекс круговорота" для всех ядер, определяемый как ядерный "коэффициент удлинения", разделенный на "Nuc 1/(форм-фактор)", где значение 1.0 указывает на идеальный круг.
ПРИМЕЧАНИЕ: "Ядерное удлинение" и "Nuc 1/(форм-фактор)" являются двумя дискретными мерами "круговости" объекта, которые оценивают взаимодополняющие, неперекрывающиеся морфометрические критерии. Первый измеряет длинные и короткие оси объекта, в то время как второй сравнивает длину периметра объекта с длиной его области. Для укрепления определения ядерной круговорота, используемого в данном протоколе, эти два измерения были объединены в единый "индекс круговорота". Предыдущий подход к оценке ядерной ploidy с использованием описанной методологии используется только ядерное удлинение17. Хотя с помощью этого подхода были получены приемлемые результаты, авторы отметили, что составной "индекс круговорота" улучшает дискриминацию вручную отобранных ядер от моноядерных и двухядерных гепатоцитов (данные не показаны). - Классифицировать ядра с индексом круговой связи 0,8 как "сложные", а те, которые "должны быть" 0,8, как "простые".
- Рассчитайте "индекс круговорота" для всех ядер, определяемый как ядерный "коэффициент удлинения", разделенный на "Nuc 1/(форм-фактор)", где значение 1.0 указывает на идеальный круг.
- Оцените "минимальное" содержание ДНК (м) для всех "простых" ядер.
- Рассчитайте ядерный радиус (r) с помощью формулы:

- Рассчитайте ядерный объем (v) с использованием объема формулы сферы:
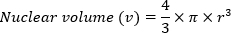
- Создание относительного значения для минимального содержания ДНК (м) с помощью формулы:

- Рассчитайте ядерный радиус (r) с помощью формулы:
- Откалибровать набор данных с помощью ядер NPC (HNF4-) в качестве внутреннего элемента управления 2х4N.
ПРИМЕЧАНИЕ: NPCs имеют содержание ДНК 2'4N в зависимости от состояния клеточного цикла. Таким образом, среднее значение NPC "минимальное" содержание ДНК (NPCм) увеличивается с травмой(рисунок 4A). Ошибка калибровки сведена к минимуму путем установления верхнего предела NPCm, представляющего порог 4c(рисунок 4B).- В таблице выберите только ядра NPC со значениями для "m", которые находятся в пределах 1 стандартного отклонения (SD) режима (это отфильтровывает шум от возможной ошибки сегментации).
- В пределах этого отфильтрованного диапазона, изучить ядерных областях и их соответствующих средних интенсивности Hoechst(Рисунок 4C).
- Оцените наименьшую ядерную область в пределах этого отфильтрованного диапазона с максимальной ядерной интенсивностью Hoechst (т.е. точкой, в которой линия кривой изменяет направление в фильтрованном наборе данных, как показано красным кругом на рисунке 4C). Это значение представляет собой переходное состояние 2N'4N (t), над которым выборка ядер 4c преобладает над ядрами 2c, что приводит к максиме средней интенсивности Hoechst.
ПРИМЕЧАНИЕ: Это значение автоматически определяется программным обеспечением; однако пользователи электронных таблиц могут вручную выбрать этот пункт в качестве переходного размера. - Рассчитайте минимальное содержание ДНК, представленное этим переходным размером (тм),следующим шагом 6.3.4.2.
- Чтобы оценить 4N плечо набора данных NPCm, добавьте 1 SD к значению tm. В результате число (Рисунок 4B) описывает верхний предел NPC минимальное содержание ДНК, которые будут использоваться для расслоения ядерной ploidy (S4c).
- Повторите шаги 6.3.4.3.1-6.3.3.3.5 для всех «контрольных» образцов.
ПРИМЕЧАНИЕ: Например, на рисунке 3невредимые контрольные печени ("Control-d0") используются в качестве контрольного состояния. - Рассчитайте средний порог стратификации 4c (S4c)для "контрольных" образцов и используйте это для экстраполировать 2c (S2c)и 8c (S8c)границы для минимального содержания ДНК (м). Пороги стратификации автоматически генерируются и сохраняются программным обеспечением (шаг 6.3.3.3).
ПРИМЕЧАНИЕ: В зависимости от конструкции исследования, средние значения порога стратификации могут быть рассчитаны для каждого состояния или для конкретных условий (например, здоровой контрольной печени). Тем не менее, программное обеспечение для анализа ядерной ploidy требует, чтобы один из набора из 2 файлов был обозначен как "контроль" для целей расчета относительных значений ploidy. - Рассчитайте значение ploidy для всех ядер, используя значение S2c, генерируемое в шаге 6.3.4.3.7 в каждом:

- Стратить "простые" гепатоциты (HNF4) ядра в 2c/4c/8c/'gt;8c кронштейны в соответствии со следующими критериями: "2c" HNF4 " "4c" HNF4" No 2 "lt; р- 4; "8c" HNF4" No 4 "lt; р No 8; "Зgt;8c" HNF4" No 8
- Чтобы реконструировать пространственное узор подгрупп, разделите ядерные данные в каждой таблице выборки в соответствии с соответствующими полями, в которых они были приобретены. Затем используйте связанные ядерные x/y координаты (от шага 5.5) для создания подгрупп по сюжету в 2D(рисунок 5C).
- Отделите ядра на «простые» или «сложные» в соответствии с ядерной морфометрией.
- Скачать и установить программное обеспечение.
Access restricted. Please log in or start a trial to view this content.
Результаты
Этот метод был использован для измерения воздействия холестатической травмы на печень взрослой мыши, кормя животных в течение 0-21 дней с гепатотоксическим питанием, содержащим 0,1% 3,5-диэтитоксикарбонил-1,4-дигидроколлайдин (DDC)17. Хроническое кормление DDC при...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Описан высокопосевной, высокопроизводительный подход к анализу ремоделирования тканей и оценке гепатоцитов ядерной флоиди в печени мурин. После того, как пользователь ознакомился с процедурой, пользователь может обрабатывать, изображения и анализировать несколько образцов в течени?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была профинансирована испанским правительством MINECO гранты BFU2014-58686-P (LAN) и SAF-2017-84708-R (DJB). LAN была поддержана национальным MINECO Рамон у Cajal стипендий RYC-2012-11700 и план GenT награду (Comunitat Valenciana, CDEI-05/20-C), и FMN регионального ValI-D студентов Валенсии Generalitat ACIF/2016/020. РП хотела бы отметить профессора Эву К. Палух за финансирование. Мы благодарим д-ра Алисию Мартинес-Ромеро (CIPF Cytometry service) за помощь в платформе IN Cell Analyzer.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 3,5-diethoxycarboxynl-1,4-dihydrocollidine diet (DDC) | TestDiet | 1810704 | Modified LabDiet mouse diet 5015 with 0.1% DDC |
| Alexa Fluor 488 donkey anti-goat IgG (H+L) | Invitrogen | A11055 | Dilution 1:500 |
| Bovine Serum Albumin | Sigma-Aldrich | A7906 | |
| Cryostat Leica CM1850 UV | Leica biosystems | CM1850 UV | Tissue sectioning |
| Fluorescent Mounting medium | Dako | S3023 | |
| GraphPad Prism | GraphPad Software | Prism 8 | Statistical software for graphing data |
| Hoechst 33342 | Sigma-Aldrich | B2261 | Final concentration 5 µg/mL |
| IN Cell Analyzer 1000 | GE Healthcare Bio-Sciences Corp | High-Content Cellular Imaging and Analysis System | |
| MATLAB | MathWorks | R2019a | Data analytics software for automated analysis of nuclear ploidy |
| Microscope coverslides | VWR International | 630-2864 | Size of 24 x 60 mm |
| Microsoft Office Excel | Microsoft | Speadsheet software | |
| OCT Tissue Tek | Pascual y Furió | 4583 | |
| Paraformaldehyde | Panreac AppliChem | 141451.121 | |
| Pen for immunostaining | Sigma-Aldrich | Z377821-1EA | 5mm tip width |
| Polysine Microscope Slides | VWR International | 631-0107 | |
| Rabbit polyclonal Anti-HNF4α | Thermo Fisher Scientific | PA5-79380 | Dilution 1:250 (alternative) |
| Rabit polyclonal Anti-HNF4α | Santa Cruz Biotechnology | sc-6556 | Dilution 1:200 (antibody used in the study) |
| Tween 20 | Sigma-Aldrich | P5927 |
Ссылки
- Gentric, G., Desdouets, C. Polyploidization in liver tissue. American Journal of Pathology. 184 (2), 322-331 (2014).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Gentric, G., Desdouets, C. Liver polyploidy: Dr Jekyll or Mr Hide? Oncotarget. 6 (11), 8430-8431 (2015).
- Wilkinson, P. D., et al. The Polyploid State Restricts Hepatocyte Proliferation and Liver Regeneration in Mice. Hepatology. 69 (3), 1242-1258 (2019).
- Wilkinson, P. D., et al. Polyploid Hepatocytes Facilitate Adaptation and Regeneration to Chronic Liver Injury. The American Journal of Pathology. 189 (6), 1241-1255 (2019).
- Zhang, S., et al. The Polyploid State Plays a Tumor-Suppressive Role in the Liver. Developmental Cell. 44 (4), 447-459 (2018).
- Chao, H. W., et al. Circadian clock regulates hepatic polyploidy by modulating Mkp1-Erk1/2 signaling pathway. Nature Communications. 8 (1), 2238(2017).
- Celton-Morizur, S., Merlen, G., Couton, D., Margall-Ducos, G., Desdouets, C. The insulin/Akt pathway controls a specific cell division program that leads to generation of binucleated tetraploid liver cells in rodents. Journal of Clinical Investigation. 119 (7), 1880-1887 (2009).
- Wang, M. J., Chen, F., Lau, J. T. Y., Hu, Y. P. Hepatocyte polyploidization and its association with pathophysiological processes. Cell Death & Disease. 8 (5), e2805(2017).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Toyoda, H. Changes to hepatocyte ploidy and binuclearity profiles during human chronic viral hepatitis. Gut. 54 (2), 297-302 (2005).
- Miyaoka, Y., et al. Hypertrophy and Unconventional Cell Division of Hepatocytes Underlie Liver Regeneration. Current Biology. 22 (13), 1166-1175 (2012).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. , (2019).
- Danielsen, H., Lindmo, T., Reith, A. A method for determining ploidy distributions in liver tissue by stereological analysis of nuclear size calibrated by flow cytometric DNA analysis. Cytometry. 7 (5), 475-480 (1986).
- Guidotti, J. E., et al. Liver Cell Polyploidization: A Pivotal Role for Binuclear Hepatocytes. Journal of Biological Chemistry. 278 (21), 19095-19101 (2003).
- Severin, E., Meier, E. M., Willers, R. Flow cytometric analysis of mouse hepatocyte ploidy - I. Preparative and mathematical protocol. Cell and Tissue Research. 238 (3), 643-647 (1984).
- Manzano-Núñez, F., et al. Insulin resistance disrupts epithelial repair and niche-progenitor Fgf signaling during chronic liver injury. PLoS Biology. 17 (1), e2006972(2019).
- Morales-Navarrete, H., et al. A versatile pipeline for the multi-scale digital reconstruction and quantitative analysis of 3D tissue architecture. eLife. 4, e11214(2015).
- Baratta, J. L., et al. Cellular organization of normal mouse liver: A histological, quantitative immunocytochemical, and fine structural analysis. Histochemistry and Cell Biology. 131 (6), 713-726 (2009).
- Pandit, S. K., et al. E2F8 is essential for polyploidization in mammalian cells. Nature Cell Biology. 14 (11), 1181-1191 (2012).
- Vinogradov, A. E., Anatskaya, O. V., Kudryavtsev, B. N. Relationship of hepatocyte ploidy levels with body size and growth rate in mammals. Genome. 44 (3), 350-360 (2001).
- Tanami, S., et al. Dynamic zonation of liver polyploidy. Cell and Tissue Research. 368 (2), 405-410 (2017).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv B Cell Pathology Including Molecular Pathology. 64 (1), 387-393 (1993).
- Gentric, G., Celton-Morizur, S., Desdouets, C. Polyploidy and liver proliferation. Clinics and Research in Hepatology and Gastroenterology. 36 (1), 29-34 (2012).
- Uhlén, M., et al. Tissue-based map of the human proteome. Science. 347 (6220), 1260419(2015).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены