Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Изучение митохондриального энергетического метаболизма одиночных 3D-сфероидов микротизюса с использованием анализа внеклеточного потока
В этой статье
Erratum Notice
Резюме
Эти протоколы помогут пользователям исследовать энергетический метаболизм митохондрий в 3D-сфероидах, полученных из клеточных линий рака, с использованием анализа внеклеточного потока Seahorse.
Аннотация
Трехмерные (3D) клеточные агрегаты, называемые сфероидами, стали авангардом клеточной культуры in vitro в последние годы. В отличие от культивирования клеток в виде двумерных одноклеточных монослоев (2D-культура), сфероидная клеточная культура способствует, регулирует и поддерживает физиологическую клеточную архитектуру и характеристики, которые существуют in vivo, включая экспрессию белков внеклеточного матрикса, клеточную сигнализацию, экспрессию генов, производство белка, дифференцировку и пролиферацию. Важность 3D-культуры была признана во многих областях исследований, включая онкологию, диабет, биологию стволовых клеток и тканевую инженерию. За последнее десятилетие были разработаны усовершенствованные методы получения сфероидов и оценки их метаболической функции и судьбы.
Анализаторы внеклеточного потока (XF) использовались для изучения митохондриальной функции в 3D-микротизюсах, таких как сфероиды, с использованием либо пластины захвата островков XF24, либо сфероидной микропластины XFe96. Однако отдельные протоколы и оптимизация зондирования митохондриального энергетического обмена у сфероидов с использованием технологии XF подробно не описаны. В данной статье представлены подробные протоколы зондирования митохондриального энергетического обмена в одиночных 3D-сфероидах с использованием сфероидных микропластин с анализатором XFe96 XF. Используя различные линии раковых клеток, технология XF способна различать клеточное дыхание в 3D-сфероидах не только разных размеров, но и разных объемов, количества клеток, содержания и типа ДНК.
Оптимальные концентрации митохондриальных эффекторных соединений олигомицина, БАМ15, ротенона и антимицина А используются для исследования специфических параметров митохондриального энергетического метаболизма в 3D-сфероидах. В этой статье также обсуждаются методы нормализации данных, полученных от сфероидов, и рассматриваются многие соображения, которые следует учитывать при изучении сфероидного метаболизма с использованием технологии XF. Этот протокол поможет стимулировать исследования в передовых сфероидных моделях in vitro .
Введение
Достижения в области моделей in vitro в биологических исследованиях быстро прогрессировали за последние 20 лет. Такие модели теперь включают модальности «орган на чипе», органоиды и 3D-сфероиды микротизю, все из которых стали общим фокусом для улучшения трансляции между исследованиями in vitro и in vivo. Использование передовых моделей in vitro, особенно сфероидов, охватывает несколько областей исследований, включая тканевую инженерию, исследования стволовых клеток, рак и биологию заболеваний 1,2,3,4,5,6,7, и тестирование безопасности, включая генетическую токсикологию 8,9,10, токсикологию наноматериалов11, 12,13,14, и тестирование безопасности и эффективности лекарств 8,15,16,17,18,19.
Нормальная морфология клеток имеет решающее значение для биологического фенотипа и активности. Культивирование клеток в 3D-сфероиды микротизуса позволяет клеткам принимать морфологию, фенотипическую функцию и архитектуру, более похожую на ту, что наблюдается in vivo , но ее трудно захватить с помощью классических методов монослойной клеточной культуры. Как in vivo , так и in vitro клеточная функция напрямую зависит от клеточной микросреды, которая не ограничивается клеточной связью и программированием (например, образования клеточно-клеточных переходов, возможности формирования клеточных ниш); воздействие на клетки гормонов и факторов роста в непосредственной среде (например, воздействие клеточных цитокинов как часть воспалительной реакции); состав физических и химических матриц (например, выращиваются ли клетки в пластической культуре жесткой ткани или в эластичной тканевой среде); и, самое главное, как на клеточный метаболизм влияют питание и доступ к кислороду, а также переработка метаболических отходов, таких как молочная кислота.
Анализ метаболических потоков является мощным способом изучения клеточного метаболизма в определенных системах in vitro . В частности, технология XF позволяет анализировать живые изменения в клеточной биоэнергетике интактных клеток и тканей в режиме реального времени. Учитывая, что многие внутриклеточные метаболические события происходят в течение порядка секунд до минут, функциональные подходы в реальном времени имеют первостепенное значение для понимания изменений клеточного метаболического потока в реальном времени в неповрежденных клетках и тканях in vitro.
В данной статье представлены протоколы культивирования раковых клеточных линий A549 (аденокарцинома легких), HepG2/C3A (гепатоцеллюлярная карцинома), MCF-7 (аденокарцинома молочной железы) и SK-OV-3 (аденокарцинома яичников) в виде 3D-сфероидных моделей in vitro с использованием подходов принудительной агрегации (рисунок 1). В нем также (i) подробно описывается, как исследовать митохондриальный энергетический метаболизм одиночных 3D-сфероидов с помощью анализатора Agilent XFe96 XF, (ii) выделяются способы оптимизации анализов XF с использованием одиночных 3D-сфероидов и (iii) обсуждаются важные соображения и ограничения зондирования метаболизма 3D-сфероидов с использованием этого подхода. Самое главное, в этой статье описывается, как собираются наборы данных, которые позволяют рассчитать скорость потребления кислорода (OCR) для определения окислительного фосфорилирования и, следовательно, митохондриальной функции в клеточных сфероидах. Хотя это не анализируется для этого протокола, скорость внеклеточного подкисления (ECAR) является еще одним параметром, который измеряется вместе с данными OCR в экспериментах XF. Однако ECAR часто плохо или неправильно интерпретируется из наборов данных XF. Мы предоставляем комментарий относительно ограничений расчета ECAR в соответствии с основными подходами производителя технологии.
Access restricted. Please log in or start a trial to view this content.
протокол
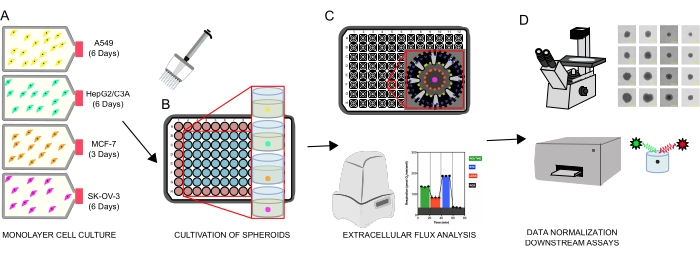
Рисунок 1: Графический рабочий процесс для генерации клеточных сфероидов, анализа внеклеточного потока и последующих анализов. Четыре линии раковых клеток были селективно культивированы как монослои (А), отделены от колб тканевых культур и засеяны в сверхнизкие прикрепленные 96-луночные микропластины с образованием сфероидов (В). A549 Легочная карцинома, HepGG2/C3A печеночная карцинома, SK-OV-3 аденокарцинома яичников и MCF-7 клетки карциномы молочной железы были посеяны через 1 × 103-8 × 103 клеток / хорошо и выращены до 7 дней для формирования одиночных сфероидов и оптимизации плотности посева сфероидов и времени культивирования путем непрерывного наблюдения и планиметрических измерений. После образования одиночные сфероиды промывали в безсыворочную XF-среду и тщательно сеяли в сфероидные микропластины, предварительно покрытые поли-D-лизином (C). Сфероиды подвергали внеклеточному анализу потока с использованием анализатора XFe96 с использованием нескольких протоколов для решения: (1) оптимального размера сфероида для ответа базального митохондриального дыхания; (2) оптимизированное титрование митохондриальных респираторных ингибиторов; (3) Оптимизация размещения сфероидов в микропластинчатых скважинах. (D) Для нормализации данных и других последующих анализов in vitro использовались анализы после XF, фазоконтрастная микроскопия и количественная оценка сфероидной ДНК. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
1. Культивирование линий раковых клеток в виде 3D сфероидов in vitro
| Клеточная линия | Описание | Культуральная среда | Источник |
| А549 | Клеточная линия карциномы легких | РПИ 1640 | Европейская коллекция аутентифицированных клеточных культур (ECACC) |
| Пируват натрия (1 мМ) | |||
| Пенициллин-Стрептомицин - (100 Ед/мл – 100 мг/мл) | |||
| 10 % (об/об) FBS | |||
| HepG2/C3A | Клеточная линия печеночной карциномы, клональное производное родительской клеточной линии HepG2 | ДМЭМ | Американская коллекция культуры тканей (ATCC) |
| Пенициллин-Стрептомицин - (100 Ед/мл – 100 мг/мл) | |||
| 10 % (об/об) FBS | |||
| МКФ7 | Клеточная линия аденокарциномы молочной железы | РПИ 1640 | Европейская коллекция аутентифицированных клеточных культур (ECACC) |
| Пируват натрия (1 мМ) | |||
| Пенициллин-Стрептомицин - (100 Ед/мл – 100 мг/мл) | |||
| 10 % (об/об) FBS | |||
| СК-ОВ-3 | Клеточная линия аденокарциномы яичников | РПИ 1640 | Европейская коллекция аутентифицированных клеточных культур (ECACC) |
| Пируват натрия (1 мМ) | |||
| Пенициллин-Стрептомицин - (100 Ед/мл – 100 мг/мл) | |||
| 10 % (об/об) FBS | |||
| Компонент | Среда для анализа RPMI (конечный объем 50 мл) | ||
| Базовый средний | Agilent Seahorse XF RPMI, pH 7.4 | ||
| Глюкоза (1 М стерильного вещества) | 11 мМ (стоковый раствор 0,55 мл) | ||
| L-глютамин (стерильный материал 200 мМ) | 2 мМ (0,5 мл запасного раствора) | ||
| Пируват натрия (стерильный материал 100 мМ) | 1 мМ (0,5 мл запасного раствора) |
Таблица 1: Раковые клеточные линейные среды и композиции XF-сред.
- Культивируйте все клеточные линии с использованием стандартной асептической техники культивирования тканей и подтвердите, что они свободны от микоплазмы, используя подходящий набор для анализа.
- Культивируйте клеточные линии в колбах для культивирования тканей Т75 или эквиваленте, используя рекомендуемую среду (таблица 1). Культивируйте клеточные линии до 65-80% слияния и проходите их регулярно максимум до 25 проходов.
- Промыть колбы клеточной культуры дважды в модифицированном фосфатно-буферном физиологическом растворе Dulbecco (DBPS).
- Отделяют клетки от колб 3 мл клеточного диссоциационного реагента (см. Таблицу материалов) в течение 5 мин при 37 °C и подтверждают отслоение микроскопией.
- Аккуратно аспирировать отсоединенную клеточную суспензию, чтобы обеспечить одноклеточную суспензию и дезактивировать реагент диссоциации клеток 7 мл полной тканевой питательной среды.
- Соберите клетки путем центрифугирования при 300 × г в течение 5 мин, выбросьте супернатант и повторно сведите клетки в полную среду.
- Подсчитайте клетки с помощью гемоцитометра или автоматизированного счетчика клеток и титруйте до желаемой плотности клеток, необходимой для посева.
ПРИМЕЧАНИЕ: Для посева всей 96-луночной пластины при 100 мкл/лунке при 4 × 103 клетках/лунке клетки следует титровать до 4 × 104 клеток/мл в рекомендуемом объеме 12 мл. - Декантируйте клеточную суспензию в стерильный резервуар и дозируйте 100 мкл клеточной суспензии в каждую лунку клеточно-репеллентной микропластины с помощью многоканального пипеттера.
ПРИМЕЧАНИЕ: Только внутренние 60 лунок микропластины должны быть засеяны, а остальные заполнены DPBS. Это сформирует барьер испарения, обеспечит однородность сфероидов по всей пластине и минимизирует эффекты края пластины. - Центрифужные сфероидные микропластины при 300 × г в течение 15 мин для форсирования клеток в рыхлые агрегаты.
- Инкубируйте пластины при 37 °C, 5% CO2 в течение минимум 3 дней, чтобы обеспечить образование сфероидов.
- Выполняйте фазово-контрастную микроскопию с использованием стандартизированных лабораторных практик для мониторинга роста сфероидов. Пополняйте клеточную культуральную среду каждые 3 дня или два раза в неделю, выполняя обмен средой в половину объема.
2. Зондирование митохондриального энергетического метаболизма одиночных сфероидов с использованием технологии Extracellular Flux (XF)
- Подготовка к анализу (за день до этого)
- Проверьте жизнеспособность сфероида с помощью инвертированного светового микроскопа с фазовым контрастом при 4-кратном увеличении, чтобы обеспечить неповрежденную сфероидную структуру, морфологию и общую однородность между образцами.
- Увлажните картридж датчика.
- Аликвота ~20 мл калибранта в коническую трубку.
- Поместите коническую трубку, содержащую калибрант, в инкубатор без CO2 37 °C на ночь.
- Извлеките содержимое из набора для анализа.
- Извлеките картридж датчика из вспомогательной пластины и поместите его вверх ногами на столешницу рядом с вспомогательной пластиной.
- Пипетка 200 мкл стерильного ddH2Oв каждую скважину картриджной пластины датчика с помощью многоканальной пипетки P300.
- Поместите картридж датчика поверх вспомогательной пластины.
- Убедитесь, что уровень воды в каждой скважине достаточно высок, чтобы погрузить датчики.
- Перенесите собранный картридж датчика в инкубатор без CO2 37 °C и оставьте на ночь.
ПРИМЕЧАНИЕ: Этот этап может быть выполнен за 12-72 ч до начала анализа.
- Микропластина сфероидного анализа покрытия
- Используя асептические методы, добавляют 30 мкл/лунку стерильного раствора поли-D-лизина (0,1 мг/мл) к сфероидной микропластине и инкубируют ее в течение 30 мин при комнатной температуре.
- Аспирируйте раствор из каждой лунки сфероидной микропластины, переверните пластину и плотно постучите ее по папиросной бумаге, чтобы удалить любой остаточный раствор.
- Дважды промыть пластину 200 мкл/лунку стерильным ddH2O.
- После окончательной стирки переверните микропластинку и плотно постучите ее на папиросную бумагу, чтобы удалить остаточную воду.
- Дайте пластине высохнуть на воздухе в течение 30 минут перед использованием или хранением при температуре 4 °C для будущего использования.
ПРИМЕЧАНИЕ: Микропластина сфероидного анализа должна быть покрыта молекулярным клеем, чтобы гарантировать, что сфероиды закреплены в нижней части микропластины. Без молекулярного клея сфероиды могут смещаться и мешать результатам анализа. Другие молекулярные клеи также могут быть использованы в качестве альтернативы Poly-D-лизину для пластин предварительного покрытия. Предварительно покрытые пластины могут храниться при температуре 4 °C, но их следует оставить для выравнивания до комнатной температуры до начала анализа.
- Подготовка среды для анализа XF
- Подготовка среды XF RPMI, как описано в таблице 1, и стерильного фильтра со шприцевым фильтром 0,22 мкм
- Подготовка к анализу (за 1 ч до анализа)
- Предварительно прогрейте добавленную XF RPMI пробирную среду до 37 °C.
- Предварительно прогрейте микропластинку сфероидного анализа с покрытием в инкубаторе безСО2 37 °C или сухой ванне.
- Подготовьте картридж датчика.
- Извлеките коническую трубку, содержащую калибрант и картридж датчика, из воздушного инкубатора.
- Извлеките картридж датчика из вспомогательной пластины и поместите его вверх ногами на рабочую поверхность.
- Используя многоканальную пипетку P300, аспирируйте воду с опорной плиты и выбросьте ее.
- Залейте раствор калибранта в резервуар для стерильных реагентов и добавьте 200 мкл/лунку предварительного калибранта к вспомогательной пластине с помощью многоканальной пипетки P300.
- Возьмите картридж датчика и поместите его обратно на верхнюю часть вспомогательной пластины, чтобы датчики были хорошо погружены в калибр.
- Перенесите собранный картридж датчика обратно в инкубатор без CO2 при 37 °C до готовности к загрузке растворов для впрыска в порт.
- Промыть сфероиды пробирной средой.
- Извлеките пластину для культуры сфероидов из инкубатора 37 °C, 5% CO2 и наблюдайте за сфероидами под микроскопом, чтобы обеспечить их целостность перед этапами переноса сфероидов.
- Загрузите все скважины сфероидной пластины 180 мкл/лунку предварительной пробирной среды, включая любые скважины фоновой коррекции.
- Частично заполните чашку Петри размером 7 см 3 мл пробирной среды.
- Используя многоканальную пипетку, нагруженную широкими наконечниками отверстий пипетки, переведите сфероиды с 96-луночной культуральной пластины в чашки Петри 7 см, установив пипетку на объем аспирации 10-50 мкл.
- Семена сфероидов в предварительно покрытую сфероидную микропластину.
- Используя микроскоп для рассечения и аппарат лайтбокса, перенесите сфероиды из чашки Петри на микропластину сфероидного анализа, как описано ниже.
- Установите объем одноканального пипеттера, оснащенного широким наконечником отверстия пипетки, на 20 мкл и тщательно аспирировать один сфероид. Поместите наконечник непосредственно в центр каждой лунки микропластины сфероидного анализа и позвольте гравитации элюировать один сфероид в центр каждой лунки, т. е. не выталкивайте какую-либо среду из кончика пипетки и позвольте капиллярному действию вывести сфероид из кончика пипетки. Для подтверждения элюирования содержимое пипетатора можно пипетировать обратно в 7 см чашку Петри под микроскопом.
ПРИМЕЧАНИЕ: Гравитационное элюирование одного сфероида обычно занимает 15-30 с в зависимости от размера/плотности сфероида. В течение этого времени пипетку не следует снимать. Любые лунки фоновой коррекции должны быть свободны от сфероидов и содержать только пробирную среду. Под микроскопом подтвердите положение каждого сфероида. Каждый сфероид в идеале должен быть расположен в центре каждой скважины. - После того, как все сфероиды были перенесены на микропластину сфероидного анализа, перенесите пластину в инкубатор без CO2 при 37 °C в течение как минимум 1 ч до анализа.
- Установите объем одноканального пипеттера, оснащенного широким наконечником отверстия пипетки, на 20 мкл и тщательно аспирировать один сфероид. Поместите наконечник непосредственно в центр каждой лунки микропластины сфероидного анализа и позвольте гравитации элюировать один сфероид в центр каждой лунки, т. е. не выталкивайте какую-либо среду из кончика пипетки и позвольте капиллярному действию вывести сфероид из кончика пипетки. Для подтверждения элюирования содержимое пипетатора можно пипетировать обратно в 7 см чашку Петри под микроскопом.
- Используя микроскоп для рассечения и аппарат лайтбокса, перенесите сфероиды из чашки Петри на микропластину сфероидного анализа, как описано ниже.
3. Подготовка и загрузка соединений в картридж датчика для XF анализов
| Инъекционная стратегия | Соединение (порт) | Начальный объем микролунки XFe96 (мкл) | Желаемая конечная концентрация скважины | Объем порта (мкл) | Конечный объем микролунки XFe96 после впрыска (мкл) | Концентрация рабочего капитала |
| 1 | Олигомицин (А) | 180 | 3 мкг/мл | 20 | 200 | 30 мкг/мл |
| Ротенон (B) | 200 | 2 мкМ | 20 | 220 | 22 мкМ | |
| Антимицин А (В) | 200 | 2 мкМ | 20 | 220 | 22 мкМ | |
| 2 | БАМ15 (А) | 180 | 5 мкМ | 20 | 200 | 50 мкМ |
| Ротенон (B) | 200 | 2 мкМ | 20 | 220 | 22 мкМ | |
| Антимицин А (В) | 200 | 2 мкМ | 20 | 220 | 22 мкМ |
Таблица 2: Концентрации митохондриальных соединений для зондирования митохондриального энергетического метаболизма одиночных 3D-сфероидов с помощью анализатора XFe96.
- Подготовьте концентрации рабочего материала каждого соединения, как указано в таблице 2 , используя полностью дополненную, предварительно высушенную среду для анализа XF RPMI.
- Ориентируйте картриджную пластину (соединенную с полезной пластиной) по колоннам, 1-12 слева направо.
- Если используется направляющая заряжания, поместите ее на пластину картриджа в соответствии с процедурой хорошо нагружа, например, если порт А загружается первым, убедитесь, что А виден в левом верхнем углу направляющей.
- Перенесите рабочий раствор каждого соединения в подходящий резервуар и, используя калиброванную многоканальную пипетку P100, дозируйте 20 мкл во все соответствующие порты. Повторите для каждого соединения в остальные порты.
ПРИМЕЧАНИЕ: Если на пластине картриджа датчика не используются какие-либо порты, их можно оставить пустыми или заполнить пробирной средой. Если используется только выбор определенной буквы порта, убедитесь, что другие порты, соответствующие этой букве, загружены средой анализа; в противном случае воздух будет закачиваться в скважину, что ставит под угрозу результаты в этих скважинах. - После загрузки порта снимите направляющие для загрузки пластин (если они используются) и подготовьте анализатор к загрузке картриджа датчика.
ПРИМЕЧАНИЕ: Если анализ не выполняется сразу после загрузки портов, поместите крышку обратно на картридж датчика и поместите пластину обратно в воздушный инкубатор при температуре 37 °C до тех пор, пока она не будет готова к загрузке в машину.
4. Проектирование анализов, стратегии инъекций и сбор данных
- Проведение анализа
- Включите анализатор и подключите к контроллеру (компьютеру).
ПРИМЕЧАНИЕ: Это можно проверить по состоянию подключения прибора на панели виджетов программного обеспечения Wave Controller. - Перейдите на страницу шаблонов в программном обеспечении WAVE, найдите файл шаблона анализа для эксперимента и дважды щелкните его, чтобы открыть.
ПРИМЕЧАНИЕ: Если шаблон анализа не отображается в представлении Шаблоны , импортируйте файл шаблона в папку шаблона с общего сетевого диска или USB-устройства флэш-памяти. - Чтобы начать анализ, перейдите на вкладку Выполнить анализ .
ПРИМЕЧАНИЕ: Если определения групп были правильно распределены на карте пластин, анализ будет готов к выполнению, как указано зеленой галочкой в правой части страницы. На этом этапе любая дополнительная информация может быть введена на странице сводки анализа или на странице, оставленной пустой; перейдите к следующему шагу. В связи с задержкой проникновения митохондриальных модуляторов в 3D-сфероиды микротизов (рисунок 2) используют информацию протокола измерения, описанную в таблице 3.
- Включите анализатор и подключите к контроллеру (компьютеру).
| Период измерения | Номер впрыска и порт | Детали измерений | Продолжительность периода (ч:мин:с) |
| Калибровка | Не применимо | Анализаторы XF всегда выполняют эту калибровку, чтобы убедиться, что измерения точны | 00:20:00 (это среднее значение и может варьироваться в зависимости от машины) |
| Уравновешивание | Не применимо | Уравновешивание происходит после калибровки и рекомендуется. | 00:10:00 |
| Базальный | Не применимо | Циклы = 5 | 00:30:00 |
| Микс = 3:00 | |||
| Ожидание = 0:00 | |||
| Мера = 3:00 | |||
| Олигомицин / БАМ15 | Инъекция 1 (порт A) | Циклов = 10 | 01:00:00 |
| Микс = 3:00 | |||
| Ожидание = 0:00 | |||
| Мера = 3:00 | |||
| Ротенон + антимицин А | Инъекция 2 (порт B) | Циклов = 10 | 01:00:00 |
| Микс = 3:00 | |||
| Ожидание = 0:00 | |||
| Мера = 3:00 | |||
| Общее время: | 03:00:00 | ||
Таблица 3: Настройка протокола для зондирования митохондриального энергетического метаболизма одиночных 3D-сфероидов с помощью анализатора XFe96.
- Нажмите кнопку Начать запуск , чтобы открыть диалоговое окно расположение сохранения .
- Введите место сохранения результирующего файла и поместите собранный картридж датчика в термоотлив, который появляется из дверцы на боковой стороне анализатора. Подождите, пока тепловой лоток откроется автоматически и на экране отобразится сообщение Load Calibrant Utility Plate . Прежде чем следовать подсказкам на экране, убедитесь: i) правильное размещение картриджа датчика на пластине Утилиты, ii) снята крышка с картриджа датчика и iii) правильная ориентация картриджа датчика на вспомогательной пластине.
- Следуйте экранным командам, чтобы начать калибровку картриджа датчика.
ПРИМЕЧАНИЕ: Время, необходимое для завершения калибровки, составляет приблизительно 10-20 мин (для анализов при 37 °C). - После калибровки картриджа датчика загрузите сфероидную микропластинку в анализатор, следуя инструкциям на экране на Wave Controller, чтобы инициировать 12-минутный шаг равновесия.
ПРИМЕЧАНИЕ: Зеленые поля с белыми галочками указывают на «хорошую» калибровку для этого колодца. Если какие-либо скважины не обеспечивают «хорошую» калибровку, они будут обозначены красным прямоугольником и белым крестом. Такие скважины должны быть отмечены и исключены из любого анализа после завершения анализа с использованием вкладки модификационного анализа . - Подождите, пока анализатор автоматически начнет получать базовые измерения после того, как машина завершит этап равновесия (как указано в протоколе прибора).
- Чтобы завершить эксперимент, следуйте экранным командам на контроллере WAVE.
ПРИМЕЧАНИЕ: После того, как сфероидная микропластина была удалена из анализатора, отбросьте картридж датчика и отложите сфероидную пластину для дальнейшего анализа, если это необходимо (например, двухцепочечная (ds) количественная оценка ДНК). Если микропластинка не требуется для дальнейшего анализа, ее можно выбросить вместе с картриджем датчика. - Дождитесь появления диалогового окна анализа и просмотрите результаты или вернитесь в представление шаблонов .
5. Стратегии нормализации и анализа данных - нормализация после анализа и последующие анализы (необязательные шаги)
- Нормализация данных
- Чтобы нормализовать данные сфероидов, обратитесь к серии протоколов, относящихся к стратегиям нормализации данных для расчета размера и объема сфероидов и количественной оценки dsDNA в сфероидных анализах. Они были включены в качестве дополнительных файлов; см. Дополнительный файл 1 и Дополнительный файл 2.
- Анализ данных
- Чтобы экспортировать данные в один из генераторов автоматизированного анализа, выполните команды экспорта данных на контроллере WAVE и выберите генератор экспорта, соответствующий типу анализа. Кроме того, можно экспортировать файл данных и загрузить его в аналитику Seahorse.
ПРИМЕЧАНИЕ: Недостатком генераторов отчетов и аналитики Seahorse является то, что анализ данных ограничен тем, как разработан анализ XF, и не позволяет измерять средние значения по циклам измерения. Ручной экспорт наборов данных из программного обеспечения прибора позволяет использовать предпочтения пользователя в этом отношении. Учитывая, что стратегия инъекций для оценки митохондриального дыхания 3D-сфероидов, вероятно, будет отличаться от типичного теста «MitoStress», была разработана серия шаблонов электронных таблиц, чтобы помочь проанализировать эти наборы данных, специфичные для 3D-клеточных культур, и будет предоставлена по запросу. Эти файлы шаблонов данных предоставят данные о ключевых параметрах митохондриальных дыхательных путей, подробно описанных и объясненных на рисунке 2. - Чтобы проанализировать данные, экспортируйте данные в виде отчета электронной таблицы из программного обеспечения контроллера WAVE и используйте независимый шаблон электронной таблицы для анализа.
- Чтобы экспортировать данные в один из генераторов автоматизированного анализа, выполните команды экспорта данных на контроллере WAVE и выберите генератор экспорта, соответствующий типу анализа. Кроме того, можно экспортировать файл данных и загрузить его в аналитику Seahorse.
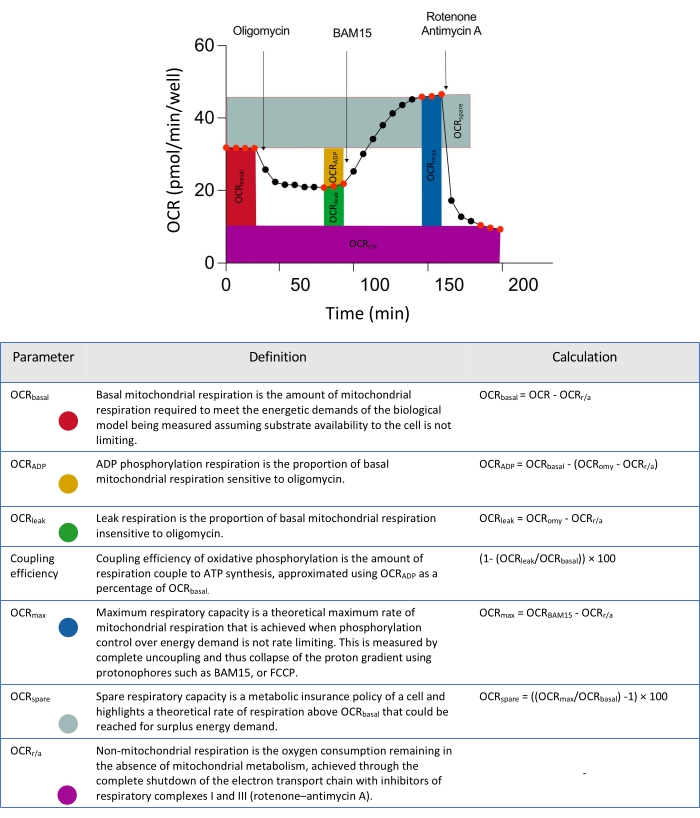
Рисунок 2: Схематические дескрипторы для параметров, полученных в результате анализа данных о внеклеточном потоке. Аббревиатура: OCR = норма потребления кислорода. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Access restricted. Please log in or start a trial to view this content.
Результаты
Для получения хорошо сформированных, компактных сфероидов каждую клеточную линию оптимизировали индивидуально по плотности посева и продолжительности культивирования (рисунок 3). Клеточные линии A549, HepG2/C3A и SK-OV-3 первоначально образовывали рыхлые агрегаты, которые не п...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Основные выводы и результаты
В этой статье представлен подробный протокол для исследования митохондриального энергетического метаболизма отдельных 3D-сфероидов с использованием серии клеточных линий, полученных из рака, с помощью анализатора XFe96 XF. Разработан и описан спос...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
У авторов нет конфликта интересов, о которых можно было бы заявить.
Благодарности
N.J.C был поддержан премией BBSRC MIBTP CASE Award с Sygnature Discovery Ltd (BB/M01116X/1, 1940003)
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| A549 | ECACC | #86012804 | Lung carcinoma cell line |
| Agilent Seahorse XF RPMI Medium, pH 7.4 | Agilent Technologies Inc. | 103576-100 | XF assay medium with 1 mM HEPES, without phenol red, sodium bicarbonate, glucose, L-glutamine, and sodium pyruvate |
| Agilent Seahorse XFe96 Extracellular Flux Analyzer | Agilent Technologies Inc. | - | Instrument for measuring rates of spheroid oxygen uptake in single spheroids |
| Antimycin A | Merck Life Science | A8674 | Mitochondrial respiratory complex III inhibitor |
| BAM15 | TOCRIS bio-techne | 5737 | Mitochondrial protnophore uncoupler |
| Black-walled microplate | Greiner Bio-One | 655076 | For fluorescence-based assays |
| CELLSTAR cell-repellent surface 96 U well microplates | Greiner Bio-One | 650970 | Microplates for generating spheroids |
| CellTiter-Glo 3D Cell Viability Assay | Promega | G9681 | Assay for the determination of cell viability in 3D microtissue spheroids |
| Cultrex Poly-D-Lysine | R&D Systems a biotechne brand | 3439-100-01 | Molecular cell adhesive for coating XFe96 spheroid microplates to facillitate attachment of spheroids |
| D-(+)-Glucose | Merck Life Sciences | G8270 | Supplement for cell culture growth and XF assay medium |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 11885084 | Culture medium for HepG2/C3A spheroids |
| EVOS XL Core Imaging System | Thermo Fisher Scientific | AMEX1000 | Phase-contrast imaging microscope |
| EZ-PCR Mycoplasma test kit | Biological Industries | 20-700-20 | Mycoplasma screening in cell cultures |
| FIJI Is Just Image J | Analysis of collated images | ||
| Foetal bovine serum | Merck Life Science | F7524 | Supplement for cell culture medium |
| HepG2/C3A | ATCC | #CRL-10741 | Hepatic carcinoma cell line, a clonal derivative of the parent HepG2 cell line |
| Lactate-Glo | Promega | J5021 | Assay for measurement of lactate within spheorid culture medium |
| L-glutamine (200 mM solution) | Merk Life Sciences | G7513 | Supplement for cell culture growth and XF assay medium |
| M50 Stereo microscope | Leica Microsytems | LEICAM50 | Stereo dissection micrscope; used for spheorid handling |
| MCF-7 | ECACC | #86012803 | Breast adenocarcinoma cell line |
| Oligomycin from Streptomyces diastatochromogenes | Merck Life Science | O4876 | ATP Synthase Inhibitor |
| Penicilin-Streptomycin | Gibco | 15140122 | Antibiotics added to cell culture medium |
| Quant-iT PicoGreen dsDNA Assay Kit | Initrogen | P7589 | Analysis of dsDNA in spehroids |
| Rotenone | Merck Life Science | R8875 | Mitochondrial Respiratory Complex I Inhibitor |
| RPMI 1640 | Gibco | 21875091 | Culture medium for A549, MCF7, and SK-OV-3 spheroids |
| Seahorse Analytics | Agilent Technologies Inc. | Build 421 | https://seahorseanalytics.agilent.com |
| Seahorse XFe96 Spheroid FluxPak | Agilent Technologies Inc. | 102905-100 | Each Seahorse XFe96 Spheroid FluxPak contains: 6 Seahorse XFe96 Spheroid Microplates (102978-100), 6 XFe96 sensor cartridges, and 1 bottle of Seahorse XF Calibrant Solution 500 mL (100840-000) |
| Serological pipette: 5, 10, and 25 mL | Greiner Bio-One | 606107; 607107; 760107 | Consumables for cell culture |
| SK-OV-3 | ECACC | #HTB-77 | Ovarian adenocarcinoma cell line |
| Sodium pyruvate (100 mM solution) | Merck Life Science | S8636 | Supplement for cell culture growth and XF assay medium |
| T75 cm2 cell culture flask | Greiner Bio-One | 658175 | Tissue culture treated flasks for maintaining cell cultures |
| TrypLExpress | Gibco | 12604-021 | Cell dissociation reagent |
| Wave controller software | Agilent Technologies Inc. | - | |
| Wide orifice tip | STARLAB International GmbH | E1011-8400 | Pipette tips with wide opening for spheroid handling |
Ссылки
- Correa de Sampaio, P., et al. A heterogeneous in vitro three dimensional model of tumour-stroma interactions regulating sprouting angiogenesis. PLoS One. 7 (2), 30753(2012).
- Amann, A., et al. Development of an innovative 3D cell culture system to study tumour-stroma interactions in non-small cell lung cancer cells. PLoS One. 9 (3), 92511(2014).
- Russell, S., Wojtkowiak, J., Neilson, A., Gillies, R. J. Metabolic profiling of healthy and cancerous tissues in 2D and 3D. Scientific Reports. 7 (1), 15285(2017).
- Zanoni, M., et al. 3D tumor spheroid models for in vitro therapeutic screening: a systematic approach to enhance the biological relevance of data obtained. Scientific Reports. 6, 19103(2016).
- Song, Y., et al. Patient-derived multicellular tumor spheroids towards optimized treatment for patients with hepatocellular carcinoma. Journal of Experimental and Clinica Cancer Research. 37 (1), 109(2018).
- Courau, T., et al. Cocultures of human colorectal tumor spheroids with immune cells reveal the therapeutic potential of MICA/B and NKG2A targeting for cancer treatment. Journal for ImmunoTherapy of Cancer. 7 (1), 74(2019).
- Ivanova, E., et al. Use of ex vivo patient-derived tumor organotypic spheroids to identify combination therapies for HER2 mutant non-small cell lung cancer. Clinical Cancer Research. 26 (10), 2393-2403 (2020).
- Mandon, M., Huet, S., Dubreil, E., Fessard, V., Le Hegarat, L. Three-dimensional HepaRG spheroids as a liver model to study human genotoxicity in vitro with the single cell gel electrophoresis assay. Scientific Reports. 9 (1), 10548(2019).
- Stampar, M., et al. Hepatocellular carcinoma (HepG2/C3A) cell-based 3D model for genotoxicity testing of chemicals. Science of the Total Environment. 755, 143255(2020).
- Coltman, N. J., et al. Application of HepG2/C3A liver spheroids as a model system for genotoxicity studies. Toxicology Letters. 345, 34-45 (2021).
- Tchoryk, A., et al. Penetration and uptake of nanoparticles in 3D tumor spheroids. Bioconjugate Chemistry. 30 (5), 1371-1384 (2019).
- Leite, P. E. C., et al. Suitability of 3D human brain spheroid models to distinguish toxic effects of gold and poly-lactic acid nanoparticles to assess biocompatibility for brain drug delivery. Partical Fibre Toxicology. 16 (1), 22(2019).
- Elje, E., et al. Hepato(Geno)toxicity assessment of nanoparticles in a HepG2 liver spheroid model. Nanomaterials. 10 (3), 545(2020).
- Conway, G. E., et al. Adaptation of the in vitro micronucleus assay for genotoxicity testing using 3D liver models supporting longer-term exposure durations. Mutagenesis. 35 (4), 319-330 (2020).
- Wang, Z., et al. HepaRG culture in tethered spheroids as an in vitro three-dimensional model for drug safety screening. Journal of Applied Toxicology. 35 (8), 909-917 (2015).
- Proctor, W. R., et al. Utility of spherical human liver microtissues for prediction of clinical drug-induced liver injury. Archives of Toxicology. 91 (8), 2849-2863 (2017).
- Basharat, A., Rollison, H. E., Williams, D. P., Ivanov, D. P. HepG2 (C3A) spheroids show higher sensitivity compared to HepaRG spheroids for drug-induced liver injury (DILI). Toxicology and Applied Pharmacology. 408, 115279(2020).
- Benning, L., Peintner, A., Finkenzeller, G., Peintner, L. Automated spheroid generation, drug application and efficacy screening using a deep learning classification: a feasibility study. Scientific Reports. 10 (1), 11071(2020).
- Mittler, F., et al. High-content monitoring of drug effects in a 3D spheroid model. Frontiers in Oncology. 7, 293(2017).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. The Biochemical Journal. 435 (2), 297-312 (2011).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Kasianowicz, J., Benz, R., McLaughlin, S. The kinetic mechanism by which CCCP (carbonyl cyanide m-chlorophenylhydrazone) transports protons across membranes. The Journal of Membrane Biology. 82 (2), 179-190 (1984).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2013).
- Mitchell, P. Coupling of phosphorylation to electron and hydrogen transfer by a chemi-osmotic type of mechanism. Nature. 191, 144-148 (1961).
- Alexopoulos, S. J., et al. Mitochondrial uncoupler BAM15 reverses diet-induced obesity and insulin resistance in mice. Nature Communications. 11 (1), 2397(2020).
- Chen, S. -Y., et al. Mitochondrial uncoupler SHC517 reverses obesity in mice without affecting food intake. Metabolism - Clinical and Experimental. 117, 154724(2021).
- Goedeke, L., Shulman, G. I. Therapeutic potential of mitochondrial uncouplers for the treatment of metabolic associated fatty liver disease and NASH. Molecular Metabolism. 46, 101178(2021).
- Hill, B. G., et al. Integration of cellular bioenergetics with mitochondrial quality control and autophagy. Biological chemistry. 393 (12), 1485-1512 (2012).
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795(2019).
- Wang, J., et al. Uncoupling effect of F16 is responsible for its mitochondrial toxicity and anticancer activity. Toxicological Sciences. 161 (2), 431-442 (2018).
- Tretter, L., Chinopoulos, C., Adam-Vizi, V. Plasma membrane depolarization and disturbed Na+ homeostasis induced by the protonophore carbonyl cyanide-p-trifluoromethoxyphenyl-hydrazon in isolated nerve terminals. Molecular Pharmacology. 53 (4), 734-741 (1998).
- Connop, B. P., Thies, R. L., Beyreuther, K., Ida, N., Reiner, P. B. Novel effects of FCCP [carbonyl cyanide p-(trifluoromethoxy) phenylhydrazone] on amyloid precursor protein processing. Journal of neurochemistry. 72 (4), 1457-1465 (1999).
- Stöckl, P., et al. Partial uncoupling of oxidative phosphorylation induces premature senescence in human fibroblasts and yeast mother cells. Free Radical Biology and Medicine. 43 (6), 947-958 (2007).
- Firsov, A. M., et al. Protonophoric action of BAM15 on planar bilayers, liposomes, mitochondria, bacteria and neurons. Bioelectrochemistry. 137, 107673(2021).
- Dranka, B. P., Hill, B. G., Darley-Usmar, V. M. Mitochondrial reserve capacity in endothelial cells: The impact of nitric oxide and reactive oxygen species. Free Radical Biology and Medicine. 48 (7), 905-914 (2010).
- Eilenberger, C., Rothbauer, M., Ehmoser, E. K., Ertl, P., Kupcu, S. Effect of spheroidal age on sorafenib diffusivity and toxicity in a 3D HepG2 spheroid model. Scientific Reports. 9 (1), 4863(2019).
- vanden Brand, D., Veelken, C., Massuger, L., Brock, R. Penetration in 3D tumor spheroids and explants: Adding a further dimension to the structure-activity relationship of cell-penetrating peptides. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1860 (6), 1342-1349 (2018).
- Niora, M., et al. Head-to-head comparison of the penetration efficiency of lipid-based nanoparticles into tumor spheroids. ACS Omega. 5 (33), 21162-21171 (2020).
- Millard, M., et al. Drug delivery to solid tumors: the predictive value of the multicellular tumor spheroid model for nanomedicine screening. International Journal of Nanomedicine. 12, 7993-8007 (2017).
- Ruas, J. S., et al. Underestimation of the maximal capacity of the mitochondrial electron transport system in oligomycin-treated cells. PLoS One. 11 (3), 0150967(2016).
- Benton, G., DeGray, G., Kleinman, H. K., George, J., Arnaoutova, I. In vitro microtumors provide a physiologically predictive tool for breast cancer therapeutic screening. PLoS One. 10 (4), 0123312(2015).
- Hirpara, J., et al. Metabolic reprogramming of oncogene-addicted cancer cells to OXPHOS as a mechanism of drug resistance. Redox Biology. 25, 101076(2019).
- Ware, M. J., et al. Generation of homogenous three-dimensional pancreatic cancer cell spheroids using an improved hanging drop technique. Tissue Engineering. Part C, Methods. 22 (4), 312-321 (2016).
- Song, Y., et al. TGF-β-independent CTGF induction regulates cell adhesion mediated drug resistance by increasing collagen I in HCC. Oncotarget. 8 (13), 21650-21662 (2017).
- Wrzesinski, K., et al. HepG2/C3A 3D spheroids exhibit stable physiological functionality for at least 24 days after recovering from trypsinisation. Toxicology Research. 2 (3), 163-172 (2013).
- Gaskell, H., et al. Characterization of a functional C3A liver spheroid model. Toxicology Research. 5 (4), 1053-1065 (2016).
- Takahashi, Y., et al. 3D spheroid cultures improve the metabolic gene expression profiles of HepaRG cells. Bioscience Reports. 35 (3), 00208(2015).
- Hendriks, D. F. G., Puigvert, L. F., Messner, S., Mortiz, W., Ingelman-Sundberg, M. Hepatic 3D spheroid models for the detection and study of compounds with cholestatic liability. Scientific Reports. 6, 35434(2016).
- Leung, B. M., Lesher-Perez, S. C., Matsuoka, T., Moraes, C., Takayama, S. Media additives to promote spheroid circularity and compactness in hanging drop platform. Biomaterials Science. 3 (2), 336-344 (2015).
- Cavo, M., et al. A synergic approach to enhance long-term culture and manipulation of MiaPaCa-2 pancreatic cancer spheroids. Scientific Reports. 10 (1), 10192(2020).
- Carlsson, J., Yuhas, J. M. Liquid-overlay culture of cellular spheroids. Recent Results in Cancer Research. 95, 1-23 (1984).
- Costa, E. C., Gaspar, V. M., Coutinho, P., Correia, I. J. Optimization of liquid overlay technique to formulate heterogenic 3D co-cultures models. Biotechnology and Bioengineering. 111 (8), 1672-1685 (2014).
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Zhang, X. D., et al. The use of strictly standardized mean difference for hit selection in primary RNA interference high-throughput screening experiments. Journal of Biomolecular Screening. 12 (4), 497-509 (2007).
- Yepez, V. A., et al. OCR-Stats: Robust estimation and statistical testing of mitochondrial respiration activities using Seahorse XF Analyzer. PLoS One. 13 (7), 0199938(2018).
- Silva, L. P., et al. Measurement of DNA concentration as a normalization strategy for metabolomic data from adherent cell lines. Analytical Chemistry. 85 (20), 9536-9542 (2013).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis
Posted by JoVE Editors on 3/11/2022. Citeable Link.
An erratum was issued for: Exploring Mitochondrial Energy Metabolism of Single 3D Microtissue Spheroids using Extracellular Flux Analysis. The Representative Results section was updated.
Figure 5 was updated from:
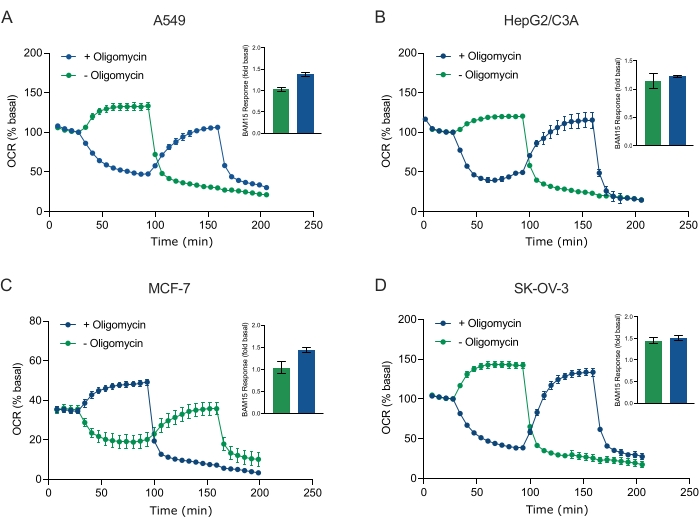
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
to:
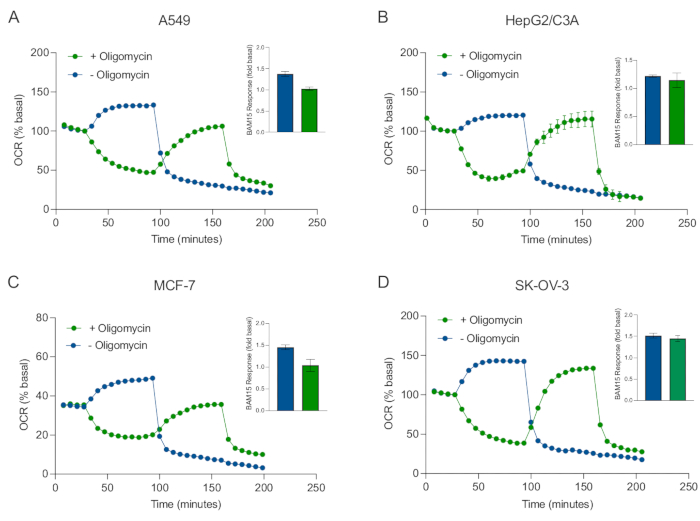
Figure 5: Single or sequential injection of mitochondrial respiratory compounds. Cancer-cell-derived spheroids of MCF-7, HEPG2/C3A, SK-OV-3, and A549 were placed into wells of an XFe96 spheroid microplate in XF RPMI and probed for OCR using the Agilent Seahorse XFe96 analyzer. OCR was measured 5x, after which 2 µg/mL oligomycin (injection Port A: green trace) or 5 µM BAM15 (injection Port A: blue trace or injection port B: green trace) to inhibit the mitochondrial ATP synthase and determine maximal respiratory capacity, respectively. Kinetic OCR data are expressed as % basal (A-D). Maximal respiratory capacity (OCRmax) was calculated as a factor of basal OCR by the equation: OCRmax = OCRBAM15 / OCRbasal. OCRmax was obtained from OCR averages across measurement cycles 8-10 post BAM15 injection with (green bars) and without (blue bars) oligomycin. Data are averages ± SEM from 3-8 individual well replicates across the spheroid assay microplate. Abbreviations: OCR = oxygen consumption rate. Please click here to view a larger version of this figure.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены