Method Article
Высокомасштабируемый подход к проведению экологических исследований самоуправляемых нематод caenorhabditis
В этой статье
Резюме
Этот протокол может быть использован для проведения крупномасштабных экологических обследований самопоглощающихся нематод Caenorhabditis . Основным преимуществом этого метода является эффективная организация и анализ экологических и молекулярных данных, связанных с нематодами, собранными из природы.
Аннотация
Caenorhabditis elegans является одним из основных модельных организмов в биологии, но только недавно исследователи сосредоточились на его естественной экологии. Относительная разреженность информации о C. elegans в ее естественном контексте исходит из проблем, связанных с идентификацией маленькой нематоды в природе. Несмотря на эти проблемы, растущее внимание к экологии C. elegans вызвало множество новой информации о его жизни за пределами лаборатории. Интенсивные поиски C. elegans в природе способствовали открытию многих новых видов Caenorhabditis и показали, что родственные нематоды часто сосуществуют в дикой природе, где они питаются микробным цветением, связанным с гниющим растительным материалом. Идентификация новых видов также показала, что андродиозная система спаривания самцов и самооплодотворяющихся гермафродитов трижды независимо развивалась в пределах Caenorhabditis. Два других самопоглощающихся вида, C. briggsae и C. tropicalis, разделяют экспериментальные преимущества C. elegans и позволили провести сравнительные исследования механистической основы важных признаков, включая самооплодотворение. Несмотря на эти достижения, многое еще предстоит узнать об экологии и природном разнообразии этих важных видов. Например, нам все еще не хватает функциональной информации для многих их генов, которая может быть достигнута только через понимание их естественной экологии. Чтобы облегчить экологические исследования самоуправляемых нематод Caenorhabditis , мы разработали высокомасштабируемый метод сбора нематод из дикой природы. Наш метод использует мобильные платформы сбора данных, облачные базы данных и программную среду R для повышения способности исследователей собирать нематод из дикой природы, записывать связанные экологические данные и идентифицировать диких нематод с помощью молекулярных штрих-кодов.
Введение
Последние два десятилетия принесли повышенный интерес к экологии нематод Caenorhabditis. Из этих исследований мы знаем, что свободно живущие виды Caenorhabditis могут быть выделены из эфемерных микросред обитания как в умеренных, так и в тропических регионах, где они питаются микробным цветением, связанным с разлагающимся растительным материалом, иногда в симпатрии1,2,3,4,5,6,7,8 . Мы также узнали, что конвергентная эволюция самооплодотворения происходила в роде три раза, и самолечение является доминирующим способом размножения для C. briggsae, C. elegans и C. tropicalis9,10. Среди этих селферов C. elegans является одним из наиболее широко изученных животных на Земле и использовался исследователями для достижения критических успехов в биологии. Важно отметить, что другие самостоятельные виды Caenorhabditis разделяют многие экспериментальные преимущества C. elegans и быстро продвигают сравнительные исследования рода. Однако загадочная природа этих нематод в дикой природе затрудняет изучение их экологии и природного разнообразия, что имеет решающее значение для понимания биологических функций их генов и способов, которыми эволюция сформировала генетическое разнообразие среди видов10,11.
Самой большой проблемой для изучения экологии самодельных нематод Caenorhabditis в дикой природе являются их небольшие размеры; взрослые нематоды часто имеют длину 1 мм или меньше. Эта задача требует, чтобы исследователи брали образцы субстратов из дикой природы и пытались отделить интересующих нематод от субстратов в лаборатории без возможности наблюдать за животными в дикой природе. Поскольку даже обученным экспертам трудно отличить самоуправляемых нематод Caenorhabditis от других свободноживущих нематод под микроскопом, нематод обычно удаляют из субстрата, изолируют и оставляют размножаться, прежде чем они будут идентифицированы по идентичности последовательности с использованием установленных молекулярных штрих-кодов3,12,13,14 . Время и усилия, необходимые для обработки каждой нематоды таким образом, представляют собой организационную проблему, поскольку исследователи должны быть в состоянии проследить идентичность каждой нематоды, изолированной в лаборатории, до точного субстрата и связанных с ним экологических данных, отобранных в полевых условиях. Здесь мы описываем пошаговый процесс эффективного сбора и идентификации самоуправляемых нематод Caenorhabditis с поля и достоверно связывают эти изоляты с соответствующими пространственными и экологическими данными в высоком масштабе.
Этот метод сбора увеличивает масштаб и точность экологических обследований за счет использования мобильных платформ сбора данных, облачных баз данных и программной среды R. Fulcrum - это настраиваемая платформа сбора данных, которая работает с большинством мобильных устройств и позволяет пользователям создавать пользовательские приложения для сбора и организации данных на основе местоположения (https://www.fulcrumapp.com). Этот протокол предоставляет подробные инструкции о том, как использовать специализированные приложения для сбора данных для организации пространственно явных экологических данных с поля и точной увязки этих данных с идентификацией нематод, изолированных в лаборатории. Протокол также объясняет, как эффективно идентифицировать самоуправляемых нематод Caenorhabditis с использованием установленных молекулярных штрих-кодов. Данные этих методов могут быть обработаны просто и воспроизводимо с помощью сопутствующего пакета программного обеспечения R easyFulcrum15 для изучения экологии и генетического разнообразия природных популяций Caenorhabditis.
протокол
1. Подготовка коллекции
- Определите место для обследования нематод Caenorhabditis .
ПРИМЕЧАНИЕ: В большинстве регионов с умеренным климатом C. elegans и C. briggsae могут быть легко изолированы от связанных с человеком мест обитания, таких как сельскохозяйственные поля или сельские и городские сады1. В субтропических и тропических регионах C. briggsae, C. elegans и C. tropicalis можно найти в связанных с человеком местах обитания, перечисленных выше, иногда в непосредственной близости друг от друга. Тем не менее, C. elegans, по-видимому, предпочитает более прохладные и сухие места обитания, чем другие виды в тропических местах обитания7,8. Каждый из видов также может быть изолирован от диких мест обитания, которые не связаны с людьми, но эти места обитания отбираются реже. - Создайте проект Fulcrum для организации сбора и изоляции данных с помощью мобильных приложений для сбора данных.
- Создайте учетную запись в Fulcrum онлайн, используя бесплатное образовательное соглашение16. Добавьте приложение Nematode Field Sampling в учетную запись Fulcrum, нажав кнопку ADD APP17.
- Добавьте приложение Nematode Isolation в учетную запись, нажав кнопку ADD APP18.
ПРИМЕЧАНИЕ: Рекомендуется, чтобы каждая поездка в место была организована как его коллекционный проект с использованием соглашения об именовании «YearMonthLocation», например, 2020FebruaryAustralia.
- Добавьте пользователей в учетную запись Fulcrum, чтобы предоставить им доступ к проекту коллекции. Убедитесь, что каждый пользователь загружает мобильное приложение Fulcrum для участия в проекте.
- Распечатайте набор этикеток с QR-кодом для отслеживания коллекций (C-меток) и изоляций нематод (S-меток) с помощью мобильного приложения. Прикрепите этикетки C к пластиковым пакетам с застежкой-молнией, сверните маркированные пакеты в группы по 25 штук и оберните их резинкой для упаковки. Храните комплект S-этикеток для использования в лаборатории.
ПРИМЕЧАНИЕ: На протяжении всего этого протокола коллекции (субстраты из полевых условий) содержатся в пакетах или на пластинах и маркируются С-метками. Изолированные нематоды маркируются S-метками. C-метки используются для идентификации уникальных коллекций, а S-метки используются для идентификации уникальных изолятов нематод. Эти два типа меток используются для установления связи между определенной коллекцией (C-метка) и нематодами, изолированными от этой коллекции (S-метки) в базе данных Fulcrum. Печатайте в два раза больше S-этикеток, чем C-этикеток для проекта коллекции, потому что в среднем две нематоды изолированы в каждой коллекции. При необходимости позже можно распечатать дополнительные S-этикетки. В дополнении представлено 2 500 уникальных C-меток (Дополнительный файл 1) и 5 000 уникальных S-меток (Дополнительный файл 2). - Подготовьте 10 см пластины NGMA для коллекций и 3,5 см пластины NGMA для изоляции нематод. Сделайте одну пластину размером 10 см и не менее двух пластин по 3,5 см на коллекцию21. Эти пластины засеваются штаммом Escherichia coli OP50 в соответствии с установленными протоколами. Храните пластины перед использованием при температуре 4 °C не более 1 месяца.
2. Полевой сбор
ПРИМЕЧАНИЕ: Нематоды Caenorhabditis чаще всего выделяют из гниющего растительного материала, включая фрукты, орехи, семена, стручки, цветы, стебли, растительную подстилку и компост1,5,6,8. Лучшие субстраты гнилые и почти неузнаваемые как плоды или цветы; избегайте слишком сухих или влажных субстратов (рисунок 1). Субстраты наиболее эффективно собираются с поля, работая парами. Человек с бесконтактным инфракрасным термометром выберет подложку для сбора и сбора образца, в то время как его партнер использует приложение Nematode Field Sampling в Fulcrum для записи данных сбора. Пара коллекторов будет повторять этот процесс до тех пор, пока не будет собрано желаемое количество образцов. Список материалов, необходимых для полевых работ, приведен в (Дополнительная таблица 1).
- Откройте мобильное приложение Fulcrum, выберите в раскрывающемся меню пункт Выборка поля нематоды. Нажмите + , чтобы начать новую запись в проекте (рисунок 2A). Сфотографируйте подложку.
- Нажмите на поле в верхнем центре, чтобы выбрать правильный проект коллекции, выполненный на шаге 1.2 (рисунок 2B). Нажмите на поле C-label в нижней части записи коллекции и выберите Сканировать, когда появится приглашение. Отсканируйте штрих-код на сумке для сбора с помощью камеры мобильного устройства, затем нажмите «Готово» в правом верхнем углу экрана.
- Нажмите на поле «Подложка » и выберите тип подложки из раскрывающегося меню. Добавьте заметки о подложке, коснувшись поля «Заметки подложки» и вручную введя заметки.
- Выберите альбомную ориентацию в раскрывающемся меню. Выберите ландшафт, который лучше всего представляет место отбора проб.
- Выберите вид на небо. При выборе вида на небо опишите видимость неба на месте отбора проб (например, вид на полное небо без препятствий с деревьев или других структур = полный).
- Измерьте температуру поверхности подложки с помощью бесконтактного термометра и запишите значение в поле температуры подложки.
ПРИМЕЧАНИЕ: Держите бесконтактный термометр не более чем на расстоянии 14 дюймов от подложки во время регистрации температуры. - Измерьте температуру и влажность окружающей среды с помощью портативного устройства и запишите эти данные в соответствующие поля.
ПРИМЕЧАНИЕ: Убедитесь, что устройство температуры и влажности окружающей среды не находится на удержании. Единица измерения изменится при отпускании кнопки. Держите устройство во внешнем кармане, чтобы избежать нерегулярных показаний. - Сохраните запись в точке опоры, нажав кнопку Сохранить в левом верхнем углу экрана.
- Соберите около столовой ложки субстрата без палочек или других твердых кусочков, перевернув мешок для сбора, чтобы использовать его в качестве «перчатки», чтобы подобрать подложку, а затем запечатайте пакет. Положите бумажное полотенце в пакет, если образец особенно влажный.
ПРИМЕЧАНИЕ: В жарком климате поместите пакеты в мягкие охладители с более прохладными пакетами, чтобы сохранить коллекции прохладными. - После того, как все образцы были собраны за день, очистите оборудование для сбора, выньте батареи из зондов, перезарядите батареи, повторно заморозьте морозильные пакеты. Синхронизируйте данные коллекции Fulcrum, нажав кнопку «Синхронизировать» в левом верхнем углу приложения Nematode Field Sampling .
ПРИМЕЧАНИЕ: Загрузка может занять несколько минут без сильного сотового соединения, поэтому может быть лучше подождать доступа к Wi-Fi. Данные останутся на мобильных устройствах и будут синхронизированы с облаком. - Отправьте образцы в домашнее учреждение, поместив их в ночную коробку для доставки. Сведите к минимуму время, в течение которого образцы подвергаются воздействию температур менее 11 °C или выше 25 °C, путем отправки упаковок в дни транспортировки груза.
ПРИМЕЧАНИЕ: Большинство судоходных предприятий не отправляют грузы ночью в выходные дни в отдаленных местах.
3. Покрытие полевых коллекций в лаборатории
ПРИМЕЧАНИЕ: В этом разделе подробно описывается, как организовать передачу образцов из маркированных пакетов для сбора на маркированные пластины. Образцы могут прибыть из ночной партии или непосредственно с месторождения.
- Получите отправление коллекций и осмотрите на наличие сломанных сумок или других доказательств повреждения. Если мешки сломаны, выбросьте материал и очистите неповрежденные мешки для сбора 70% этанолом; избегайте C-этикетки на пакете с этанолом, так как это обесцвечивает этикетку и затрудняет ее чтение.
- Для каждой сумки с застежкой-молнией обратите внимание на этикетку C на сумке и прикрепите соответствующую C-метку к крышке 10-сантиметровой пластины, обнаруженной бактериями OP50.
ПРИМЕЧАНИЕ: Маркированные 10-сантиметровые пластины для остальной части протокола называются «С-пластинами». Самый простой способ организовать образцы - поместить мешки для сбора на лабораторный стенд с соответствующей C-пластиной сверху (рисунок 3). - Для каждого сбора переложите около одной столовой ложки образца из пакета для сбора на С-пластину с помощью чистой пластиковой ложки. Добавьте образец вокруг бактериального газона в форме полумесяца или кольца, не покрывайте полностью бактериальный газон (рисунок 4).
ПРИМЕЧАНИЕ: Держите столовую ложку в чистоте, помещая ее в стакан с 95% этанолом, когда она не используется. Используйте бумажное полотенце, чтобы высушить столовую ложку перед переносом дополнительных образцов. - Запишите время, в течение которого коллекции были перенесены из мешков для сбора в С-пластины, и держите С-пластины при комнатной температуре (RT) в течение не менее 24 часов, прежде чем пытаться изолировать нематод в разделе 4.
4. Выделение нематод из коллекций
- Откройте приложение Fulcrum на мобильном устройстве и выберите Nematode Isolation в меню приложения (рисунок 5A). Создайте новую запись изоляции, коснувшись значка + в правом нижнем углу (рисунок 5B).
- На экране новой записи изоляции подтвердите правильный проект коллекции, проверив имя проекта, отображаемое в поле в центре верхнего края. Если отображается неправильный проект, коснитесь имени проекта, чтобы переключиться на правильный проект (рисунок 5C).
- Нажмите на кнопку Select под полем C-label , чтобы найти C-метку, связанную с образцом, из которого изолируются нематоды (рисунок 5D). Нажмите на значок поиска , затем нажмите на значок сканирования , чтобы отсканировать QR-код C-метки на C-пластине с камерой устройства. После сканирования QR-кода в поле C-label появится запись C-label .
- Нажмите на значок «Камера» в поле «Фотографии », чтобы открыть камеру устройства, и используйте его, чтобы сфотографировать образец на C-пластине с видимым QR-кодом (рисунок 5E). Нажмите «Готово», чтобы вернуться на экран изоляции.
ПРИМЕЧАНИЕ: Эти фотографии записи изоляции могут быть использованы для изучения конкретных атрибутов подложки в более позднее время. - Используйте рассекающий микроскоп для поиска нематод на С-пластине. Нажмите на поле Черви на образце , чтобы записать присутствие нематод на образце (рисунок 5F). Нажмите «Да », если нематоды присутствуют на С-пластине, и нажмите « Нет », если нематод нет.
- Нажимайте на треки , если присутствуют только следы нематод. Если нематод нет, парафильмируйте С-пластину и утилизируйте ее в контейнер для биологической опасности.
ПРИМЕЧАНИЕ: Переверните С-пластину над мусорным баком биологической опасности и осторожно постучите по задней части пластины, чтобы выбить все отобранные субстраты. Этот шаг облегчает поиск и изоляцию нематод, которые могут находиться под подложкой на С-пластине. - Если нематоды присутствуют, изолируйте до пяти нематод от С-пластины. Чтобы изолировать нематоду, перенесите одну нематоду с С-пластины на S-пластину с помощью платиновой проволочной кирки. Изолируйте здоровых, гравидных взрослых, если это возможно. Тем не менее, изолируйте другие стадии, если взрослые не найдены.
ПРИМЕЧАНИЕ: После изоляции до пяти S-пластин будут иметь по одной нематоде. Держите эти S-пластины с изолированными нематодами из одной и той же C-пластины, организованными вместе в аккуратную стопку вдали от других S-пластин, пока они не будут введены в точку опоры. - Нажмите на поле «Пластины с S-меткой», чтобы ввести S-пластины, используемые для этой изоляции. Нажмите на + в правом нижнем углу. Нажмите на S-метку , а затем нажмите «Сканировать », чтобы открыть камеру устройства. Используйте камеру устройства для сканирования QR-кода S-метки на S-пластине.
ПРИМЕЧАНИЕ: Убедитесь, что код S-метки совпадает с кодом на пластине. Если он совпадает, нажмите «Готово». Если это не так, нажмите «Отмена» и повторно сканируйте, пока он не совпадет, затем нажмите «Готово». Иногда QR-коды близлежащих пластин случайно сканируются. - После ввода каждой S-пластины сохраните запись с помощью кнопки Сохранить в правом верхнем углу. Запись будет утеряна, если она не будет сохранена. Нажмите на + в правом нижнем углу, чтобы добавить больше S-маркированных пластин, если это необходимо, пока не будут введены все нематоды, выделенные из C-пластины. После добавления всех пластин с S-меткой для записи изоляции нажмите кнопку < в левом верхнем углу, чтобы вернуться к экрану записи изоляции.
ПРИМЕЧАНИЕ: Чтобы отменить запись изоляции, поскольку ошибки не могут быть устранены, нажмите кнопку Отмена в левом верхнем углу. На этом шаге откроется диалоговое окно с вопросом, можно ли удалить запись без сохранения. При желании нажмите Да, Отменить. - Нажмите на кнопку «Сохранить» в правом верхнем углу после того, как в запись изоляции будет правильно добавлена вся информация. Затем парафильмируйте S-пластины с изолированными нематодами и откладывайте их в область, предназначенную для хранения S-пластин с нематодами.
- Парафильмируйте С-пластину и выбросьте ее в бункер биологической опасности. Нажмите на значок Синхронизация , чтобы загрузить все данные в Fulcrum.
- Отсортируйте все S-пластины в буквенно-цифровом порядке, затем поместите S-пластины в картонные коробки. Убедитесь, что S-образные пластины опущены вниз и парафильмированы. Сложите до четырех S-пластин в одну позицию в коробке и пометьте картонную коробку названием проекта, датой, временем и уникальным номером коробки.
- Храните маркированные коробки на RT. Эти изоляты будут проверяться на пролиферацию через 48 ч и снова через 168 ч, если это необходимо.
5. Экспорт S-пластин из Fulcrum
ПРИМЕЧАНИЕ: В этом разделе подробно описывается, как экспортировать S-метки, используемые в процессе изоляции, из базы данных проекта Fulcrum. Эти S-метки будут использоваться для отслеживания распространяющихся изофемальных линий, пока они идентифицируются по идентичности последовательности в разделах 6-9.
- Войдите на веб-сайт Fulcrum и выберите приложение Nematode Isolation . Нажмите «Экспортер» в левой части экрана. Щелкните, чтобы выбрать нужный проект, и установите флажок Изоляция нематоды . Нажмите кнопку Далее , чтобы загрузить файл .zip, содержащий файл 'nematode_isolation_s_labeled_plates.csv'.
- Откройте файл 'nematode_isolation_s_labeled_plates.csv' и отсортируйте его по столбцу 'S-label' в порядке возрастания (самая маленькая S-метка будет сверху). Выделите все S-метки и скопируйте их из электронной таблицы.
- Перейдите к шаблону google sheet (wild_isolate_genotyping_template) с помощью веб-браузера19.
- Сделайте копию этого листа Google, щелкнув правой кнопкой мыши на вкладке Шаблон генотипирования , а затем выбрав опцию Копировать в новую электронную таблицу . Выберите Открыть электронную таблицу , чтобы просмотреть новую таблицу Google.
- Назовите этот новый лист именем проекта точки опоры, за которым следует «wild_isolate_genotyping», например, «2020FebruaryAustralia_wild_isolate_genotyping».
ПРИМЕЧАНИЕ: Этот лист называется «листом генотипирования» во всем остальном протоколе.
- Вставьте S-метки, скопированные из столбца 'nematode_isolation_s_labeled_plates.csv' 's_label', в столбец листа генотипирования под названием 's_label'. Проверьте столбец «s_label_repeat_error» на «1». Значение '1' в этом столбце означает, что S-метка дублируется где-то на листе генотипирования. При обнаружении дублирования исследуйте и исправляйте их, прежде чем двигаться дальше.
- Заполните столбец «isolation_box_number» листа генотипирования для всех S-меток.
6. Проверьте наличие распространения на S-пластинах
- Проверьте наличие размножающихся животных на S-пластинах через 48 ч после изоляции (используйте дату и время последней изоляции на коробке с шага 4.11, чтобы определить время).
ПРИМЕЧАНИЕ: Размножающиеся нематоды характеризуются потомством на S-пластине. - Если S-пластина распространяется, введите «1» в столбец proliferation_48 на листе генотипирования, затем переместите S-пластину в коробку с надписью «48-часовое распространение, вставка 1». Поместите максимум 88 S-пластин в ящик для распространения, затем начните заполнять новую коробку с надписью «48-часовое распространение, коробка 2». Убедитесь, что S-метки организованы в буквенно-цифровом порядке в 48-часовых ящиках распространения.
ПРИМЕЧАНИЕ: Не утилизируйте нераспространяющиеся S-пластины; эти пластины будут проверены снова через 168 часов после изоляции. При желании консолидируйте эти S-пластины в числовом порядке в полях с надписью «48 ч нераспространения, коробка X», но не забудьте записать, когда на новом поле должна произойти отметка 168 ч. - После идентификации всех распространяющихся S-меток через 48 ч перейдите к разделу 7 для S-пластин с пролиферацией через 48 ч.
- Проверьте S-пластины, которые не размножались через 48 ч после изоляции, снова через 168 ч после изоляции.
- Если S-пластина в настоящее время распространяется, введите «1» в столбец proliferation_168 на листе генотипирования, а затем переместите S-пластину в коробку с надписью «168-часовое распространение, вставка 1».
- Поместите максимум 88 S-пластин в ящик для распространения, а затем начните заполнять новую коробку с надписью «168 ч распространения, коробка 2». Обязательно организуйте S-метки в буквенно-цифровом порядке в коробках распространения 168 ч.
- Выбросьте S-пластины, которые не имеют распространения через 168 ч. Переходим к разделу 7 для S-пластин с разрастанием через 168 ч.
7. Лизис изофемальных линий
ПРИМЕЧАНИЕ: На этом шаге будет использоваться инструмент фильтрации данных в листах Google, чтобы помочь распечатать листы лизиса для S-пластин в полях распространения. Целью листов лизиса является предоставление персоналу правильных положений для S-меток в лизисных ленточных трубках на стенде.
- Откройте лист генотипирования для нужного проекта и выделите все ячейки, введя Cmd+A. Щелкните Data > Создать фильтр, чтобы добавить кнопку фильтра в заголовок каждого столбца. Используйте кнопки Фильтр для отображения только S-пластин, которые будут генотипизированы. Например, если все S-пластины с разрастанием через 48 ч должны быть лизированы: нажмите кнопку «Фильтр» в столбце «proliferation_48» и выберите «1».
- После того, как лист Google генотипирования был отфильтрован, просмотрите список отображаемых S-меток, чтобы убедиться, что они являются S-метками, которые будут напечатаны на листе.
- В столбце «strip_tube_number» листа Google для генотипирования введите уникальный номер каждые 11 строк.
- Введите номера трубок полосы для проекта в последовательном порядке, начиная с 1 и никогда не дублируя. В поле «strip_tube_position» введите от 2 до 12 для каждого номера трубки полосы.
ПРИМЕЧАНИЕ: Используйте 12-трубные ленточные трубки для лизиса. Первой позицией (strip_tube_position 1) будет контроль, но элементы управления не добавляются на листы лизиса (добавляются только strip_tube_positions, 2-12). Во время лизиса положительный контрольный штамм 'N2' будет добавлен в положение 1 каждой четной ленточной трубки в качестве положительного контроля. Никакие черви не добавляются в положение 1 каждой трубки с нечетной полосой в качестве отрицательного контроля.
- Отфильтруйте лист Google для генотипирования, чтобы включить только S-метки в один ящик для распространения, которые должны быть лизированы, затем выберите столбцы «s_label» через «lysis_notes». Распечатайте лист лизиса для каждой коробки распространения, подлежащей лизированию.
- Нажмите на раскрывающееся меню в поле Печать и выберите Выбранные ячейки. Нажмите «Далее » в правом верхнем углу, затем используйте диалоговое окно, чтобы распечатать лист лизиса для поля распространения.
- Повторите шаги 7.3-7.5, чтобы напечатать лист лизиса для каждой коробки распространения.
ПРИМЕЧАНИЕ: Каждый ящик для пролиферации вмещает до 88 S-пластин, что соответствует восьми 12-луночным ленточным трубам. - Подготовьте 12-луночные ленточные трубки для всех образцов, которые будут лизироваться. Пометьте ленточную трубку уникальным «strip_tube_number», назначенным на листе лизиса. Эта этикетка должна быть написана на полоске колпачка и ленточной трубке, чтобы избежать путаницы, если они разделены. Трубы с полосой EVEN имеют положительный контроль (черви N2) в положении 1. Трубы с полосой ODD имеют отрицательный контроль (без червей) в положении 1.
- Внесите достаточный буфер лизиса (100 мМ KCl, 20 мМ Tris pH 8,2, 5 мМ MgCl2, 0,9% IGEPAL, 0,9% Tween 20, 0,02% желатина с добавлением протеиназы K к конечной концентрации 0,4 мг/мл) для всех образцов и добавьте 5% дополнительно для погрешности пипетки. Масштабируйте по мере необходимости.
ПРИМЕЧАНИЕ: Буфер лизиса лучше всего готовить путем объединения всех ингредиентов, кроме протеиназы К и замораживания в 10-50 мл аликвот при -20 °C. Разморозить аликвоты и держать при температуре 4 °C перед использованием; непосредственно перед употреблением добавляют протеиназу К и тщательно перемешивают. Держите буфер лизиса на льду во время работы. - Расположите S-пластины для конкретной ленточной трубки по порядку, используя печатный лист лизиса в качестве руководства.
- Снимите крышку с одной ленточной трубки и добавьте 8 мкл буфера лизиса к каждому колпачку с помощью повторного пипетка. Добавьте буфер лизиса в одну полоску колпачков за раз, потому что буфер лизиса испарится, если его оставить в RT и открыть. Выберите 3-5 животных из исходных пластин (S-образная пластина или N2 для положительного контроля) в соответствующие положения крышки, указанные на листе лизиса. Запишите заметки для любой S-пластины с менее чем 5 червями, отобранными для лизиса, в lysis_notes разделе рабочего листа лизиса.
- После загрузки нематод в каждое положение ленточной трубки поместите полоску колпачка обратно на ленточную трубку. Сопоставьте отмеченный колпачок (позиция 1) с маркированной трубкой (позиция 1). После закрытия центрифугируйте ленточную трубку ненадолго, пока нематоды не окажутся в нижней части трубки.
- Поместите полоску в морозильную камеру при температуре -80 °C до полного замораживания (не менее 10 мин). Повторяйте шаги с 7.9 по 7.11 до тех пор, пока во все полоски не будут добавлены нематоды для лизиса. Организуйте полоски трубок в числовом порядке.
- Снимите наборы ленточных трубок и запустите программу лизиса в термоциклере: 1 ч при 60 °C, 15 мин при 95 °C, держите при 12 °C. Когда программа лизиса завершена, раскрутите образцы при 300 x g в течение 15 с на RT и храните лизаты при -80 °C в течение 1 недели.
- Организуйте полосы трубок в числовом порядке с помощью держателей пластин с 96 скважинами и включите этикетку с номером коробки распространения, диапазоном номеров трубок полосы, датой и инициалами исследователя. Обновите столбцы листа генотипирования «lysis_date» и «lysis_notes» информацией из листа лизиса.
8. ПЦР последовательностей SSU и ITS2
ПРИМЕЧАНИЕ: В этом разделе содержатся инструкции о том, как выполнить две отдельные ПЦР для каждой лизированной S-пластины. Первый набор праймеров усиливает 500-bp фрагмент 18S рДНК малого субъединичного гена (SSU); oECA1271 = прямая грунтовка TACAATGGAAGGCAGCAGGC, oECA1272 = обратная грунтовка CCTCTGACTTTCGTTCTTGATTAA 12. Эта ПЦР используется для проверки качества шаблонной ДНК. ПЦР усиливает область SSU почти для всех видов нематод. Если ПЦР СГУ не может усилиться, этот результат говорит о том, что качество лизиса плохое и лизис должен быть повторен для этой S-пластины. Второй набор праймеров усиливает 2000-битный фрагмент внутренней транскрибированной спейсерной области между генами рДНК 5,8S и 28S (ITS2); oECA1687 = прямая грунтовка CTGCGTTACTTACCACGAATTGCARAC, oECA202 = обратная грунтовка GCGGTATTTGCTACTACCAYYAMGATCTGC3. Продукт ПЦР ITS2 секвенирован Сэнгером, и последовательность используется для идентификации нематод рода Caenorhabditis до видового уровня по сходству последовательностей.
- Используйте инструмент фильтрации на листе генотипирования, чтобы просмотреть только S-метки, которые будут использоваться для ПЦР.
- Обновите pcr_plate_number и pcr_well столбцы на листе генотипирования. Для предотвращения деградации лизизного материала одновременно проводятся ПКР SSU и ITS2.
- Используйте один и тот же pcr_plate_number для ПЦР ITS2 и SSU, даже если это отдельные реакции в отдельных пластинах. Они будут различаться маркировкой «SSU» или «ITS2».
- Назначают pcr_plate_number до восьми или менее ленточных трубок (по одной ленточной трубке на ряд 96-луночной ПЦР-пластины, расположенной в порядке возрастания, например, с наименьшим номером трубки полосы сверху). Затем назначьте pcr_plate_well на каждую S-метку в ленточных тубах.
ПРИМЕЧАНИЕ: Ленточные трубы расположены в порядке возрастания, причем наименьший номер трубы полосы присваивается строке A, а наибольший номер - строке H. Позиция 1 всех ленточных труб назначена колонке 1. Поэтому ленточной трубке No1, позиция 1 будет присвоена ПЦР-пластине No1, колодец А01. - Маркировка 96-луночной ПЦР-пластины (пластин) для размещения образцов, которые будут использоваться для ПЦР. Пометьте каждую пластину ПЦР следующей информацией: название проекта, тип ПЦР, номер пластины ПЦР и дату ПЦР (например, 2020FebruaryAustralia_SSU_1_20200304). Кроме того, пометьте пластину номерами трубок, которые будут загружены в каждый ряд.
- Извлеките материал лизиса из морозильной камеры при температуре -80 °C и разморозьте ленточные трубки, содержащие материал лизиса, на льду. Пока материал лизиса оттаивает, приготовьте мастер-миксы ITS2 и SSU в отдельных трубках на льду. Рецепты ПЦР SSU и ITS2 приведены в дополнительной таблице 2.
ПРИМЕЧАНИЕ: Подготовьте 100 реакций мастер-смеси ПЦР для каждой 96-луночной пластины, чтобы учесть ошибку пипетирования. Используйте коническую смесь объемом 15 мл или 50 мл для удержания основной смеси, если необходимо использовать большие объемы. - Осторожно вращайте мастер-микс до тех пор, пока Taq не будет распределен по всему миксу. После смешивания аликвота 38 мкл мастер-смеси в соответствующие лунки ПЦР-пластин на льду. Используйте стерильные одноразовые v-образные желоба и 12-луночную многоканальную пипетку для переноса основной смеси на пластины ПЦР.
- Открутите размороженные лизисные ленточные трубки, чтобы удалить материал лизиса из колпачков. Осторожно снимите крышки всех ленточных трубок, которые будут загружены в первую ПЦР-пластину. Используйте малообъемную многоканальную пипетку (либо 12-луночную, либо 8-луночную), чтобы добавить 2 мкл лизата в соответствующую скважину в пластине ПЦР. Аккуратно выпейте лизат вверх и вниз один раз, прежде чем удалить 2 мкл.
ПРИМЕЧАНИЕ: Проверьте советы, чтобы убедиться, что они содержат лизис перед переносом. Не забудьте изменить подсказки между строками или столбцами. - Накройте пластину ПЦР клейкой фольгой ПЦР и используйте ролик для создания плотного уплотнения. После нанесения фольги кратковременно раскрутите пластины ПЦР в центрифуге. Держите пластину на льду до тех пор, пока она не будет готова к работе в термоциклере.
- Запустите PCR с соответствующей программой термоциклера. Подробную информацию о программах ПЦР SSU и ITS2 см. в Дополнительной таблице 2 .
- Повторяйте шаги 8.4-8.8 до тех пор, пока не будут запущены все PCR.
- Пока идут реакции ПЦР, влейте 100 мл 1,5% агарозного геля. Каждый гель будет содержать образцы или одну пластину ПЦР.
- Добавьте 1,5 г агарозы в колбу объемом 500 мл, затем добавьте 100 мл 1x буфера TAE (дополнительная таблица 3) и закрутите для смешивания. Микроволновая печь для растворения и охлаждения геля.
- Как только раствор остынет, добавьте 5 мкл 10 мг/мл раствора бромида этидия и перемешайте до однородной массы. Вылейте раствор в литейный лоток с четырьмя 25-луночными гребнями, чтобы гель мог вместить 96 образцов плюс лестницу для каждого ряда в геле.
ПРИМЕЧАНИЕ: Бромид этидия является мощным мутагеном. При обращении с бромидом этидия используйте лабораторный халат, химически стойкие перчатки и очки химической безопасности.
- Непосредственно перед завершением ПЦР добавьте 6-кратный загрузочный краситель в одноразовый желоб и используйте многоканальную пипетку, чтобы добавить 2 мкл 6-кратного загрузочного красителя в каждую скважину новой 96-луночной ПЦР-пластины. Эта пластина будет использоваться для загрузки образцов в гель. Сделайте достаточно этих пластин, чтобы вместить все образцы.
- Когда ПЦР сделаны, удалите пластины ПЦР и кратковременно центрифугируйте их при 300 х г в течение 15 с на RT. Храните пластины ПЦР на льду, пока продукты ПЦР не будут исчерпаны на геле.
- Чтобы запустить продукты на геле, используйте 12-луночную многоканальную пипетку, чтобы добавить 5 мкл каждого образца в соответствующий колодец 96-луночной пластины, содержащей 2 мкл 6-кратного загрузочного красителя.
- Затем загрузите 6 мкл этой смеси в каждую лунку недавно отлитого геля. Загрузите 6 мкл по 1 КБ плюс лестница в первый колодец каждого ряда геля.
ПРИМЕЧАНИЕ: Чтобы заполнить лунки гелем, может потребоваться вставить ряд А и ряд В из пластины ПЦР в первый ряд геля. Чтобы избежать путаницы, запишите gel_number и gel_position в лист генотипирования для каждого образца ПЦР.
- Поместите новую фольгированную крышку на оставшуюся ПЦР в пластине (пластинах) и храните их при 4 °C. Эти реакционные продукты будут использоваться для секвенирования на этапе 9.
- Запустите продукты ПЦР на гель при 120 В в течение 20 мин. Визуализируйте гель и запишите, какие S-метки дают продукты ITS2 и/или SSU PCR в столбцах «pcr_product_its2» и «prc_product_ssu» листа генотипирования. Отметьте наличие группы с помощью '1'; отметьте '0' для группы без группы.
9. Идентификация нематод с помощью секвенирования И последовательности BLAST Сэнгера
ПРИМЕЧАНИЕ: В этом разделе приведены инструкции по секвенированию ампликонов ITS2 из S-меток, выравниванию этих последовательностей с базой данных Национального центра биотехнологической информации (NCBI) с использованием алгоритма BLAST и анализу результатов BLAST для идентификации нематод на S-пластинах.
- Для каждого образца, который является ITS2-положительным, используйте оставшийся продукт ITS2 PCR для секвенирования Сэнгера с использованием прямой праймера oECA306 (CACTTTCAAGCAACCCGAC). Организуйте, чтобы выходные файлы секвенирования были легко связаны с S-меткой, записав столбцы «sequencing_plate» и «sequencing_well» каждой S-метки в листе генотипирования.
- Получите выходные файлы .seq для каждой S-метки с платформы виртуализации. Расположите SEQ-файлы для проекта в одном каталоге с SEQ-файлами для каждого пакета виртуализации, расположенными в подкаталогах.
- Откройте средство интерфейса командной строки и перейдите в верхний каталог, содержащий SEQ-файлы, введя команду: cd . Если он еще не существует, создайте объединенную FASTA для всех файлов .seq, введя следующую команду: для dir в */; do cd $dir; для файла в *.seq; do echo ">"$file; cat $file; done >>.. /all_seqs.фа; кд..; сделано.
ПРИМЕЧАНИЕ: Этот код создаст объединенный файл FASTA с именем 'all_seqs.fa' из всех файлов .seq в каталоге проекта. Этот файл может быть использован в онлайн-инструменте NCBI для быстрого выравнивания последовательности ITS2 каждой S-метки по базе данных последовательностей NCBI. - В веб-браузере перейдите на веб-сайт NBCI BLAST20 и нажмите кнопку Выбрать файл . Выберите только что созданный файл all_seqs.fa, затем нажмите кнопку Несколько похожих последовательностей (BLASTn).. Нажмите кнопку BLAST в нижней части страницы, чтобы начать поиск BLAST.
- Обновите лист генотипирования результатами BLAST для каждой S-метки. Используйте инструмент фильтра, чтобы упростить обновление листа Google для генотипирования. Щелкните Данные > Создать фильтр , чтобы добавить кнопку фильтра в заголовок каждого столбца. Отфильтруйте столбец sequencing_plate, чтобы выбрать пластины последовательности, которые должны быть обновлены с помощью результатов BLAST.
- Используйте раскрывающееся меню на странице результатов NCBI BLAST, чтобы проверить результаты для каждой последовательности ITS2 S-пластины (рисунок 6).
- Проверьте, нет ли попаданий BLAST. Идентификатор последовательности в раскрывающемся списке с префиксом * не имеет поражений взрыва. Для этих S-меток введите «no hit < current date>» в столбце manual_blast_notes листа генотипирования.
- Проверьте возможный новый вид Caenorhabditis . Нажмите на ссылку в верхней части хита, чтобы визуализировать выравнивание (рисунок 6). Если верхним попаданием является (1) вид Caenorhabditis , (2) выравнивание содержит более пяти несоответствий в центре последовательности, и (3) охват запросов превышает 50%, этот результат предполагает, что изолят может быть новым видом Caenorhabditis (рисунок 7). Для этих S-пластин входят виды верхнего BLAST, попавшего в столбец «species_id», введите 1 в столбце «possible_new_caeno_sp» и «возможный новый Caeno sp.» в столбец «manual_blast_notes» вместе с процентной идентичностью (например, «возможная новая идентичность Caeno sp. 89%).
- Для последовательностей S-пластин, которые ВЗРЫВАЮТся к виду Caenorhabditis , введите полный род и название вида верхнего хита BLAST в столбце «species_id». Например, «Caenorhabditis elegans».
- Для последовательностей S-пластин, которые ВЗРЫВАЮТся к виду, не относящемуся к Caenorhabditis , введите только род верхнего удара BLAST, за которым следует «sp» в столбце «species_id». Эта нотация означает, что изолят является неизвестным видом в пределах названного рода. Например, 'Oscheius sp.'.
ПРИМЕЧАНИЕ: Последовательность ITS2 не может быть использована для надежной идентификации изолятов до видового уровня за пределами рода Caenorhabditis3,13. - Введите 1 в столбец «make_strain_name» листа генотипирования, если «species_id» = «Caenorhabditis elegans», «Caenorhabditis briggsae» или «Caenorhabditis tropicalis», ИЛИ «possible_new_caeno_sp» = 1.
- Назовите штаммы уникальными названиями в соответствии с номенклатурными соглашениями Caenorhabditis , то есть уникальным лабораторным обозначением, состоящим из 2-3 прописных букв, за которыми следует число для каждого уникального штамма23. Введите имена штаммов в столбец 'strain_name'.
- После того, как штаммы названы, они могут быть криоконсервированы с использованием установленных протоколов 24.
10. Обработка данных коллекции с помощью пакета easyFulcrum в R
ПРИМЕЧАНИЕ: На этом шаге описывается, как связать данные сбора (C-метки) и данные изоляции нематод (S-метки) вместе с помощью пакета easyFulcrum R. Программное обеспечение содержит функции, которые будут далее объединять данные Fulcrum с данными генотипирования из листа генотипирования, так что идентификаторы видов S-меток и названия штаммов организованы в единый фрейм данных.
- Создайте новый каталог с именем для проекта коллекции. Расположите структуру папок в каталоге в соответствии с требованиями, описанными в пакете R easyFulcrum15.
- Перейдите на веб-сайт Fulcrum и войдите в систему. Экспортируйте необработанные данные проекта из базы данных Fulcrum с помощью инструмента экспорта данных веб-сайта Fulcrum слева и установите следующие флажки: проект, включить фотографии, включить данные GPS, выборку полей и изоляцию.
ПРИМЕЧАНИЕ: Данные Fulcrum для проекта будут экспортированы в виде пяти файлов с разделителями-запятыми (.csv). Полные данные проекта будут объединены в единый фрейм данных с помощью пакета easyFulcrum в R. - Переместите пять .csv файлов, экспортированных из Fulcrum, в каталог проекта, созданный на шаге 10.1, как указано в vignette easyFulcrum21.
- Откройте сеанс Rstudio и установите пакет easyfulcrum в R, введя следующие команды в консоли R 'install.packages("devtools")' и 'devtools::install_github("AndersenLab/easyfulcrum")'.
- Откройте новый сценарий R и следуйте инструкциям в виньетке easyfulcrum для обработки данных коллекции21.
Результаты
Этот протокол использовался для сбора нематод Caenorhabditis из нескольких мест, включая Гавайи и Калифорнию. Уровень успешности изоляции для нематод Caenorhabditis варьируется в зависимости от места сбора, климата, опыта отбора проб и типов субстратов. Протокол был использован для широкого отбора проб на Гавайских островах, где в течение нескольких лет и сезонов было проведено девять проектов по сбору. Показатели успешности изоляции для самостоятельных видов Caenorhabditis почти идентичны для C. briggsae (162 из 4 506 образцов, 3,6%) и C. elegans (163 из 4 506 образцов, 3,6%) и намного ниже для C. tropicalis (26 из 4 506 образцов, 0,58%)8. Каждый из самоплавающихся видов обогащается гниющими плодовыми и цветочными субстратами относительно других категорий субстратов. Возьмите образцы гниющих фруктов и цветочных субстратов, если исследователь пытается максимизировать вероятность успеха, а не охарактеризовать предпочтения субстрата. Однако показатель успешности варьируется в зависимости от качества выбранной подложки. Например, среди фруктовых и цветочных субстратов те субстраты, которые слишком сухие, влажные или свежие, скорее всего, не дадут нематод Caenorhabditis .
Масштабируемость этого протокола сбора данных очевидна из количества коллекций, которые одна пара исследователей может собрать из дикой природы. Например, в октябре 2018 года пара исследователей, использующих этот протокол сбора, смогла собрать в общей сложности более 1000 образцов за 7 дней из нескольких мест на двух Гавайских островах. Эта полевая команда отправила образцы в течение ночи в лабораторию, где команда из восьми исследователей выделила более 2000 нематод из образцов по мере их прибытия. Ключевым преимуществом этого протокола является то, что он сводит к минимуму затраты, связанные с отбором проб в отдаленных местах, за счет сокращения оборудования и персонала, необходимых на местах. Используя этот протокол, небольшая полевая команда может сосредоточиться на отборе проб, в то время как изоляционная команда может обрабатывать образцы в своем домашнем учреждении, используя хрупкое и тяжелое оборудование, такое как рассекающие микроскопы и агаровые пластины для выделения нематод. Кроме того, внедрение мобильного приложения для сбора данных позволяет напрямую связывать все полевые данные, связанные с образцами, с C-меткой, что позволяет группе изоляции работать независимо от полевой команды при обработке образцов.
Исследователи, использующие этот протокол сбора, должны учитывать усилия, необходимые для изоляции нематод до начала проекта сбора. Этапы изоляции и идентификации ограничивают скорость, и небольшая команда сбора может быстро перегрузить изоляторы образцами. Кроме того, лабораторное пространство, необходимое для обработки многих коллекций, может помешать текущим исследованиям (рисунок 3). Кроме того, некоторые изолированные нематоды требуют дополнительных усилий для генотипирования. Например, примерно 2% изолятов не могут амплироваться с помощью набора праймеров SSU PCR после первой попытки лизиса и должны быть повторно лизированы, чтобы гарантировать, что материал лизиса пригоден для амплификации с помощью набора праймеров ITS2 (рисунок 8). Кроме того, примерно 3% изолятов не могут получить качественные последовательности после начального раунда секвенирования Сэнгера. Для этих изолятов часто требуется еще один раунд лизиса, ПЦР ITS2 и секвенирование Сэнгера, что может увеличить время обработки для команды изоляции. Важно отметить, что идентичность последовательности сама по себе не является достаточным доказательством для оправдания нового вида Caenorhabditis (рисунок 7). Чтобы должным образом оправдать выращивание изолята в качестве нового вида Caenorhabditis , необходимо приложить дополнительные усилия для проведения экспериментов по спариванию и создания типизированного экземпляра13. Формальное морфологическое описание типизированного образца также является предпочтительным, но не обязательным3. Вместе эти соображения предполагают, что исследователи, принимающие этот протокол сбора, выиграют от пробных тестов шагов изоляции и идентификации, чтобы обеспечить правильное распределение ресурсов до начала проекта сбора. Важно отметить, что даже небольшие проекты по сбору данных могут извлечь выгоду из этого протокола, поскольку процесс обладает высокой воспроизводимостью, и данные могут быть легко проверены для целей контроля качества в лабораторных группах.

Рисунок 1: Примеры субстрата. (А) Идеальный гниющий плод показан в центре изображения (1), плод почти неузнаваем. Менее гнилые плоды показаны рядом; избегайте дегустации свежевыпавших фруктов (2). (B) Идеально разложившийся цветок показан вверху (3). Избегайте отбора проб свежевыпавших цветов (4). (C) Темные листья под верхним слоем сухих листьев идеально подходят при отборе проб для самостоятельных нематод Caenorhabditis (5). Избегайте отбора проб сухого опавшего листа (6). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 2: Мобильное приложение Nematode Field Sampling. (A) Начальный экран после открытия приложения Nematode Field Sampling на устройстве Apple в Fulcrum. Красная стрелка в правом нижнем углу указывает на кнопку + , используемую для создания новой записи коллекции. (B) Пример новой записи коллекции, показанной на устройстве Apple. Красная стрелка указывает на поле «Проект» в верхней части экрана записи коллекции. Обязательно выберите правильный проект при выборке в поле. Поле проекта по умолчанию будет последним проектом, используемым при создании последующих записей коллекции. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 3: Коллекционные мешки и коллекционные пластины, организованные до выкладки образцов. На этом рисунке показаны образцы в пакетах для коллекций с маркировкой C слева. Каждая сумка для коллекций имеет соответствующую 10-сантиметровую пластину с маркировкой C. Справа расположены 10 см коллекционные пластины, которые содержат образец материала после того, как он был перенесен из коллекционных пакетов. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 4: Коллекционная пластина (С-пластина) с правильно перенесенным образцом. 10 см С-пластина с разлагающимися плодами, помещенная на край бактериального газона. C-метка крепится к крышке пластины. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
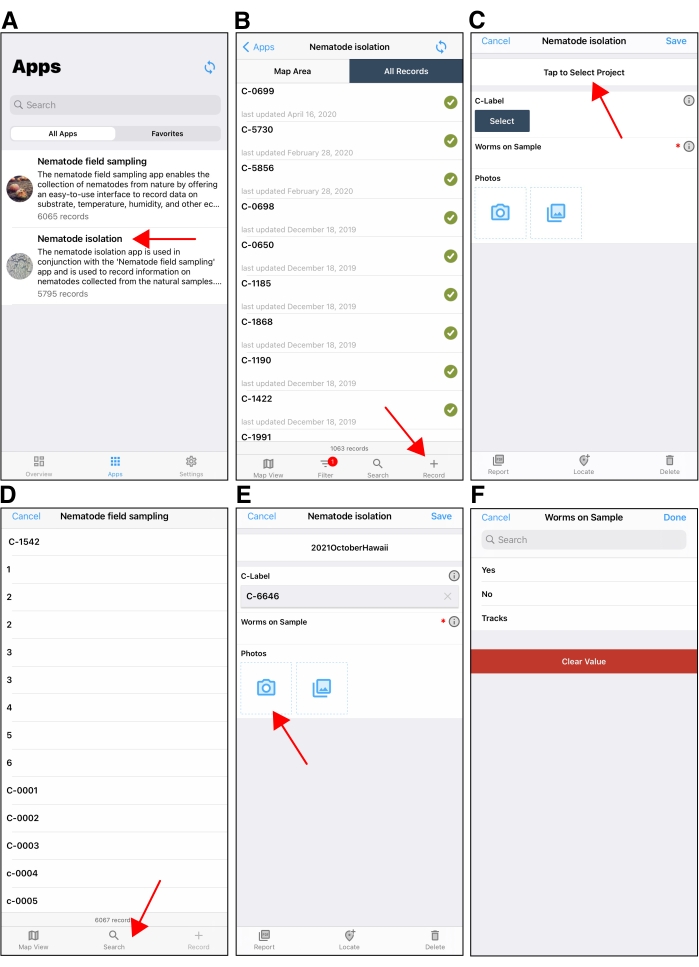
Рисунок 5: Мобильное приложение изоляции Nematode. (A) Экран выбора приложения в мобильном приложении Fulcrum. Красная стрелка указывает на приложение «Изоляция нематод ». (B) Начальный экран после открытия приложения Nematode Isolation на устройстве Apple в точке опоры. Красная стрелка в правом нижнем углу указывает на кнопку + , используемую для создания новой записи изоляции. (C) Пример новой записи изоляции, показанной на устройстве Apple. Красная стрелка указывает на поле «Проект» в верхней части экрана записи изоляции. Обязательно выберите правильный проект при изоляции. Поле проекта по умолчанию будет использоваться для последнего проекта, используемого при создании последующих записей изоляции. (D) После нажатия на поле «Выбрать » под C-меткой пользователи нажимают кнопку поиска (красная стрелка), чтобы найти C-метку, из которой они изолируют нематод. (E) После выбора C-метки пользователи будут фотографировать C-пластину с помощью камеры устройства. (F) Затем пользователи вводят, есть ли нематоды на С-пластине или нет. S-метки добавляются к записи изоляции, если есть нематоды, подлежащие изоляции. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
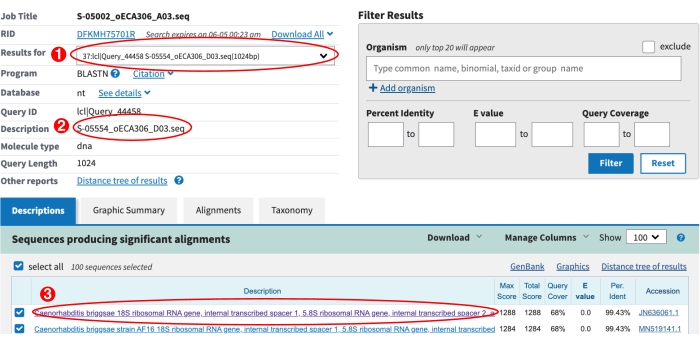
Рисунок 6: Страница результатов NCBI BLAST. (1) Раскрывающееся меню, используемое для просмотра результатов BLAST для всех последовательностей. (2) Описание текущей последовательности, выбранной из раскрывающегося списка. В этом случае показаны результаты для S-метки S-05554. (3) Показан верхний удар BLAST для S-05554. Фиолетовый текст указывает на то, что ссылка для визуализации этого выравнивания была щелкнута. Пожалуйста, не забудьте осмотреть выравнивания на глаз, чтобы определить возможные новые виды Caenorhabditis , см. шаг 9.8 выше. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.

Рисунок 7: Примеры визуализации выравнивания NCBI BLAST. (A) Пример последовательности запросов ITS2 изолята, выровненной по последовательности субъекта C. kamaaina . (1) Процент идентичности выравнивания (89%), который является низким для верхнего удара BLAST. (2) Несоответствие между запросом и последовательностью субъекта (от G до A). (3) Зазор в четыре пары оснований в предметной последовательности, сделанный алгоритмом выравнивания; пробелы в запросе или теме указывают на плохое выравнивание. (4) Обобщенная область в центре выравнивания со многими несоответствиями и пробелами. Регион, подобный этому, предполагает, что последовательность запросов может исходить от нового вида Caenorhabditis . Показан фактический пример выравнивания нового вида, C. oiwi, который был обнаружен в 2017 году. (B) Пример хорошего выравнивания между последовательностью запросов ITS2 изолята и последовательностью субъекта. (5) Процент идентичности выравнивания (99%), что обычно означает, что последовательность запросов происходит от изолята того же вида, что и субъект. (6) Центральная область выравнивания с совершенной идентичностью. Регион, подобный этому, предполагает, что изолят запроса, вероятно, является тем же видом, что и субъект. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
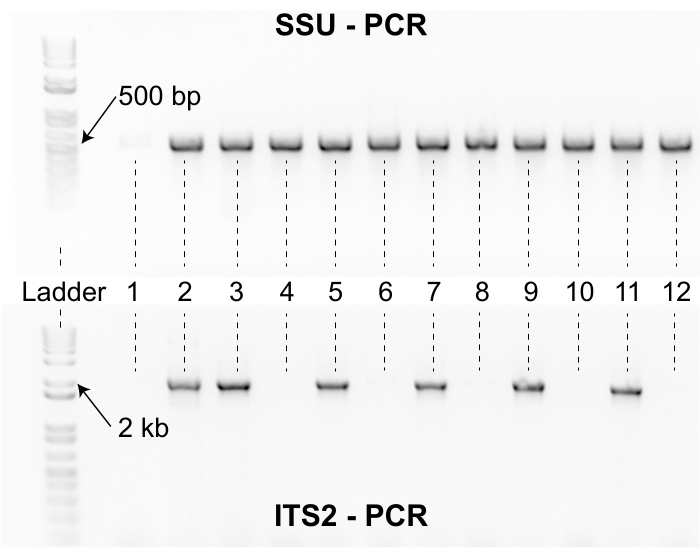
Рисунок 8: Продукты SSU и ITS2 PCR. Верхний гель показывает продукты ПЦР, полученные с помощью праймера SSU для 12 репрезентативных образцов. Лестница ДНК включена слева в качестве ссылки. Продукты SSU PCR для нематод Caenorhabditis имеют длину около 500 bp. Образцы 2-12 усилены набором праймеров СБУ, но образец один не был. Отсутствие ампликона SSU 500 bp для образца говорит о том, что материал лизиса был низкого качества и образец должен быть повторно лизирован. Нижний гель показывает продукты ПЦР, полученные с помощью праймера ITS2 для тех же 12 образцов, показанных в верхнем геле. Лестница и образцы находятся в одинаковой ориентации для обоих гелей. Шесть из 12 образцов не усиливались с помощью набора праймеров ITS2. Образцы с полосами SSU и ITS2 секвенированы Сэнгером и идентифицированы по сходству последовательностей с использованием алгоритма NCBI BLAST. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный файл 1: C-метки. PDF-файл, содержащий 2500 уникальных C-меток. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: S-метки. PDF-файл, содержащий 5000 уникальных S-меток. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительная таблица 1: Полевые материалы. Упаковочный лист материалов, используемых в полевых условиях для отбора проб нематод. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 2: Рецепты ПЦР и условия термоциклера. Таблица рецептов ПЦР и термоциклерных условий для ПЦР ITS2 и SSU. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица 3: Рецепты буфера электрофореза. Рецепт 0,5 М рН 8,0 раствора этилендиаминтетрауксусной кислоты (ЭДТА) и буферного раствора TRIS-ацетат-ЭДТА (TAE). Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Обсуждение
Этот протокол содержит критические шаги, которые должны выполняться с осторожностью. Например, важно, чтобы полевые и изоляционные группы тщательно выбирали правильный проект сбора в заявке до сбора образцов с поля или выделения нематод из образцов в лаборатории. В случае, если выбран неправильный проект сбора, ошибочные записи данных лучше всего исправить в базе данных Fulcrum с помощью онлайн-инструментов редактирования записей. Этот процесс может быть утомительным для многих неуместных записей. Однако база данных сохраняет любые изменения в записях, чтобы можно было провести полный аудит записей сбора и изоляции. Другие критические шаги в этом протоколе включают обработку образцов с поля и нематод, выделенных из этих образцов. Для обеспечения того, чтобы нематоды Caenorhabditis выдержали этапы отбора проб и отгрузки, температура образцов должна поддерживаться в диапазоне от 4 °C до 25 °C. Температура выше 25 °C может вызвать стерильность c. elegans14. Убедитесь, что образцы переносятся из коллекционных пакетов на коллекционные пластины в течение пяти дней, когда это возможно, чтобы свести к минимуму потери для нематод. После того, как нематоды изолированы, очень важно, чтобы они генотипировались и криоконсервировались, прежде чем они погибнут. Трудно найти живых нематод на S-пластинах, которым более двух-трех недель, потому что грибковое и бактериальное загрязнение может сделать S-пластины негостеприимными.
Этот протокол может быть легко изменен для размещения различных типов данных, которые исследователи могут захотеть собрать, находясь в полевых условиях. Например, легко настроить приложение «Выборка поля Нематоды» с новыми полями ввода данных, используя онлайн-графический интерфейс Fulcrum для редактирования приложения. Кроме того, пакет анализа данных, easyFulcrum, может учитывать эти изменения при обработке новых данных15. Еще одна модификация, которую пользователи могут счесть привлекательной, заключается в использовании другого метода выборки на местах. Вместо того, чтобы отбирать дискретные субстраты, исследователи могут захотеть отобрать большие области, содержащие несколько типов субстратов. Эти более крупные образцы лучше всего обрабатывать в лаборатории с использованием методов экстракции воронки или лотка Baermann13. Важно отметить, что использование C-меток и S-меток по-прежнему применимо к этим методам и, следовательно, совместимо с мобильными приложениями.
Основные ограничения этого протокола связаны со временем обработки нематод до выделения в лаборатории. Во-первых, время задержки между сбором проб и выделением нематод делает невозможным регистрацию стадий развития нематод на данном образце во время сбора. Во-вторых, частота самцов и ауткроссинг в природе являются ключевыми эволюционными вопросами для самопоглощающихся нематод Caenorhabditis10. Этот метод не очень хорошо подходит для решения этих вопросов, потому что нематоды, вероятно, прошли через несколько поколений до изоляции. Отсроченная изоляция означает, что прямые доказательства мужской частоты в природе невозможны. Кроме того, задержка нескольких поколений во время этапов генотипирования означает, что геномные доказательства ауткроссинга (гетерозиготности) будут разрушены до того, как штамм нематоды может быть секвенирован. Для выявления гетерозиготности в природе для секвенирования используют потомство, произведенное непосредственно изолированной от природы нематодой2. Другим потенциальным ограничением этого протокола является то, что он предвзято относится к идентификации самопоглощающегося канорхабдита. Это связано с тем, что изолированные нематоды самодовольных видов имеют больше шансов на размножение, чем облигатные ауткроссеры, которые будут размножаться только в том случае, если оплодотворенная самка изолирована.
Этот метод сбора основан на существующих протоколах сбора13,14. Основным достижением этого метода является использование мобильных технологий и специализированного программного обеспечения для облегчения организации огромных объемов экологических и молекулярных данных, связанных с крупномасштабными проектами сбора. Экологические данные, полученные с использованием этого протокола сбора, могут быть использованы для решения нерешенных вопросов для естественных популяций видов Caenorhabditis. Например, данные, полученные с помощью этого метода, были использованы для выявления нишевых предпочтений для видов на Гавайских островах. Более того, секвенируя геномы криоконсервированных нематод, исследователи могут исследовать, как паттерны генетической изменчивости коррелируют с экологическими данными. Исследования такого рода могут выявить признаки местной адаптации в популяциях Caenorhabditis и дать важное представление об актуальности генетических вариаций в естественных контекстах8. Чтобы получить функциональное понимание многих генов у нематод Caenorhabditis, вероятно, потребуются экологические исследования11. Даже для C. elegans большая часть генов не имеет функциональных аннотаций, несмотря на то, что это первое многоклеточное животное, которое было секвенировано, и одно из наиболее тщательно изученных животных на Земле. Этот протокол сбора был разработан, чтобы помочь устранить этот пробел в знаниях, способствуя сбору диких нематод Caenorhabditis и изучению их экологии и природного генетического разнообразия.
Раскрытие информации
Авторы сообщают об отсутствии конфликта интересов.
Благодарности
Это исследование было поддержано стартовыми фондами Северо-Западного университета и премией Национального научного фонда CAREER Award (IOS-1751035), присужденной E.C.A.
Материалы
| Name | Company | Catalog Number | Comments |
| Adhesive labels | Avery | 61533 | Printing the C-labels and S-labels |
| Agarose | Thomas Scientific | C748D75 | agarose gel preparation |
| Backpack | A backpack for each team member to hold equipment, samples, food, and water for the day. | ||
| Cardboard boxes | Fisher Scientific | AS261 | Storage of S-plates |
| Centrifuge | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Collection bags | Zip-lock | NA | Collection of substrates |
| Digital temperature/humidity meter | Preciva | HT-86 | Measurement of ambient temperature and humidity |
| Disecting scope | NA | NA | Nematode isolation, lysis |
| Disposible reservoirs | USA Scientific | 1306-1010 | Aliquoting PCR master mixes and loading dye |
| DNA ladder, 1 KB plus | Thermo Scientific | SM0243 | Visualizing SSU and ITS2 PCR products |
| dNTPs | Thomas Scientific | CB4430-2 | PCR master mix component |
| easyFulcrum R package | NA | NA | An R package for processing collection data http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html |
| EDTA solution (0.5 M pH 8.0) | NA | NA | See supplemental table 3 for recipe |
| Ethanol (95%) | NA | NA | Plating out samples, spoon sterilization |
| Ethidium bromide solution (10 mg/mL) | VWR | 97064-602 | Visualizing SSU and ITS2 PCR products |
| External battery charger for mobile device | NA | NA | External battery charger and charging cable for iPhone or Android |
| Flask (500 mL) | Fisher Scientific | FB501500 | agarose gel preparation |
| Food | NA | NA | Pack accordingly |
| Gel electrophoresis apparatus | Thermo Scientific | B2 | Visualizing SSU and ITS2 PCR products |
| Gel electrophoresis power supply | BioRad | 1645050 | Visualizing SSU and ITS2 PCR products |
| Gel loading dye, 6x | NEB | B7022S | Visualizing SSU and ITS2 PCR products |
| ITS2 primer set | NA | NA | oECA1687 = forward primer CTGCGTTACTTACCACGAATTG CARAC, oECA202 = reverse primer GCGGTATTTGCTACTACCAYYAM GATCTGC |
| ITS2 sequencing primer | NA | NA | oECA306 = forward primer CACTTTCAAGCAACCCGAC |
| Lysis buffer | NA | NA | Nematode lysis, see protocol for recipe |
| Microwave | NA | NA | agarose gel preparation |
| Mobile device | NA | NA | Charged phone in airplane mode. The Fulcrum app GPS positions are inaccurate compared to the GPS positions extracted from pictures. This setting ensures that you use less power and get more precise GPS measurements. |
| NGMA plates, 10 cm | Fisher Scientific | FB0875713 | C-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| NGMA plates, 3.5 cm | Greiner Bio-One | 5662-7102Q | S-plates, see Andersen et al. 2014 for NGMA plate protocol. |
| Non-contact infrared thermometer | Etekcity | B00837ZGRY | Measurement of substrate temperature |
| Paper towels | NA | NA | Paper towels for absorbing excess moisture in a bagged sample. |
| Parafilm | Fisher Scientific | 13-374-12 | Plate sealing |
| PCR adhesive foil | VWR | 60941-126 | PCR adhesive foil |
| PCR buffer (10x) | Thomas Scientific | CB3702-7 | PCR master mix component |
| PCR plates (96-well) | Thomas Scientific | 1149K04 | SSU and ITS2 PCRs |
| Pencil | NA | NA | |
| Plastic spoons | NA | NA | Plating out samples |
| Platinum pick | NA | NA | Nematode isolation, lysis |
| Pre-labeled ziplock collection bags | NA | NA | Bundles of zip-lock plastic bags pre-labelled with C-label barcodes. Each bundle should contain 25 barcoded bags wrapped with a rubber band. |
| Proteinase K | Sigma | 3115879001 | Nematode lysis, added to lysis buffer just prior to use |
| R software | NA | NA | R software: A language and environment for statistical computing https://www.R-project.org/ |
| Soft cooler bag and ice pack | NA | NA | Collection coolers and cooler packs to keep samples cool when ambient temperature is above 25 °C. |
| Spare batteries | NA | NA | Extra batteries for sampling equipment |
| SSU primer set | NA | NA | oECA1271 = forward primer TACAATGGAAGGCAGCAGGC, oECA1272 = reverse primer CCTCTGACTTTCGTTCTTGATTAA |
| Strip tube caps (12-well) | Thomas Scientific | 1159V29 | Nematode lysis |
| Strip tubes (12-well) | Thomas Scientific | 1159V31 | Nematode lysis |
| TAE buffer (1x) | NA | NA | Visualizing SSU and ITS2 PCR products. See supplemental table 3 for recipe |
| Taq polymerase | Thomas Scientific | C775Y45 | PCR master mix component |
| Thermocycler | NA | NA | Neamtode lysis, SSU and ITS2 PCR |
| Water | NA | NA | Pack accordingly |
Ссылки
- Frézal, L., Félix, M. -A. C. elegans outside the Petri dish. eLife. 4, 05849(2015).
- Sivasundar, A., Hey, J. Sampling from natural populations with RNAI reveals high outcrossing and population structure in Caenorhabditis elegans. Current Biology: CB. 15 (17), 1598-1602 (2005).
- Kiontke, K. C., et al. A phylogeny and molecular barcodes for Caenorhabditis, with numerous new species from rotting fruits. BMC Evolutionary Biology. 11, 339(2011).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Richaud, A., Zhang, G., Lee, D., Lee, J., Félix, M. -A. The local coexistence pattern of selfing genotypes in Caenorhabditis elegans natural metapopulations. Genetics. 208 (2), 807-821 (2018).
- Ferrari, C., Salle, R., Callemeyn-Torre, N., Jovelin, R., Cutter, A. D., Braendle, C. Ephemeral-habitat colonization and neotropical species richness of Caenorhabditis nematodes. BMC Ecology. 17 (1), 43(2017).
- Crombie, T. A., et al. Deep sampling of Hawaiian Caenorhabditis elegans reveals high genetic diversity and admixture with global populations. eLife. 8, 50465(2019).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. bioRxiv. , (2021).
- Noble, L. M., et al. Selfing is the safest sex for Caenorhabditis tropicalis. eLife. 10, 62587(2021).
- Cutter, A. D., Morran, L. T., Phillips, P. C. Males, Outcrossing, and Sexual Selection in Caenorhabditis Nematodes. Genetics. 213 (1), 27-57 (2019).
- Petersen, C., Dirksen, P., Schulenburg, H. Why we need more ecology for genetic models such as C. elegans. Trends in Genetics: TIG. 31 (3), 120-127 (2015).
- Haber, M., Schüngel, M., Putz, A., Müller, S., Hasert, B., Schulenburg, H. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2005).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and Related Nematodes. WormBook. , https://www.ncbi.nlm.nih.gov/books/NBK19764/ (2018).
- Poullet, N., Braendle, C. Sampling and isolation of C. elegans from the natural habitat. Methods in Molecular Biology. 1327, 221-229 (2015).
- Di Bernardo, M., Crombie, T. A., Cook, D. E., Andersen, E. C. easyFulcrum: An R package to process and analyze ecological sampling data generated using the Fulcrum mobile application. PloS one. 16 (10), 0254293(2021).
- Mobile data collection & workflow automation in NCAP. , Available from: https://www.fulcrumapp.com/ (2021).
- Nematode Isolation Application. , Available from: https://www.fulcrumapp.com/apps/nematode-isolation (2021).
- Nematode Field Sampling Application. , Available from: https://www.fulcrumapp.com/apps/nematode-field-sampling (2021).
- JOVE wild_isolate-genotyping-template. , Available from: https://docs.google.com/spreadsheets/d/1oEP40jy6_K6Wkwn7Qm4iF58rTUe4JWk2zhJ20-keoOk/edit (2021).
- Nucleotide BLAST: Search nucleotide databases using a nucleotide query. , Available from: https://blast.ncbi.nim.nih.gov/Blast.cgi?PAGE_TYPE=BlastSearch (2021).
- easyFulcrum. , Available from: http://andersenlab.org/easyfulcrum/articles/easyFulcrum.html (2021).
- Andersen, E. C., Bloom, J. S., Gerke, J. P., Kruglyak, L. A variant in the neuropeptide receptor npr-1 is a major determinant of Caenorhabditis elegans growth and physiology. PLoS Genetics. 10 (2), 1004156(2014).
- Tuli, M. A., Daul, A., Schedl, T. WormBook: Caenorhabditis Nomenclature. WormBook. , (2018).
- Stiernagle, T. WormBook: Maintenance of C. Elegant. WormBook. , (2006).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены