Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Высокопроизводительный скрининг микробных изолятов с воздействием на здоровье Caenorhabditis elegans
* Эти авторы внесли равный вклад
В этой статье
Резюме
Кишечные микробы могут положительно или отрицательно влиять на здоровье своего хозяина через определенные или сохраненные механизмы. Caenorhabditis elegans является удобной платформой для скрининга таких микробов. Настоящий протокол описывает высокопроизводительный скрининг 48 бактериальных изолятов на предмет воздействия на стрессоустойчивость нематод, используемых в качестве прокси для здоровья червей.
Аннотация
Благодаря своему небольшому размеру, короткой продолжительности жизни и легкой генетике, Caenorhabditis elegans предлагает удобную платформу для изучения влияния микробных изолятов на физиологию хозяина. Он также флуоресцирует синим цветом при смерти, обеспечивая удобное средство точного определения смерти. Это свойство было использовано для разработки высокопроизводительных анализов выживания C. elegans без маркировки (LFASS). Они включают в себя покадровую флуоресцентную регистрацию популяций червей, установленных в многолуночных пластинах, из которых может быть получено медианное время смерти популяции. В настоящем исследовании используется подход LFASS для скрининга нескольких микробных изолятов одновременно на воздействие на восприимчивость C. elegans к сильным тепловым и окислительным стрессам. Такой микробный скрининговый конвейер, который, в частности, может быть использован для предварительного скрининга пробиотиков, используя сильную стрессоустойчивость в качестве прокси для здоровья хозяина, сообщается здесь. Протокол описывает, как выращивать как коллекции изолятов кишечной микробиоты C. elegans , так и синхронные популяции червей в многоязычных массивах, прежде чем комбинировать их для анализов. Приведенный пример охватывает тестирование 47 бактериальных изолятов и одного контрольного штамма на двух штаммах червей в двух параллельных анализах напряжения. Тем не менее, конвейер подходов легко масштабируется и применим к скринингу многих других модальностей. Таким образом, он обеспечивает универсальную установку для быстрого обследования многопараметрического ландшафта биологических и биохимических условий, которые влияют на здоровье C. elegans .
Введение
Человеческое тело содержит около 10-100 триллионов живых микробных клеток (бактерий, грибов архей), которые в основном находятся в кишечнике, коже и слизистой оболочке1. В здоровом состоянии они обеспечивают преимущества для своего хозяина, включая выработку витаминов, созревание иммунной системы, стимуляцию врожденных и адаптивных иммунных реакций на патогены, регуляцию жирового обмена, модуляцию стрессовых реакций и многое другое, с влиянием на рост и развитие, начало заболевания и старение 2,3,4,5 . Микробиота кишечника также значительно развивается на протяжении всей жизни. Наиболее радикальная эволюция происходит в младенчестве и раннем детстве6, но значительные изменения также происходят с возрастом, включая снижение численности бифидобактерий и увеличение Clostridium, Lactobacillus, Enterobacteriaceae и Enterococcus видов 7. Образ жизни может еще больше изменить микробный состав кишечника, что приводит к дисбактериозу (потеря полезных бактерий, чрезмерный рост условно-патогенных бактерий), что приводит к различным патологиям, таким как воспалительные заболевания кишечника, диабет и ожирение5, но также способствует развитию болезней Альцгеймера и Паркинсона 8,9,10,11.
Это осознание критически способствовало уточнению концепции оси кишечник-мозг (GBA), где взаимодействия между физиологией кишечника (теперь включая микробы внутри него) и нервной системой считаются основным регулятором метаболизма животных и физиологических функций12. Однако точная роль микробиоты в передаче сигналов кишечником и мозгом и связанные с ней механизмы действия далеко не полностью поняты13. Поскольку микробиота кишечника является ключевым фактором, определяющим здоровое старение, то, как бактерии модулируют процесс старения, стало предметом интенсивных исследований и споров 6,14,15.
С демонстрацией того, что круглый червь Caenorhabditis elegans содержит добросовестную микробиоту кишечника, в которой преобладают, как и у других видов, Bacteroidetes, Firmicutes и Actinobacteria 16,17,18,19,20, его быстрый рост в качестве экспериментальной платформы для изучения комменсальных взаимодействий между кишечником хозяина и кишечника 21,22,23,24 ,25,26 значительно расширил наш следственный арсенал 26,27,28,29. В частности, высокопроизводительные экспериментальные подходы, доступные для C. elegans для изучения взаимодействий ген-диета, ген-лекарство, ген-патоген и т. Д., Могут быть адаптированы для быстрого изучения того, как бактериальные изоляты и коктейли влияют на здоровье и старение C. elegans.
Настоящий протокол описывает экспериментальный конвейер для одновременного скрининга массивов бактериальных изолятов или смесей, установленных в многолуночных пластинах, на предмет воздействия на стрессоустойчивость C. elegans в качестве прокси для здоровья, который может быть использован для идентификации пробиотиков. В нем подробно описывается, как выращивать большие популяции червей и обрабатывать бактериальные массивы в форматах пластин с 96 и 384 лунками перед обработкой червей для автоматизированного анализа стрессоустойчивости с использованием считывателя флуоресцентных пластин (рисунок 1). Этот подход основан на автоматизированных анализах выживания без маркировки (LFASS)30 , которые используют явление флуоресценциисмерти 31, в результате чего умирающие черви производят всплеск синей флуоресценции, который может быть использован для точного определения времени смерти. Синяя флуоресценция испускается глюкозильными эфирами антраниловой кислоты, хранящимися в кишечных гранулах C. elegans (тип органелл, связанных с лизосомами), которые лопаются, когда некротический каскад запускается в кишечнике червя после смерти31.
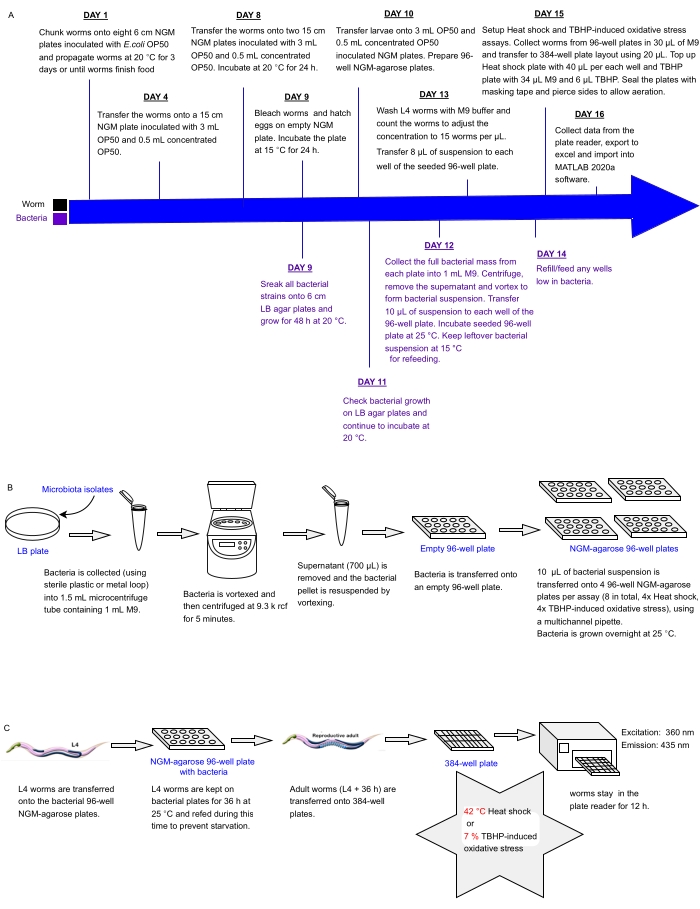
Рисунок 1: Экспериментальный рабочий процесс для высокопроизводительного скрининга бактериальных изолятов с воздействием на устойчивость C. elegans к стрессу. (A) Временная шкала для поддержания червей и бактерий и настройки анализа. (B) Установка и обработка 96-луночной бактериальной пластинчатой решетки. (C) Установка червячной пластины на 384 скважины. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
Двумя штаммами C. elegans , используемыми параллельно для настоящего исследования, были Bristol N2 wild type и HT1890: daf-16 (mgDf50), которые растут с одинаковой скоростью. Тем не менее, протокол может быть воспроизведен с любой комбинацией двух штаммов, которые имеют сходные темпы роста. Отметим, что при параллельном тестировании других штаммов (например, диких типов и медленно растущих мутантов daf-2 ) необходимо учитывать различные темпы роста, и соответственно, протокол нуждается в корректировке. Временные рамки и количество червей и бактерий в следующем протоколе оптимизированы для параллельного тестирования 48 бактериальных изолятов на двух штаммах червей в двух анализах LFASS в тетрапликатах. Корректировки потребуются для параллельного тестирования дополнительных условий. Кишечная палочка Штамм бактерий OP50 был получен из Центра генетики caenorhabditis (CGC) Университета Миннесоты. 48 бактериальных изолятов были получены из лаборатории Шуленбурга и поддерживались на агаре LB.
1. Культивирование C. elegans на OP50 (Дни 1 - 8)
ПРИМЕЧАНИЕ: Текущий подход направлен на выращивание гермафродитов C. elegans на твердой среде на всех стадиях и позволяет избежать ненужных диетических изменений (т. Е. Использование альтернативных быстрорастущих штаммов E. coli, таких как NA22, или более богатых питательных сред, таких как яичные пластины), чтобы оставаться как можно ближе к стандартным условиям роста32,33, которые все еще широко используются. Температура роста червя (здесь установленная на уровне 15 ° C) зависит от используемого штамма (штаммов) C. elegans и может нуждаться в корректировке (например, чтобы избежать или вызвать экспрессию термочувствительного фенотипа или биомаркера). Для получения информации о разведении червей, пожалуйста, смотрите ссылку33.
- Подготовьте восемь пластин NGM диаметром 6 см (10 мл агара среды для роста нематод, NGM, дополнительный файл 1)32,33 на штамм червя и дайте им высохнуть в течение 1 дня при комнатной температуре.
- Приготовьте насыщенную жидкую культуру бактерий E. coli OP50 путем посева одного бактериального клона из свежевыращенной пластины агара Lysogeny Broth (LB agar, Дополнительный файл 1) в 25 мл среды OP50 (Дополнительный файл 1) в коническую трубку объемом 50 мл. Выращивайте культуру в течение ночи при температуре 37 °C в шейкерном инкубаторе.
- Инокулируют восемь пластин NGM по 6 см на штамм 100 мкл насыщенной жидкой культуры E. coli OP50 на пластину и держат пластины при 20 °C в течение 2 дней перед использованием.
- Используя скальпель, вырежьте и переложите квадратный кусок агара размером 0,5 см с червями из недавно истощенной пластины NGM на каждую из восьми привитых 6 см пластин NGM и высиживайте эти пластины при 20 ° C в течение 3 дней (или до тех пор, пока черви не закончат пищу).
- Подготовьте пять пластин NGM по 15 см на штамм червя (30 мл среды NGM на пластину) и привите 3 мл OP50. Дайте пластинам высохнуть перед инкубацией при 37 °C в течение ночи. Держите пластины при температуре 20 °C до использования на последующих этапах.
- Используя пипетку P-1000, добавьте до 3 мл стерильного буфера M9 (дополнительный файл 1) к пластинам NGM 6 см (шаг 1.1.) для повторного суспендирования червей и соберите раствор червя со всех восьми пластин на штамм в одной конической трубке объемом 15 мл.
- Центрифуга при 142 х г в течение 2 мин при 4 °C. Осторожно удалите супернатант с помощью пипетки P-5000 или водяного насоса, оснащенного стерильной пипеткой или наконечником Пастера. Добавьте 10 мл стерильного буфера M9 для промывки гранул червя. Повторите 2x.
- Удалите супернатант (насколько это возможно) и перенесите червей на 15-сантиметровую привитую пластину NGM OP50 (шаг 1.5.) с помощью пипетки. Добавить 0,5 мл концентрированной культуры OP50.
- Чтобы сделать концентрированный OP50, привите каждую из четырех бутылок LB по 1 л 2 мл закваски OP50 (приготовленной на этапе 1.2.) и выращивайте в встряхивающем инкубаторе в течение 6 ч при 37 °C и 160 х г. Гранулируют бактерии при 3057 х г и 20 °C в течение 15 мин. Выбросьте супернатант, повторно суспендируйте бактериальные гранулы с 6 мл среды OP50 и соберите в стерильную коническую трубку объемом 50 мл.
ПРИМЕЧАНИЕ: Бактерии могут храниться при температуре 4 °C до 1 недели.
- Выращивайте каждый штамм червя на пластине NGM диаметром 15 см в течение 3-4 дней при 15 °C, повторно кормя червей 0,5 мл концентрированного OP50 ежедневно.
- После того, как черви почти закончили пищу, соберите и промыте в буфере M9 (шаг 1.6.1.), перенесите культуру каждого штамма червя на две 15-сантиметровые пластины NGM (шаг 1.5.) и размножите червей при 20 ° C до ~ 95% популяции взрослых особей (для дикого типа Bristol N2 это займет около 24 ч).
ПРИМЕЧАНИЕ: Взрослые особи гравидов характеризуются наличием яиц внутри червя, и идеальная тарелка также должна иметь обилие невылупившихся яиц, отложенных на тарелке без слишком большого количества личинок33.
- После того, как черви почти закончили пищу, соберите и промыте в буфере M9 (шаг 1.6.1.), перенесите культуру каждого штамма червя на две 15-сантиметровые пластины NGM (шаг 1.5.) и размножите червей при 20 ° C до ~ 95% популяции взрослых особей (для дикого типа Bristol N2 это займет около 24 ч).
2. Поддержание коллекций изолятов кишечной микробиоты (День 9)
- Распределите 48 бактериальных изолятов на отдельных пластинах агара 6 см LB и вырастайте в течение 48 ч при 20 °C.
ПРИМЕЧАНИЕ: Бактерии могут быть выращены при 25 ° C в течение 24-36 ч, если это необходимо раньше, но более длительный рост на 20 ° C позволяет обнаружить потенциальные загрязняющие вещества. - Синхронизируйте большое количество C. elegans.
- Отбеливайте взрослых червей, следуя стандартному способу подготовки яиц33 , и переносите яйца на две несеянные 15 см пластины NGM в течение 24 ч при 15 °C, чтобы все личинки L1 вылуплялись и росли синхронно на последующих этапах.
ВНИМАНИЕ: Будьте осторожны при обращении с отбеливающими растворами.
- Отбеливайте взрослых червей, следуя стандартному способу подготовки яиц33 , и переносите яйца на две несеянные 15 см пластины NGM в течение 24 ч при 15 °C, чтобы все личинки L1 вылуплялись и росли синхронно на последующих этапах.
3. Выращивание крупных культур C. elegans (День 10)
- После вылупления соберите личинок L1 (из Шага 2.2.1.) в 3-4 мл M9 в чистую коническую трубку 15 мл. Пипетку четыре капли 10 мкл раствора червя на предметное стекло или пластину и подсчитывайте количество червей в каждой капле под стереомикроскопом при 16-кратном увеличении. Определить концентрацию раствора червячком можно путем усреднения количества личинок из всех капель раствора червя. Умножьте это значение на оставшийся объем и оцените общее количество червей для каждого штамма.
ПРИМЕЧАНИЕ: На этом этапе требуется 46 000-50 000 личинок L1 на штамм, чтобы позже заполнить 384-луночную пластину или две полуплиты.- Для каждого штамма переложите все личинки L1 на две 15-сантиметровые пластины NGM (23 000-25 000 L1 на пластину), предварительно привитые 3 мл OP50 (этап 1,5.) и повторно засеянные 0,5 мл концентрированного OP50.
- Инкубировать при 15 °C, доливая 0,5 мл концентрированного OP50 ежедневно по мере необходимости, пока черви не достигнут стадии L4.
ПРИМЕЧАНИЕ: Стадия L4 характеризуется немного более темным кишечником и белым пятном в форме полудиска или полумесяца, где вульва в конечном итоге образует32,33. - Подготовьте 96-луночные NGM-агарозные пластины, следуя приведенным ниже шагам.
- Подготовьте восемь 96-луночных NGM-агарозных пластин, заполнив каждую скважину 125 мкл NGM-агарозы (четыре пластины на анализ).
ПРИМЕЧАНИЕ: Рекомендуется накрыть некоторые дополнительные пластины, если некоторые из них загрязняются на последующих этапах. Для параллельного проведения анализов потребуются два считывателя пластин, но они также могут быть запущены последовательно, начиная с анализа теплового стресса, поскольку он может выполняться всего 6 часов. Для этих пластин <4% золяной агар заменяют агарозой (см. Таблицу материалов), что позволяет медленнее и более равномерно высыхать через пробки NGM и уменьшать закапывание червей для лучшего восстановления. - Убедитесь, что скважины заполнены равномерно и без пузырьков. Используйте тепловой блок, установленный при 70 °C (при медленной теплопередаче через пластик многолуночной пластины NGM-агароза может нагреваться только до примерно 55-60 °C), чтобы предотвратить затвердевание смеси во время процесса. Чтобы удалить пузырьки внутри лунок, используйте стерильную пламенную иглу.
- Позвольте плитам из 96 скважин установить комнатную температуру в стерильной среде перед инвертированием (крышкой вниз, чтобы предотвратить конденсацию) и храните при 4 °C в чистой коробке до тех пор, пока это не понадобится.
- Подготовьте восемь 96-луночных NGM-агарозных пластин, заполнив каждую скважину 125 мкл NGM-агарозы (четыре пластины на анализ).
- На 11-й день проверьте червей из Шага 3.2., убедившись, что никаких загрязнений не появилось, и черви все еще изобилуют.
- На 12-й день проверьте червей из Шага 3.1., убедившись, что никаких загрязнений не появилось, и черви все еще изобилуют. Кроме того, проверьте стадию развития червей.
ПРИМЕЧАНИЕ: Пол/ штамм и стадия развития червя, такие как используемые черви L4 или L4 + 24 часа, зависят от лечения, которому подвергаются черви. Здесь гермафродиты дикого типа подвергались воздействию бактериальных изолятов из L4 в течение 36 ч.
4. Подготовка коллекций изолятов кишечной микробиоты для повторного кормления червей
- Мониторинг роста бактерий на пластинах агара LB из Шага 2.1. и продолжать инкубировать при температуре 20°С.
ПРИМЕЧАНИЕ: Хотя это не идеально, в случае, если некоторые клоны не растут или не обнаруживают загрязнений, бактерии могут быть повторно перенесены из чистых запасов на пластины LB 6 см и выращены при 25-28 ° C в течение 24 часов, чтобы быть готовыми к эксперименту. - Определить схему массива из 96 скважин для тестируемого бактериального сбора, облегчая систематическое заполнение пластин и анализ данных на последующих этапах (Дополнительная таблица 1).
- Соберите бактериальную массу с каждой бактериальной пластины размером 6 см (шаг 4.1.) и перенесите ее в меченую микроцентрифужную трубку объемом 1,5 мл, содержащую 1 мл буфера M9. Выполните это, используя либо одноразовую стерильную пластиковую петлю диаметром 2 мм, либо металлическую петлю диаметром 5 мм. Стерилизуйте металлическую петлю между бактериальными штаммами, погружая в 100% этанол, пылая и охлаждаясь в течение 5 с.
- Вращайте трубки микроцентрифуги до тех пор, пока бактериальные гранулы не будут полностью повторно суспендированы (в зависимости от бактериального штамма, это может занять ~ 1-10 с).
- Открутите при 9 300 х г в течение 5 мин при комнатной температуре, удалите 700 мкл супернатанта и повторно суспендируйте бактериальную гранулу путем вихря.
- Перенесите 200 мкл каждой бактериальной суспензии в одну лунку пустой стерильной 96-луночной пластины в соответствии с схемой, изложенной на этапе 4.2.
- Из этой пластины инокулируют восемь 96-луночных NGM-агарозных пластин (приготовленных на стадии 3.3.) 10 мкл бактериального раствора с использованием многоканальной пипетки и инкубируют с крышкой при 25 °C в течение 24 ч. Не запечатывайте пластины, чтобы обеспечить высыхание пластин и бактериальный аэробный рост, а также избежать избыточной конденсации.
- Запечатайте 96-луночную суспензионную пластину, подготовленную на этапе 4.6. с чистой клейкой уплотнительной пленкой (см. Таблицу материалов) и хранить при температуре 15 °C до 5 дней. Это будет использоваться для повторного кормления червей по мере необходимости.
5. Установка теплового шока и окислительного анализа LFASS (дни 13 - 14)
- Взглянув на пластины из Шага 3.5., оцените стадию развития червей. Как только >90% червей достигнут L434, соберите червей в 10 мл стерильного раствора M9 в конические трубки объемом 15 мл.
- Тщательно вымойте червей (по крайней мере, в 4 раза), вращаясь при 142 х г в течение 2 мин при 4 °C, удаляя супернатант и добавляя 10 мл свежего стерильного M9 между каждой стиркой, чтобы избавиться от бактерий OP50. Повторно суспендировать червячную гранулу в 10 мл M9.
- Переложить 50 мкл раствора червя в низкоповерхностную связующую трубку (см. Таблицу материалов), содержащую 950 мкл M9. После осторожного перемешивания содержимого трубки, чтобы избежать осаждения червей, быстро используйте смачиваемый наконечник пипетки с низким связыванием, чтобы перенести 3-4 отдельные капли 10 мкл на стеклянный предмет или пластину NGM и подсчитать номера червей под стереомикроскопом (см. Таблицу материалов) при 16-кратном увеличении. Усредните количество от 3-4 капель и определите количество червей на микролитр в растворе червя (см. Шаг 3.1.).
- Отрегулируйте концентрацию червя в пробирке 10 мл, чтобы достичь ~ 120 червей в 8 мкл. Если раствор приготовлен на шаге 5.2. недостаточно концентрирован, раскрутит червей вниз и удалит M9 соответственно, чтобы достичь 120 червей на 8 мкл.
- Перенесите 8 мкл раствора червя (~120 червей) в каждую из скважин восьми 96-луночных NGM-агарозных пластин из Шага 4.7., используя многоканальный пипет или повторную пипетку. Убедитесь, что вы используете наконечники с низким уровнем удержания, чтобы ограничить потерю червей. Также может потребоваться обрезать концы кончиков, чтобы позволить крупным взрослым червям ограничить механическую нагрузку на взрослых червей.
ПРИМЕЧАНИЕ: Анализ требует минимум 30 живых здоровых червей для надежной работы, но лучше всего работает с примерно 100 червями на скважину. - Инкубировать червячные и бактериальные 96-луночные NGM-агарозные пластины при 25 °C в течение 36 ч.
- Проверьте пластины между 12-24 часами, убедившись, что черви остаются изобилующими на протяжении всего времени. Если требуется повторное кормление, повторно суспендируйте бактерии в 96-луночной пластине бактериального массива, хранящейся при 15 °C на этапе 4.8., и добавьте до 10 мкл соответствующего бактериального раствора в 96-луночные NGM-агарозные пластины, где черви подвергаются риску голодания до конца 36-часового инкубационного периода (голодающие черви будут давать совершенно разные результаты, так что это очень важно).
ПРИМЕЧАНИЕ: Следующие шаги должны быть выполнены на 15-й день. Перед началом анализа может потребоваться оптимизировать высоту считывания. Оптимальное показание будет достигнуто на 20-50 мкм выше дна скважины. Это будет зависеть от модели считывателя пластин. Некоторые предлагают возможность Z-сканирования, в то время как другие допускают ручной ввод высоты. Установите оптимальную высоту на уровне, на котором обнаружен самый высокий сигнал синей флуоресценции (365 нм/430 нм). Некоторые считыватели пластин могут работать на фиксированной высоте, оптимизированной для анализа адгезивных клеток, и могут быть не идеальными для анализов LFASS. - Через 36 ч дозируйте 30 мкл M9 в каждую лунку 96-луночной пластины.
ПРИМЕЧАНИЕ: Для анализа тепловых напряжений считыватель пластин должен достичь требуемой температуры для выполнения анализа и, возможно, потребуется включить его заранее. Текущий протокол использует 42 ° C для максимизации скорости уничтожения, но подход применяется к другим температурам выше 30 ° C. - Перенесите червей (около 20 мкл) на плиту из 384 скважин в соответствии с установленными схемами, используя наконечники с низким удержанием (подумайте о том, чтобы отрезать конец наконечников, чтобы позволить крупным червям уменьшить механическое напряжение для взрослых червей).
ПРИМЕЧАНИЕ: Для настоящего исследования для двух описанных здесь анализов используются две различные настройки считывателя пластин (тепловой стресс и окислительный стресс), и, таким образом, образцы, предназначенные для этих двух анализов, не должны быть покрыты одной и той же 384-луночной пластиной. - Убедитесь, что считыватели пластин настроены правильно (таблица 1).
- Пополните плиты из 384 скважин большим количеством M9, стремясь к конечному объему 60 мкл на скважину. Для анализа теплового напряжения добавьте 40 мкл M9, а для t-BHP-индуцированного окислительного стресса добавьте 34 мкл M9 в 6 мкл t-BHP (см. Таблицу материалов).
- Начните анализ в течение 2 мин после добавления t-BHP (в идеале все черви должны подвергаться воздействию t-BHP одновременно, время разрешения анализа составляет 2 мин). Если это невозможно, используйте таймер для оценки времени, затраченного на пипетку t-BHP до начала анализа, чтобы впоследствии можно было скорректировать медианное время смерти.
- Закройте пластины прозрачной крышкой. Запечатайте края 384-луночных пластин маскировочной лентой (наклеиванием на пластину и крышку), следя за тем, чтобы лента не проходила через крышку или под пластину. Разрезайте ленту между крышкой и пластиной с интервалами, используя скальпель, чтобы обеспечить воздухообмен при минимизации испарения во время анализа.
- Вставьте пластину в считыватель пластин (см. Таблицу материалов) и начните запуск. Стремитесь возбуждать при 365 нм и обнаруживать излучение при 435 нм каждые 2 мин в течение 6-12 ч (таблица 1).
ПРИМЕЧАНИЕ: Как правило, 6 ч достаточно для анализа теплового стресса при 42 °C и 8 ч для анализа окислительного стресса 7% t-BHP.
6. Обработка данных считывателя пластин
- Сохраните необработанные флуоресцентные данные из устройства чтения пластин в форматах .txt, .csv или .xls /.xlsx, разделенных запятыми или табуляцией, а затем преобразуйте их в формат xls /.xlsx. В зависимости от формата данных реорганизуйте их в соответствии с макетом листа Excel, необходимым для анализа LFASS. Следуйте подробным инструкциям, приведенным в ссылке30.
ПРИМЕЧАНИЕ: В то время как данные могут быть проанализированы вручную, нормализуя каждый временной ряд и ища время, когда флуоресценция смерти достигает половины максимума, автоматизированный анализ может быть проведен в Matlab, выполняя процедуру LFASS30. - Загрузите и установите Matlab (версия 2014a или выше) и пакет программного обеспечения LFASS с https://github.com/ABA80/LFASS. Следуйте приведенным в нем рекомендациям и аннотациям.
ПРИМЕЧАНИЕ: На рисунке 1С дано краткое описание подхода. Matlab необходим для выполнения процедуры LFASS. Альтернативно, код Matlab может быть переведен в Oracle, за исключением функции fitting, которая является проприетарной. Новые функции сглаживания и сигмовидной кислоты могут быть переписаны для обеспечения возможности использования в полностью открытой платформе. - Между анализами LFASS переместите данные и результаты в новое место, так как анализ LFASS обработает все файлы в папке данных и перезапишет файлы в папке Results.
7. Проверка данных
- Откройте файл excel и пометьте ряды в соответствии с положением скважины на пластине из 384 скважин. В дополнительном файле 2 приведен пример файла Excel необработанных флуоресцентных данных, полученных для анализа теплового удара. Используйте положение колодца на 384-луночной пластине, чтобы маркировать штаммы червей и бактерий.
- Перед анализом Matlab визуально осмотрите данные в Excel, построив интенсивность флуоресценции с течением времени для репрезентативного колодца. В зависимости от используемого считывателя пластин данные могут быть шумными, но должны отображать четкий пик. В частности:
- Определите значение флуоресценции, ниже которого пик не будет существенно отличаться от шума (установка такого порога в LFASS ускорит анализ, исключив пустые скважины).
- Обратите внимание на самый ранний момент времени, когда флуктуации флуоресценции ослабевают перед подъемом (животные могут энергично метаться до 30 минут, что приводит к быстрым колебаниям показаний синей флуоресценции).
ПРИМЕЧАНИЕ: Пиковая установка может быть улучшена путем исключения этих ранних временных точек из окна установки кривой. - Обратите внимание на временные точки, между которыми, как ожидается, будут падать минимальные и максимальные значения флуоресценции (посмотрите на несколько скважин, чтобы определить эти диапазоны), поскольку они будут использоваться для подгонки кривой.
- Проверьте, значительно ли изменяются амплитуды пиков флуоресценции между скважинами, нормализуйте данные перед дальнейшим анализом по следующей формуле:
Нормализованная флуоресцентная скважинаn (t) = (Флуоресцентная скважинаn [t] - минимальная флуоресцентная скважина [Dt]) / (максимальная флуоресцентная скважина [Dt] - минимальная флуоресцентная скважина [Dt])
где «n» — текущее число скважины, «t» — временная точка, а «Dt» — ряд временных точек для анализа.
8. Обработка данных LFASS
ПРИМЕЧАНИЕ: Подробная информация приводится в https://github.com/ABA80/LFASS и в дополнительных материалах Ссылки30.
- Создайте две вложенные папки в папке LFASS, одну для анализируемых данных и одну для результатов, например «мои данные» и «результаты».
- Скопируйте файл данных Excel в подпапку LFASS "мои данные" после проверки данных.
- Запустите MATLAB, перейдите в папку LFASS , введите и запустите fitfolder в командном окне (дополнительный файл 3). Затем следуйте инструкциям на экране.
- Набрав «fitfolder», система запрашивает имя папки, в которой находится файл excel, например, «my data». Введите имя папки с данными (в данном примере "my data").
- Следуйте инструкциям на экране, указав различные запрашиваемые параметры.
- Введите «2» для временного интервала между последовательными измерениями в текущем протоколе (указание этого позволяет выражать результаты в минутах, а не в единицах точки времени).
ПРИМЕЧАНИЕ: Временной интервал может быть изменен для более или менее частого выполнения флуоресцентных измерений (для уменьшения или увеличения временного разрешения), а также в зависимости от возможностей считывателя пластин (т.е. временной интервал может потребоваться увеличить для считывателей пластин, которые не могут выполнять достаточно быстрые измерения). Убедитесь, что экспериментальный временной интервал всегда совпадает с процедурой LFASS. - Назначьте верхний порог допуска, набрав «0,95» (его можно изменить по мере необходимости для улучшения посадки) и нижний порог допуска, набрав «0,05» (это может быть изменено по мере необходимости для улучшения посадки), чтобы ограничить подгонку сигмовидной кишки.
ПРИМЕЧАНИЕ: Другие параметры времени основаны на заметках пользователя из проверки данных (шаг 7.2.).
- Введите «2» для временного интервала между последовательными измерениями в текущем протоколе (указание этого позволяет выражать результаты в минутах, а не в единицах точки времени).
- Выберите, следует ли отображать или неподготовленные и сглаженные кривые, введя «y» для YES или «n» для NO при появлении запроса. Чтобы визуально осмотреть сходящиеся припадки, выберите первый.
ПРИМЕЧАНИЕ: Последний полезен для визуализации всех сглаженных данных, но обычно не выбирается, потому что он генерирует слишком много всплывающих графиков. После этого Matlab выполнит процедуру LFASS, которая может занять 1-10 минут, если несколько файлов Excel обрабатываются за один раз. Всплывающие окна с кривыми будут отображаться в соответствии с выбором в шаге 8.6. В дополнительном файле 4A показан пример подходящей кривой. - Выберите, следует ли (1) анализировать кривые, идентифицированные как шум, или (2) переоборудовать плохо подогнанные кривые с опцией [y/n]. Введите y для утверждения и n для отклонения.
ПРИМЕЧАНИЕ: Рекомендуется одобрить переоборудование, особенно если есть много плохо подогнанных или неподготовленных кривых. Это позволит пользователю предоставлять индивидуальные параметры подгонки кривых для каждой кривой по мере их появления на экране и запрашивать только более ранние и более поздние границы для подгонки сигмовидной кости. Его можно попробовать столько раз, сколько потребуется. - После анализа данных закройте Matlab и откройте папку LFASS .
- Нажмите на вложенную папку LFASS Мои результаты, так как файлы результатов автоматически сохраняются в папке Результаты как .txt.
ПРИМЕЧАНИЕ: Matlab генерирует три файла .txt: "Batch-fitted.txt", "Batch and noise-fitted.txt" и "Refitted.txt". Первые два сохраняются в качестве меры предосторожности в случае сбоя компьютера или ошибки пользователя во время переоборудования. Файлом, содержащим наиболее точный полный анализ, является "Refitted.txt". - Откройте файл Refitted.txt с помощью Microsoft Excel и сохраните как .xls для дальнейшей обработки. Дополнительный файл 4B показывает пример такого результирующего файла.
ПРИМЕЧАНИЕ: Для каждой скважины (организованной в ряды) в столбцах, дающих оценки медианного времени гибели популяции червей, приводятся три значения: "Raw": сообщает о времени пересечения на половинном максимуме пика экспериментальных данных; "Batch-fitted": сообщает время пересечения на половинном максимуме кривой пакетной установки; "Переоборудованный": сообщает о времени пересечения на полумаксимате переоборудованной кривой. - Сохраните файл в .xls формате как копию в безопасном месте. Если этого не сделать, вы рискуете перезаписать файлы во время следующего запуска процедуры LFASS.
ПРИМЕЧАНИЕ: Затем результаты могут быть дополнительно обработаны для построения графиков или статистического анализа.
Результаты
Анализы LFASS обеспечивают надежный, высокопроизводительный и быстрый скрининг нескольких тестовых состояний одновременно, таких как скрининг многочисленных генетических и микробиотных параметров, которые способствуют стрессоустойчивости и старению. Эксперименту требуется всего 2-3 ?...
Обсуждение
C. elegans предлагает множество преимуществ для быстрого скрининга нескольких экспериментальных параметров одновременно, благодаря своим небольшим размерам, прозрачности, быстрому развитию, короткому сроку службы, недороговизне и простоте обращения. Его значительно более простой г...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы благодарим CGC Minnesota (Madison, США, NIH - P40 OD010440) за предоставление штаммов червей и OP50 и Pr. Hinrich Schulenburg (CAU, Киль, Германия) за предоставление всех экологических микробных изолятов, изображенных здесь. Эта работа финансировалась за счет гранта UKRI-BBSRC ab (BB/S017127/1). JM финансируется стипендией FHM PhD Ланкастерского университета.
Материалы
| Name | Company | Catalog Number | Comments |
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Ссылки
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood - Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer's disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson's disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer's disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson's disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены