Method Article
Индустриализированная лазерная микродиссекция с искусственным интеллектом для микромасштабированного протеомного анализа микроокружения опухоли
* Эти авторы внесли равный вклад
В этой статье
Резюме
Этот протокол описывает высокопроизводительный рабочий процесс для сегментации на основе искусственного интеллекта интересующих областей, подтвержденных патологией, из окрашенных изображений тонких участков тканей для обогащения популяций клеток с гистологическим разрешением с использованием лазерной микродиссекции. Эта стратегия включает в себя новый алгоритм, позволяющий передавать демаркации, обозначающие интересующие клеточные популяции, непосредственно на лазерные микроскопы.
Аннотация
Микроокружение опухоли (TME) представляет собой сложную экосистему, состоящую из десятков различных типов клеток, включая опухоли, стромы и популяции иммунных клеток. Чтобы охарактеризовать вариации протеомного уровня и гетерогенность опухоли в масштабе, необходимы высокопроизводительные методы для селективной изоляции дискретных клеточных популяций при злокачественных новообразованиях солидных опухолей. Этот протокол описывает высокопроизводительный рабочий процесс, обеспечиваемый искусственным интеллектом (ИИ), который сегментирует изображения окрашенных гематоксилином и эозином (H & E), тонких участков тканей в подтвержденные патологией области, представляющие интерес для селективного сбора популяций клеток с гистологическим разрешением с использованием лазерной микродиссекции (LMD). Эта стратегия включает в себя новый алгоритм, позволяющий передавать области, обозначающие интересующие клеточные популяции, аннотированные с помощью программного обеспечения для цифровых изображений, непосредственно в лазерные микроскопы, что позволяет более легко собирать данные. Была выполнена успешная реализация этого рабочего процесса, демонстрирующая полезность этого гармонизированного метода для селективного сбора популяций опухолевых клеток из TME для количественного, мультиплексированного протеомного анализа с помощью масс-спектрометрии высокого разрешения. Эта стратегия полностью интегрируется с рутинным обзором гистопатологии, используя анализ цифровых изображений для поддержки обогащения интересующих клеточных популяций и является полностью обобщаемой, что позволяет гармонизировать сбор клеточных популяций из TME для многоомного анализа.
Введение
TME представляет собой сложную экосистему, населенную очень разнообразным набором типов клеток, таких как опухолевые клетки, стромальные клетки, иммунные клетки, эндотелиальные клетки, другие типы мезенхимальных клеток и адипоциты, наряду со сложным внеклеточным матриксом1. Эта клеточная экосистема варьируется внутри и между различными участками органов заболевания, что приводит к сложной гетерогенности опухоли 2,3. Недавние исследования показали, что гетерогенные опухоли и опухоли с низкой клеточностью опухоли (низкой чистотой) часто коррелируют с плохим прогнозом заболевания 2,3.
Чтобы понять молекулярное взаимодействие между популяциями опухолевых и неопухолевых клеток в пределах TME в масштабе, стандартизированные и высокопроизводительные стратегии необходимы для селективного сбора различных клеточных популяций, представляющих интерес для последующего многоомного анализа. Количественная протеомика представляет собой быстро развивающийся и все более важный метод для дальнейшего понимания биологии рака. На сегодняшний день преобладание исследований, использующих протеомику, сделало это с белками, извлеченными из цельных препаратов опухолевой ткани (например, криопульверизированными), что привело к скудности в понимании гетерогенности протеомного уровня в TME 4,5,6.
Разработка стратегий сбора образцов, которые легко интегрируются и используют информацию из рабочих процессов клинической патологии, позволит создать новое поколение протеомики с гистологическим разрешением, которые в значительной степени дополняют рабочие процессы диагностической патологии золотого стандарта. LMD обеспечивает прямой и селективный сбор клеточных субпопуляций или областей, представляющих интерес (ROI), путем микроскопического обследования гистологически окрашенных тонких участковткани 7. Недавние крупные достижения в области цифровой патологии и анализа с поддержкой ИИ продемонстрировали способность автоматически идентифицировать уникальные композиционные особенности и ROI в TME, многие из которых коррелируют с молекулярными изменениями и клиническими особенностями заболевания, такими как устойчивость к терапии и прогноз заболевания8.
Рабочий процесс, описанный в протоколе, представленном здесь, использует коммерческие программные решения для выборочной аннотации ROI опухоли в цифровых гистопатологических изображениях и использует программные инструменты, разработанные собственными силами для передачи этих ОПУХОЛЕВЫХ ROI на лазерные микроскопы для автоматизированного сбора дискретных клеточных популяций, представляющих интерес, который легко интегрируется с последующими рабочими процессами мультиомного анализа. Эта интегрированная стратегия значительно сокращает время оператора LMD и сводит к минимуму продолжительность, в течение которой ткани должны находиться при температуре окружающей среды. Интеграция автоматизированного отбора признаков и сбора LMD с высокопроизводительной количественной протеомикой демонстрируется путем дифференциального анализа TME из двух репрезентативных гистологических подтипов эпителиального рака яичников: высокодифференцированного серозного рака яичников (HGSOC) и прозрачно-клеточной карциномы яичников (OCCC).
протокол
Все протоколы исследований были одобрены для использования в соответствии с одобренным Западным IRB протоколом «Интегрированный молекулярный анализ рака эндометрия и яичников для выявления и проверки клинически информативных биомаркеров», который считается освобожденным в соответствии с федеральным постановлением США 45 CFR 46.102 (f). Все экспериментальные протоколы, включающие человеческие данные в этом исследовании, соответствовали Хельсинкской декларации. Информированное согласие было получено от всех субъектов, участвующих в исследовании.
ВНИМАНИЕ: Следующие реагенты, используемые во всем протоколе, являются известными или подозреваемыми канцерогенами и /или содержат опасные материалы: этанол, воду DEPC, раствор гематоксилина Майера, раствор Эозина Y, метанол, ацетонитрил и муравьиную кислоту. Надлежащее обращение, как описано в соответствующих паспортах безопасности (SDS), и использование соответствующих средств индивидуальной защиты (СИЗ) является обязательным.
1. Создание файла данных списка фигур по умолчанию (.sld), содержащего фидуциалы калибратора
ПРИМЕЧАНИЕ: Этапы протокола, описанные в этом разделе, специфичны для использования с инвертированным лазерным микроскопом и связанным с ним программным обеспечением (см. Таблицу материалов). Создание файла .sld по умолчанию необходимо только один раз для каждого лазерного микроскопа. Полученный файл может быть использован для резки фидуциалов во всех слайдах PEN, используемых впоследствии. Приблизительное время: 5 мин (только один раз).
- Откройте программное обеспечение LMD и загрузите мембрану полиэтиленнафталата (PEN) на ступень LMD лицевой стороной вниз, с этикеткой, ближайшей к пользователю. Снимите флажок Закрыть строки в правой части окна программы.
- Используйте функцию PtoP (точка-точка) при большом увеличении (63x), чтобы нарисовать три стрелки «V», которые служат в качестве фидуциалов калибровки. Начиная с одной внешней точки на V, проведите линию до средней точки V и одним щелчком мыши. Затем нарисуйте вторую линию от центральной точки V до конца второй внешней точки V и дважды щелкните, чтобы создать одну незакрытую V-образную фигуру из двух линий.
ПРИМЕЧАНИЕ: Эти калибровочные фидуциалы должны быть размещены в трех углах слайда: передний правый, задний правый, задний левый. - Выберите параметр автофокусировки (автофокусировка) перед вырезанием . Вырежьте слайд в позиции 1, переместите его в каждое из оставшихся положений слайда и точно проследите калибровочные разрезы.
- Сохраните файл .sld и выберите параметр Сохранить без калибровки во всплывающем диалоговом окне, чтобы избежать разрезания фидуциалов калибровки на мембрану.
ПРИМЕЧАНИЕ: Репрезентативный файл .sld, содержащий стандартные фидуциалы калибратора для четырех положений слайдов, представлен в дополнительном файле 1.
2. Подготовка слайдов LMD
ПРИМЕЧАНИЕ: Этапы протокола, описанные в этом разделе, специфичны для использования с инвертированным лазерным микроскопом и связанным с ним программным обеспечением (см. Таблицу материалов). Приблизительное время: 5 мин.
- Убедитесь, что затвор полностью высох, прежде чем резать опорные калибровочные фидуциалы. Откройте программное обеспечение LMD и откройте SLD-файл калибровки по умолчанию под параметром Импорт фигур .
- Выберите параметр автофокусировки (автофокусировка) перед вырезанием . Загрузите слайд(ы) тканью, обращенной вниз, и наклейку скользить ближе к оператору в держатель слайда на ступени LMD.
- Используя лазерный микроскоп и калибровочный файл .sld по умолчанию, врежьте фидуциалы калибровки в мембрану PEN.
- ДОПОЛНИТЕЛЬНО: Врежьте калибровочные фидуциалы в мембрану PEN либо до, либо после того, как участок (секции) ткани будет помещен на слайд. Если калибровочные фидуциалы разрезаются до размещения ткани, убедитесь, что ткань и/или фиксатор не перекрываются с калибраторами при размещении ткани на слайде на этапе 2.5. Если калибровочные фидуциалы разрезаются после помещения ткани, остановитесь после завершения этапа 2.4 и перейдите к разделу 3.
- Просмотрите все калибраторы по отдельности, чтобы убедиться, что каждый разрез является полным и видимым.
ПРИМЕЧАНИЕ: Используйте функцию «Переместить и вырезать», чтобы вручную направить лазер на любые калибровочные фидуциалы, которые не полностью прорезали мембрану PEN. - Поместите замороженный или закрепленный формалином, парафиновый (FFPE) участок ткани на слайд, содержащий калибровочные фидуциалы.
3. Окрашивание тканей
ПРИМЕЧАНИЕ: Приблизительное время: 30 мин.
- Зафиксируйте замороженные тканевые слайды LMD в 70% этаноле (EtOH), содержащем коктейльные реагенты ингибитора фосфатазы, в течение 5 минут.
- Промыть горки в диэтилпирокарбоматной (DEPC) воде, содержащей коктейльные реагенты ингибитора фосфатазы, в течение 1 мин.
- Мойте горки в воде DEPC в течение 1 мин.
- Инкубируйте слайды в растворе гематоксилина Майера в течение 3 минут.
- Промойте горки в воде DEPC в течение 3 минут.
- Промойте горки свежим обменом воды DEPC в течение 1 мин.
- Инкубируйте слайды в водном растворе Eosin Y в течение 1 с.
- Промойте слайды 2 x 5 с в 95% EtOH.
- Промойте слайды 3 x 10 с в 100% EtOH.
- Протрите лишний EtOH с задней части слайдов и дайте слайдам высохнуть на воздухе.
- Храните слайды при температуре -80 °C, если LMD не должен быть выполнен немедленно.
4. Изображение слайдов
ПРИМЕЧАНИЕ: Шаги протокола, описанные в этом разделе, относятся к отсканированным слайдам (см. Таблицу материалов) и результирующим изображениям, сохраненным в виде файлов .svs. Используйте любой сканер и связанное с ним программное обеспечение, которое генерирует файлы изображений в формате, который может открыть программное обеспечение для анализа изображений (см. Таблицу материалов). Поддерживаемые типы файлов, использующие пирамидальные tiff, включают JPG, TIF, MRXS, QPTIFF, компонент TIFF, SVS, AFI, SCN, LIF, DCM, OME. TIFF, ND2, VSI, NDPI, NDPIS, CZI, BIF, KFB и ISYNTAX. Приблизительное время: 5 мин.
- Включите сканер и откройте программное обеспечение сканера слайдов. Загрузите слайд тканью, обращенной вверх, на одну ступень слайда в сканере. Убедитесь, что горка полностью сухая, и аккуратно поместите обшивку поверх ткани. Не используйте этанол или погружное масло под крышкой.
- Сделайте снимок на микрофотографии с помощью настроек, откалиброванных для настройки мембраны PEN вместо стеклянного фона и для игнорирования окрашивания фоновой мембраны в соответствии с инструкциями производителя.
- Отрегулируйте область изображения, перетащив и изменив размер внутреннего зеленого периметра, чтобы захватить всю область мембраны PEN по мере необходимости. Добавьте четыре точки фокусировки на ткань, дважды щелкнув по обзорному изображению снимка, и три точки фокусировки на мембране рядом с фидуциалами калибровки (по одной точке фокусировки на каждую из трех калибровочных фидуциалов).
ПРИМЕЧАНИЕ: Четыре точки фокусировки могут быть размещены практически в любом месте на участке ткани, хотя размещение на ткани, которая слишком темно окрашена и кажется черной, может привести к сбою сканирования. - В меню Вид выберите Видеомонитор. Вручную отрегулируйте фокусировку с помощью ползунка тонкой и/или макрофокусировки, по мере необходимости, для каждой точки вокруг ткани LMD. Захват сканирования изображения при большом увеличении (20x). Убедитесь, что все фидуциалы калибровки видны и четки на сохраненном изображении.
5. Автоматический выбор функций с помощью программного обеспечения для анализа изображений
- Для целых опухолевых коллекций (приблизительное время: 5 мин; в зависимости от случая):
- Откройте программное обеспечение для анализа изображений (см. Таблицу материалов). Выберите «Открыть изображения» и во всплывающем окне выберите SVS-файл изображения, созданный при сканировании слайда на сканере AT2.
ПРИМЕЧАНИЕ: Репрезентативный файл изображения .svs представлен в дополнительном файле 2. - Перейдите на вкладку Аннотации . Выберите инструмент «Перо» на панели инструментов «Аннотация» и нарисуйте фигуру вокруг ткани.
- Выберите фигуру и щелкните изображение правой кнопкой мыши. В раскрывающемся меню Дополнительно выберите Секционирование (плитка). Установите для параметра Размер плитки и пространство между 500 и 40 соответственно и нажмите кнопку ОК , чтобы создать плитки. Выберите и удалите фигуру периметра, используемую для создания плиток на шаге 5.1.2.
- Выберите раскрывающееся меню Действия слоя | Экспортируйте, чтобы сохранить мозаичные аннотации в виде файла .annotation.
ПРИМЕЧАНИЕ: Репрезентативный файл .annotation для всей коллекции опухолевой ткани представлен в дополнительном файле 3. - Создайте папку для сеанса или проекта и сохраните файл ANNOTATION во вложенной папке, помеченной уникальным идентификатором слайда.
- Перейдите на вкладку Аннотации . Выберите раскрывающийся список Действия слоя | Удалить все слои , чтобы удалить все аннотации с изображения. Выберите инструмент «Перо» и нарисуйте короткую линию от внутреннего кончика стрелки для каждого фидуциала калибровки. Рисуйте линии от меток в следующем порядке: вверху слева, сверху справа, внизу справа.
- Выберите раскрывающееся меню Действия слоя | Экспортируйте, чтобы сохранить аннотации строк в виде файла .annotation. Добавьте _calib к имени файла и поместите файл во вложенную папку, содержащую координаты для мозаичных фигур.
ПРИМЕЧАНИЕ: Репрезентативный файл _calib.annotation представлен в Дополнительном файле 4. - Скопируйте адрес основного проекта или папки сеанса. Откройте скрипт, генерирующий импорт XML, "Malleator" (доступен через https://github.com/GYNCOE/Mitchell.et.al.2022), используя интегрированную среду разработки IDLE, и вставьте адрес папки проекта между кавычками в нижней части скрипта.
- Выберите раскрывающееся меню Выполнить | Запустите модуль для выполнения скрипта.
ПРИМЕЧАНИЕ: Файл импорта .xml LMD будет сгенерирован внутри вложенной папки, созданной для изображения/слайда. Репрезентативный файл .xml представлен в дополнительном файле 5.
- Откройте программное обеспечение для анализа изображений (см. Таблицу материалов). Выберите «Открыть изображения» и во всплывающем окне выберите SVS-файл изображения, созданный при сканировании слайда на сканере AT2.
- Только для коллекций, обогащенных LMD (приблизительное время: 15 мин; в зависимости от случая):
- Откройте программное обеспечение для анализа изображений (см. Таблицу материалов). Выберите «Открыть изображения» и во всплывающем окне выберите SVS-файл изображения, созданный при сканировании слайда.
- Перейдите на вкладку Аннотации . Выделите и используйте инструмент « Прямоугольная аннотация», чтобы нарисовать поле вокруг ткани.
- Выберите аннотацию поля и щелкните правой кнопкой мыши на изображении. Выберите раскрывающееся меню Дополнительно | Параметр «Секционирование (мозаичный ).» Установите для параметра Размер плитки и пространство между 500 и 40 соответственно и нажмите кнопку ОК , чтобы создать плитки. Выберите и удалите аннотацию поля периметра, используемую для создания плиток на шаге 5.2.2.
- Выберите раскрывающееся меню Действия слоя | Экспортируйте , чтобы сохранить мозаичные аннотации в виде файла .annotation.
- Поместите сохраненную копию алгоритма Python "Dapọ" (доступного через https://github.com/GYNCOE/Mitchell.et.al.2022), разработанного для объединения слоев аннотаций, классифицированных AI, в ту же папку, что и файл мозаичных аннотаций.
- Скопируйте имя файла мозаичной аннотации. Откройте программу Python с помощью интегрированной среды разработки IDLE и вставьте имя файла мозаичной аннотации между цитатами в нижней части программы.
- Выберите раскрывающееся меню Выполнить | Запустите модуль. Подождите, пока будет создан новый файл, в котором все мозаичные аннотации будут объединены под одним слоем.
- Откройте программное обеспечение для анализа изображений и перейдите на вкладку Аннотации . Выберите раскрывающееся меню Действия слоя | Удалить все слои , чтобы удалить все аннотации с изображения.
- Выберите раскрывающееся меню Действия слоя | Импорт файла локальной аннотации. Во всплывающем окне выберите объединенный файл .annotation, созданный сценарием. Убедитесь, что все импортированные плитки находятся под одним слоем аннотаций.
- Перейдите на вкладку Классификатор и следуйте инструкциям производителя, чтобы создать фигуры для ROI. Перед запуском классификатора выберите нужный слой (слои) аннотации (т. е. слой опухоли или стромы), установив флажок ROI или поля на вкладке Аннотации в разделе Дополнительные параметры классификатора. Используйте параметр Слой аннотаций в меню Действия классификатора для запуска классификатора.
- После завершения анализа классификатора перейдите на вкладку Аннотации и выберите слой аннотаций, созданный в результате анализа. Выберите раскрывающееся меню Действия слоя | Удалите все слои, кроме текущих , чтобы удалить все остальные слои аннотаций с изображения.
- Выберите раскрывающееся меню Действия слоя | Экспортируйте, чтобы сохранить аннотации в виде файла .annotation. Создайте папку для сеанса или проекта и сохраните файл ANNOTATION во вложенной папке, помеченной уникальным идентификатором слайда.
ПРИМЕЧАНИЕ: Репрезентативный файл .annotation для классифицированной коллекции тканей, обогащенных LMD, представлен в Дополнительном файле 6. - Перейдите на вкладку Аннотации, выберите раскрывающийся список Действия слоя | Удалить все слои, чтобы удалить все аннотации с изображения. Выберите инструмент «Перо» и нарисуйте короткую линию от каждого метода калибровки. Рисуйте линии из меток в следующем порядке: вверху слева, сверху справа, внизу справа.
- Выберите раскрывающееся меню Действия слоя | Экспортируйте , чтобы сохранить аннотации строк в виде файла .annotation. Добавьте _calib к имени файла и поместите файл во вложенную папку, содержащую координаты для мозаичных фигур.
- Скопируйте адрес основного проекта или папки сеанса. Откройте скрипт, генерирующий импорт XML, "Malleator" (доступен через https://github.com/GYNCOE/Mitchell.et.al.2022), используя интегрированную среду разработки IDLE, затем вставьте адрес папки проекта между кавычками в нижней части скрипта.
- Выберите раскрывающееся меню Выполнить | Запустите модуль для выполнения скрипта.
ПРИМЕЧАНИЕ: Файл импорта .xml LMD будет сгенерирован внутри вложенной папки, созданной для изображения/слайда.
6. Лазерная микродиссекция
ПРИМЕЧАНИЕ: Этапы протокола, описанные в этом разделе, специфичны для использования с инвертированным лазерным микроскопом и связанным с ним программным обеспечением (см. Таблицу материалов). Приблизительное время: 2 ч; зависит от случая.
- Загрузите отмеченный мембранный слайд (содержащий калибровочные фидуциалы) тканью, обращенной вниз, и стороной этикетки ближе к оператору в держатель слайдов на сцене лазерного микроскопа.
- Выберите «Импортировать фигуры» в раскрывающемся меню «Файл ». Выберите .xml файл импорта LMD , созданный для слайда. Выберите «Нет » во всплывающем окне, чтобы избежать загрузки опорных точек из файла, и «Нет » во втором всплывающем окне, чтобы избежать использования ранее сохраненных контрольных точек для калибровки.
- Следуйте подсказкам приложения LMD и выровняйте калибровочный крест по каждому из трех фидуциалов калибровки на слайде. Ищите фидуциалы калибровки, которые появляются в левом верхнем, правом верхнем и правом нижнем углу изображения слайда в программном обеспечении для анализа изображений, которые будут соответствовать контрольным точкам в переднем правом, заднем правом и заднем левом углах, соответственно, перевернутого слайда LMD на сцене микроскопа. Переключайтесь между использованием объектива 5x для определения местоположения и объектива 63x для выравнивания каждой физической калибровки. Выберите «Нет » во всплывающем окне, чтобы избежать сохранения опорных точек в файл, и «ОК» во втором всплывающем окне, чтобы убедиться, что слайд вставлен.
- Переместите объектив 5x в нужное положение и выберите Да во всплывающем окне, чтобы использовать фактическое увеличение. Как только появятся импортированные фигуры, сфокусируйте камеру на ткани.
- Выделите и выделите все фигуры в окне «Список фигур », перетащите их на место, используя одну или две аннотации в поле зрения в качестве ссылок, и выровняйте вертикальную ось Z для резки лазером.
- Просмотрите импортированные фигуры и назначьте их в соответствующее положение трубки для сбора. Нажмите Start Cut , чтобы запустить лазер.
ПРИМЕЧАНИЕ: Импортированные фигуры в .xml файле будут автоматически назначены позиции «без шапки» в окне «Список фигур ». Для сбора ткани импортированные фигуры должны быть переназначены в положение, содержащее загруженную трубку.
7. Переваривание белка по технологии цикла давления (PCT)
ПРИМЕЧАНИЕ: Приблизительное время: 4 ч (3 ч без высыхания вакуумной центрифуги).
- Поместите 0,5 мл пробирок, содержащих закрытые микротрубки PCT, содержащие собранную ткань LMD, в 20 мкл 100 мМ TEAB/10% ацетонитрила в термоциклер и нагревайте при 99 °C в течение 30 мин, затем охладите до 50 °C в течение 10 мин.
- Вращайте трубки в течение 30 с при 4000 × г , а затем извлекайте микротрубки из пробирок объемом 0,5 мл. С помощью инструмента MicroCap удалите и удалите MicroCaps из микротрубок PCT. Добавьте трипсин (см. Таблицу материалов) в соотношении 1 мкг на 30 мм2 ткани и вставьте MicroPestle в MicroTube с помощью инструмента MicroCap.
- Переложите микротрубки в бароцикловый картридж и соберите весь картридж. Поместите картридж в бароциклерную барокамеру и закрепите крышку. Бароцикл при 45 000 фунтов на квадратный дюйм в течение 50 с и атмосферное давление в течение 10 с при 50 °C в течение 60 циклов.
- После завершения бароциклирования переведите микротрубки в микроцентрифужную трубку объемом 0,5 мл и центрифугу в течение 2 мин при 4000 × г.
- Извлеките микротрубку из микроцентрифужной трубки объемом 0,5 мл с помощью инструмента «Колпачок». Осторожно удалите пестик с помощью колпачкового инструмента и промойте нижнюю половину пестика 20 мкл жидкой воды класса хроматографии-масс-спектрометрии (LC-MS) и соберите промывку в чистую микроцентрифужную трубку объемом 0,5 мл.
- Осторожно постучите по Микротрубке на столешнице, чтобы переместить жидкость на дно и перенести весь раствор из Микротрубки в трубку микроцентрифуги объемом 0,5 мл.
- Добавьте 20 мкл воды класса LC-MS в MicroTube и осторожно постучите по столешнице. Перенесите раствор для мытья в пробирку объемом 0,5 мл и повторите этот этап промывки еще раз.
- Вакуумная центрифуга для сушки образцов до ~2 мкл и добавления 100 мкл 100 мМ TEAB, рН 8,0.
- Определить концентрацию пептида с помощью колориметрического анализа (анализ бицинхониновой кислоты (BCA); см. Таблицу материалов) в соответствии с протоколом производителя.
8. Маркировка тандемных массовых тегов (TMT) и очистка EasyPep
ПРИМЕЧАНИЕ: Приблизительное время: 7 ч 20 мин (2 ч 20 мин без вакуумной центрифуги время сушки).
- Доведите изобарические реагенты для маркировки ТМТ до температуры окружающей среды перед открытием. Добавьте 500 мкл 100% ацетонитрила в каждый флакон ТМТ (5 мг). Инкубировать в течение 10 мин со случайными вихрями.
- Растворите 5 мкг пептидного образца в 100 мкл 100 мМ TEAB, рН 8,0 и добавьте 10 мкл данного реагента TMT. Создание и включение эталонных пулов, представляющих каждый отдельный образец в эксперименте, в каждый набор образцов мультиплексов TMT для облегчения количественной оценки образцов в нескольких мультиплексах TMT9. Инкубировать реакции в течение 1 ч при температуре окружающей среды со случайным встряхиванием/постукиванием.
- Погасите реакцию маркировки ТМТ путем добавления 10 мкл 5% гидроксиламина и инкубируйте в течение 30 мин при температуре окружающей среды с периодическим постукиванием. После закалки смешайте образцы, меченые ТМТ, в одну пробирку и высушите примерно до 200 мкл.
- Добавьте 1,800 мкл 0,1% муравьиной кислоты. Проверьте pH с помощью pH-бумаги: если pH ~3, добавьте 1 мл 0,1% муравьиной кислоты; если рН >3, добавляют 10-20 мкл 5% муравьиной кислоты до рН ~3. Добавьте 0,1% муравьиной кислоты, чтобы довести до конечного объема 3 мл.
- Снимите вкладку в нижней части колонки очистки пептидов, снимите колпачок и поместите его в коническую трубку объемом 15 мл. Перенесите образец с маркировкой TMT в столбец и продолжите очистку в соответствии с протоколом производителя.
- Вакуумная центрифуга для сушки элюированных пептидов до ~20 мкл, переноса во флакон LC с использованием бикарбоната аммония 25 мМ для конечного объема 80 мкл и перехода к автономному фракционированию.
9. Фракционирование и объединение мультиплексных образцов TMT
ПРИМЕЧАНИЕ: Приблизительное время: 3 ч 30 мин (1 ч 30 мин без вакуумной центрифуги время сушки).
- Фракционировать меченые ТМТ пептидные мультиплексы с помощью основной обратнофазной хроматографии на 96 фракций путем развития увеличивающегося линейного градиента (0,69% мин-1) подвижной фазы В (ацетонитрила) в подвижную фазу А (10 мМ NH4HCO3, рН 8,0).
- Генерация 36 сцепленных фракций путем объединения пробных скважин. Вакуумная центрифуга для сухих фракций до ~2 мкл и повторного суспендирования в бикарбонате аммония 25 мМ (конечная концентрация 1,5 мкг/10 мкл), центрифуги при 15 000 × г в течение 10-15 мин и переноса во флаконы ЛК для анализа РС.
10. Жидкостная хроматография тандемная масс-спектрометрия (LC-MS/MS)
ПРИМЕЧАНИЕ: Приблизительное время: В зависимости от приборного метода и экспериментального проектирования.
- Откалибруйте масс-спектрометр в соответствии с инструкциями/протоколами производителя.
- Подготовьте свежие подвижные фазы и стандарты и выполните соответствующие предварительные приготовления LC (включая, но не ограничиваясь, продувкой растворителей, промывным воздухом и сценариями испытаний на утечку для указанного прибора [см. Таблицу материалов]). Уравновешивайте предварительные и аналитические колонны и цикл отбора проб перед началом анализа.
- До и между последовательными мультиплексными анализами TMT проверьте, соответствует ли система LC-MS ранее протестированным показателям производительности, используя гарантию качества / контроль качества (QA / QC) TMT-меченые пептидные дайджесты и (например, MSPE (см. Таблицу материалов), HeLa).
- Загрузите флаконы автосэмплера в соответствующие позиции в автосамплере LC. Анализируйте отдельные фракции с помощью соответствующего метода градиента/МС. Чередуйте «промывочный» прогон со стандартом пептида (например, калибровка времени удержания пептида [PRTC]) примерно один раз в день для оценки хроматографических и масс-спектральных характеристик. После анализа каждой серии мультиплексных фракций TMT запустите эталонные стандарты QA/QC TMT для оценки производительности системы.
- Запустите процедуры оценки масс-спектрометра после эталонных стандартов QA/QC TMT для оценки производительности после отбора проб, а затем откалибруйте систему, как указано на шаге 10.1 для следующего набора образцов.
11. Биоинформационный анализ данных
ПРИМЕЧАНИЕ: Приблизительное время: Экспериментальный дизайн зависит.
- Перенесите все образцы данных (например, файлы .raw) на соответствующий сетевой накопитель/компьютерный диск.
- Поиск всех фракций вместе с использованием приложения для анализа требуемых данных (например, Proteome Discover, Mascot) с использованием соответствующих параметров9 в справочной базе данных видового белка для генерации пептидных спектральных совпадений (PSM) и извлечения интенсивности сигнала ионов репортера TMT. Фильтрация PSM на основе соответствующих показателей контроля качества и агрегированная нормализованная медиана логарифмического 2-преобразованного коэффициента репортерных ионов TMT в глобальные уровни содержания белка, как описано ранее 3,9.
- Сравните изменения белка в интересующих условиях с помощью желаемого программного обеспечения для дифференциального анализа.
Результаты
Свежезамороженные тонкие участки тканей у двух пациентов с HGSOC и двумя ocCC были проанализированы с использованием этого интегрированного процесса идентификации ROI тканей на основе ИИ, сегментации, LMD и количественного протеомного анализа (рисунок 1). Репрезентативные H&E-окрашенные участки ткани для каждой опухоли были рассмотрены сертифицированным патологоанатомом; клеточность опухоли колебалась от 70% до 99%. Ткани были тонко сечены на слайдах мембраны PEN (Дополнительный файл 2) и предварительно вырезаны с помощью калибраторных фидуциалов (Дополнительный файл 1), что позволило интегрировать данные позиционной ориентации из аннотаций, сгенерированных в программном обеспечении для анализа изображений (см. Таблицу материалов) с декартовой ориентацией координат в программном обеспечении LMD. После окрашивания H&E были получены изображения с высоким разрешением (20x) слайдов PEN, содержащих ткань плюс калибраторы.
Популяции опухолей и стромальных клеток на микроснимках были сегментированы с использованием программного обеспечения для анализа изображений (см. Таблицу материалов) для селективного сбора LMD, наряду с урожаями, представляющими весь тонкий участок ткани (например, всю опухолевую ткань) (рисунок 1). Недискриминационные аннотации для целых коллекций опухолевых тканей были получены путем разделения всего участка ткани плитками размером 500мкм2, оставляя зазор в 40 мкм между плитками для поддержания целостности мембраны PEN и предотвращения скручивания мембраны во время LMD. На слайдах для обогащения LMD с гистологическим разрешением классификатор ИИ в программном обеспечении для анализа изображений (см. Таблицу материалов) был обучен различать опухолевые и стромальные клетки вместе с пустым стеклянным фоном слайда. Репрезентативные области опухоли, стромы и пустого стекла были выделены вручную, и инструмент классификатора использовался для сегментации этих ROI по всему участку ткани. Сегментированные слои, представляющие целую ткань, эпителий опухоли и строму, были сохранены отдельно как отдельные файлы .annotation (Дополнительный файл 3 и Дополнительный файл 6). В отдельной копии файла изображения (без секционированных аннотаций ROI) короткая строка от самого центрального наконечника каждого из трех фидуциальных калибраторов была аннотирована и сохранена в виде файла .annotation с тем же именем файла, что и каждый из файлов слоя аннотаций LMD, но с добавлением суффикса «_calib» (Дополнительный файл 4). Эти линии использовались для совместной регистрации положения калибраторов мембраны PEN с данными списка форм аннотаций, полученными в программном обеспечении для анализа изображений.
Настоящее исследование предоставляет два алгоритма, «Malleator» и «Dapọ» на Python для поддержки этого рабочего процесса LMD на основе ИИ, которые доступны в https://github.com/GYNCOE/Mitchell.et.al.2022. Алгоритм Malleator извлекает определенные декартовы координаты для всех отдельных аннотаций (окупаемость инвестиций в ткани и калибраторы) из парных файлов .annotation и объединяет их в один файл импорта XML (дополнительный файл 5). В частности, алгоритм Malleator использует имя каталога из родительской папки в качестве входных данных для поиска во всех папках подкаталогов и создает .xml файлы для всех вложенных папок, которые еще не имеют .xml объединенного файла. Алгоритм Malleator объединяет все слои аннотаций в программном обеспечении для анализа изображений (см. Таблицу материалов) в один слой и преобразует сгенерированные ИИ данные списка фигур, которые сохраняются как проприетарный тип файла .annotation, в формат .xml, совместимый с программным обеспечением LMD. После объединения файлов аннотаций и калибратора сгенерированный алгоритмом .xml файл сохраняется и импортируется в программное обеспечение LMD. Небольшие корректировки необходимы для ручной регулировки выравнивания аннотаций, что также служит для регистрации вертикального (z-плоскостного) положения слайд-сцены на лазерном микроскопе. Алгоритм Dapọ используется специально для коллекций, обогащенных LMD. Секционированные плитки автоматически назначаются отдельным слоям аннотаций программным обеспечением для анализа изображений. Алгоритм Dapọ объединяет все секционированные плитки в один слой аннотаций перед использованием инструмента Classifier, тем самым уменьшая время выполнения анализа Classifier для коллекций, обогащенных LMD.
Вся опухоль и образцы тканей, обогащенные LMD, были переварены, помечены реагентами TMT, мультиплексированы, фракционированы в автономном режиме и проанализированы с помощью количественной протеомики на основе MS, как описано ранее9. Средний выход пептидов (43-60 мкг) и извлечение (0,46-0,59 мкг/мм2) для образцов, собранных с использованием этого рабочего процесса, управляемого ИИ, были сопоставимы с предыдущими отчетами 9,10. В общей сложности 5 971 белок был совместно количественно определен во всех образцах (дополнительная таблица S1). Неконтролируемая иерархическая кластеризация с использованием 100 наиболее изменчивых белков привела к сегрегации гистотипов HGSOC и OCCC от обогащенных LMD и цельных опухолевых образцов (рисунок 2A), аналогично описанному ранее11. Напротив, образцы стромы, обогащенные LMD, из HGSOC и OCCC сгруппированы вместе и независимо от обогащенных LMD образцов опухоли и целой опухоли. Среди 5 971 количественного белка 215 были значительно изменены (LIMMA adj. p < 0,05) между целыми опухолевыми коллекциями из образцов HGSOC и OCCC (дополнительная таблица S2). Эти измененные белки сравнивали с теми, которые были идентифицированы для дифференциации опухолевой ткани HGSOC и OCCC Hughes et al.11. Из 76 сигнатурных белков, количественно оцененных Hughes et al., 57 были совместно количественно определены в этом наборе данных и были сильно коррелированы (Spearman Rho = 0,644, p < 0,001) (рисунок 2B).
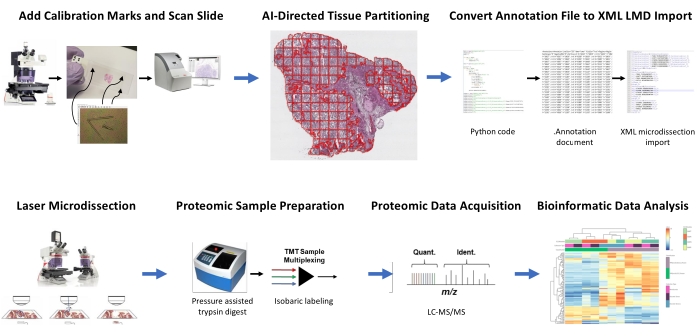
Рисунок 1: Сводка интегрированного рабочего процесса для автоматизированного выбора интересующей области ткани для лазерной микродиссекции для последующей количественной протеомики. Калибровочные фидуциалы вырезаются на слайдах мембраны PEN для совместного регистрации данных позиционной ориентации из производных ИИ сегментов roI тканей в программном обеспечении для анализа изображений HALO с горизонтальным позиционированием на микроскопе LMD. Алгоритм Malleator используется для объединения аннотированных данных сегментации во всех слоях аннотации слайда с _calib ссылочным файлом и для преобразования его в файл .xml, совместимый с программным обеспечением LMD. LMD-собранная ткань для протеомного анализа переваривается и анализируется с помощью высокопроизводительной количественной протеомики, как описано ранее9. Сокращения: LMD = лазерная микродиссекция; ROI = интересующий регион; TMT = тандемный массовый тег; Квант. = количественная оценка; Идентификация. = идентификация; LC-MS/MS = жидкостная хроматография-тандемная масс-спектрометрия. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
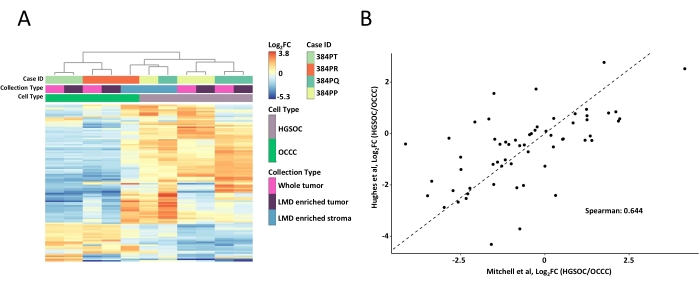
Рисунок 2: Анализ белков в образцах опухолей, обогащенных LMD, и цельных опухолей. (A) Неконтролируемый иерархический кластерный анализ 100 наиболее изменчиво распространенных белков в образцах HGSOC и OCCC LMD и целых опухолевых образцов. (B) Корреляция логарифмического2-кратного изменения содержания белка между сборами целых опухолей HGSOC и OCCC в настоящем исследовании (Mitchell et al., x-axis) и аналогичном исследовании Hughes et al. (y-axis)11. Сокращения: LMD = лазерная микродиссекция; HGSOC = серозный рак яичников высокой степени; OCCC = клироклеточная карцинома яичников; log2FC = log2-преобразованное протеомное изобилие. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительная таблица S1: Обилие 5 971 белка, совместно количественно оцененных во всех обогащенных LMD и цельных образцах опухоли из образцов тканей HGSOC и OCCC. Сокращения: LMD = лазерная микродиссекция; HGSOC = серозный рак яичников высокой степени; OCCC = прозрачно-клеточная карцинома яичников. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительная таблица S2: Дифференциально экспрессированные белки (215) в целых опухолевых коллекциях из HGSOC против OCCC (LIMMA adj. p < 0,05). Сокращения: HGSOC = серозный рак яичников высокой степени; OCCC = прозрачно-клеточная карцинома яичников. Пожалуйста, нажмите здесь, чтобы загрузить эту таблицу.
Дополнительный файл 1: Файл репрезентативного списка фигур (.sld), содержащий стандартные фидуциалы калибратора для четырех позиций слайдов. Файл может быть импортирован в программное обеспечение LMD. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: Репрезентативный файл изображения .svs окрашенного H&E участка ткани высокого разрешения (20x). Файл можно открыть и просмотреть с помощью программного обеспечения для анализа изображений или программного обеспечения LMD. Аббревиатура: H&E = гематоксилин и эозин; LMD = лазерная микродиссекция. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: Репрезентативный файл .annotation секционированных целых сегментов опухоли. Файл может быть импортирован в программное обеспечение для анализа изображений. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 4: Репрезентативный файл _calib.annotation калибратора фидуциальных сегментов. Координатная информация представляет собой восточное позиционирование коротких калибраторных линий, нарисованных от каждого наконечника стрелки. Файл может быть импортирован в программное обеспечение для анализа изображений. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 5: Репрезентативный файл расширяемого языка разметки (.xml), сгенерированный алгоритмом Malleator. Файл может быть импортирован в программное обеспечение лазерной микродиссекции. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 6: Репрезентативный файл .annotation секционированных сегментов, классифицированных AI для коллекций, обогащенных LMD. Файл может быть импортирован в программное обеспечение для анализа изображений. Сокращения: ИИ = искусственный интеллект; LMD = лазерная микродиссекция. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Несмотря на то, что существует множество прецедентов исследований, направленных на разработку и/или совершенствование рабочих процессов обогащения целевых клеточных субпопуляций из FFPE и/или свежезамороженных тканей, а также методологий поддержания качества образцов во время обработки 9,12,13,14,15, существует существенная потребность в разработке автоматизированных стратегий подготовки образцов клинических тканей к молекулярному анализу для снижения изменчивости и повысить воспроизводимость. Этот рабочий процесс описывает стандартизированный, полуавтоматизированный протокол, который интегрирует существующие программные инструменты анализа изображений (см. Таблицу материалов) для сбора дискретных клеточных популяций с гистологическим разрешением с помощью LMD из образцов клинических тканей.
Пространственно-разрешенное обогащение LMD ROI, захватывающее дискретные клеточные популяции, представляет собой этап обработки тканей следующего поколения до многоомного анализа для улучшения молекулярной характеристики и идентификации и облегчения обнаружения селективных биомаркеров. Этот протокол улучшает существующие методологии, уменьшая часто длительное воздействие участков тканей на окружающую среду, что связано с ручной сегментацией ROI гистологом (что может занять >1-2 ч до сбора LMD). Вместо этого этот рабочий процесс позволяет предварительно идентифицировать ROI с помощью классификации и сегментации под руководством ИИ. Ограничение времени пребывания тканей уменьшит ложные вариации в оценках высоколабильных молекулярных мишеней, таких как фосфопептиды и мРНК, или для аналитических методов на основе антител, которые полагаются на то, что целевой белок находится в своей нативной конформации для обнаружения.
Вырезание аккуратных фидуциалов калибратора на слайде мембраны PEN, которые хорошо видны на отсканированном изображении слайда, является одним из ключевых компонентов, обеспечивающих интеграцию программного обеспечения для анализа изображений (см. Таблицу материалов) с рабочим процессом LMD. Обеспечение того, чтобы калибраторы имели точную («чистую») точку в нижней части V-образной формы, позволяет выбрать точную точку в программном обеспечении для анализа изображений для калибраторных линий, из которых должны быть взяты линии, как описано в шагах 5.1.6 и 5.2.13. Выравнивание этих точек во время импорта в программное обеспечение LMD имеет решающее значение для правильного наложения аннотаций (облегчается путем создания совместимого файла .xml с использованием алгоритмов «Malleator» и / или «Dapọ») на соответствующую рентабельность инвестиций в ткани на физическом слайде LMD. Необходимо выделить все формы и коллективно «перетащить» на место, даже если выравнивание точно при импорте в программное обеспечение LMD, чтобы зарегистрировать вертикальное (z-плоскостное) положение слайд-сцены на лазерном микроскопе. Незначительные корректировки в позиционировании аннотаций над ROI ткани также могут быть сделаны на этом этапе, если это необходимо.
Ограничение текущей версии алгоритма Malleator заключается в том, что он несовместим с предопределенными инструментами формы аннотаций, предоставляемыми программным обеспечением для анализа изображений (см. Таблицу материалов), хотя будущие обновления / версии алгоритма будут направлены на улучшение этой совместимости. Файл .annotation для фигур, нарисованных с помощью этих инструментов, содержит только два набора парных координат x и y для каждой аннотации без полной пространственной ориентации вокруг этих точек. Текущее использование этих инструментов приводит к тому, что аннотации преобразуются в прямые линии, определенные только двумя точками в процессе импорта. Ручное определение сегментов ROI ткани требуется для успешного преобразования в формат XML и импорта LMD. Это может быть выполнено либо путем ручного определения каждого ROI с индивидуальными полигональными аннотациями свободной руки, специфичными для целевой области, либо путем применения приближенной круговой или прямоугольной аннотации ко всем сегментам ROI ткани, если это необходимо, и будет совместимо с этим рабочим процессом.
В то время как рабочий процесс, представленный здесь, был продемонстрирован для протеомного анализа свежезамороженных образцов ткани рака человека, этот рабочий процесс LMD на основе ИИ может быть эквивалентно использован с тканями FFPE, нераковыми типами тканей и теми, которые получены из нечеловеческих источников. Он также может поддерживать другие последующие рабочие процессы молекулярного профилирования, включая транскриптомный, геномный или фосфопротеомный анализ. Этот рабочий процесс может также использовать другие виды использования программного обеспечения для анализа изображений (см. Таблицу материалов), в том числе с возможностями, связанными с подсчетом клеток или другими аналитическими модулями, включая модуль «Multiplex IHC» или «Дополнение тканевого микрочипа (TMA)». Будущие приложения этого рабочего процесса также могут извлечь выгоду из предварительного определения количества клеток на сегмент ROI, тем самым обеспечивая эквивалентные клеточные входы в нескольких коллекциях, или используя альтернативные методы определения клеточных ROI, представляющих интерес, такие как иммуногистохимия или клеточная социология.
Раскрытие информации
T.P.C. является членом SAB компании ThermoFisher Scientific, Inc и получает финансирование исследований от AbbVie.
Благодарности
Финансирование этого проекта было частично предоставлено Программой оборонного здравоохранения (HU0001-16-2-0006 и HU0001-16-2-00014) Университету военных служб для Центра передового опыта гинекологического рака. Спонсоры не играли никакой роли в разработке, исполнении, интерпретации или написании исследования. Отказ: Мнения, выраженные в настоящем документе, являются мнениями авторов и не отражают официальную политику Министерства армии/флота/ВОЕННО-воздушных сил, Министерства обороны или Правительства США.
Материалы
| Name | Company | Catalog Number | Comments |
| 1260 Infinity II System | Agilent Technologies Inc | Offline LC system | |
| 96 MicroCaps (150uL) in bulk | Pressure Biosciences Inc | MC150-96 | |
| 96 MicroPestles in bulk | Pressure Biosciences Inc | MP-96 | |
| 96 MicroTubes in bulk (no caps) | Pressure Biosciences Inc | MT-96 | |
| 9mm MS Certified Clear Screw Thread Kits | Fisher Scientific | 03-060-058 | Sample vial for offline LC frationation and mass spectrometry |
| Acetonitrile, Optima LC/MS Grade | Fisher Chemical | A995-4 | Mobile phase solvent |
| Aperio AT2 | Leica Microsystems | 23AT2100 | Slide scanner |
| Axygen PCR Tubes with 0.5 mL Flat Cap | Fisher Scientific | 14-222-292 | Sample tubes; size fits PCT tubes and thermocycler |
| Barocycler 2320EXT | Pressure Biosciences Inc | 2320-EXT | Barocycler |
| BCA Protein Assay Kit | Fisher Scientific | P123225 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Easy-nLC 1200 | Thermo Fisher Scientific | Liquid Chromatography | |
| EasyPep Maxi Sample Prep Kit | Thermo Fisher Scientific | NCI5734 | Post-label sample clean up column |
| EASY-SPRAY C18 2UM 50CM X 75 | Fisher Scientific | ES903 | Analytical column |
| Eosin Y Solution Aqueous | Sigma Aldrich | HT110216 | |
| Formic Acid, 99+ % | Thermo Fisher Scientific | 28905 | Mobile phase additive |
| ggplot2 version 3.3.5 | CRAN | https://cran.r-project.org/web/packages/ggplot2/ | |
| HALO | Indica Labs | Image analysis software | |
| IDLE (Integrated Development and Learning Environment) | Python Software Foundation | ||
| iheatmapr version 0.5.1 | CRAN | https://cran.r-project.org/web/packages/iheatmapr/ | |
| iRT Kit | Biognosys | Ki-3002-1 | LC-MS QAQC Standard |
| limma version 3.42.2 | Bioconductor | https://bioconductor.org/packages/release/bioc/html/limma.html | |
| LMD Scanning stage Ultra LMT350 | Leica Microsystems | 11888453 | LMD stage model outfitted with PCT tube holder |
| LMD7 (software version 8.2.3.7603) | Leica Microsystems | LMD apparatus (microscope, laser, camera, PC, tablet) | |
| Mascot Server | Matrix Science | Data analysis software | |
| Mass Spec-Compatible Human Protein Extract, Digest | Promega | V6951 | LC-MS QAQC Standard |
| Mayer’s Hematoxylin Solution | Sigma Aldrich | MHS32 | |
| PEN Membrane Glass Slides | Leica Microsystems | 11532918 | |
| Peptide Retention Time Calibration Mixture | Thermo Fisher Scientific | 88321 | LC-MS QAQC Standard |
| Phosphatase Inhibitor Cocktail 2 | Sigma Aldrich | P5726 | |
| Phosphatase Inhibitor Cocktail 3 | Sigma Aldrich | P0044 | |
| Pierce LTQ Velos ESI Positive Ion Calibration Solution | Thermo Fisher Scientific | 88323 | Instrument calibration solution |
| PM100 C18 3UM 75UMX20MM NV 2PK | Fisher Scientific | 164535 | Pre-column |
| Proteome Discoverer | Thermo Fisher Scientific | OPTON-31040 | Data analysis software |
| Python | Python Software Foundation | ||
| Q Exactive HF-X | Thermo Fisher Scientific | Mass spectrometer | |
| R version 3.6.0 | CRAN | https://cran-archive.r-project.org/bin/windows/base/old/2.6.2/ | |
| RColorBrewer version 1.1-2 | CRAN | https://cran.r-project.org/web/packages/RColorBrewer/ | |
| Soluble Smart Digest Kit | Thermo Fisher Scientific | 3251711 | Digestion reagent |
| TMTpro 16plex Label Reagent Set | Thermo Fisher Scientific | A44520 | isobaric TMT labeling reagents |
| Veriti 60 well thermal cycler | Applied Biosystems | 4384638 | Thermocycler |
| Water, Optima LC/MS Grade | Fisher Chemical | W6-4 | Mobile phase solvent |
| ZORBAX Extend 300 C18, 2.1 x 12.5 mm, 5 µm, guard cartridge (ZGC) | Agilent Technologies Inc | 821125-932 | Offline LC trap column |
| ZORBAX Extend 300 C18, 2.1 x 150 mm, 3.5 µm | Agilent Technologies Inc | 763750-902 | Offline LC analytical column |
Ссылки
- Motohara, T., et al. An evolving story of the metastatic voyage of ovarian cancer cells: cellular and molecular orchestration of the adipose-rich metastatic microenvironment. Oncogene. 38 (16), 2885-2898 (2019).
- Aran, D., Sirota, M., Butte, A. J. Systematic pan-cancer analysis of tumour purity. Nature Communications. 6, 8971 (2015).
- Hunt, A. L., et al. Extensive three-dimensional intratumor proteomic heterogeneity revealed by multiregion sampling in high-grade serous ovarian tumor specimens. iScience. 24 (7), 102757 (2021).
- Dou, Y., et al. Proteogenomic characterization of endometrial carcinoma. Cell. 180 (4), 729-748 (2020).
- Zhang, H., et al. Integrated proteogenomic characterization of human high-grade serous ovarian cancer. Cell. 166 (3), 755-765 (2016).
- Gillette, M. A., et al. Proteogenomic characterization reveals therapeutic vulnerabilities in lung adenocarcinoma. Cell. 182 (1), 200-225 (2020).
- Silvestri, A., et al. Protein pathway biomarker analysis of human cancer reveals requirement for upfront cellular-enrichment processing. Laboratory Investigation. 90 (5), 787-796 (2010).
- Echle, A., et al. Deep learning in cancer pathology: a new generation of clinical biomarkers. British Journal of Cancer. 124 (4), 686-696 (2021).
- Lee, S., et al. Molecular analysis of clinically defined subsets of high-grade serous ovarian cancer. Cell Reports. 31 (2), 107502 (2020).
- Xuan, Y., et al. Standardization and harmonization of distributed multi-center proteotype analysis supporting precision medicine studies. Nature Communications. 11 (1), 5248 (2020).
- Hughes, C. S., et al. Quantitative profiling of single formalin fixed tumour sections: proteomics for translational research. Scientific Reports. 6 (1), 34949 (2016).
- Espina, V., et al. A portrait of tissue phosphoprotein stability in the clinical tissue procurement process. Molecular & Cellular Proteomics. 7 (10), 1998-2018 (2008).
- Espina, V., Heiby, M., Pierobon, M., Liotta, L. A. Laser capture microdissection technology. Expert Review of Molecular Diagnostics. 7 (5), 647-657 (2007).
- Havnar, C. A., et al. Automated dissection protocol for tumor enrichment in low tumor content tissues. Journal of Visualized Experiments. (169), e62394 (2021).
- Mueller, C., et al. One-step preservation of phosphoproteins and tissue morphology at room temperature for diagnostic and research specimens. PLoS One. 6 (8), (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены