Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Создание представления транскрипционной регуляции транскриптомных признаков для задачи прогнозирования и обнаружения темных биомаркеров на небольших наборах данных
В этой статье
Резюме
Здесь мы представляем протокол для преобразования транскриптомных данных в представление mqTrans, позволяющий идентифицировать темные биомаркеры. Несмотря на то, что эти биомаркеры не экспрессируются дифференциально в обычном транскриптомном анализе, они демонстрируют дифференциальную экспрессию в представлении mqTrans. Этот подход служит дополнением к традиционным методам, выявляя ранее упущенные биомаркеры.
Аннотация
Транскриптом представляет собой уровни экспрессии многих генов в образце и широко используется в биологических исследованиях и клинической практике. Исследователи обычно сосредотачивались на транскриптомных биомаркерах с дифференциальными представлениями между фенотипической группой и контрольной группой образцов. В этом исследовании была представлена многозадачная структура обучения сети графового внимания (GAT) для изучения сложных межгенных взаимодействий эталонных образцов. Демонстрационная референсная модель была предварительно обучена на здоровых образцах (HealthModel), которая может быть непосредственно использована для создания представления количественной транскрипционной регуляции на основе модели (mqTrans) независимых тестовых транскриптомов. Сгенерированный mqTrans вид транскриптомов был продемонстрирован задачами прогнозирования и детектирования темных биомаркеров. Термин «темный биомаркер» возник из-за его определения, согласно которому темный биомаркер демонстрирует дифференциальное представление в представлении mqTrans, но не дифференциальную экспрессию на исходном уровне экспрессии. Темный биомаркер всегда упускался из виду в традиционных исследованиях обнаружения биомаркеров из-за отсутствия дифференциальной экспрессии. Исходный код и руководство по конвейеру HealthModelPipe можно скачать с http://www.healthinformaticslab.org/supp/resources.php.
Введение
Транскриптом состоит из экспрессии всех генов в образце и может быть профилирован с помощью высокопроизводительных технологий, таких как микрочип и РНК-секвенирование1. Уровни экспрессии одного гена в наборе данных называются транскриптомным признаком, а дифференциальное представление транскриптомного признака между фенотипом и контрольной группами определяет этот ген как биомаркер этого фенотипа 2,3. Транскриптомные биомаркеры широко используются в исследованиях диагностики заболеваний4, биологического механизма5, анализа выживаемости 6,7 и т.д.
Паттерны активности генов в здоровых тканях несут важнейшую информацию о жизни 8,9. Эти закономерности дают неоценимую информацию и служат идеальными справочными материалами для понимания сложных траекторий развития доброкачественных заболеваний10,11 и смертельных заболеваний12. Гены взаимодействуют друг с другом, и транскриптомы представляют собой конечные уровни экспрессии после их сложных взаимодействий. Такие паттерны формулируются как транскрипционная регуляционная сеть13 и метаболическая сеть14 и др. Экспрессия матричных РНК (мРНК) может транскрипционно регулироваться транскрипционными факторами (ТФ) и длинными межгенными некодирующими РНК (линкРНК)15,16,17. Традиционный анализ дифференциальной экспрессии игнорировал такие сложные взаимодействия генов с предположением о независимости между признаками18,19.
Недавние достижения в области графовых нейронных сетей (GNN) демонстрируют необычайный потенциал в извлечении важной информации из данных, основанных на OMIC, для исследованийрака20, например, идентификация модулей коэкспрессии21. Врожденная способность GNN делает их идеальными для моделирования сложных взаимоотношений и зависимостей между генами22,23.
Биомедицинские исследования часто сосредоточены на точном прогнозировании фенотипа по сравнению с контрольной группой. Такие задачи обычно формулируются в виде бинарных классификаций 24,25,26. Здесь две метки классов обычно кодируются как 1 и 0, true и false или даже positive и negative27.
Это исследование было направлено на предоставление простого в использовании протокола для создания представления транскрипционной регуляции (mqTrans) набора данных транскриптома на основе предварительно обученной эталонной модели сети графового внимания (GAT). Для преобразования транскриптомных признаков в признаки mqTrans был использован многозадачный фреймворк GAT из ранее опубликованной работы26 . Большой набор данных здоровых транскриптомов из платформы Xena28 Калифорнийского университета в Санта-Крузе (UCSC) был использован для предварительного обучения референсной модели (HealthModel), которая количественно измеряла регуляции транскрипции от регуляторных факторов (ТФ и линкРНК) до целевых мРНК. Сгенерированное представление mqTrans может быть использовано для построения моделей прогнозирования и обнаружения темных биомаркеров. В этом протоколе в качестве иллюстративного примера используется набор данных пациентов с аденокарциномой толстой кишки (COAD) из базы данных29 Атласа генома рака (TCGA). В этом контексте пациенты на I или II стадиях классифицируются как отрицательные образцы, в то время как пациенты на III или IV стадиях считаются положительными образцами. Также сравнивается распределение темновых и традиционных биомаркеров по 26 типам рака TCGA.
Описание конвейера HealthModel
Методология, используемая в этом протоколе, основана на ранее опубликованной структуре26, как показано на рисунке 1. Для начала пользователям необходимо подготовить входной набор данных, передать его в предлагаемый конвейер HealthModel и получить функции mqTrans. Подробные инструкции по подготовке данных приведены в разделе 2 раздела протокола. После этого у пользователей есть возможность комбинировать признаки mqTrans с исходными транскриптомными признаками или продолжать только с сгенерированными признаками mqTrans. Затем полученный набор данных подвергается процессу выбора признаков, при этом пользователи могут выбрать предпочтительное значение для k в k-кратной перекрестной проверке для классификации. Основным оценочным показателем, используемым в этом протоколе, является точность.
HealthModel26 классифицирует транскриптомные признаки по трем отдельным группам: TF (транскрипционный фактор), lincRNA (длинная межгенная некодирующая РНК) и mRNA (матричная РНК). Признаки TF определяются на основе аннотаций, доступных в Атласе белков человека30,31. В данной работе используются аннотации линкРНК из набора данных GTEx32. Гены, принадлежащие к путям третьего уровня в базе данных KEGG33, рассматриваются как признаки мРНК. Стоит отметить, что если признак мРНК проявляет регуляторную роль для гена-мишени, как это задокументировано в базе данных TRRUST34, он реклассифицируется в класс TF.
Этот протокол также вручную генерирует два файла примеров для идентификаторов генов регуляторных факторов (regulatory_geneIDs.csv) и мРНК-мишеней (target_geneIDs.csv). Матрица попарных расстояний между регуляторными признаками (ТФ и линкРНК) вычисляется с помощью коэффициентов корреляции Пирсона и кластеризуется с помощью популярного инструментального взвешенного сетевого анализа генной коэкспрессии (WGCNA)36 (adjacent_matrix.csv). Пользователи могут напрямую использовать конвейер HealthModel вместе с этими примерами файлов конфигурации для создания представления mqTrans набора транскриптомных данных.
Технические характеристики HealthModel
HealthModel представляет сложные взаимосвязи между ТФ и линкРНК в виде графа, где входные объекты служат вершинами, обозначенными V , и матрицей межвершинных ребер, обозначенной как E. Каждый образец характеризуется К-регуляторными признаками, обозначаемыми как VK×1. В частности, набор данных включал 425 ТФ и 375 линкРНК, в результате чего размерность выборки составила K = 425 + 375 = 800. Для установления матрицы кромок E в этой работе использовался популярный инструмент WGCNA35. Попарный вес, связывающий две вершины, представленные как 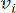 и
и 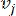 , определяется коэффициентом корреляции Пирсона. Генная регуляторная сеть имеет безмасштабную топологию36, характеризующуюся присутствием генов-концентраторов с ключевыми функциональными ролями. Мы вычисляем корреляцию между двумя объектами или вершинами,
, определяется коэффициентом корреляции Пирсона. Генная регуляторная сеть имеет безмасштабную топологию36, характеризующуюся присутствием генов-концентраторов с ключевыми функциональными ролями. Мы вычисляем корреляцию между двумя объектами или вершинами, 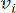 и
и 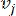 , используя меру топологического перекрытия (TOM) следующим образом:
, используя меру топологического перекрытия (TOM) следующим образом:
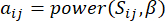 (1)
(1)
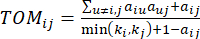 (2)
(2)
Мягкий пороговый β вычисляется с помощью функции 'pickSoft Threshold' из пакета WGCNA. Применяется степенная экспоненциальная функция a ij, где 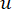 представляет ген, исключая i и j, и
представляет ген, исключая i и j, и 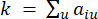 представляет связность вершин. WGCNA кластеризует профили экспрессии транскриптомных признаков в несколько модулей, используя широко используемую меру несходства (
представляет связность вершин. WGCNA кластеризует профили экспрессии транскриптомных признаков в несколько модулей, используя широко используемую меру несходства (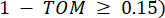 37.
37.
Фреймворк HealthModel изначально разрабатывался как многозадачная архитектура обучения26. Этот протокол использует только задачу предварительного обучения модели для построения транскриптомного представления mqTrans. Пользователь может дополнительно усовершенствовать предварительно обученную модель HealthModel в многозадачной графовой сети внимания с помощью дополнительных транскриптомных образцов, специфичных для конкретной задачи.
Технические сведения о выборе и классификации функций
Пул выбора признаков реализует одиннадцать алгоритмов выбора признаков (FS). Среди них три алгоритма ФС на основе фильтров: выбор K лучших признаков с использованием максимального коэффициента информации (SK_mic), выбор K признаков на основе FPR MIC (SK_fpr) и выбор K объектов с наибольшим уровнем ложного обнаружения MIC (SK_fdr). Кроме того, три древовидных алгоритма ФС оценивают отдельные признаки с помощью дерева решений с индексом Джини (DT_gini), адаптивного дерева решений (AdaBoost) и случайного леса (RF_fs). Пул также включает в себя два метода-оболочки: рекурсивное исключение признаков с помощью классификатора линейных опорных векторов (RFE_SVC) и рекурсивное исключение признаков с классификатором логистической регрессии (RFE_LR). Наконец, включены два алгоритма внедрения: линейный классификатор SVC с самыми ранжированными значениями важности признаков L1 (lSVC_L1) и классификатор логистической регрессии с самыми ранжированными значениями важности признаков L1 (LR_L1).
Пул классификаторов использует семь различных классификаторов для построения моделей классификации. Эти классификаторы включают в себя линейный метод опорных векторов (SVC), наивный байесовский метод Гаусса (GNB), классификатор логистической регрессии (LR), k-ближайшего соседа, с k, установленным в 5 по умолчанию (KNN), XGBoost, случайный лес (RF) и дерево решений (DT).
Случайное разбиение датасета на обучающие: тестовые подмножества можно задать в командной строке. В приведенном примере используется соотношение train: test = 8:2.
Access restricted. Please log in or start a trial to view this content.
протокол
ПРИМЕЧАНИЕ: Следующий протокол описывает детали аналитической процедуры информатики и команд Python основных модулей. На рисунке 2 показаны три основных шага с примерами команд, используемых в этом протоколе, и ссылки на ранее опубликованные работы26,38 для получения более подробной технической информации. Выполните следующие действия по следующему протоколу под обычной учетной записью пользователя в компьютерной системе и избегайте использования учетной записи администратора или root. Это вычислительный протокол, в котором отсутствуют биомедицинские опасные факторы.
1. Подготовьте среду Python
- Создайте виртуальную среду.
- В этом исследовании использовался язык программирования Python и виртуальная среда Python (VE) с Python 3.7. Выполните следующие действия (Рисунок 3A):
conda create -n healthmodel python=3.7
conda create - это команда для создания нового VE. Параметр -n указывает имя новой среды, в данном случае healthmodel. А python=3.7 указывает устанавливаемую версию Python. Выберите любое предпочтительное имя и версию Python, поддерживающую приведенную выше команду. - После выполнения команды результат будет аналогичен рисунку 3B. Введите y и дождитесь завершения процесса.
- В этом исследовании использовался язык программирования Python и виртуальная среда Python (VE) с Python 3.7. Выполните следующие действия (Рисунок 3A):
- Активация виртуальной среды
- В большинстве случаев активируйте созданную VE следующей командой (рисунок 3C):
Conda Активация HealthModel - Следуйте инструкциям для активации VE для конкретной платформы, если некоторые платформы требуют, чтобы пользователь загрузил файлы конфигурации для конкретной платформы для активации.
- В большинстве случаев активируйте созданную VE следующей командой (рисунок 3C):
- Установка PyTorch 1.13.1
- PyTorch — это популярный пакет Python для алгоритмов искусственного интеллекта (ИИ). В качестве примера используйте PyTorch 1.13.1 на основе платформы программирования графического процессора CUDA 11.7. Другие версии можно найти на https://pytorch.org/get-started/previous-versions/. Используйте следующую команду (рисунок 3D):
PIP3 Установить фонарик TorchVision TorchAudio
ПРИМЕЧАНИЕ: Настоятельно рекомендуется использовать PyTorch версии 1.12 или более поздней. В противном случае установка необходимого пакета torch_geometric может быть сложной задачей, как отмечено на официальном сайте torch_geometric: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch — это популярный пакет Python для алгоритмов искусственного интеллекта (ИИ). В качестве примера используйте PyTorch 1.13.1 на основе платформы программирования графического процессора CUDA 11.7. Другие версии можно найти на https://pytorch.org/get-started/previous-versions/. Используйте следующую команду (рисунок 3D):
- Установка дополнительных пакетов для резака-геометрии
- Следуя рекомендациям, приведенным в https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, установите следующие пакеты: torch_scatter, torch_sparse, torch_cluster и torch_spline_conv с помощью команды (рисунок 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- Следуя рекомендациям, приведенным в https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, установите следующие пакеты: torch_scatter, torch_sparse, torch_cluster и torch_spline_conv с помощью команды (рисунок 3E):
- Установите пакет torch-geometric .
- Для этого исследования требуется специальная версия 2.2.0 пакета факел-геометрия . Выполните команду (рисунок 3F):
pip install torch_geometric==2.2.0
- Для этого исследования требуется специальная версия 2.2.0 пакета факел-геометрия . Выполните команду (рисунок 3F):
- Установите другие пакеты.
- Такие пакеты, как pandas, обычно доступны по умолчанию. Если нет, установите их с помощью команды pip. Например, чтобы установить pandas и xgboost, выполните:
pip install pandas
pip install xgboost
- Такие пакеты, как pandas, обычно доступны по умолчанию. Если нет, установите их с помощью команды pip. Например, чтобы установить pandas и xgboost, выполните:
2. Использование предварительно обученной модели HealthModel для генерации признаков mqTrans
- Скачайте код и предварительно обученную модель.
- Загрузите код и предварительно обученную модель HealthModel с веб-сайта: http://www.healthinformaticslab.org/supp/resources.php, который называется HealthModel-mqTrans-v1-00.tar.gz (рисунок 4A). Загруженный файл можно распаковать по указанному пользователем пути. Подробную формулировку и вспомогательные данные внедренного протокола можно найти в26.
- Введите параметры для запуска HealthModel.
- Во-первых, измените рабочий каталог на папку HealthModel-mqTrans в командной строке. Используйте следующий синтаксис для выполнения кода:
python main.py <папка данных> <папка модели> <выходная папка>
Ниже приведены подробные сведения о каждом параметре, а также о папках данных, модели и выходных данных.
Папка данных: Это папка исходных данных, и каждый файл данных имеет формат csv. Эта папка данных содержит два файла (см. подробное описание в шагах 2.3 и 2.4). Эти файлы должны быть заменены персональными данными.
data.csv: Файл транскриптомной матрицы. В первой строке перечислены идентификаторы признаков (или генов), а в первом столбце — идентификаторы образцов. Список генов включает регуляторные факторы (ТФ и линкРНК), а также регулируемые гены мРНК.
label.csv: образец файла этикетки. В первом столбце перечислены идентификаторы образцов, а в столбце с именем "label" указан образец метки.
model folder: Папка для сохранения информации о модели:
HealthModel.pth: предварительно обученная модель HealthModel.
regulatory_geneIDs.csv: Идентификаторы регуляторных генов, использованные в этом исследовании.
target_geneIDs.csv: Гены-мишени, используемые в этом исследовании.
adjacent_matrix.csv: Соседняя матрица регуляторных генов.
Выходная папка: Выходные файлы записываются в эту папку, созданную кодом.
test_target.csv: Значение экспрессии генов-мишеней после Z-нормализации и импутации.
pred_target.csv: Прогнозируемое значение экспрессии генов-мишеней.
mq_target.csv: Прогнозируемое значение экспрессии генов-мишеней.
- Во-первых, измените рабочий каталог на папку HealthModel-mqTrans в командной строке. Используйте следующий синтаксис для выполнения кода:
- Подготовьте файл транскриптомной матрицы в формате csv.
- Каждая строка представляет выборку, а каждый столбец — ген (рис. 4B). Назовите файл транскриптомной матрицы как data.csv в папке данных .
ПРИМЕЧАНИЕ: Этот файл может быть сгенерирован путем ручного сохранения матрицы данных в формате .csv из программного обеспечения, такого как Microsoft Excel. Транскриптомная матрица также может быть сгенерирована с помощью компьютерного программирования.
- Каждая строка представляет выборку, а каждый столбец — ген (рис. 4B). Назовите файл транскриптомной матрицы как data.csv в папке данных .
- Подготовьте файл этикетки в формате csv.
- Как и в случае с файлом транскриптомной матрицы, назовите файл меток как label.csv в папке данных (рисунок 4C).
ПРИМЕЧАНИЕ: В первом столбце приведены имена образцов, а метка класса каждого образца указана в столбце label. Значение 0 в столбце метки означает, что эта выборка отрицательная, 1 означает положительную выборку.
- Как и в случае с файлом транскриптомной матрицы, назовите файл меток как label.csv в папке данных (рисунок 4C).
- Сгенерируйте объекты mqTrans.
- Выполните следующую команду, чтобы сгенерировать объекты mqTrans и получить выходные данные, показанные на рисунке 4D. Функции mqTrans генерируются в виде файла ./output/mq_targets.csv, а файл меток пересохраняется как файл ./output/label.csv. Для удобства дальнейшего анализа исходные значения экспрессии генов мРНК также извлекаются в виде файла ./output/ test_target.csv.
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- Выполните следующую команду, чтобы сгенерировать объекты mqTrans и получить выходные данные, показанные на рисунке 4D. Функции mqTrans генерируются в виде файла ./output/mq_targets.csv, а файл меток пересохраняется как файл ./output/label.csv. Для удобства дальнейшего анализа исходные значения экспрессии генов мРНК также извлекаются в виде файла ./output/ test_target.csv.
3. Выберите функции mqTrans
- Синтаксис кода выбора признаков
- Во-первых, измените рабочий каталог на папку HealthModel-mqTrans . Используйте следующий синтаксис:
python ./FS_classification/testMain.py
Подробная информация о каждом параметре приведена ниже:
in-data-file: Файл входных данных
in-label-file: Метка файла входных данных
Выходная папка: В этой папке сохраняются два выходных файла, в том числе Output-score.xlsx (метод выбора признаков и точность соответствующего классификатора) и Output-SelectedFeatures.xlsx (выбранные имена объектов для каждого алгоритма выбора признаков).- select_feature_number: выберите количество объектов в диапазоне от 1 до количества объектов файла данных.
- test_size: Задайте соотношение испытуемого образца к разделению. Например, 0,2 означает, что входной набор данных случайным образом разбивается на тестовые подмножества train: в соотношении 0,8:0,2.
- combine: Если true, объединять два файла данных для выбора признаков, т.е. исходные значения выражений и объекты mqTrans. Если false, то для выбора признаков используется только один файл данных, т.е. исходные значения выражения или функции mqTrans.
- combine file: Если combine имеет значение true, укажите это имя файла для сохранения объединенной матрицы данных.
ПРИМЕЧАНИЕ: Этот конвейер предназначен для демонстрации того, как сгенерированные объекты mqTrans работают в задачах классификации, и он напрямую использует файл, сгенерированный разделом 2, для следующих операций.
- Во-первых, измените рабочий каталог на папку HealthModel-mqTrans . Используйте следующий синтаксис:
- Запустите алгоритм выбора признаков для выбора признаков mqTrans.
- Включите combine =False , если пользователь выбирает функции mqTrans или исходные функции.
- Во-первых, выберите 800 исходных объектов и разделите набор данных на train: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - Включите combine =True, если пользователь хочет объединить объекты mqTrans с исходными значениями выражения для выбора объектов. Здесь демонстративный пример состоит в том, чтобы выбрать 800 признаков и разбить набор данных на train: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
ПРИМЕЧАНИЕ: На рисунке 5 показана выходная информация. Дополнительные файлы, необходимые для этого протокола, находятся в папке HealthModel-mqTrans-v1-00.tar (Supplementary Coding File 1).
Access restricted. Please log in or start a trial to view this content.
Результаты
Оценка представления mqTrans транскриптомного набора данных
В тестовом коде используются одиннадцать алгоритмов выбора признаков (FS) и семь классификаторов для оценки того, как сгенерированное mqTrans-представление транскриптомного набора данных вносит вклад в задачу классифик?...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Раздел 2 (Использование предварительно обученной модели HealthModel для создания функций mqTrans) протокола является наиболее важным шагом в этом протоколе. После подготовки вычислительной рабочей среды в разделе 1, раздел 2 генерирует представление mqTrans транскриптомного набора данных на основ...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была поддержана Старшей и Младшей группой по технологическим инновациям (20210509055RQ), Научно-техническими проектами провинции Гуйчжоу (ZK2023-297), Научно-техническим фондом Комиссии по здравоохранению провинции Гуйчжоу (gzwkj2023-565), Научно-техническим проектом Департамента образования провинции Цзилинь (JJKH20220245KJ и JJKH20220226SK), Национальным фондом естественных наук Китая (U19A2061), Ключевой лабораторией интеллектуальных вычислений больших данных провинции Цзилинь (20180622002JC) и Фонды фундаментальных исследований для центральных университетов, JLU. Мы выражаем нашу искреннюю признательность редактору обзора и трем анонимным рецензентам за их конструктивную критику, которая сыграла важную роль в существенном повышении строгости и ясности этого протокола.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Ссылки
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены