Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Измерение длины хвоста Poly A из головного мозга и клеточной линии личинки дрозофилы
В этой статье
Резюме
Протокол описывает эффективный и надежный метод количественной оценки длины поли(А) интересующего гена из нервной системы дрозофилы , который может быть легко адаптирован к тканям или типам клеток других видов.
Аннотация
Полиаденилирование является важнейшей посттранскрипционной модификацией, которая добавляет поли(А)-хвосты к 3'-концу молекул мРНК. Длина поли(А)-хвоста жестко регулируется клеточными процессами. Дисрегуляция полиаденилирования мРНК связана с аномальной экспрессией генов и различными заболеваниями, включая рак, неврологические расстройства и аномалии развития. Таким образом, понимание динамики полиаденилирования имеет жизненно важное значение для разгадки сложностей процессинга мРНК и посттранскрипционной регуляции генов.
В данной работе представлен метод измерения длины поли(А)-хвоста в образцах РНК, выделенных из головного мозга личинок дрозофилы и клеток S2 Drosophila Schneider. Мы использовали подход гуанозин/инозин (G/I), который включает ферментативное добавление остатков G/I на 3'-конце мРНК с использованием дрожжевой поли(А)-полимеразы. Эта модификация защищает 3'-конец РНК от ферментативной деградации. Защищенные полноразмерные поли(А)-хвосты затем ретранскрибируются с помощью универсального антисмыслового праймера. Впоследствии ПЦР-амплификация выполняется с использованием ген-специфического олиго, нацеленного на интересующий ген, а также олиго универсальной последовательности, используемого для обратной транскрипции.
В результате образуются ПЦР-продукты, охватывающие поли(А)-хвосты интересующего гена. Поскольку полиаденилирование не является однородной модификацией и приводит к образованию хвостов различной длины, продукты ПЦР имеют диапазон размеров, что приводит к образованию мазка на агарозном геле. Наконец, продукты ПЦР подвергают электрофорезу в капиллярном геле высокого разрешения с последующим количественным определением с использованием размеров продуктов поли(А) ПЦР и ген-специфического продукта ПЦР. Этот метод предлагает простой и надежный инструмент для анализа длины поли(А)-хвоста, что позволяет нам получить более глубокое представление о сложных механизмах, управляющих регуляцией мРНК.
Введение
Большинство эукариотических мРНК посттранскрипционно полиаденилированы на своем 3'-конце в ядре путем добавления нематричных аденозинов каноническими поли(А)-полимеразами. Неповрежденный поли(А)-хвост имеет решающее значение на протяжении всего жизненного цикла мРНК, поскольку он необходим для ядерного экспорта мРНК1, облегчает взаимодействие с поли(А)-связывающими белками для повышения трансляционной эффективности2 и придает устойчивость к деградации3. В некоторых случаях поли(А)-хвост также может подвергаться удлинению в цитоплазме, чему способствуют неканонические поли(А)полимеразы4. В цитоплазме длина поли(А)-хвоста динамически изменяется и влияет на продолжительность жизни молекулы мРНК. Известны многочисленные полимеразы и деаденилазы, модулирующие длину хвоста 5,6,7. Например, укорочение поли(А)-хвостов коррелирует с трансляционным подавлением, тогда как удлинение поли(А)-хвостов усиливает трансляцию 8,9.
Накопление геномных исследований продемонстрировало фундаментальное значение длины поли(А)-хвоста в различных аспектах биологии эукариот. Это включает в себя роль в развитии зародышевых клеток, раннем эмбриональном развитии, синаптической пластичности нейронов для обучения и памяти, а также воспалительной реакции10. Было разработано множество методов и анализов для измерения длины поли(А)-хвоста. Например, анализ РНКазы H/oligo(dT) использует преимущества РНКазы H в присутствии или отсутствии олиго(dT) для изучения длины поли(A) хвоста11,12. Другие методы изучения поли(А)-хвоста включают ПЦР-амплификацию 3'-концов, такую как экспресс-амплификация поли(А) концов кДНК (RACE-PAT)12,13 и лигаз-опосредованный поли(А) тест (LM-PAT)14. Дальнейшие модификации анализа PAT включают ePAT15 и sPAT16. Ферментативный G-хвост17,18 или G/I-хвост 3'-конца являются другими вариантами анализа PAT. Дальнейшая модификация этих методов включает использование флуоресцентно меченных праймеров вместе с электрофорезом в капиллярном геле для анализа с высоким разрешением, называемым поли(А) тестом высокого разрешения (Hire-PAT)19. Эти ПЦР-анализы позволяют быстро и высокочувствительно количественно определить длину поли(А).
С развитием секвенирования нового поколения высокопроизводительный метод секвенирования, такой как PAL-seq20 и TAIL-seq21, позволяет проводить анализ полиаденилирования в масштабе транскриптома. Однако эти методы обеспечивают только короткие считывания 36-51 нуклеотидов. Поэтому FLAM-Seq22 был разработан для глобального профилирования длины хвоста полноразмерной мРНК и обеспечивает длинные чтения. Технология нанопор23 обеспечивает независимое от ПЦР прямое секвенирование РНК или прямой кДНК для оценки длины хвоста поли(А). Однако эти высокопроизводительные методы не лишены ограничений. Они требуют большого количества исходных материалов, являются дорогостоящими и трудоемкими. Кроме того, анализ редких транскриптов может быть чрезвычайно сложным при использовании высокопроизводительных методов, а низкопроизводительные методы, основанные на ПЦР, по-прежнему дают преимущество, когда необходимо проанализировать небольшое количество транскриптов, для пилотных экспериментов и валидации других методов.
Недавно мы продемонстрировали, что мРНК Dscam1 содержат короткие поли(А)-хвосты у дрозофилы, что обуславливает необходимость неканонического связывания цитоплазматического поли(А)-связывающего белка на Dscam1 3'UTR с использованием метода хвостов G/I24. Здесь мы предлагаем оптимизированную процедуру подготовки тканей и количественной оценки длины поли(А) мРНК из нервной системы дрозофилы и клеток Drosophila S2.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Выращивание и отбор личинок дрозофилы
- Поддерживать/культивировать штамм мух (w1118, дикий тип) на стандартной кормовой среде для мух при 25 °C во увлажненном инкубаторе.
- Отобрать 10 блуждающих личинок3-го возраста через 72 ч после откладки яиц.
- Поместите личинок в пустую чашку Петри диаметром 35 мм и аккуратно промойте их, переложив личинок в новую чашку с водопроводной водой с помощью щипцов. Сделайте это 2 раза, чтобы удалить остатки пищи.
2. Выделение мозга из личинок дрозофилы (рис. 1)
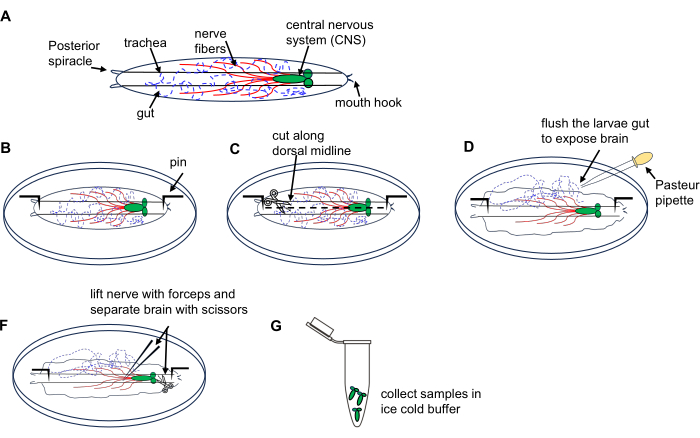
Рисунок 1: Вскрытие головного мозга личинки дрозофилы из блуждающей стадии3-го возраста. (А) Схематические рисунки личинки дрозофилы. (Б-Г) Вскрытие личинок. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
- Поместите 10 личинок на чашку для вскрытия, содержащую ледяной PBS.
- Положите личинку дорсальной стороной вверх (идентифицируется по трахейным трубкам, идущим по ее длине) и приколите каждый конец к дну чашки, а затем сделайте небольшой надрез на стенке тела на заднем конце.
- Разрежьте стенку тела по дорсальной средней линии по направлению к переднему концу с помощью ножниц для микродиссекции.
- Кратковременно промойте внутреннюю часть личинки с помощью пипетки Пастера 3 раза с PBS в чашке, чтобы обнажить мозг.
- Найдите и приподнимите мозг с помощью щипцов и осторожно изолируйте его с помощью ножниц для микродиссекции.
- Перенесите препарированный мозг в микроцентрифужную пробирку объемом 1,5 мл, наполненную ледяным PBS на льду; Соберите мозги всех личинок. Приступайте к экстракции РНК с помощью набора для микроподготовки РНК, как описано в разделе 4.
ПРИМЕЧАНИЕ: Проведите вскрытие 10 личинок в течение 15 минут, чтобы предотвратить повреждение тканей и деградацию РНК.
3. Клетки Шнайдера дрозофилы S2
- Выращивание клеток дрозофилы S2 в среде дрозофилы Шнайдера с добавлением 10% фетальной бычьей сыворотки (FBS) при 25 °C во увлажненном инкубаторе до плотности 8 × 10 6-10 × 106 клеток/мл с минимальной жизнеспособностью 90%.
- В стерильной конической пробирке объемом 50 мл разбавляют клетки до 2,5 × 106 клеток/мл средой дрозофилы Шнайдера, добавленной 10% FBS, предварительно подогретой до 25 °C.
- Переносят 8 мл клеточной суспензии (20 × 106 клеток) в культуральную планшет диаметром 100 мм и добавляют 4 мл среды, чтобы получить 12 мл (1-й день).
- Инкубируют культивируемые клетки при температуре 25 °C во влажном инкубаторе.
ПРИМЕЧАНИЕ: Клетки неплотно прилипают к пластине через 12-16 ч (2-й день). - Трансфицируют клетки соответствующими плазмидами ДНК24.
- Инкубируют в течение 48 ч в увлажненном инкубаторе.
- После инкубации соберите клетки, добавив 5 мл ледяного PBS щадящим пипетированием (4-й день).
- Переложите клетки в пробирку объемом 15 мл.
- Гранулируют клетки центрифугированием при 1 000 × г в течение 5 мин при 4 °C.
- Промойте клетки 2 раза ледяным PBS путем осторожного пипетирования и соберите клетки, центрифугируя их при 1 000 × g в течение 5 мин при 4 °C.
- Выполните экстракцию РНК с помощью набора для минипрепа РНК.
ПРИМЕЧАНИЕ: Выполните следующие действия внутри стерильного ламинарного колпака.
4. Выделение общей РНК из головного мозга личинок дрозофилы и клеток S2
- Мозг личинки: Удалите PBS кратковременным центрифугированием (8 с коротким отжимом при 5 000 × г).
- Добавьте 600 мкл буфера для лизиса РНК и гомогенизируйте 10 раз с помощью пластикового пестика. Визуально осмотрите пробирку под стереомикроскопом, чтобы убедиться в полном лизисе.
- Центрифугу при 1 000 × г в течение 5 мин при 4 °C для удаления остатков ткани. Перелейте очищенную надосадочную жидкость в безнуклеазную микроцентрифужную пробирку.
- Изолируйте РНК с помощью набора для микроподготовки РНК в соответствии с инструкциями производителя.
ПРИМЕЧАНИЕ: Использование набора для микропреппирования РНК имеет важное значение для образцов мозга личинок из-за небольшого количества РНК, присутствующего в образцах. - Ячейки S2: Удалите PBS и изолируйте РНК в соответствии с инструкциями производителя.
- Измерение выхода и качества РНК методами спектрофотометрии и электрофореза в агарозном геле.
- Определяют чистоту и количество экстрагированной РНК, измеряя оптическую плотность выделенной РНК при А260 нм и А280 нм соответственно. Убедитесь, что соотношение A260 нм/A 280 нм составляет ≥2,0, а концентрация РНК составляет >350 нг/мкл для последующих применений.
ПРИМЕЧАНИЕ: Типичный выход РНК из мозга 10 личинок дрозофилы составляет ~500-800 нг/мкл или 2,5-4 мкг в 5 мкл. Для клеток S2 выход составляет ~2-3 мкг/мкл (15-30 мкг в 15 мкл). Изолированную РНК можно хранить при температуре -80 °C для длительного хранения.
- Определяют чистоту и количество экстрагированной РНК, измеряя оптическую плотность выделенной РНК при А260 нм и А280 нм соответственно. Убедитесь, что соотношение A260 нм/A 280 нм составляет ≥2,0, а концентрация РНК составляет >350 нг/мкл для последующих применений.
5. Приготовление РНК-геля и электрофорез
- 1,5% гель денатурирующей РНК (100 мл)
ПРИМЕЧАНИЕ: Формальдегид токсичен при контакте с кожей и вдыхании паров; Обрабатывайте его в химическом вытяжном шкафу.- Растворите три агарозные таблетки (1,5 г) в 82 мл буфера MOPS (дополнительный файл 1) до полного распада таблеток с образованием мелких частиц.
- Нагрейте агарозную суспензию в микроволновой печи до тех пор, пока раствор не станет прозрачным и все частицы полностью не растворятся.
- Охладите раствор до ~60 °C.
- Добавьте 18 мл 37% формальдегида, затем осторожно перемешайте. Вылейте раствор в заливочный лоток и дайте ему застыть в вытяжном шкафу.
- Подготовка образцов РНК и электрофорез
- Разбавьте образец РНК до 200 нг (в 5 мкл) и добавьте 5 мкл красителя с 2-кратной загрузкой РНК.
- Нагрейте образцы при 70 °C в течение 5 мин в сухой ванне.
- Загрузите 2 мкл РНК-лестницы в первую полосу и 10 мкл образцов в соседние полосы.
- Проводят электрофорез в буфере MOPS при 100 В в течение 60 мин для геля размером 5 х 6 см.
ПРИМЕЧАНИЕ: Отрегулируйте условия электрофореза в зависимости от размеров ампликона. - Визуализируйте гель на УФ-трансиллюминаторе.
ПРИМЕЧАНИЕ: Наличие одной полосы размером ~600 нуклеотидов указывает на интактный препарат РНК (см. рис. 2А).
6. Измерение длины хвоста Poly (A)
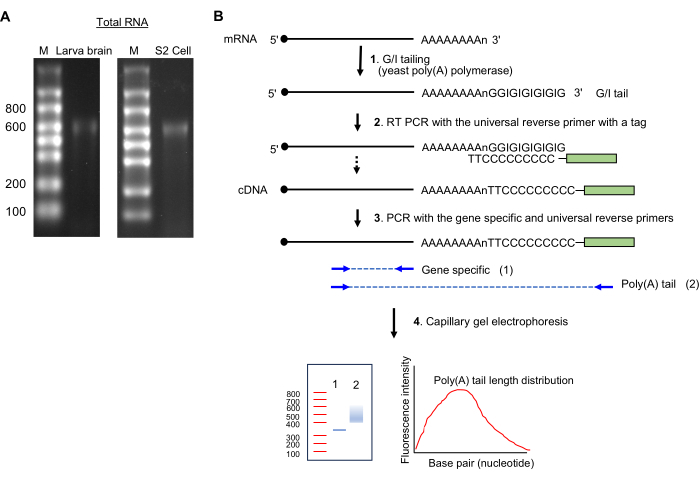
Рисунок 2: Подготовка образца РНК и анализ поли(А)-хвоста. (A) Изображения РНК-геля показывают общую РНК из мозга личинки дрозофилы (слева) и клеток S2 (справа) на 1,5% агарозном геле формальдегида. Размеры лестницы одноцепочечной РНК показаны в нуклеотидах на полосе M. Обратите внимание на основную полосу РНК при ~600 нт, которая исходит от рРНК. (Б) Схема поли(А)-хвостового анализа. Аббревиатура: G/I = гуанозин/инозин. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
- Хвосты GI (Рисунок 2B)
- Выдерживая реагенты на льду, готовят следующую смесь (20 мкл): до 14 мкл общего образца РНК (1 мкг), 4 мкл 5-кратной буферной смеси Tail и 2 мкл смеси ферментов 10x Tail.
- Инкубировать при 37 °C в течение 60 мин в амплификаторе.
- Добавьте 1,5 мкл раствора хвостового упора; Держать на льду 2 мин.
ПРИМЕЧАНИЕ: Приступайте к обратной транскрипции или храните образцы РНК с GI-хвостом при -80 °C до тех пор, пока не будете готовы приступить к обратной транскрипции.
- Обратная транскрипция и амплификация ПЦР
- Синтезируйте кДНК, приготовив смесь и инкубируя ее в условиях, описанных в Дополнительном файле 1.
- Разбавьте образцы кДНК и выполните ПЦР для амплификации ДНК, как указано в Дополнительном файле 1.
7. ПЦР-анализ продукта методом электрофореза в агарозном геле
- Анализируют небольшую порцию (2-5 мкл) продуктов ПЦР из стадии 6.2.2 на 2,5% агарозном геле электрофорезом при 100 В в течение 45 мин для контроля качества.
- Проверьте специфичность ПЦР для геноспецифических и хвостоспецифических реакций путем секвенирования ПЦР-полос, экстрагированных гелем.
8. Капиллярный электрофорез
- Гель-электрофорез высокого разрешения на 1 мкл продуктов ПЦР (0,5-5 нг/мкл) из геноспецифической и поли(А)-специфической ПЦР с использованием биоанализатора с высокочувствительным набором ДНК. Ищите хорошо разрешенные пики, указывающие на успешное выполнение.
9. Анализ данных: измерение длины поли (А) хвоста (Рисунок 3)
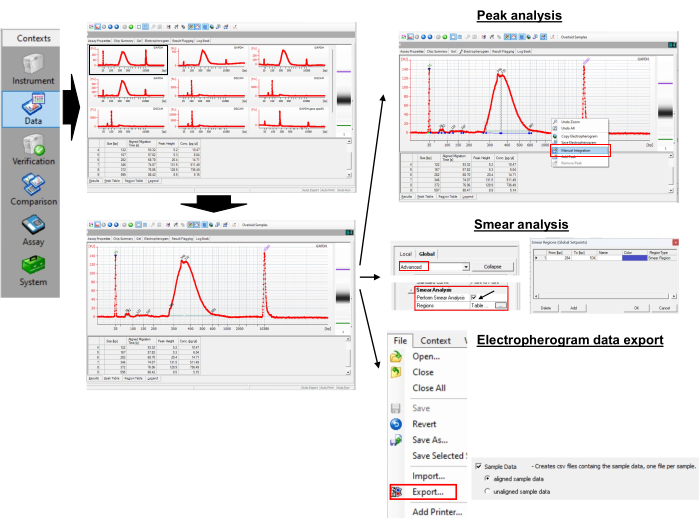
Рисунок 3: Измерение длины хвоста Poly(A) и пикового значения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Доступ к данным
- Чтобы получить доступ к данным, откройте файл xad в программном обеспечении.
- Выберите имя образца или многозвенной цепи на панели древовидного списка.
ПРИМЕЧАНИЕ: Результат будет показан в виде электроферограмм или гелеобразных изображений для выбранных образцов. Нижняя пара оснований 35 и верхняя отметка 10 380.н. являются внутренними стандартами, используемыми для выравнивания данных лестницы (50-7 000.н.) с данными из скважин для отбора проб. - Увеличивайте и уменьшайте масштаб электроферограмм и гелеобразных изображений, чтобы отобразить детали.
- Выполнение анализа пиков и мазков
- Чтобы получить размеры пиков, откройте электроферограмму выбранного образца.
- Щелкните правой кнопкой мыши по электроферограмме и выберите ручное интегрирование , чтобы вручную выбрать пики, перетащив горизонтальную линию.
- Обратите внимание на пиковые значения в таблице Пик. Определите пик с наибольшей высотой пика. Это пик длины хвоста poly(A) для отдельного образца. В приведенном примере это 346.н.
- Включите значок «Показать/скрыть уставки » в верхнем меню и подождите, пока справа не появится новая панель.
- Выберите «Дополнительно», прокрутите вниз, чтобы найти «Выполнить анализ мазка», и установите флажок. Это добавит таблицу Region на вкладку электроферограммы.
- Выберите электроферограмму из образца и перейдите в таблицу Region, в которой отображаются меню From [bp] и To [bp]. Чтобы установить начальную и конечную [bp], щелкните правой кнопкой мыши по электроферограмме и выберите «Регион», чтобы добавить «Добавить область».
- Щелкните правой кнопкой мыши любую ячейку в таблице «Регион» и выберите «Изменить регионы », чтобы открыть небольшое новое окно, в котором можно задать пользовательские области.
ПРИМЕЧАНИЕ: Например, мы использовали область от 300 до 550 б.. для GAPDH. Геноспецифическая ПЦР GAPDH показала пик в 265.н. Универсальный праймер (табл. 1) увеличивает длину поли(А) ПЦР на 35.о. путем отжига до G/I-хвостовых РНК. Таким образом, первый адениновый нуклеотид на РНК GAPDH начинается с 300.н. (265 + 35). Мы произвольно ограничили максимальную длину поли(А) хвоста до 250 (300 + 250 = 550). Из таблицы region программа возвращает средний размер в пределах региона как 387 bp. - Используйте уравнение (1) для вычисления длины хвоста поли(А) на интересующей мРНК:
Длина хвоста Poly(A) = (A - B - 35) (1)
Где A – среднее значение поли(А)-специфического продукта ПЦР по электроферограмме (т.е. 387.о. для GAPDH), В – пиковое значение геноспецифического продукта ПЦР из электроферограммы (т.е. 265.о. для GAPDH), а «35» – длина универсальной метки обратного праймера.
ПРИМЕЧАНИЕ: Из приведенных выше расчетов следует, что средняя длина хвостового оперения GAPDH составляет 387 - 265 - 35 = 87.н.
10. Визуализация распределения длины хвоста poly(A)
- Экспорт данных в виде csv-файла в разделе Файл | Экспорт | Sample Data для получения образцов данных.
ПРИМЕЧАНИЕ: Экспортированный csv-файл отображает время выполнения , а не bp по оси X. - Перейдите к электроферограмме образца и установите флажок «Показать размеры » на вкладке «Электроферограмма », чтобы автоматически преобразовать таблицу регионов в таблице областей из bp во время выполнения. В этом примере от 70,39 до 86,28 с соответствуют от 300 до 550.о.
- Откройте CSV-файл и выберите значения от 70,39 с до 86,28 с времени выполнения, чтобы построить график. Чтобы визуализировать размеры ад по оси X на графике, экспортируйте электроферограмму с размерами ад в виде файла изображения и наложите ее на график, сгенерированный в электронной таблице. Это будет соответствующим образом соответствовать размерам bp на распределении хвостов poly(A).
Access restricted. Please log in or start a trial to view this content.
Результаты
В данной работе мы проанализировали длину поли(А)-хвоста Dscam1 и GAPDH из головного мозга личинок дрозофилы (рис. 4). Выделенные РНК визуализировали на агарозном геле для контроля качества. Одна полоса РНК размером около 600 нуклеотидов указывает на интактную под...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
В этом протоколе мы описываем технику вскрытия мозга личинки дрозофилы из блуждающей стадии3-го возраста, а также подготовку образца из клеток Drosophila S2. Из-за лабильного характера мРНК сбор образцов требует особой осторожности. При вскрытии головного мозга личинок мозг н...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
У авторов нет конфликта интересов, который можно было бы раскрыть.
Благодарности
Это исследование было поддержано Национальным институтом неврологических расстройств и инсульта R01NS116463 Дж.К., а также Центром клеточной и молекулярной визуализации в Университете Невады в Рино, который был поддержан грантом Национальных институтов здравоохранения P20GM103650 и использовался для исследований, представленных в этом исследовании.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| 3-(N-morpholino) propanesulfonic acid (MOPS) | Research Product Internation (RPI) | M92020 | |
| Agilent High Sensitivity DNA Kit | Agilent Technologies | 5067-4626 | |
| Agilent software 2100 expert free download demo | Agilent Technologies | https://www.agilent.com/en/product/automated-electrophoresis/bioanalyzer-systems/bioanalyzer-software/2100-expert-software-228259 | |
| Apex 100 bp-Low DNA Ladder | Genesee Scientific | 19-109 | |
| Bioanalyzer | Agilent 2100 Bioanalyzer G2938C | ||
| Diethyl pyrocarbonate (DEPC) | Research Product Internation (RPI) | D43060 | |
| DNA dye (Gel Loading Dye, Purple (6x) | New England biolabs | B7024S | |
| Drosophila S2 cell line | Drosophila Genomics Resource Center stock #181 | ||
| Drosophila Schneider’s Medium | Thermo Fisher Scientific | 21720024 | |
| Ehidium bromide | Genesee scientific | 20-276 | |
| Fetal bovine serum (FBS) | Sigma-Aldrich | F4135 | |
| Forceps Dumont 5 | Fine Science tools | 11254-20 | |
| Nuclease free water | Thermo Fisher Scientific | AM9932 | |
| PBS 10x | Research Product Internation (RPI) | P32200 | |
| Poly(A) Tail-Length Assay Kit | Thermo Fisher Scientific | 764551KT | |
| RiboRuler Low Range RNA Ladder | Thermo Fisher Scientific | SM1833 | |
| RNA Gel Loading Dye (2x) | Thermo Fisher Scientific | R0641 | |
| RNA microprep kit | Zymoresearch | R1050 | |
| RNA miniprep kit | Zymoresearch | R1055 | |
| Scissors-Vannas Spring Scissors - 2.5 mm Cutting Edge | Fine Science tools | 15000-08 | |
| TopVision Agarose Tablets | Thermo Fisher Scientific | R2802 | |
| Tris-Acetate-EDTA (TAE) | Thermo Fisher Scientific | B49 |
Ссылки
- Stewart, M. Polyadenylation and nuclear export of mRNAs. Journal of Biological Chemistry. 294 (9), 2977-2987 (2019).
- Machida, K., et al. Dynamic interaction of poly(A)-binding protein with the ribosome. Scientific Reports. 8 (1), 17435(2018).
- Eisen, T. J., et al. The dynamics of cytoplasmic mRNA metabolism. Molecular Cell. 77 (4), 786-799 (2020).
- Liudkovska, V., Dziembowski, A. Functions and mechanisms of RNA tailing by metazoan terminal nucleotidyltransferases. Wiley Interdisciplinary Reviews RNA. 12 (2), e1622(2021).
- Goldstrohm, A. C., Wickens, M. Multifunctional deadenylase complexes diversify mRNA control. Nature Reviews Molecular Cell Biology. 9 (4), 337-344 (2008).
- Schmidt, M. J., Norbury, C. J. Polyadenylation and beyond: emerging roles for noncanonical poly(A) polymerases. Wiley interdisciplinary reviews RNA. 1 (1), 142-151 (2010).
- Laishram, R. S. Poly(A) polymerase (PAP) diversity in gene expression - Star-PAP vs canonical PAP. FEBS Letters. 588 (14), 2185-2197 (2014).
- Salles, F. J., Lieberfarb, M. E., Wreden, C., Gergen, J. P., Strickland, S. Coordinate initiation of Drosophila development by regulated polyadenylation of maternal messenger RNAs. Science. 266 (5193), 1996-1999 (1994).
- Wreden, C., Verrotti, A. C., Schisa, J. A., Lieberfarb, M. E., Strickland, S. Nanos and pumilio establish embryonic polarity in Drosophila by promoting posterior deadenylation of hunchback mRNA. Development. 124 (15), 3015-3023 (1997).
- Passmore, L. A., Coller, J. Roles of mRNA poly(A) tails in regulation of eukaryotic gene expression. Nature Reviews Molecular Cell Biology. 23 (2), 93-106 (2021).
- Murray, E. L., Schoenberg, D. R. Assays for determining poly(a) tail length and the polarity of mRNA decay in mammalian cells. Methods in Enzymology. 448, 483-504 (2008).
- Salles, F. J., Strickland, S. Analysis of poly(a) tail lengths by PCR: The PAT assay. Methods in Molecular Biology. 118, 441-448 (1999).
- Salles, F. J., Darrow, A. L., O'Connell, M. L., Strickland, S. Isolation of novel murine maternal mRNAs regulated by cytoplasmic polyadenylation. Genes and Development. 6 (7), 1202-1212 (1992).
- Salles, F. J., Strickland, S. Rapid and sensitive analysis of mRNA polyadenylation states by PCR. Genome Research. 4 (6), 317-321 (1995).
- Janicke, A., Vancuylenberg, J., Boag, P. R., Traven, A., Beilharz, T. H. ePAT: A simple method to tag adenylated RNA to measure poly(a)-tail length and other 3' RACE applications. RNA. 18 (6), 1289-1295 (2012).
- Minasaki, R., Rudel, D., Eckmann, C. R. Increased sensitivity and accuracy of a single-stranded DNA splint-mediated ligation assay (sPAT) reveals poly(a) tail length dynamics of developmentally regulated mRNAs. RNA Biology. 11 (2), 111-123 (2014).
- Martin, G., Keller, W. Tailing and 3'-end labeling of RNA with yeast poly(A) polymerase and various nucleotides. RNA. 4 (2), 226-230 (1998).
- Kusov, Y. Y., Shatirishvili, G., Dzagurov, G., Verena, G. M. A new G-tailing method for the determination of the poly(a) tail length applied to hepatitis a virus RNA. Nucleic Acids Research. 29 (12), 57(2001).
- Bazzini, A. A., Lee, M. T., Giraldez, A. J. Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish. Science. 336 (6078), 233-237 (2012).
- Subtelny, A. O., Eichhorn, S. W., Chen, G. R., Sive, H., Bartel, D. P. Poly(a)-tail profiling reveals an embryonic switch in translational control. Nature. 508 (1), 66-71 (2014).
- Chang, H., Lim, J., Ha, M., Kim, V. N. TAIL-seq: Genome-wide determination of poly(a) tail length and 3' end modifications. Molecular Cell. 53 (6), 1044-1052 (2014).
- Legnini, I., Alles, J., Karaiskos, N., Ayoub, S., Rajewsky, N. FLAM-seq: Full-length mRNA sequencing reveals principles of poly(A) tail length control. Nature Methods. 16 (9), 879-886 (2019).
- Garalde, D. R., et al. Highly parallel direct RNA sequencing on an array of nanopores. Nature Methods. 15 (3), 201-206 (2018).
- Singh, M., Ye, B., Kim, J. H. Dual leucine zipper kinase regulates Dscam expression through a noncanonical function of the cytoplasmic poly(A)-binding protein. Journal of Neuroscience. 42 (31), 6007-6019 (2022).
- Macharia, R. W., Ombura, F. L., Aroko, E. O. Insects' RNA profiling reveals absence of "hidden break" in 28S ribosomal RNA molecule of onion thrips, Thrips tabaci. Journal of Nucleic Acids. 2015, 965294(2015).
- Miura, P., Sanfilippo, P., Shenker, S., Lai, E. C. Alternative polyadenylation in the nervous system: to what lengths will 3' UTR extensions take us. Bioessays. 36 (8), 766-777 (2014).
- Sement, F. M., et al. et al Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucleic Acids Research. 41 (14), 7115-7127 (2013).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены