Method Article
PyDesigner v1.0: Pythonic реализация конвейера DESIGNER для диффузионной магнитно-резонансной томографии
* Эти авторы внесли равный вклад
В этой статье
Резюме
Здесь мы представляем PyDesigner, основанный на Python конвейер обработки диффузионной магнитно-резонансной томографии (dMRI), способный корректировать типичные артефакты dMRI и производить диффузионно-тензорную визуализацию (DTI), диффузионную эксцессионную визуализацию (DKI), визуализацию волоконных шариков (FBI), моделирование микроструктуры (целостность белого вещества [WMTI] и белое вещество волоконного шара [FBWM]) и выходы трактографии.
Аннотация
PyDesigner — это программный пакет на основе Python, основанный на оригинальном параметре диффузии EStImation с конвейером Гиббса и NoisE Removal (DESIGNER) (Dv1) для предварительной обработки дМРТ и оценки тензора. Это программное обеспечение открыто предоставляется для некоммерческих исследований и не может использоваться для клинической помощи. PyDesigner сочетает в себе инструменты FSL и MRtrix3 для выполнения шумоподавления, коррекции звона Гиббса, вихревых токовых коррекций движения, маскирования мозга, сглаживания изображений и коррекции смещения Райса для оптимизации оценки множественных мер диффузии. Его можно использовать на разных платформах на Windows, Mac и Linux для точного получения часто используемых метрик из наборов данных DKI, DTI, WMTI, FBI и FBWM, а также ODF трактографии и файлов .fib. Он также не зависит от формата файла, принимая входные данные в формате .nii, .nii.gz, .mif и dicom. Это удобное и простое в установке программное обеспечение также выводит метрики контроля качества, иллюстрирующие графики отношения сигнал/шум, воксели выбросов и движение головы для оценки целостности данных. Кроме того, этот конвейер обработки dMRI поддерживает обработку нескольких наборов данных эхо-времени и настраивает конвейер, позволяя пользователю указывать, какие процессы используются и какие выходные данные создаются для удовлетворения различных потребностей пользователей.
Введение
Диффузионная МРТ (дМРТ) широко применяется для неинвазивного изучения микроструктурных свойств головного мозга. Несмотря на то, что было предложено множество методов дМРТ, наиболее часто используются два из них: диффузионно-тензорная визуализация (DTI) и диффузионная эксцессионная визуализация (DKI). Эти методы тесно связаны, при этом DKI является расширением DTI, которое включает количественную оценку диффузионной негауссовости1. Оба обеспечивают различные измерения скалярной диффузии и позволяют построить трактографию волокон белого вещества. Важным преимуществом DTI и DKI является то, что они имеют прочную основу в физике диффузии, так что их обоснованность не зависит от подробных предположений относительно микроструктуры тканей 2,3. Это позволяет применять DTI и DKI по всему мозгу и телу для участников любого возраста и состояния заболевания.
Поскольку необработанные диффузионно-взвешенные изображения (DWI) ухудшаются под воздействием множества факторов, включая шум сигнала, движение, звон Гиббса и вихревые токовые искажения, перед вычислением любых диффузионных величин следует использовать предварительную обработку. Предварительная обработка DWI является активной областью исследований, которая в настоящее время находится в очень развитом состоянии. Тем не менее, объединение необходимых этапов обработки в один конвейер, который дает согласованные результаты, является сложной задачей, поскольку необходимо настроить несколько пользовательских параметров в зависимости от деталей получения данных дМРТ, а также потому, что порядок, в котором выполняются шаги предварительной обработки, влияет на результат. По этой причине в 2016 году был первоначально предложен конвейер Diffusion parameter EStImation with Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) для оптимизации, стандартизации и упрощения предварительной обработки для DWI5. DESIGNER Dv1 опирается на программные инструменты, встроенные в FSL, MRtrix3, MATLAB и Python, для создания бесшовного и полного процесса DWI, который включает в себя коррекцию изображения через предварительную обработку и оценку тензора диффузии/эксцесса5. С помощью флагов управления для включения или выключения этапов предварительной обработки коррекция DWI может выполняться выборочно. DESIGNER предварительно обрабатывает в определенном порядке: (i) анализ главных компонент Марченко-Пастура (MP-PCA) для подавленияшума 6, (ii) коррекция звона Гиббса7, (iii) коррекция искажений эхо-планарной визуализации (EPI)8, коррекция вихревых токов9, коррекция движения10 и замена выбросов11, (iv) коррекция поля смещения B1, (v) генерация маски мозга, (vi) сглаживание, (vii) коррекция шума Райса и (viii) нормализация b0. Предварительная обработка в этом конкретном порядке повышает как точность, так и эффективное отношение сигнал/шум (SNR)5. Стоит отметить, что каждый шаг PyDesigner является необязательным и может быть использован или пропущен в зависимости от предпочтений пользователя. Сглаживание, например, может не быть необходимым этапом предварительной обработки для некоторых наборов данных. Хотя это помогает уменьшить количество ошибок фильтрации, для наборов данных сверхвысокого качества это может не понадобиться. Таким образом, пользователи могут использовать только те шаги, которые необходимы для обработки их данных.
Внедрение DESIGNER на разных платформах является сложной задачей из-за различий в операционных системах и настройках среды. В частности, тот факт, что DESIGNER Dv1 в основном написан на MATLAB, создает значительные проблемы переносимости, возникающие из-за сложных требований к конфигурации, необходимых для обеспечения взаимодействия Python-MATLAB. Более того, различные комбинации MATLAB, Python и версий зависимостей ставят под угрозу воспроизводимость этого конвейера. По этой причине мы разработали PyDesigner, который полностью основан на Python. Этот конвейер обработки dMRI не только обеспечивает бесшовную предварительную обработку, но и позволяет встраивать конвейер в контейнер Docker, что значительно повышает переносимость и воспроизводимость. Кроме того, заменив код MATLAB, PyDesigner позволяет избежать всех лицензионных сборов и улучшает доступность.
Этот конвейер обработки dMRI дополняет подход громкой связи, представленный в DESIGNER, добавляет несколько новых функций и включает в себя инструменты FSL и MRtrix3 для выполнения предварительной обработки. Стандартные математические библиотеки Python, такие как Numpy12, SciPy13 и CVXPY14,15, были использованы для замены частей MATLAB DESIGNER на код Python. Это программное обеспечение открыто предоставляется и доступно на Github16. Цель этого документа не в том, чтобы проверить или сравнить наше программное обеспечение с аналогичным программным обеспечением, а скорее в том, чтобы предоставить пользователям пошаговое руководство по обработке их данных с помощью PyDesigner, если они захотят это сделать.
протокол
Все данные, использованные для разработки и тестирования программного обеспечения, были собраны в соответствии с протоколом исследования, утвержденным институциональным наблюдательным советом.
ПРИМЕЧАНИЕ: PyDesigner был создан на базе MacOS 10.14 и требует подсистемы Windows для Linux (WSL) для работы в Windows. Те же команды можно использовать для систем Linux/Mac.
1. Совместимость с операционными системами
- Для систем Windows перейдите по ссылке для установки WSL17 , а затем перейдите к шагу 2.6.
- Для систем на базе Unix (таких как Linux и Mac) перейдите к разделу 2.
ПРИМЕЧАНИЕ: Системы на основе Unix могут изначально устанавливать PyDesigner и его зависимости.
2. Установите FSL
ПРИМЕЧАНИЕ: Все тестирование PyDesigner проводилось с FSL версии 6.0.2, и рекомендуется использовать версию 6.0.2 или выше.
- Перейдите на FSL wiki18 и выберите «Скачать/Установить». Для пользователей Windows перейдите к шагу 2.6.
- Выберите Загрузить FSL под заголовком Установка FSL. Затем выберите лицензию на установку программного обеспечения FSL, прочитайте соглашение и нажмите кнопку «Согласен».
- Заполните обязательные поля и выберите «Зарегистрироваться».
- Выберите ссылку Скачать FSL (первая ссылка на странице). Затем выберите ссылку с инструкцией по установке (вторая ссылка на странице). Следуйте этим инструкциям, чтобы загрузить FSL для каждой операционной системы.
ПРИМЕЧАНИЕ: Установка FSL занимает около 10 минут. - Чтобы проверить успешную установку, введите в новое окно терминала следующее:
$ flirt -version. При этом должен быть возвращен номер версии. Перейдите к шагу 3. - Для пользователей Windows следуйте руководству по установке WSL для FSL19.
3. Создание среды Conda
- Установите пакет conda для управления средами Python. Пользователи могут выбрать либо Miniconda, либо Anaconda.
- Скачать Miniconda/Anaconda20. Пользователи Mac и Linux переходят к шагу 3.2.
- Для Windows настройте conda в WSL21.
ПРИМЕЧАНИЕ: Следующие инструкции относятся к Miniconda, но те же команды работают и для Anaconda. Хотя conda больше не поддерживает Python 3.7, все еще можно создать среду conda с помощью Python 3.7, что позволяет реализовать этот протокол точно так, как описано.
- Чтобы создать среду conda, введите в терминал следующее:
$ conda create -n mri python=3.7. Используйте эту среду conda для всех действий, связанных с PyDesigner.- При появлении запроса с помощью
Proceed ([y]/n)?клавиши Entery.
ПРИМЕЧАНИЕ: -n flag: Используйте это для указания имени среды conda. В видео среда conda называется mri.
ПРИМЕЧАНИЕ:python=Рекомендуется использовать версию 3.7, так как PyDesigner был собран и протестирован на этой версии.
- При появлении запроса с помощью
4. Установите MRtrix3
- Активируйте среду conda, введя следующую команду:
$ conda activate mri - Откройте страницу загрузки MRtrix322 и следуйте инструкциям по загрузке для соответствующей операционной системы. В установочном коде будет запрошено подтверждение определенных зависимостей. Enter,
yчтобы продолжить процесс загрузки.- Распечатайте документацию MRtrix3 для подтверждения установки, введя в терминал следующее:
$ mrinfo -h
- Распечатайте документацию MRtrix3 для подтверждения установки, введя в терминал следующее:
5. Установите PyDesigner
- Откройте новое окно терминала и активируйте среду conda, введя в терминал следующее:
$ conda activate mri - Выполните команду:
$ pip install PyDesigner-DWI - Чтобы убедиться в успешности установки, выведите номер версии, введя в терминал следующее:
$ pydesigner -v- Если появляется ошибка «модуль не найден», обратитесь к разделу «Устранение неполадок» в конце протокола (раздел 10)
ПРИМЕЧАНИЕ: $ pydesigner -h распечатает справочную документацию, которая объясняет команды и синтаксис для программного обеспечения. PyDesigner также можно скачать через Github. Смотрите инструкцию по установке на сайте23.
- Если появляется ошибка «модуль не найден», обратитесь к разделу «Устранение неполадок» в конце протокола (раздел 10)
6. Подготовка данных
ПРИМЕЧАНИЕ: PyDesigner работает с несколькими типами файлов, такими как .nii, .nii.gz, .mif и DICOM. В видео DICOM сортируются и преобразуются в NIfTI перед использованием этого конвейера обработки dMRI. С файлами NIfTI легче работать, потому что они обезличены, и в каждой последовательности есть один файл.
- Чтобы отсортировать DICOM, используйте инструмент DicomSort24.
ПРИМЕЧАНИЕ: Допускается использование любого другого инструмента сортировки DICOM, который выполняет ту же задачу. - После сортировки DICOM преобразуйте их в файлы NIfTI с помощью dcm2niix25.
- Установите dcm2niix, введя в терминал следующую команду:
$ sudo apt installdcm2niix - После загрузки введите в терминал следующее, чтобы проверить установку, распечатав документацию для dcm2nii:
$ dcm2niix -h
- Установите dcm2niix, введя в терминал следующую команду:
- Чтобы запустить dcm2niix, введите в терминал следующую команду:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
ПРИМЕЧАНИЕ: -f: Чтобы указать структуру имен файлов для выходного файла, следуйте за этим тегом с любой желаемой структурой имен файлов. В видео используется: -f %s_%p_%d . %s, %p и %d являются обозначениями для серии, номера протокола и описания соответственно. Подойдет любая файловая структура. Ожидаемые выходные данные dcm2niix — файлы .bval, .bvec, .json и .nii. Если данные TOPUP были получены, то будут файлы .json и .nii TOPUP.
7. Базовое использование PyDesigner
ПРИМЕЧАНИЕ: PyDesigner может занять до 1,5 часов для одного набора данных в зависимости от параметров обработки и возможностей компьютера.
- Чтобы запустить стандартную обработку, используйте флаг
-sили-standard. Эта команда запустит рекомендуемый конвейер предварительной обработки в следующем порядке: шумоподавление6, коррекция звона Гиббса7, коррекция искажений EPI8, коррекция вихревых токов9, создание масок мозга и спинномозговой жидкости (CSF), сглаживание, коррекция смещения Райса, обнаружение выбросов11, оценка тензора и создание параметрических карт. - Чтобы выполнить базовую команду PyDesigner, сначала активируйте среду conda, введя в терминале следующую команду:
$ conda activate mri - Распечатайте справочную документацию для программного обеспечения, введя следующую команду:
$ pydesigner -h. В этой документации описаны все возможные команды и синтаксис для PyDesigner. - Используйте следующий пример для структурирования команды.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - Обратите внимание на следующие важные флажки:
--verbose: Используйте это для просмотра консоли PyDesigner во время обработки данных (не обязательно).--rpe pairs #: Используйте этот параметр, чтобы пользователи с последовательностью TOPUP могли ускорить процесс коррекции искажений EPI8 с помощью TOPUP. Символ # указывает, сколько объемов B0 из последовательности TOPUP будет использоваться. Использование #=1 предотвращает переоценку поля искажений и сокращает время, необходимое для создания этого поля. Пользователи обычно используют #=1 с надежными результатами. При использовании #=1 PyDesigner по умолчанию возьмет первый том.
8. Расширенное использование PyDesigner: адаптация программного обеспечения к индивидуальным потребностям пользователей
ПРИМЕЧАНИЕ: При настройке PyDesigner опустите -s флаг; этот флаг является командой для автоматической предварительной обработки.
- Активируйте среду conda, введя в терминал следующую команду:
$ conda activate mri - Обратитесь к веб-сайту программного обеспечения23 для получения списка флагов; каждый флаг позволяет пользователю запускать каждую часть PyDesigner по отдельности.
- Структурируйте команду. Начните с флагов для каждой последовательности, которую пользователь хотел бы выполнить. Затем направьте PyDesigner к входным и выходным файлам. Например:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - Пример 1: Запустите только тензорную аппроксимацию (без дополнительной предварительной обработки).
- Активируйте среду conda, введя в терминал следующую команду:
$ conda activate mri - Обратитесь к веб-сайту PyDesigner23 за списком флагов. В этом примере используется
-m. -m: Добавьте этот флаг, чтобы ограничить аппроксимацию тензора только вокселями мозга. Это ускоряет подгонку тензора + обнаружение выбросов.- Для структурирования команды используйте следующий пример:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
ПРИМЕЧАНИЕ: Входными данными для подгонки тензора является предварительно обработанный файл .nii.
- Активируйте среду conda, введя в терминал следующую команду:
- Пример 2: Запустите шумоподавление в одиночку (без дополнительной предварительной обработки).
- Активируйте среду conda, введя в терминал следующее:
$ conda activate mri - Обратитесь к веб-сайту PyDesigner23 за списком флагов. В этом примере используется
-n. - -n: Добавьте этот флаг для завершения шага шумоподавления. Введите предварительно обработанный файл DWI с меткой DWI_preproccessed.nii, который находится в папке вывода каждого субъекта.
- Для структурирования команды используйте следующий пример:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- Активируйте среду conda, введя в терминал следующее:
9. Будущие обновления PyDesigner
ПРИМЕЧАНИЕ: С обновлениями можно ознакомиться на сайте23.
- Чтобы обновить PyDesigner, введите в новое окно терминала следующее:
$ pip install --upgrade PyDesigner-DWI
ПРИМЕЧАНИЕ: Если PyDesigner был установлен через Github16, его также необходимо обновить с помощью git.
10. Устранение неполадок при установке
- При загрузке PyDesigner код ошибки «модуль не найден» появится, если отсутствует модуль Python.
- Исправьте недостающий модуль, введя в терминал следующее:
$ pip install nameofmodule - Затем повторите первую команду install, введя в терминал следующее:
$ pip install PyDesigner-DWI - Теперь PyDesigner должен быть полностью установлен. Пожалуйста, вернитесь к шагу 5.3. Если возникает та же ошибка, повторяйте этот процесс до тех пор, пока не будут установлены все отсутствующие модули Python.
- Исправьте недостающий модуль, введя в терминал следующее:
11. Устранение ошибок вывода
- Если возникают какие-либо проблемы, просмотрите файл log_command.json , чтобы выявить ошибки предварительной обработки. Просмотрите промежуточные файлы, чтобы определить конкретную ошибку.
- Чтобы получить помощь в устранении неполадок, отправьте запрос на странице16 GitHub.
12. Запуск PyDesigner с примером набора данных
- Скачайте пример набора данных от OpenNeuro здесь26.
ПРИМЕЧАНИЕ: Этот пример набора данных содержит как необработанные, так и предварительно обработанные данные. Необработанные данные предназначены в качестве входных данных для обработки примера PyDesigner. Предварительно обработанные данные состоят из выходных данных, сгенерированных после обработки необработанных данных с помощью конвейера обработки dMRI на iMac OS v12.4. Пользователи могут загружать необработанные данные, обрабатывать их с помощью PyDesigner и сравнивать свои выходные данные с предоставленными примерами выходных данных, чтобы убедиться, что PyDesigner работает правильно. Необработанные данные содержатся в главной папке (/PyDesigner Example Dataset/sub-01), а предварительно обработанные данные — в папке derivatives (/PyDesigner Example Dataset/derivatives/sub-01). Если не указано иное, набор данных OpenNeuro будет загружен в папку пользователя по умолчанию. - Откройте новый терминал на Mac/Linux или WSL на Windows.
ПРИМЕЧАНИЕ: Чтобы запустить стандартный конвейер обработки PyDesigner, следуйте приведенному ниже примеру структуры команд. Введите следующие команды ниже в терминал/WSL. В этом примере, где «./user_download_folder/» указывает путь к папке с данными. Этот путь будет специфичным для того, где хранятся данные примера на компьютере отдельного пользователя. - Чтобы создать новую папку для выводов, введите в терминал следующее:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - Чтобы активировать среду conda, введите в терминал следующее:
$ conda activate mri - Чтобы запустить PyDesigner на примере набора данных, введите в терминал следующее:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
ПРИМЕЧАНИЕ: В этом примере ./user_download_folder/ указывает путь к папке с данными. Этот путь будет специфичным для каждого компьютера пользователя. Команды на шагах с 12.3 по 12.5 соответствуют структуре шага 8.3. Эта команда использует флаг -s, стандартный флаг обработки PyDesigner, который выполняет следующие этапы обработки в следующем порядке: шумоподавление6, коррекция звонка Гиббса7, устранение искажений 8,9, маскировка мозга, сглаживание и коррекция смещения Райса. - Если программное обеспечение установлено правильно, все выходные данные будут отображаться во вновь созданной папке PyDesigner_Outputs. Сравните файлы предварительной обработки, созданные PyDesigner, с примером папки предварительной обработки набора данных, найденной в папке «производные», загруженной из OpenNeuro.
- Обратитесь к разделу репрезентативных результатов, чтобы сравнить их метрические карты с результатами в примере набора данных.
Результаты
Программное обеспечение PyDesigner применяет несколько этапов коррекции изображений к необработанным данным диффузии и генерирует выходные данные, используемые для повышения точности необработанных файлов при проведении анализа. Каждый этап, доступный в процессе разработки, был предварительно проверен с помощью рецензируемых публикаций 5,6,7,8,9,10,11, как обсуждалось во введении. Результаты работы программного обеспечения могут быть использованы в таких анализах, как профили трактографии, матрицы связности, воксельные анализы, анализы ROI, fODF-анализы, TBSS и фиксальные анализы.
На веб-сайтепрограммного обеспечения 23 перечислены все выходные файлы, сгенерированные в ходе конвейера предварительной обработки. После выполнения каждого запроса консоль будет выводить описание всех завершенных процессов. Существует 3 типа выходных файлов: файлы обработки, метрики и контроль качества. Структура выходных каталогов показана на рисунке 1. Эти файлы доступны при использовании стандартной предварительной обработки (см. раздел 7 протокола). Если пользователю требуется более продвинутое использование (см. раздел 8 протокола), доступные выходные файлы будут зависеть от того, какие процессы были завершены.
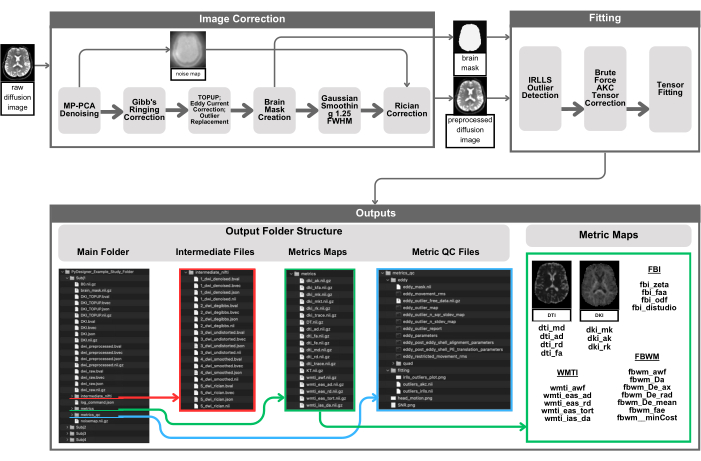
Рисунок 1: Визуальное представление конвейера PyDesigner. Предварительная обработка начинается с передачи входного 4D DWI в PyDesigner (вверху слева), который затем подвергается шумоподавлению MP-PCA для получения шумоподавленного 4D DVI и 3D-карты шума. Затем шумоподавленный 4D DWI подвергается коррекции звона Гиббса, коррекции смещения Райса, TOPUP, коррекции вихревых токов и коррекции выбросов. Затем маска мозга вычисляется для последующих шагов, обнаружения выбросов и подгонки тензора для ускорения вычислений за счет их выполнения только внутри маски мозга. Результаты можно найти в подкаталогах в основной папке обработки предмета - intermediate_nifti, метрики и metrics_qc. Обратите внимание, что скриншоты выходных данных PyDesigner не являются исчерпывающим списком всех возможных выходных данных, а скорее представляют собой схематический визуальный пример того, что могут ожидать пользователи. Выходные данные, как окончательные, так и промежуточные файлы, будут различаться в зависимости от вводимых пользователем данных и используемых флагов обработки. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обработка
Файлы обработки используются в конвейере PyDesigner и хранятся в корневом каталоге вывода. Для каждого этапа предварительной обработки промежуточные файлы DWI сохраняются в выходной папке "intermediate_nifti", как показано на рисунке 1. На эти файлы следует ссылаться при решении любых проблем с обработкой или выходными данными, чтобы оценить каждый шаг конвейера отдельно.
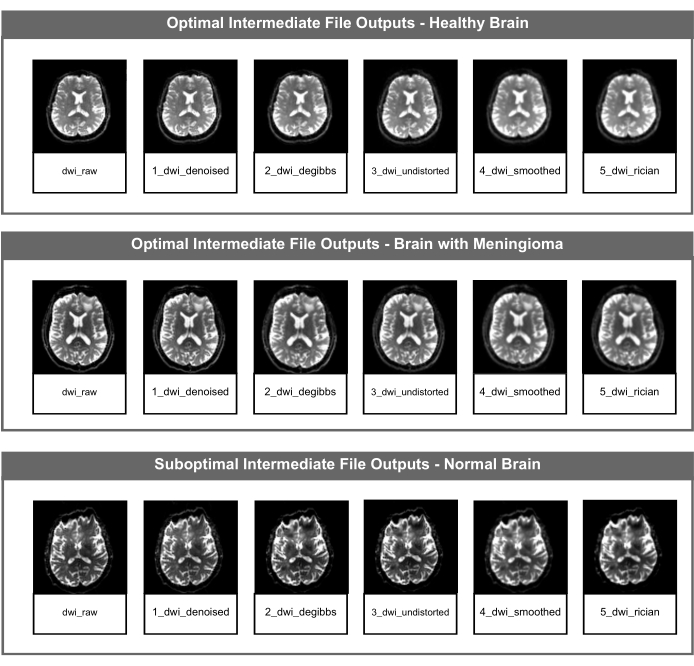
Рисунок 2: Оптимальные и неоптимальные промежуточные файлы DWI NifTI. На рисунке показан промежуточный файл NIfTI для каждого шага коррекции изображений конвейера PyDesigner. Верхняя строка представляет собой пример оптимальных промежуточных выходных файлов с использованием данных от здорового взрослого мозга, средняя строка — пример оптимальных промежуточных выходных файлов с использованием данных от патологического мозга (менингиомы), а нижняя строка отображает неоптимальные промежуточные файловые выходы с использованием данных от здорового взрослого мозга с артефактом восприимчивости, не связанным со структурой мозга или здоровьем. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Метрика
Эта папка содержит все параметрические карты, которые вычисляет PyDesigner (см. рисунок 1). Это включает в себя параметрические карты для DTI/DKI, визуализацию волоконных шариков (FBI)/белого вещества волоконных шаров (FBWM) и показатели целостности тракта белого вещества (WMTI) (Таблица 1)16.

Таблица 1: Ожидаемый диапазон значений для метрик DTI/DKI и FBI/FBWM. Таблица включает в себя список надежных метрик DTI, DKI, FBI и FBWM, сгенерированных PyDesigner, и их диапазоны ожидаемых значений. Также перечислены необходимые последовательности и b-значения (s/mm2) для получения каждой метрики. Перечислены следующие метрики DTI: FA, MD, AD и RD. Перечислены метрики DKI: MK, AK, RK и KFA. Указанный показатель ФБР — FAA. Перечисленные метрики FBWM: AWF, DA, DE_AX, DE_RAD и FAE.
Пользователи могут проводить визуальный и ценностный контроль качества (QC) метрик средней диффузии (MD), фракционной анизотропии (FA) и среднего эксцесса (MK) для выявления неоптимальных результатов. Если эти метрики не являются оптимальными по сравнению со стандартами, описанными ниже, пользователи должны просмотреть каждый промежуточный файл, описанный на рисунке 2 , чтобы определить, какой этап предварительной обработки был неудачным.
Визуальный контроль качества используется для выявления неоптимальных результатов (например, проблем с подгонкой тензора и артефактов). Мы рекомендуем использовать ImageJ для визуального контроля качества, чтобы гарантировать, что изображение не будет обработано с помощью программного обеспечения по умолчанию. В верхней строке рисунка 3 показаны типичные метрические карты MD, FA и MK с использованием биологически правдоподобного порога 0-3мкм2/мс, 0-1мкм2/мс и 0-2мкм2/мс соответственно (Рисунок 3 [Верхняя строка]). Карта МД должна иметь самые высокие значения в желудочках и высокие значения в корковом сером веществе (Рисунок 3A [Верхний ряд]). Карта FA должна иметь видимые тракты белого вещества, которые четко прослеживаются по всему мозгу (Рисунок 3B [Верхний ряд]). Карта MK должна иметь высокие значения в WM и более низкие значения в сером веществе и CSF (Рисунок 3C [Верхняя строка]). На рисунке 3D [Верхняя строка] показан пример метрической карты с проблемами подгонки тензоров, в результате которой образуются кластеры вокселей с нулевыми значениями. Если возникают какие-либо проблемы, просмотрите файл log_command.json, чтобы найти ошибки предварительной обработки. Просмотрите промежуточные файлы, чтобы определить конкретную ошибку. Чтобы получить помощь в устранении неполадок, отправьте запрос на странице GitHub PyDesigner.
Контроль качества значений используется для определения того, являются ли вокселы данной метрической карты относительно однородными между субъектами для каждого набора данных. Диапазон ожидаемых значений для каждой карты и набора данных зависит от данных и параметров PyDesigner. В нашем тестовом наборе данных мы имели устойчивые пики в диапазонах от 8000 до 10 000, от 2500 до 4000 и от 5000 до 13 000 для MD, FA и MK соответственно, используя биннинг гистограммы FSLeyes по умолчанию. В нижнем ряду рисунка 3 приведены примеры вариабельности гистограммы. В таблице 1 приведены ожидаемые значения по оси X для этих показателей. Более высокие или более низкие вокселы характерны для набора данных или указывают на артефакт или проблему предварительной обработки (Рисунок 3D [Нижняя строка]).
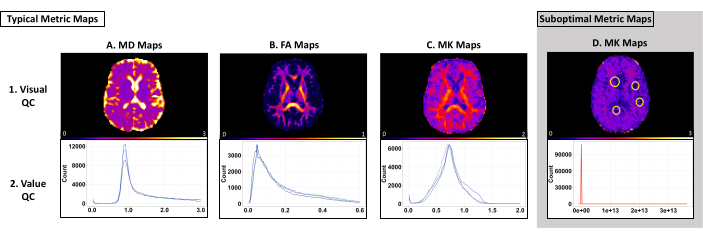
Рисунок 3: Примеры метрических карт и гистограмм из PyDesigner с оптимальными и неоптимальными результатами. В верхнем ряду показаны примеры карт MD, FA и MK с одним объектом, используемых для визуального контроля качества. В нижней строке отображаются многопредметные гистограммы, используемые для значения QC. (А-С) Типичные примеры метрических карт и гистограмм, которые проходят визуальный и ценностный контроль качества. Каждая синяя линия на гистограммах для каждого типа метрики представляет отдельный набор данных. Обратите внимание, что каждый набор данных следует одной и той же кривой и попадает в один и тот же диапазон. (D) Пример метрической карты, которая не проходит визуальный контроль или контроль качества значений. Обратите внимание, как красная линия на гистограмме показывает кривую, которая отличается от кривой A-C. Нулевые воксели, обведенные кружком на этой карте метрик, возникают из-за проблем с подгонкой тензора во время предварительной обработки (панель D, верхняя строка). Эта гистограмма является примером того, как вокселы в наборе данных обычно выше или ниже, чем ожидалось (панель D, нижняя строка). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Контроль качества
После обработки данных PyDesigner следует использовать папку metrics_qc (см. рис. 1) для определения неоптимальных наборов данных. Для каждого набора данных PyDesigner выводит три графика, используемых для контроля качества.
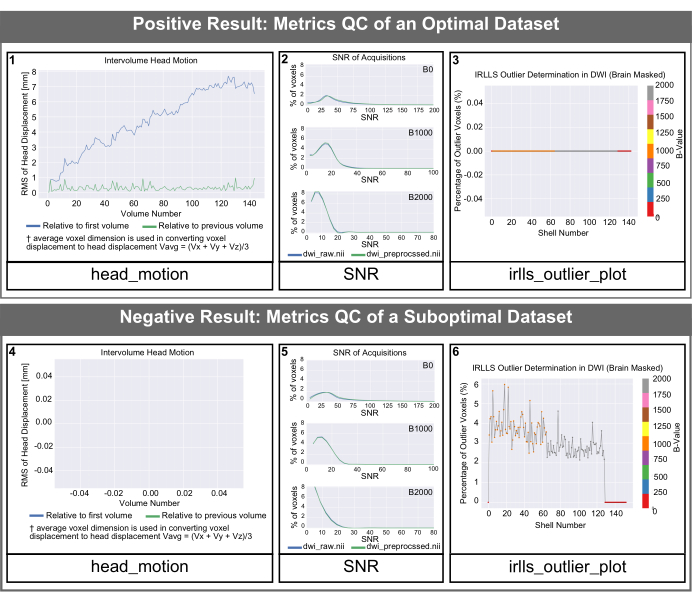
Рисунок 4: Гистограммы контроля качества, сгенерированные PyDesigner, для оптимальных и неоптимальных наборов данных. SNR, межобъемное движение головы и гистограммы выбросов, сгенерированные PyDesigner. Обе строки представляют данные здорового мозга взрослого человека. Верхняя строка представляет собой пример гистограмм контроля качества для оптимального набора данных. В нижней строке показаны результаты контроля качества неоптимального набора данных с артефактом восприимчивости, не связанным со структурой мозга или здоровьем. Обратите внимание, что размер текста меток в выводах PyDesigner по умолчанию меньше, чем они будут отображаться на этом рисунке. Мы увеличили размер текста на этом рисунке для удобочитаемости. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
На head_motion графиках на рисунке 4 показано смещение головы относительно первого объема и предыдущего объема. Как видно на рисунке 4 (панель 1), смещение головки обычно невелико, и PyDesigner корректирует эти артефакты движения в стандартном конвейере обработки с помощью программы FSL Eddy вместе с TOPUP для коррекции движения и вихревых токов9. Для неоптимальных наборов данных график head_motion может отображаться пустым, как показано на рисунке 4 (панель 4). Это указывает на то, что коррекция вихретокового движения не увенчалась успехом, поэтому PyDesigner не смог вывести график. Файлы журнала вихретоковой коррекции можно найти в подпапке eddy в папке metrics_qc (см. рис. 1). На графике отношения сигнал/шум (SNR) отображаются 3 графика. Каждый график предназначен для разного b-значения и показывает как предварительно обработанные, так и необработанные данные. Для оптимального набора данных пиковое отношение сигнал/шум необработанных данных должно составлять ≥5 (рис. 4 [панель 2]). Субоптимальные наборы данных будут иметь пиковое отношение отношение сигнал/шум необработанных данных, равное ≤3 (рис. 4 [панель 5]). В идеале пользователи должны видеть, что пик SNR для всех b-значений увеличивается незначительно, но не резко. График выбросов находится в папке подгонки в metrics_qc и показывает процент выбросов в наборе данных (рис. 4 [панель 3 и 6]). Хороший набор данных должен иметь низкий процент выбросов, обычно менее 5% (рис. 4 [панель 3]). Неоптимальный набор данных будет иметь большой процент выбросов, как показано на рисунке 4 (панель 6).
Примеры результатов набора данных
После того как PyDesigner завершит обработку примера набора данных, все выходные данные должны содержаться в папке "PyDesigner_Outputs". Эти выходные данные можно сравнить с теми, которые находятся в папке «производные», упакованной с примером набора данных, загруженного из OpenNeuro (обработанного на MacOS 12.4). Если программное обеспечение работает правильно, файловая структура «PyDesigner_Outputs» и «производных» будет точно такой же. Аналогичным образом, графики SNR, движения головы и выбросов, найденные во вложенной папке "metrics_qc", должны совпадать с графиками на рисунке 5A. Метрические карты (находятся в подпапке "metrics") можно сравнивать с помощью программного обеспечения для визуализации, такого как FSLeyes, MRIcron, ImageJ и т.д. Гистограммы значений FA, MD и MK из предварительно обработанных/метрических показателей можно увидеть на рисунке 5B. Обратите внимание, что все метрические гистограммы, показанные на рисунке 5B , масштабированы на основе предложенных шкал метрических значений в таблице 1.
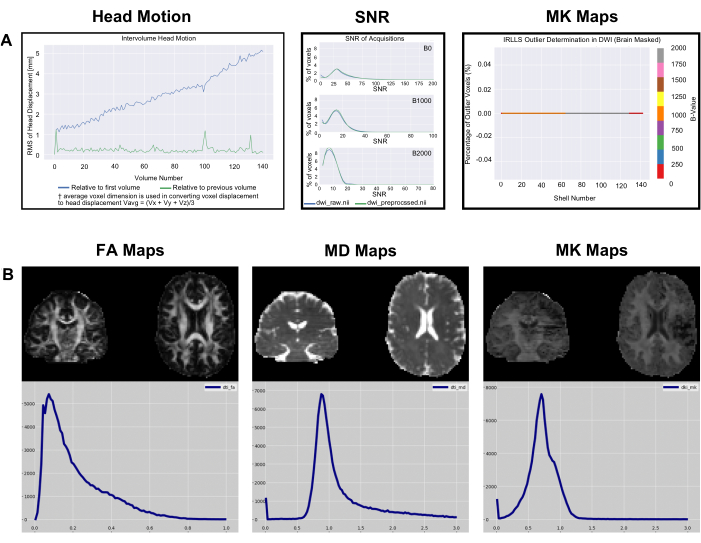
Рисунок 5: Пример гистограмм Data Metric и Metric QC. (A) SNR, межобъемное движение головы и гистограммы выбросов, сгенерированные PyDesigner для примера данных, загруженных из OpenNeuro. Обратите внимание, что размер текста на графиках на этом рисунке был увеличен для удобства чтения. (B) Метрика одного субъекта отображает гистограммы количества вокселевых значений для FA, MD и MK для одного и того же набора данных, визуализированные с помощью FSLeyes v6.0. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
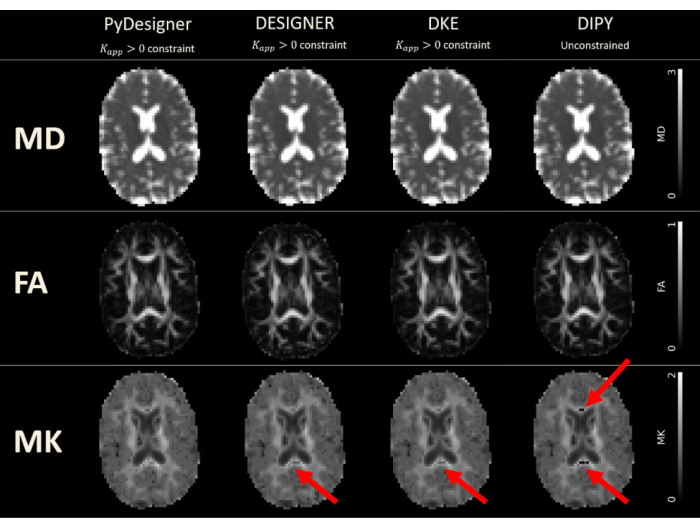
Рисунок 6: Карты DTI и DKI, полученные из PyDesigner, DESIGNER, DKE и DIPY. Тензорная аппроксимация выполнялась с ограничением Каппа > 0 в PyDesigner, DESIGNER5 и Diffusional Kurtosis Estimator (DKE)27, тогда как неограниченная аппроксимация использовалась в диффузионной визуализации в Python (DIPY)28 из-за программных ограничений. Единицами измерения МД являются квадраты микрометров в миллисекунду (мкм2/мс), в то время как другие метрики безразмерны. Выпадения из-за общей проблемы с коррекцией звона Гиббса можно увидеть на картах MK, созданных DESIGNER, DKE и DIPY. Этот рисунок был воспроизведен с разрешения Dhiman et al.29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
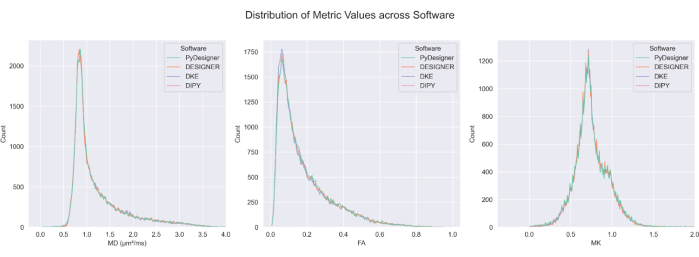
Рисунок 7: Сравнение FA, MD и MK по конвейерам. Распределение вычисленных значений для FA, MD и MK из PyDesigner, DESIGNER5, DKE27 и DIPY28 в мозге, исключенном из CSF, аналогично для большинства вокселей. Расчет карты метрик по методам сопоставим. Этот рисунок был воспроизведен с разрешения Dhiman et al.29. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обсуждение
Основной мотивацией для разработки PyDesigner было внедрение ключевых элементов DESIGNER при одновременной замене всего кода MATLAB на Python, что позволило бы повысить переносимость и доступность. PyDesigner и DESIGNER Dv1 дают почти идентичные результаты29. Тем не менее, в PyDesigner включено несколько дополнительных опций, настроек по умолчанию и незначительных исправлений ошибок. В онлайновой документации PyDesigner16 они подробно описываются.
PyDesigner также дает результаты, аналогичные широко используемым инструментам анализа Diffusional Kurtosis Estimator (DKE)27 и Diffusion Imaging in Python (DIPY)28 DKI29 (см. Рисунок 6 и Рисунок 7), но карты, созданные на основе предварительно обработанных DESIGNER дМРТ, считаются более точными из-за их алгоритма подгонки, продемонстрированного Ades-Aron et al.5. На рисунке 6 приведено сравнение метрических карт MD, FA и MK из PyDesigner, DESIGNER5, DKE27 и DIPY28. На рисунке 7 показано сравнение гистограмм MD, FA и MK из каждого трубопровода. Сочетание подгонки тензора с ограничениями, обнаружения выбросов и коррекции кажущегося коэффициента эксцесса дает более надежную и точную аппроксимацию тензора, как видно в PyDesigner и DESIGNER5.
Преимущество PyDesigner перед DESIGNER Dv1 заключается в том, что он доступен через контейнер NeuroDock Docker30, что значительно повышает портативность и упрощает установку. Этот контейнер работает на всех основных платформах ОС, совместимых с Docker, включая Windows, Mac OS и различные дистрибутивы Linux. Основываясь на отзывах пользователей, PyDesigner v2.0 будет включать обновления для контейнера PyDesigner Docker. Улучшения, представленные в новой версии PyDesigner, и введение многоступенчатого файла Dockerfile решат все существующие проблемы, с которыми сталкиваются пользователи. Если у пользователей возникают проблемы с установкой, мы рекомендуем отправлять вопросы на страницу обсуждения PyDesigner16. Контейнерная технология Docker также обеспечивает простое развертывание в высокопроизводительных кластерах (HPC) для быстрой пакетной обработки DWI в локальных кластерах, совместимых с Docker.
PyDesigner также включает расчеты микроструктурного моделирования, выходящие за рамки DKI, включая WMTI3, FBI и FBWM. Для WMTI достаточно стандартного набора данных DKI, а связанные с ним микроструктурные параметры рассчитываются по умолчанию. Однако следует подчеркнуть, что валидность WMTI ограничена областями белого вещества с высоким FA (т.е. FA ≥ 0,4). Некоторые метрики WMTI имеют ограниченную точность из-за предположения о параллельном выравнивании аксонов в любом заданном вокселе31. FBI 32,33,34 — это отдельный метод дМРТ, применимый ко всему белому веществу головного мозга, который требует высокого b-значения (т.е. b ≥ 4000 с/мм2) и данных дМРТ, отобранных с минимум 64 направлениями диффузионного кодирования (наряду с данными для b = 0). Основными выходными данными FBI являются функция плотности ориентации волокна (fODF) для каждого вокселя белого вещества, которая может быть использована для трактографии белого вещества и служит входом для FBWM, а также внутриаксональная фракционная анизотропия (FAA). FBWM использует данные дМРТ от DKI и FBI для оценки тех же параметров, что и WMTI, но с повышенной точностью, и его можно применять по всему белому веществу независимо от значения FA. Таким образом, если эти дополнительные данные доступны, то оценки FBWM предпочтительнее оценок из WMTI35. Как и в случае с ФБР, FBWM был валидирован только на белом веществе головного мозга взрослых.
В дополнение к инвариантам вращения, предоставляемым PyDesigner (ориентация RAS), файлы .fib, специфичные для DSIstudio (ориентация LPS), создаются отдельно для DTI, DKI и FBI. Файлы .fib содержат информацию о направлениях ODF для создания профилей трактографии для каждого метода. Полученные профили трактографии могут быть использованы для создания матриц связности на уровне предмета и группы. Файлы DKI и FBI .fib содержат многонаправленную информацию, передающую информацию о пересечении волокна, которая недоступна при трактографии DTI. Более того, в каждый файл .fib включены различные ротационные инварианты каждого метода, которые могут быть использованы в различных комбинациях в качестве критериев для посева, выполнения и остановки трактографии. Более подробную информацию о DSIstudio можно найти на их веб-сайте36.
Еще одной примечательной особенностью PyDesigner является многофайловый ввод, который позволяет ему обрабатывать различные файловые входы - NifTi (.nii), сжатый NifTi (.nii.gz), DICOM (.dcm) и формат файлов MRtrix (.mif). PyDesigner может автоматически определять информацию о приобретении из метаданных заголовка независимо от формата ввода и вносить соответствующие исправления, тем самым поддерживая подход невмешательства. Независимо от различий в протоколах, одна и та же команда (см. выше) может быть использована для обработки самых разных DWI. Таким образом, это программное обеспечение экономит время и усилия, сводя к минимуму ручные этапы предварительной обработки и команды. В недавно выпущенном обновлении (v1.0-RC10) эта функция была улучшена за счет обеспечения совместимости с несколькими наборами данных эхо-времени (multi-TE). Это позволяет PyDesigner выполнять этапы предварительной обработки изображений, которые в значительной степени не зависят от TE, на мульти-TE DWI для получения изображения с минимальным шумом и артефактами. Затем TE-зависимые тензорные вычисления выполняются для каждого TE отдельно для получения метрик диффузии или эксцесса.
Обратите внимание, что PyDesigner v1.0 не представляет собой конечную точку при проектировании конвейеров предварительной обработки изображений. Разработка и валидация инструментов обработки изображений является активным направлением исследований. В частности, во время подготовки данной рукописи были предложены новые разработки в области шумоподавления изображений и устранения звона Гиббса, что привело к выпуску пересмотренного конвейера Designer, Designer Dv237, который включает улучшенное шумоподавление и коррекцию артефактов Гиббса для дМРТ, полученных с помощью частичного сбора данных Фурье35, и доступен на DESIGNER Github38. Кроме того, организационное соответствие стандарту BIDS39 является важным аспектом любого конвейера анализа МРТ. Формат BIDS обеспечивает более эффективный обмен данными и реализацию конвейера за счет создания определенной структуры набора для всех типов данных МРТ. Несмотря на то, что PyDesigner в настоящее время не совместим с BIDS, в следующей версии PyDesigner будут включены выходные данные, совместимые с BIDS. Кроме того, PyDesigner изначально был написан с использованием Python версии 3.7. На момент публикации Python 3.7 уже считается устаревшим. Тем не менее, информация, содержащаяся в этой рукописи, не зависит от версии на языке Python. То есть, описанные здесь процедуры, которые являются важными функциями предварительной обработки (например, шумоподавление, коррекция звонка Гиббса и т.д.), будут следовать одному и тому же операционному и концептуальному рабочему процессу независимо от изменений в процедурах установки. Важно отметить, что, как и в случае со всем программным обеспечением на основе Python, будущие версии PyDesigner будут обновлены до новой версии Python. Мы стремимся проверять и интегрировать такие разработки с течением времени.
Для получения наиболее актуальной версии PyDesigner, включая любую новую документацию, относящуюся к обновлениям версии Python, читателям рекомендуется обратиться к веб-сайту перед началом нового анализа и принять участие в дискуссионном форуме, где можно задать вопросы о PyDesigner16. Реализация Docker для переносимости называется NeuroDock30 и содержит PyDesigner и его зависимости для обеспечения обработки данных на широком спектре платформ.
Раскрытие информации
Никакой.
Благодарности
Мы благодарны за полезные беседы с Оливией Хорн, Дэниелом Ленчем и Грэмом Уорнером.
Исследования, представленные в этой публикации, были частично поддержаны грантами Национальных институтов здравоохранения R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 и T32DC014435. Ответственность за содержание лежит исключительно на авторах и не обязательно отражает официальную точку зрения Национальных институтов здравоохранения. Дополнительное финансирование было предоставлено Фондом Литвина.
Материалы
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
Ссылки
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены