Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Экстракция ДНК и сравнение старых и свежих образцов некрофильных мух
* Эти авторы внесли равный вклад
В этой статье
Erratum Notice
Резюме
В этой статье описывается протокол экстракции свежей и старой некрофильной ДНК мухи с использованием модифицированного общего набора для экстракции ДНК.
Аннотация
В общей сложности пять образцов Chrysomya megacephala - три свежих образца, один образец, хранившийся в спирте в течение 2 лет, и один образец, хранившийся в сухом запечатанном хранилище в течение 2 лет, защищенных только от света - были отобраны для изучения того, может ли набор для экстракции ДНК крови извлечь ДНК у некрофильных мух и определить, может ли алкоголь продлить сохранение ДНК некрофильных мух. Во-первых, набор для извлечения ДНК из грудной клетки был использован для извлечения ДНК из тканей грудной клетки. Затем чистота и концентрация ДНК были исследованы с помощью считывателя микропланшетов и флуориметра. Наконец, ПЦР-амплификацию и электрофорез экстрагированной ДНК проводили с помощью некрофильных мухоспецифичных праймеров, расположенных в митохондриальной последовательности гена CO I. Результаты показали, что чистота ДНК всех образцов была выше 2,0. Концентрация ДНК наблюдалась в следующем порядке: свежие образцы > законсервированные спиртом старые образцы > необработанные, старые образцы. Все образцы имели специфические электрофоретические полосы после амплификации ПЦР. В заключение, набор для экстракции ДНК крови может быть использован для успешного извлечения ДНК из некрофильных мух, а концентрация ДНК в свежих образцах мух выше, чем в старых образцах мух. Мухи могут долго храниться в спирте.
Введение
Вывод о времени смерти всегда был одним из ключевых и сложных вопросов, требующих решения в судебной практике. В практике криминалистики для уголовного дела определение интервала вскрытия играет решающую роль в определении времени совершения преступления, установлении подозреваемого и сужении рамок расследования. Традиционные методы определения времени смерти основаны в первую очередь на ранних посмертных явлениях, которые, как правило, могут быть использованы только для определения времени смерти в течение 24 часов. Однако длительное время смерти не может быть определено посмертными явлениями. В настоящее время в судебно-медицинском сообществе общепризнано, что судебная энтомология может стать эффективным методом определения более длительного времени смерти.
Насекомые-некрофаги названы так из-за их личинок, которые питаются трупами. Среди них, когда присутствует труп, взрослые некрофильные мухи будут первыми, кто доберется до трупа и отложит свои яйца или личинки. Личинки питаются трупной тканью и превращаются в куколок, которые оперяются во взрослых особей. Некрофильные мухи играют огромную роль в практике криминалистики из-за их регулярного жизненного цикла и географического распространения, например, вычитая время смерти и определяя место смерти 1,2. Однако барьером для использования некрофильных мух в криминалистической практике является видовая идентификация. Морфологические методы до сих пор используются в качестве авторитетного метода для видовой идентификации некрофильных мух. Однако морфологическая идентификация видов требует высокой степени целостности насекомых. Если мухи находились на разных стадиях развития или из-за морфологических изменений, вызванных выбором окружающей среды, все это затрудняет морфологическое исследование. В частности, морфология куколок и панцирей куколок, которые чаще всего обнаруживаются на месте преступления, едва узнаваема. Это, наряду с нехваткой специалистов по морфологии, создает огромные трудности для морфологической идентификации видов. Таким образом, появилось применение методов молекулярной биологии для видовой идентификации мух, что снижает сложность морфологической идентификации и не зависит от стадии развития 3,4,5,6,7.
Первым шагом в идентификации видов некрофильных мух на основе методов молекулярной биологии является эффективная экстракция ДНК, в то время как специализированные наборы для экстракции ДНК насекомых в настоящее время обычно не доступны. А как использовать обычные наборы крови для извлечения ДНК некрофильных мух, становится все более актуальным с криминалистической точки зрения. Некрофильные мухи, найденные на местах преступлений, часто изуродованы или подверглись разложению и деградации ДНК. Образцы мух не доступны для извлечения ДНК сразу после получения, или, если это возможно, целые образцы должны быть сохранены из-за требований к сохранности доказательств. Исходя из вышеуказанных требований, в данном исследовании мы применили набор ДНК крови на основе расщепления протеиназы К и отобрали три свежих образца Chrysomya megacephala (Diptera, Lepidoptera) (рис. 1), один старый образец C. megacephala, помещенный в алкоголь на два года, и один образец C. megacephala , помещенный в защищенное от света хранилище на два года, взвесили каждый из пяти образцов. выбирал ткань грудной клетки насекомого для экстракции ДНК. Сравнивая качество и чистоту ДНК, выделенной из старого и нового образцов, проводили ПЦР-амплификацию и электрофорез экстрагированной ДНК с некрофильными мухоспецифичными праймерами, расположенными в митохондриальной последовательности гена CO 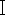 .
.
Access restricted. Please log in or start a trial to view this content.
протокол
Примечание: В общей сложности в этом протоколе было использовано пять образцов C. megacephala - три свежих образца (Fresh 1, Fresh 2 и Fresh 3), один образец хранился в спирте в течение 2 лет (Old 1) и один образец хранился в сухом герметичном хранилище в течение 2 лет, защищенном только от света (Old 2). Образцы должны быть промаркированы в соответствии с требованиями эксперимента.
1. Общее хранение и подготовка образцов
- Отбор и подготовка образцов
ПРИМЕЧАНИЕ: Обязательно работайте в вытяжном шкафу при нанесении этилацетата для уничтожения мух-некрофагов. Перед взвешиванием удалите жидкость с поверхности мухи.- Свежие образцы: Поместите три собранных образца мух в газовые флаконы после их уничтожения этилацетатом. Взвесьте и назовите образцы как Fresh 1, Fresh 2 и Fresh 3 и немедленно используйте их в экспериментах.
Примечание: Образцы свежей мухи в этом протоколе имели массу 56,8 мг, 49,3 мг и 52,1 мг. - Старый образец 1: Извлеките образцы от мух, убитые этилацетатом и хранящиеся в наполненных спиртом центрифужных пробирках в течение 2 лет в герметичной защите, защищенной от света при комнатной температуре. Взвесьте образец после адсорбции поверхностной жидкости фильтровальной бумагой; Назовем его Старый 1.
ПРИМЕЧАНИЕ: Масса Old 1 составила 42,3 мг в этом протоколе. - Старый образец 2: Извлеките убитый этилацетатом образец мухи и храните в центрифужной пробирке в течение 2 лет в герметичной защите, защищенной от света при комнатной температуре. Взвесьте и назовите его Старый 2.
- Свежие образцы: Поместите три собранных образца мух в газовые флаконы после их уничтожения этилацетатом. Взвесьте и назовите образцы как Fresh 1, Fresh 2 и Fresh 3 и немедленно используйте их в экспериментах.
- Подготовка перед экстракцией
- Добавьте небольшое количество жидкого азота для охлаждения емкости из нержавеющей стали; Дождитесь, пока весь жидкий азот испарится.
- Положите в контейнер образец от мухи; Медленно вливайте жидкий азот, чтобы жидкий азот не разбрызгивался и не попадал на образец насекомого.
- Заморозьте образцы в жидком азоте. Удаляйте образцы после того, как жидкий азот испарится.
- Отделите грудную клетку по отдельности, поместите в центрифужную пробирку объемом 2 мл и нарежьте на мельчайшие срезы.
ПРИМЕЧАНИЕ: Разрежьте образец на мелкие кусочки, чтобы его можно было полностью расщепить, и ДНК могла быть высвобождена в больших количествах. - Повторите описанные выше шаги для каждой мухи, заморозьте по отдельности, а затем переместите в новую центрифужную пробирку, чтобы предотвратить перекрестное загрязнение ДНК.
2. Экстракция ДНК
ПРИМЕЧАНИЕ: Все этапы центрифугирования должны выполняться при комнатной температуре (15-25 °C) в микроцентрифуге. Все действия должны выполняться в строгом соответствии с принципами асептики и во избежание перекрестного загрязнения.
- Лизис тканей
- Добавьте 180 мкл буфера для лизиса тканей в центрифужную пробирку объемом 2 мл, содержащую клипсированный образец.
- Добавьте 20 мкл протеиназы K. Тщательно перемешайте при вортексе при 3 000 об/мин в течение 10 с и инкубируйте при 56 °C до полного лизирования тканей насекомого.
ПРИМЕЧАНИЕ: Добавлять больше протеиназы К следует в том случае, если ткань не полностью переварена. Лизис обычно завершается за 1-3 часа, а также может продлить время лизиса на ночь.
- Осаждение ДНК
- Вихрь при 3 000 об/мин в течение 15 с. Добавьте в образец 200 мкл буфера для лизиса и тщательно перемешайте путем вортексирования при 3 000 об/мин в течение 15 с.
- Добавьте 200 μл этанола (96-100%) и снова тщательно перемешайте, перемешивая при 3 000 об/мин в течение 15 секунд.
ПРИМЕЧАНИЕ: Буфер для лизиса и этанол могут быть предварительно добавлены вместе за один шаг для экономии времени. Белый осадок может образоваться при добавлении лизисного буфера и этанола.
- Пипетируйте смесь в адсорбционную колонку ДНК, помещенную в пробирку для сбора объемом 2 мл. Центрифугируйте при 6 000 × г в течение 1 мин. Выбросьте проточную и сборную трубку.
- Поместите адсорбционную колонку ДНК из предыдущего этапа в новую пробирку для сбора объемом 2 мл, добавьте 500 мкл буфера для удаления белка и центрифугируйте при 6000 × г в течение 1 минуты. Выбросьте проточную и сборную трубку.
- Перенесите адсорбционную колонку ДНК в новую пробирку для сбора объемом 2 мл, добавьте 500 мкл опреснительного буфера и центрифугируйте при температуре 20 000 × г в течение 3 минут, чтобы высушить мембрану адсорбционной колонки ДНК. Выбросьте проточную и сборную трубку.
ПРИМЕЧАНИЕ: Эта стадия центрифугирования обеспечивает удаление остатков этанола перед последующим элюированием. Если есть остатки этанола, снова центрифугируйте при 20 000 × г в течение 1 минуты. - Поместите адсорбционную колонку ДНК в чистую микроцентрифужную пробирку объемом 2 мл, непосредственно нанесите 100 мкл буфера для элюирования ДНК на мембрану колонки, инкубируйте при комнатной температуре в течение 1 минуты, а затем центрифугируйте при 6000 × г в течение 1 минуты для элюирования.
ПРИМЕЧАНИЕ: Элюирование 100 мкл (вместо 200 мкл) увеличивает конечную концентрацию ДНК в элюате, но также снижает общий выход ДНК. Чтобы максимизировать выход ДНК, повторите элюирование один раз, как описано в шаге 2.8. - Храните образцы ДНК при температуре 4 °C (до 1 недели) или -80 °C для длительного хранения.
3. Определение концентрации ДНК
- Определите значения OD 260 и OD 280 для сравнения чистоты ДНК и концентрации нуклеиновых кислот с помощью считывателя микропланшетов. Повторите три раза на каждом образце.
- Определите концентрацию извлеченной ДНК с помощью флуориметра с помощью сопутствующего набора, с тремя репликами для каждого образца.
- В две центрифужные пробирки объемом 0,5 мл взять по 190 мкл рабочего раствора, взять по 10 мкл Стандарта #1 и Стандарта #2, добавить их в рабочие растворы и хранить в незащищенном от света месте не менее 15 минут.
ПРИМЕЧАНИЕ: Стандарт #1 и Стандарт #2 точно откалиброваны для определенной концентрации ДНК. Стандартные решения должны быть точно сконфигурированы, чтобы обеспечить формирование точной стандартной кривой. Стандарт #1 представляет собой образец двухцепочечной ДНК (дцДНК) с плотностью 0,2 нг/мкл; стандарт #2 составляет 2 000 нг/μл. Рабочим раствором является краситель, который специфически связывается с дцДНК. - Возьмите 2 μL исследуемой ДНК и смешайте его со 198 μL рабочего раствора в центрифужной пробирке объемом 0,5 мл и держите в незащищенном от света месте не менее 15 минут.
ПРИМЕЧАНИЕ: Если концентрация ДНК низкая, добавьте 5 μL исследуемой ДНК со 195 μL рабочего раствора. - Через 15 минут включите прибор и выберите «Измерение концентрации дцДНК» | Большой запас хода. Сначала протестируйте Стандарт #1, а затем Стандарт #2 для получения стандартной кривой.
ПРИМЕЧАНИЕ: Стандартное решение должно быть использовано в тот же день после его настройки; Если оставить его слишком долго, это приведет к большой ошибке в стандартной кривой. - Поместите исследуемые образцы в пробирные лунки один за другим, отрегулируйте объем спайка ДНК до 2 μл, повторите измерение три раза, возьмите среднее значение и запишите.
- В две центрифужные пробирки объемом 0,5 мл взять по 190 мкл рабочего раствора, взять по 10 мкл Стандарта #1 и Стандарта #2, добавить их в рабочие растворы и хранить в незащищенном от света месте не менее 15 минут.
4. ПЦР и электрофоретическое обнаружение
ПРИМЕЧАНИЕ: Последовательность митохондриального гена CO 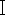 длиной 278.о. была взята для амплификации методом ПЦР с помощью прямого праймера C1-J-2495: CAGCTACTTTATGAGCTTTAGG и обратного праймера C1-N-2800: CATTTCAAGCTGTGTAAGCATC8, а затем был проведен электрофорез в 2% агарозном геле для проверки эффекта амплификации и верифицирована длина продуктов амплификации.
длиной 278.о. была взята для амплификации методом ПЦР с помощью прямого праймера C1-J-2495: CAGCTACTTTATGAGCTTTAGG и обратного праймера C1-N-2800: CATTTCAAGCTGTGTAAGCATC8, а затем был проведен электрофорез в 2% агарозном геле для проверки эффекта амплификации и верифицирована длина продуктов амплификации.
- Настройте 20 мкл системы амплификации ПЦР, добавив 10 мкл 2x смеси Taq, 1 мкл каждого праймера, 5 мкл ddH2O и 3 мкл матричной ДНК; Хорошо перемешайте в течение 10 с в центрифуге.
- Используйте следующие параметры цикла ПЦР: предденатурация 95 °C в течение 10 мин; денатурация 95 °С в течение 30 с, отжиг 48 °С в течение 30 с, растяжение 72 °С в течение 45 с, всего 40 циклов; и окончательное продление до 72 °C в течение 10 мин.
- Подвергайте продукты после ПЦР электрофорезу с 2% агарозным гелем, а после электрофореза помещайте их на систему визуализации геля для фотоанализа.
Access restricted. Please log in or start a trial to view this content.
Результаты
Мы использовали считыватель микропланшетов для измерения значений OD260 и OD280 экстрагированного раствора ДНК, а затем получили значения OD260/OD280 для оценки чистоты ДНК. OD260/OD280 всех свежих образцов и старого образца 1 был больше 2. OD260/OD280 старого образца 2 имел максимальное значение 2,187, хотя с?...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Экстракция ДНК является наиболее важным звеном в молекулярной идентификации некрофильных мух, а метод ее экстракции и качество извлеченной ДНК напрямую влияют на последующее обнаружение. В этом эксперименте мы успешно извлекли ДНК из некрофильных мух с помощью обычного набора крови, ...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов.
Благодарности
Эта работа поддержана Национальным фондом естественных наук Китая (82060341,81560304) и научно-исследовательским проектом «Инновационная платформа академика провинции Хайнань» (YSPTZX202134).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
Ссылки
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214(2021).
- Cai, J. -F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532(2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699(2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375(2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778(2023).
Access restricted. Please log in or start a trial to view this content.
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены