Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Экстракция РНК с использованием одной пробирки на одном животном для сравнения относительных уровней транскриптов с помощью qRT-ПЦР у тихоходки Hypsibius exemplaris
В этой статье
Резюме
В данной работе представлен метод быстрой экстракции РНК и сравнения уровня транскрипта для анализа экспрессии генов у тихоходки Hypsibius exemplaris. Этот высокопроизводительный метод, использующий физический лизис, требует в качестве исходного материала одну тихоходку и приводит к надежному производству кДНК для количественной полимеразной цепной реакции с обратной транскрипцией (qRT-PCR).
Аннотация
Тихоходка Hypsibius exemplaris — это новый модельный организм, известный своей способностью выживать в экстремальных условиях окружающей среды. Чтобы изучить молекулярные механизмы и генетическую основу такой экстремальной толерантности, многие исследования опираются на секвенирование РНК (РНК-секвенирование), которое может быть выполнено на популяциях от больших когорт до отдельных животных. Полимеразная цепная реакция с обратной транскрипцией (ОТ-ПЦР) и РНК-интерференция (РНК-интерференция) впоследствии используются для подтверждения результатов РНК-секвенирования и оценки генетических требований к генам-кандидатам, соответственно. Для таких исследований требуется эффективный, точный и доступный метод экстракции РНК и измерения относительных уровней транскриптов с помощью количественной ОТ-ПЦР (qRT-PCR). В этой работе представлен эффективный метод экстракции РНК с одной тихоходкой (STST), который не только надежно изолирует РНК от отдельных тихоходок, но и сокращает необходимое время и затраты на каждую экстракцию. Этот метод экстракции РНК позволяет получить количество кДНК, которое может быть использовано для амплификации и обнаружения множественных транскриптов с помощью количественной ПЦР (qRT-PCR). Метод валидирован путем анализа динамических изменений в экспрессии генов, кодирующих два белка, регулируемых тепловым шоком, Heat-Shock Protein 70 β2 (HSP70 β2) и Heat-Shock Protein 90α (HSP90α), что позволяет оценить их относительные уровни экспрессии у лиц, подвергшихся тепловому воздействию, с помощью qRT-PCR. STST эффективно дополняет существующие методы экстракции объемной и одиночной РНК тихоходки, позволяя быстро и недорого исследовать отдельные уровни транскрипции тихоходок с помощью qRT-PCR.
Введение
Тихоходки — это небольшие многоклеточные животные, известные своей способностью выживать в экстремальных условиях, которые смертельны для большинства другихформ жизни. Например, эти животные могут пережить почти в 1000 раз большую дозу ионизирующего излучения, которая смертельна для человека 2,3,4,5,6,7,8,9,10, почти полное высыхание 11,12,13,14,15, замораживание в отсутствие добавленного криопротекторы 16,17,18, а в их высохшем состоянии даже вакуум космоса 19,20. Благодаря своей уникальной способности к выживанию в экстремальных условиях, эти животные стали основополагающими моделями для понимания экстремальной толерантности у сложных многоклеточных организмов 1,21,22,23.
Стабильные генетические манипуляции с этими замечательными животными, включая трансгенез и модификацию генов зародышевой линии, до недавнего времени оставались неуловимыми24,25. Таким образом, большинство экспериментов по выявлению молекулярных механизмов экстремотолерантности проводятся с помощью транскрипционного профилирования с помощью секвенирования РНК. Существует множество ценных и информативных наборов данных по секвенированию РНК для тихоходок в различных экстремальных условиях, начиная от радиации 8,9,26,27,28, теплового стресса29, стресса замерзания 12 и высыхания 27,30,31,32,33. В некоторых из этих исследований использовались методы экстракции и очистки объемной РНК, чтобы пролить свет на наше молекулярное понимание экстремальной толерантности. Тем не менее, массовая экстракция транскриптов РНК у многих животных препятствует анализу вариаций в экспрессии генов между особями, тем самым упуская потенциальное богатство более точных наборов данных. Важно отметить, что в этих исследованиях часто анализируются гетерогенные популяции животных, которые включают в себя как животных, переживающих стрессовые факторы окружающей среды, так и тех, кто этого не делает. Таким образом, эти исследования осложняются усреднением данных экспрессии из нескольких и потенциально сильно различающихся состояний ответа. Чтобы решить эту проблему, Arakawa et al., 201634 разработали элегантный конвейер с низким входом РНК-секвенирования, в котором используется набор для экстракции РНК с последующим этапом линейной амплификации ПЦР с использованием одного 34,35,36 или нескольких 30,37,38 животных в качестве входных данных. Эти исследования были основополагающими для нашего понимания экстремальной толерантности тихоходок22. Интересно, что этот протокол также был применен к количественной ОТ-ПЦР с использованием семи животных в качестве исходного материала24.
У большинства модельных организмов, после идентификации потенциальных мишеней с помощью РНК-секвенирования, затем выполняется кОТ-ПЦР для подтверждения транскрипционных изменений, идентифицированных с помощью РНК-секвенирования, и оценки временного хода экспрессии генов-кандидатов с высоким разрешением. Для проверки функции идентифицированных генов за такими исследованиями часто следует РНК-опосредованный нокдаун молекулярных мишеней39,40 и анализ экстремотолерантной способности12,41. Эффективность каждого нокдауна РНК-интерференции обычно подтверждается с помощью кОТ-ПЦР путем непосредственного мониторинга снижения распространенности транскриптов. Тем не менее, РНК-интерференция является трудоемким процессом у тихоходок, так как каждая дцРНК должна быть доставлена путем ручной микроинъекции индивидуумов39,40. Из-за низкой пропускной способности этой стратегии быстрый и недорогой метод экстракции РНК, адаптированный для qRT-PCR от одиночных животных, будет очень ценным для исследований тихоходок. Несмотря на то, что предыдущие методы были разработаны для извлечения РНК из одиночных тихоходок, эти протоколы не объединяли их экстракцию с qRT-PCR, вместо этого полагаясь на методы, основанные на оптической плотности 12,40,41. Руководствуясь этими проблемами, мы стремились разработать протокол, который бы надежно получал РНК в таком количестве и качестве, которые можно было бы использовать для qRT-PCR из одного H. exemplaris.
Адаптированный на основе протокола экстракции РНК одного животного, разработанного для Caenorhabditis elegans42, STST оптимизирован для H. exemplaris. Метод экстракции состоит из шести быстрых этапов замораживания-размораживания, физически разрушающих кутикулу, что позволяет экстракционировать РНК и последующий синтез кДНК. Метод STST сокращает время экстракции более чем в 24 раза по сравнению с методами экстракции объемной РНК, как описано в Boothby, 201843, и на 30% по сравнению с наборами для экстракции одиночной РНК тихоходки, как описано в Arakawa et al., 201634. Кроме того, количество взаимодействий образца и экспериментатора уменьшается с 5 до 1 по сравнению с препаратами наборов для экстракции РНК, что снижает риск загрязнения экзогенными рибонуклеазами. При запросе высокоэкспрессированных генов метод STST производит достаточное количество кДНК для 25 количественных реакций ОТ-ПЦР на одну тихоходку, требуя всего 1 мкл из общего объема кДНК в 25 мкл на одну реакцию. Тем не менее, матричные концентрации должны быть определены эмпирически для транскриптов с более низкой численностью.
Эффективность метода STST для анализа динамических изменений экспрессии генов оценивали путем исследования дифференциальной экспрессии генов, кодирующих белок теплового шока-90α (HSP90α) и белок теплового шока 70β2 (HSP70β2), в ответ на кратковременный тепловой шок при 35°C в течение 20 минут. Как HSP70β2, так и HSP90α у большинства эукариотических организмов быстро активируются после кратковременного воздействия теплового шока (20 минут)42. Анализ H. exemplaris показал, что как HSP70β2, так и HSP90α-кодирующие РНК, выделенные из одиночных термически обработанных тихоходок, показали статистически значимое увеличение экспрессии после кратковременного теплового воздействия. Эти результаты демонстрируют, что протокол STST может быть использован для анализа динамических изменений экспрессии генов у отдельных животных с течением времени.
Метод экстракции STST должен дополнять существующие экспериментальные методы, такие как РНК-секвенирование, облегчая быструю и недорогую экстракцию РНК и последующее сравнение уровней транскриптов с помощью qRT-PCR. Этот метод также будет полезен для оценки эффективности и пенетрантности РНК-интерференции у людей, введенных вручную, более количественно, чем только оптическая плотность. Наконец, благодаря схожим структурам кутикуляра и физическим характеристикам, вполне вероятно, что этот метод будет также эффективен для анализа экспрессии генов у других видов тихоходок44.
протокол
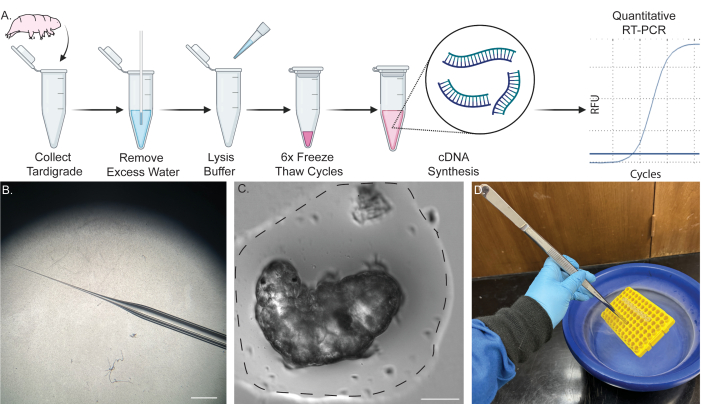
Рисунок 1: Однотрубочный конвейер для экстракции РНК у одной тихоходки. (A) Схема, показывающая протокол для экстракции РНК у одной тихоходки, включающий шесть циклов замораживания-размораживания и последующий синтез кДНК. В дальнейшем образцы могут быть использованы для проведения ОТ-ПЦР и кОТ-ПЦР. (B) Изображение конуса микропипетки, используемого для удаления воды. Масштабная линейка: 2 мм. (C) Яркое изображение тихоходки в небольшом объеме воды (пунктирная линия). Удаление большей части воды в указанном объеме необходимо для успешной экстракции и предотвращает разбавление буфера для лизиса. Масштабная линейка: 50 μм. (D) Изображение, показывающее погружение образцов в жидкий азот с использованием длинных щипцов для быстрого замораживания и безопасного размораживания образцов. Часть контента была создана в BioRender. Кирк, М. (2022) BioRender.com/d93s511 Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
ПРИМЕЧАНИЕ: На рисунке 1А показана схема процедуры. Для получения подробной информации о процедурах культивирования тихоходок и водорослей см. ранее опубликованные отчеты 45,46,47.
1. Стерилизация родниковой воды
- Налейте 2 л родниковой воды из 5-галлонного кувшина для воды (см. Таблицу материалов для подробностей) в стеклянную бутылку объемом 2 л, пригодную для автоклава.
- Наденьте крышку на безопасную для автоклава бутылку и закройте ее небольшим количеством автоклавной ленты. Не затягивайте бутылку; Сверху наденьте колпачок.
- Автоклав родниковой воды в течение 50 минут во влажном цикле без этапа сушки.
- Дайте воде нагреться до комнатной температуры (RT) и плотно закройте крышку перед хранением при RT.
2. Стеклянная микропипетка для вытягивания (с помощью съемника для пипетки)
- Закрепите стеклянную микропипетку (Наружный диаметр: 1 мм, Внутренний диаметр: 0,58 мм, Длина: 10 см) на съемнике микропипетки. Избегайте контакта с нагревательной нитью, так как это может изменить форму пипетки и повредить нить.
- Определите вытягивание пипетки опытным путем для каждой нити накала и съемника пипетки. Тем не менее, чтобы служить отправной точкой для оптимизации, используйте температуру 78 °C и один шаг вытягивания с весом 182,2 г.
- Дайте нити нагреться и под действием силы тяжести разделите стеклянную микропипетку на две стеклянные микропипетки с острыми концами (рис. 1B).
- Храните эти стеклянные микропипетки в закрытой 100-миллиметровой чашке Петри с воском или глиной, чтобы удерживать их на месте и предотвратить поломку острых кончиков.
3. Стеклянное вытягивание микропипеток (без съемника пипетки)
- Зажгите горелку Бунзена или другой контролируемый источник пламени на слабом уровне.
- Возьмите стеклянную микропипетку с одним концом в каждой руке.
- Держите центр стеклянной микропипетки над пламенем, пока стекло не начнет плавиться. Затем быстро раздвиньте два конца. Так получится два очень нежных острых кончика.
- Слегка сломайте кончик парой стерильных тонких щипцов.
- Храните эти стеклянные микропипетки в закрытой 100-миллиметровой чашке Петри с воском или глиной, чтобы удерживать их на месте и предотвратить поломку острых кончиков.
4. Экстракция РНК
- Получите 0,5 л жидкого азота в криобезопасной емкости.
ВНИМАНИЕ: Жидкий азот является криогенным и может вызвать ожоги при контакте с кожей или глазами. При работе используйте защитную одежду, брызговики, нитриловые перчатки, криоперчатки, лабораторный халат и обувь с закрытыми носками. Перед транспортировкой жидкости убедитесь, что контейнер безопасен для жидкого азота. Также можно использовать ванну с этанолом и сухим льдом для этого этапа. - Приготовьте мастер-смесь для синтеза кДНК: раствор объемом 10 мкл, содержащий 1 мкл праймера случайного гексамера, 2 мкл ДНКазы, 4 мкл 5x RT буфера, 1 мкл ферментной смеси, 1 мкл H2O и 1 мкл 10 мл dNTPs. Хранят такой раствор на льду.
- Приготовьте буфер для лизиса тихоходок (5 мМ Трис (pH = 8), 0,5% (v/v) Моющее средство 1, 0,5% (v/v) Моющее средство 2, 0,25 mM EDTA в стерильной воде, не содержащей нуклеазы).
ПРИМЕЧАНИЕ: Этот раствор можно хранить на столе в течение 6 месяцев. Тем не менее, поддерживайте стерильность и избегайте потенциальных источников, загрязняющих РНК. - Аликвота достаточна буфера для лизиса для экстракции (2 μл/тихоходка).
- Добавьте ингибитор РНКазы в буферный раствор для лизиса тихоходок до конечной концентрации 4 Ед/мкл.
- Сделайте вихрь и раскрутите раствор в RT на настольной центрифуге со скоростью 2000 x g в течение 5 с перед хранением раствора на льду.
- Удалите из культуры столько тихоходок, сколько необходимо для эксперимента, с помощью стерильной пипетки P1000 с фильтром и поместите их в стерильную чашку Петри диаметром 35 мм.
ПРИМЕЧАНИЕ: Таким образом может быть обработано любое количество тихоходок. Обычно для экстракции обрабатывают трех тихоходок в одном состоянии. - Промойте тихоходок три раза, используя 1 мл стерильной родниковой воды и стерильную пипетку P1000 с фильтрующим наконечником. Медленное пипетирование вверх и вниз помогает удалить водорослевые загрязнения.
- Используя препарирующий микроскоп с увеличением от 25 до 50 раз, перенесите одну тихоходку из этой промытой культуры в новую стерильную чашку Петри диаметром 35 мм с помощью стерильной пипетки P10 с фильтрующим наконечником.
- Используйте стерильную пипетку P200 с фильтрующим наконечником, чтобы промыть одиночную тихоходку в 100 μл стерильной воды, не содержащей нуклеаз.
ПРИМЕЧАНИЕ: Этот этап промывки используется для дальнейшего удаления загрязнений, включая рибонуклеазы. - Переложите промытую тихоходку на дно чистой стерильной ПЦР-пробирки в 1-2 мкл стерильной воды без нуклеаз с помощью стерильной пипетки P10 с фильтрующим наконечником, тщательно следя за тем, чтобы тихоходка не прилипла к боковой стороне наконечника.
- Визуализируйте тихоходку под препарирующим микроскопом при 25-кратном увеличении.
- Чтобы облегчить удаление воды, слегка разбейте кончик стеклянной микропипетки наружу пробирки. Убедитесь, что отверстие достаточно большое, чтобы поднимать воду, но не тихоходку.
- Используя капиллярное действие вытянутой стеклянной микропипетки, удаляйте воду до тех пор, пока животное не будет окружено небольшим пузырем воды диаметром примерно две тихоходки.
- Контролируйте процесс удаления воды с помощью препарирующего эндоскопа, чтобы убедиться, что уровень воды соответствует действительности и тихоходка остается гидратированной.
ПРИМЕЧАНИЕ: На рисунке 1C приведен пример того, сколько воды необходимо удалить. Это критически важный шаг. Тихоходка будет окружена небольшим пузырем воды, чтобы предотвратить ее высыхание, но следует удалить как можно больше лишней воды, чтобы предотвратить разбавление буфера лизиса. Пример остальных уровней воды приведен на рисунке 1C. - Сразу после удаления воды добавьте на дно пробирки 2 мкл буфера для тихоходного лизиса, кратковременно завихрите, и центрифугируйте пробирку при RT в течение 5 с при 2000 x g на настольной центрифуге.
- Немедленно поместите образцы, содержащие тихоходок, в штатив для ПЦР-пробирок и убедитесь, что они плотно удерживаются штативом.
- Возьмитесь за штатив с помощью пары длинных грубых щипцов и осторожно опустите штатив с образцами в жидкий азот до полного замерзания (Рисунок 1D).
- Снимите решетку с жидким азотом и сразу же поставьте ее на лед. Дайте образцу оттаять (требуется от ~45 с до 1 минуты). Контролируйте образец каждые 15 с, извлекая его изо льда и осматривая на видном месте. Как только образец станет заметно прозрачным, переходите к следующему шагу.
- Повторите шаги 4.18-4.19 еще пять раз. Для максимального лизиса и экстракции требуется в общей сложности шесть циклов замораживания-размораживания (рис. 2A, B).
- После завершения замораживания-размораживания поместите образцы на лед и сразу же переходите к следующему этапу. Не замораживайте образцы на этом этапе для хранения, так как это уменьшит доступную РНК для получения кДНК.
5. Синтез кДНК
- Добавьте 2 мкл мастер-смеси для синтеза кДНК в ПЦР-пробирку, содержащую лизат тихоходки. Кратковременно переверните пробирку и вращайте ее при RT при 2000 x g в течение 5 с с помощью настольной центрифуги, прежде чем заменять образцы на лед.
- Поместите образцы в амплификатор и инкубируйте при 25 °C в течение 10 мин до отжига праймеров, при 55 °C в течение 30 мин для выполнения обратной транскрипции и, наконец, нагрейте инактивированные ферменты при 85 °C в течение 5 мин.
- После инкубации немедленно поместите пробирку на лед и разбавьте образец до общего объема 25 μл, добавив 21 μл стерильной воды, не содержащей нуклеаз. Для транскриптов с низким числом копий измените этот шаг разбавления в соответствии с эмпирическим путем.
6. Количественная ПЦР
- Определяют температуру отжига набора грунтовок с использованием общей РНК, полученной из большего количества тихоходок, например, методом объемной экстракции, представленным в Boothby, 201843.
- Запустите градиент температуры ПЦР для определения оптимальной температуры отжига перед запуском qRT-PCR (все настройки ПЦР, используемые в этом протоколе, см. в Таблице 1 и Таблице 2).
- Разморозьте один тюбик индикаторного красителя, смешайте на льду и изолируйте от света. Положите на лед 96-луночный планшет для количественной ПЦР и поместите 5 μл суперсмеси, 2 μл воды, 1 μл каждого праймера (10 μM) и 1 μл продукта кДНК в нужное количество лунок.
- Запечатайте ПЦР-планшет пломбой и запустите qRT-PCR с температурой отжига, подходящей для набора праймеров (все настройки qRT-PCR, используемые в этой статье, см. в таблице 3).
7. Количественная оценка и интерпретация результатов
- Сравните результаты количественно с одним или несколькими контрольными генами, экспрессия которых, как ожидается, будет постоянной в течение заданных условий. Для этого исследования был использован ген актина.
- Получите и сравнитезначения C t или порог цикла для каждой скважины со значениями Ct реакций контрольных генов. Рассчитайте кратное изменение экспрессии генов с помощью следующего уравнения:
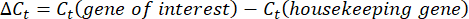
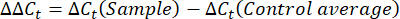
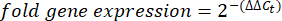
ПРИМЕЧАНИЕ: Экспрессия генов фолда построена для каждого транскрипта и тихоходки в виде 2-(ΔΔCt)48. - Чтобы получить приблизительную оценку числа транскриптов по t-значению C, используйте следующее уравнение:
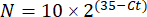
Где N — количество транскриптов, а 2 — предполагаемая эффективность ПЦР или кратное увеличение флуоресценции за цикл ПЦР48.
Результаты
Разработка и оптимизация экстракции одиночной тихоходки на РНК
Адаптируя протокол из Ly et al., 201542 для экстракции РНК у тихоходок, система STST оптимизирована для максимального увеличения количества и качества препарата (рис. 1A). ОТ-...
Обсуждение
В этом исследовании представлен эффективный метод экстракции РНК для qRT-PCR одиночной тихоходки. Прямое сравнение методологии STST с существующим набором для экстракции одиночной тихоходки показало, что экстракция STST РНК дает в >200 раз большее количество транскриптов ак...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Мы хотим выразить признательность стипендии NIH Ruth Kirschstein Fellowship # 5F32AG081056-02 и стипендии Эрретта Фишера, которая поддержала доктора Молли #2R01HD081266#R01GM143771Дж. Авторы также отмечают использование Лаборатории биологических наноструктур в Калифорнийском институте наносистем при поддержке Калифорнийского университета в Санта-Барбаре и Калифорнийского университета, канцелярии президента.
Материалы
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
Ссылки
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены