Method Article
Белковая инженерия с помощью дисплея поверхности дрожжей
В этой статье
Резюме
В этом протоколе описаны основные этапы проведения кампаний по выбору поверхности дрожжей для обогащения вариантов белка, связывающихся с представляющим интерес антигеном.
Аннотация
Белковая инженерия позволяет улучшить существующие функции данного белка или генерировать новые функции. Одним из наиболее широко используемых и универсальных инструментов в области белковой инженерии является отображение поверхности дрожжей, где пул рандомизированных белков экспрессируется на поверхности дрожжей. Сцепление фенотипа (например, связывание белка, отображаемого дрожжами, с интересующим антигеном) и генотипа (плазмида, кодирующая вариант белка) позволяет выбрать эту библиотеку по желаемым свойствам и последующее секвенирование обогащенных вариантов. Сочетая селекцию магнитных шариков с проточной цитометрической сортировкой, можно отбирать и обогащать варианты белков с усиленным связыванием с целевым антигеном. Примечательно, что в дополнение к созреванию аффинности, связывание с мишенью также может быть достигнуто без какого-либо начального аффинности связывания. Здесь мы предоставляем пошаговый протокол, который охватывает все основные части кампании по выбору дрожжевой поверхности и дает примеры типичных результатов дрожжевой поверхности. Мы демонстрируем, что отображение поверхности дрожжей является широко применимым и надежным методом, который может быть установлен в любой молекулярно-биологической лаборатории, имеющей доступ к проточной цитометрии.
Введение
Отображение поверхности дрожжей является одной из ключевых технологий в области белковой инженерии. Это позволяет выбирать варианты белка с желаемыми свойствами, такими как повышенное сродство или стабильность. Впервые представленный в 1997году, он является одной из наиболее часто используемых технологий отображения наряду с фаговым дисплеем 2,3, рибосомным дисплеем4 и дисплеем клеток млекопитающих 5,6,7. Представляющий интерес белок (POI) отображается на поверхности дрожжевых клеток путем его слияния с якорными белками. Существует целый ряд различных якорных белков, и чаще всего POI сливается с С-концом белка спаривания агглютинина дрожжей Aga2p 1,8. Кроме того, POI обычно окружен двумя метками, такими как метка гемагглютинина (HA-метка) и метка c-myc, что позволяет определять уровень отображения с помощью флуоресцентно меченных антител и проточной цитометрии (рис. 1A). Типичные кампании по отбору дрожжей включают в себя комбинацию отбора магнитных гранул и проточной цитометрической сортировки. Отбор шариков позволяет обрабатывать большое количество клеток и обогащать варианты белка, связывающиеся с антигеном-мишенью, также с низким сродством, поскольку поливалентные взаимодействия с нагруженными антигеном шариками приводят к эффектам авидности и, следовательно, предотвращают потерю вариантов с низким сродством (рис. 1B). Проточный цитометрический анализ и отбор дают преимущество визуализации связывания отображаемых вариантов POI с меченым антигеном. Следовательно, связывающие популяции могут быть отсортированы и культивированы, что приводит к обогащению вариантов белка с желаемыми характеристиками в течение нескольких раундов сортировки. Кроме того, могут быть проведены дополнительные раунды случайного мутагенеза для дальнейшего увеличения разнообразия и, следовательно, вероятности обнаружения дополнительных мутаций, которые способствуют аффинности и/или стабильности белка.
Отображение поверхности дрожжей имеет определенные преимущества, такие как (а) механизм экспрессии эукариот, обеспечивающий окислительное сворачивание белка, а также посттрансляционные модификации эукариот (такие как N-гликозилирование), (б) нормализация экспрессии за счет обнаружения двух пептидных меток, фланкирующих белок, (в) визуальный контроль хода отбора с помощью проточной цитометрии (например, процент связывающих клеток и интенсивность связывания) и (г) возможность анализа отдельных белковых мутантов на дрожжевые дрожжи (например, анализирующие термостабильность, а также аффинность), представляющие собой экономящую время альтернативу трудоемкой экспрессии и очистке белков9. Фактически, как сродство (значения KD), так и стабильность (значения T50) белков, отображаемых на поверхности дрожжей, показали хорошие корреляции с данными, полученными с использованием биофизических методов и растворимых белков 9,10,11,12. Поверхностный дисплей дрожжей был использован для конструирования различных белков, например, фрагментов антител 13,14,15,16, фибронектинового домена10-го типа III типа17,18, rcSso7d19,20 или кнотинов21. Аналогичным образом, были проведены обширные исследования по оптимизации дизайна библиотек дрожжей путем изменения рандомизированных положений, а также использования кодонов аминокислот 17,22,23. Дрожжевая поверхностная индикация оказалась успешной для обеспечения стабильности 14,15,24,25, аффинности 18,26,27, ферментативной активности 28,29,30,31 и экспрессии белка 32. Кроме того, более сложные приложения, такие как условное связывание в присутствии или отсутствии малой молекулы, были реализованы с использованием дисплея поверхностидрожжей 20.
В этом протоколе мы описываем все основные этапы селекционной кампании с отображением поверхности дрожжей на примере библиотеки G4 (основанной на10-м домене фибронектина III типа, Fn3), отобранной против антигена человеческого ретинолсвязывающего белка 4 (hRBP4) в присутствии малой молекулы A112020. Этот отбор был проведен для получения белок-белкового взаимодействия, которое зависит от небольшой молекулы, которая может быть использована в качестве молекулярного переключателя. Следует отметить, что в то время как альтернативные подходы возможны при отображении поверхности дрожжей, типичный выбор дрожжей обычно направлен на связывание с целевым антигеном без какого-либо предыдущего сродства к связыванию. Мы охватываем все этапы кампании по отбору дрожжей, включая культивирование дрожжевой библиотеки, отбор гранул, проточную цитометрическую сортировку и созревание аффинности с помощью подверженной ошибкам ПЦР (эпПЦР). Таким образом, данный протокол дополняет предыдущие протоколы отображения поверхности дрожжей33,34 и может быть использован в качестве основы для выбора поверхностного отображения дрожжей (рис. 1) с любой заданной библиотекой дрожжей и целевым антигеном по выбору.
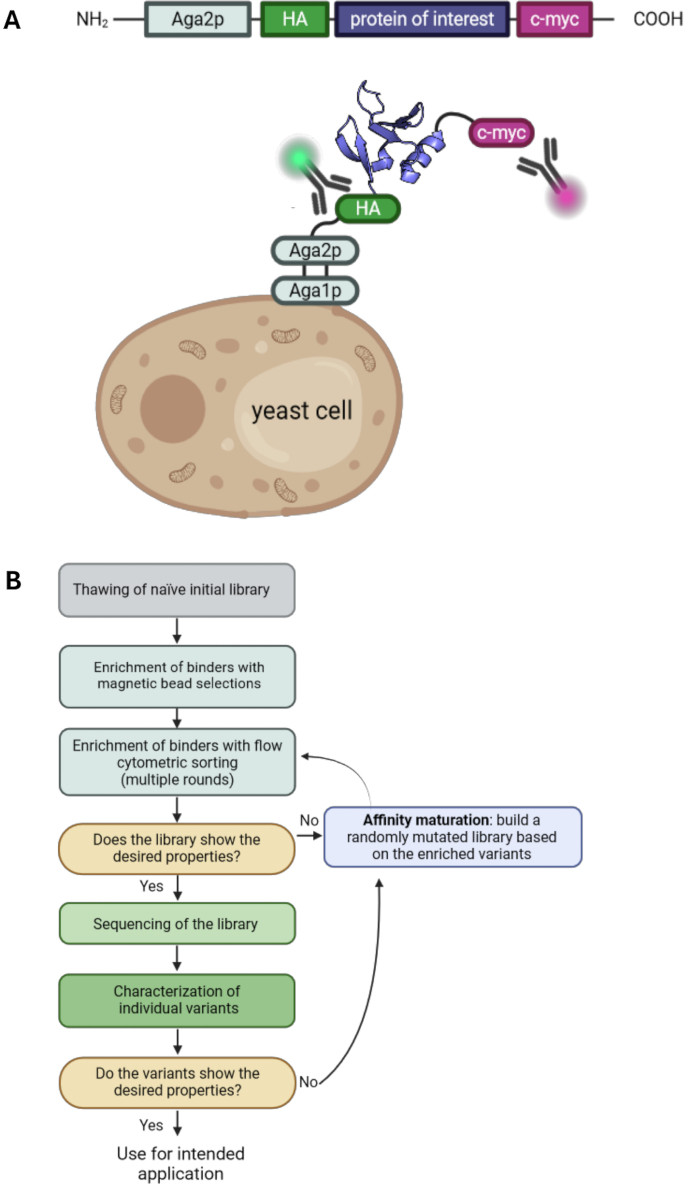
Рисунок 1: Принцип отображения дрожжевой поверхности и типичный рабочий процесс для выбора дрожжевой поверхности. (A) POI клонируется в вектор отображения поверхности дрожжей и, как правило, окружен N-концевой HA- и C-концевой c-myc-меткой. Конструкция сплавлена с дрожжевым брачным белком Aga2p для отображения на поверхности. Изображенный белок представляет собой сконструированное связующее вещество "RS3" из PDB ID: 6QBA20. (B) Блок-схема, иллюстрирующая типичный рабочий процесс для кампаний по выбору дрожжевых поверхностей, которые сочетают обогащение вариантов белка желаемыми свойствами путем отбора гранул и проточной цитометрической сортировки, а также эпПЦР для аффинного созревания. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
протокол
1. Размораживание и выращивание дрожжевых библиотек
- Разморозьте достаточное количество аликвот библиотеки замороженных дрожжей в RT и немедленно разбавьте в SD-CAA (см. Таблицу 1) до OD600 , равного примерно 1 (это соответствует 1 x 107 клеток/мл).
ПРИМЕЧАНИЕ: Убедитесь, что вы охватываете разнообразие библиотеки как минимум в 10 раз. Таким образом, если размер библиотеки составляет 2,5 x 108 клонов (таково разнообразие библиотеки G4, используемой в данном примере), инокулируйте минимум 2,5 x 109 клеток в 250 мл SD-CAA, чтобы получить внешний диаметр600 из 1. Этот протокол также может быть начат с жидких культур (дрожжевых клеток, только что преобразованных с помощью библиотеки). - Оцените количество жизнеспособных клеток.
- Возьмите аликвоту объемом 100 мкл из этой дрожжевой суспензии (при наружном диаметре600 из 1) и выполните серию разведения 1:10 с 900 мкл среды SD-CAA в микроцентрифужных пробирках.
- Приготовьте разведения до 10-5 и наберите 100 мкл разведений 10-3, 10-4 и 10-5 на планшетах SD-CAA (табл. 1). Выдерживайте пластины при температуре 30 °C в течение 2-3 дней. Убедитесь, что количество жизнеспособных ячеек после размораживания превышает разнообразие библиотеки как минимум в 10 раз.
- Тем временем продолжайте использовать оставшуюся клеточную суспензию и культивируйте дрожжевую культуру, содержащую библиотеку G4, в стеклянных колбах в течение ночи при температуре 30 °C при встряхивании (180 об/мин). После ночной инкубации продолжайте работу с разделом 2 (Индукция экспрессии белка на поверхности дрожжей).
- Через 2-3 дня инкубации подсчитайте колонии на планшетах SD-CAA путем визуального осмотра. При расчете окончательного числа жизнеспособных клеток после размораживания учитывайте коэффициент разведения и соотношение покрытого (т.е. 100 мкл) и исходного объема культуры (т.е. 250 мл).
Жизнеспособные клетки/мл = число колоний на пластине × коэффициенте разведения
Жизнеспособные клетки (всего) = (жизнеспособные клетки/мл) × объем культуры
| Среда/буфер | Компонент | Концентрация [г/л] | Комментарии/Описание | |||
| SD-CAA | D-глюкоза | 20 | Растворите все компоненты среды в 1000 мл ddH,2O и стерильно фильтруйте с помощью одноразовых стерильных фильтров 0,22 мкм. | |||
| Азотистая основа дрожжей | 6.7 | |||||
| Кислоты касминоса | 5 | |||||
| Моногидрат лимонной кислоты | 7.4 | |||||
| Дигидрат цитрата натрия дигидрат | 10.83 | |||||
| SG-CAA | D-галактоза | 20 | Растворите все компоненты среды в 1000 мл ddH,2O и стерильно фильтруйте с помощью одноразовых стерильных фильтров 0,22 мкм. | |||
| D-глюкоза | 2 | |||||
| Азотистая основа дрожжей | 6.7 | |||||
| Казаминокислоты | 5 | |||||
| динатрия гидрофосфат гептагидрат | 10.2 | |||||
| Натрия дигидрофосфата моногидрат | 8.56 | |||||
| Пластины SD-CAA | Сорбит | 182 | Растворить сорбит, динатрия гидрофосфат гептагидрат, натрия дигидрофосфата моногидрат и агар-агар в 900 мл ддН2О и автоклав. Растворите и стерильно отфильтруйте оставшиеся компоненты в 100 мл ddH2O и добавляйте, когда автоклавная среда станет теплой. | |||
| динатрия гидрофосфат гептагидрат | 10.2 | |||||
| Натрия дигидрофосфата моногидрат | 7.44 | |||||
| Агар-агар | 15 | |||||
| D-глюкоза | 20 | |||||
| Азотистая основа дрожжей | 6.7 | |||||
| Казаминокислоты | 5 | |||||
| YPD | Пептон | 20 | Приготовьте 10-кратный запас D-глюкозы (200 г/л) и стерильный фильтрат с помощью одноразовых стерильных фильтров 0,22 мкм. Растворите пептон и дрожжевой экстракт в 900 мл ddH2O и автоклав. Когда тепло потеплеет, добавьте 100 мл 10x D-глюкозы. | |||
| Дрожжевой экстракт | 10 | |||||
| D-глюкоза | 20 | |||||
| Пластины YPD | Пептон | 20 | Приготовьте 10-кратный запас D-глюкозы (200 г/л) и стерильный фильтрат с помощью одноразовых стерильных фильтров 0,22 мкм. Пептон, дрожжевой экстракт и агар-агар растворить в 900 мл ddH2O и автоклав. Когда тепло потеплеет, добавьте 100 мл 10x D-глюкозы. | |||
| Дрожжевой экстракт | 10 | |||||
| D-глюкоза | 20 | |||||
| Агар-агар | 15 | |||||
| PBSA | БСА | 1 | Растворите БСА в PBS и стерильно фильтруйте с помощью одноразовых стерильных фильтров 0,22 мкм. | |||
Таблица 1: Состав среды и буфера.
2. Индукция экспрессии белка на поверхности дрожжей
- После ночной инкубации дрожжевые культуры обычно достигают наружного диаметра600 в диапазоне от 2 до 20. Измерьте наружный диаметр600 с помощью фотометра, чтобы определить плотность дрожжевой культуры.
ПРИМЕЧАНИЕ: Измерения наружного диаметра являются линейными только в диапазоне 0,1-1. Поэтому выполняйте разведение (обычно 1:20 и 1:50) в SD-CAA, чтобы определить внешний диаметр600.- Разбавьте элементы в SD-CAA до наружного диаметра600 1 и убедитесь, что разнообразие покрыто в 10 раз (т.е. 2,5 x 109 элементов используются для разбавления библиотеки G4). Продолжайте инкубацию при температуре 30 °C при встряхивании, чтобы клетки достигли фазы экспоненциального роста.
- Приготовьте запасы глицерина (охватывающие разнообразие в 50 раз) библиотек дрожжей (в SD-CAA, содержащем 15% глицерина) из ночной культуры. Для этого центрифугируют необходимое количество дрожжевых клеток (2000 x g, 3 мин, 4 °C), отбрасывают надосадочную жидкость и ресуспендируют клеточную гранулу в 1 мл среды SD-CAA с добавлением 15% глицерина. Немедленно заморозьте запасы глицерина при температуре -80 °C.
ПРИМЕЧАНИЕ: Жизнеспособность дрожжевых клеток увеличивается, если дрожжевые культуры выращиваются до неподвижной фазы перед замораживанием. В более поздних раундах отбора, когда разнообразие библиотек меньше, обычно достаточно использовать 700 мкл культуры дрожжей и смешать ее с 300 мкл SD-CAA с добавлением 50% глицерина. Здесь дрожжевые культуры обычно показывают значения наружного диаметра600 15 в стационарной фазе, но фактическое значение может зависеть от используемого фотометра. Таким образом, стационарная фаза может быть определена путем повторных измерений наружного диаметра600 . Если значение наружного диаметра600 не увеличивается в течение инкубационного периода в 2 ч, достигается стационарная фаза.
- Центрифугируйте соответствующий объем клеток, чтобы покрыть разнообразие в 10 раз при 2000 x g в течение 5 мин, через 4-6 ч после разведения культуры дрожжей (типичные значения OD600 находятся в диапазоне 4-6), и выбросьте надосадочную жидкость.
- Повторно суспендируйте клеточную гранулу в SG-CAA (см. Таблицу 1) до получения наружного диаметра600 из 1 и переложите ее в свежую стеклянную колбу.
Примечание: Ресуспендия в среде, содержащей галактозу, индуцирует экспрессию белка на поверхности с помощью индуцируемого галактозой промотора. - Инкубируйте дрожжевые культуры в течение ночи при температуре 20 °C и встряхивайте со скоростью 180 об/мин, чтобы вызвать поверхностную экспрессию POI. После ночной индукции дрожжевые культуры обычно достигают значений OD600 в диапазоне от 2 до 5; если температура индукции повышается до 37 °C, ожидайте значения наружного диаметра600 от 4 до 10.
ПРИМЕЧАНИЕ: Снижение температуры до 20°C замедляет рост клеток и, как правило, приводит к улучшению уровня отображения. Тем не менее, индукция при 37 °C может быть полезной для увеличения давления отбора по отношению к стабилизированным мутантам35.
3. Первый раунд отбора валиков из библиотек дрожжей (положительный отбор)
ПРИМЕЧАНИЕ: Стандартная процедура выбора валика включает в себя 6 этапов (Таблица 2).
| День | Шаг | |
| 0 | Ночная культура | |
| 1 | Индукция экспрессии белка на поверхности дрожжевых клеток | |
| 2 | Первый выбор валика с 1 положительным выбором | |
| 3 | Удаление гранул, пассирование, индукция экспрессии белка на поверхности дрожжевых клеток и замораживание библиотеки | |
| 4 | Второй выбор бусин с 3 отрицательными и 1 положительным выбором | |
| 5 | Удаление бусин и заморозка библиотеки | |
Таблица 2: Типичный график проведения отбора бусин в библиотеке дрожжей.
- Подготовка бусин
- Подготовьте бусины (см. Таблицу материалов) для первого выбора бусин: Для каждой пробирки ресуспендируйте 10 мкл магнитных шариков биотинового связующего (4 × 105 бусин/мкл) в 990 мкл PBSA (Таблица 1) для промывки, поместите тубу на магнитную решетку на 2 мин с открытой крышкой. Осторожно удалите надосадочную жидкость. Повторите этап промывки с 1 мл PBSA.
ПРИМЕЧАНИЕ: Если необходимо несколько пробирок с шариками с одним и тем же биотинилированным антигеном, их можно приготовить в одной пробирке на этом этапе для получения однородного раствора антиген-гранул. В этом случае количество антигена нужно соответственно увеличить на следующем этапе. - Ресуспендируйте гранулы в общем объеме 1 мл PBSA с 6,7-33 пмоль биотинилированного антигена (здесь: 6,86 мкл hRBP4 с концентрацией 0,125 мг/мл) в микроцентрифужной пробирке объемом 1,5 мл.
ПРИМЕЧАНИЕ: В то время как 6,7 пмоль антигена достаточно, следует использовать 33 пмоль, если антиген не является ограничивающим. Если отбор осуществляется для антигена, связанного с низкомолекулярным лекарственным препаратом или лигандом, как в данном случае, этот препарат/лиганд должен присутствовать во время отбора и должен быть добавлен на этом этапе (здесь: 5 μM A1120). - Инкубировать при температуре 4 °C в течение 2 ч с легким встряхиванием или вращением.
ПРИМЕЧАНИЕ: На этом этапе можно подготовить клетки. - После инкубации и перед добавлением в клетки поместите пробирку на магнитную решетку на 2 мин с открытой крышкой. Удалите надосадочную жидкость и промойте наполненные антигеном шарики 1 мл PBSA, как описано выше. Повторите этап стирки.
ПРИМЕЧАНИЕ: Если выбран антиген, связанный с лекарственным средством или лигандом, промывочный буфер должен содержать этот препарат или лиганд в избытке (здесь: 5 мкМ А1120). - Повторно суспендируйте нагруженные антигеном гранулы в 50 мкл PBSA (в присутствии препарата или лиганда, если применимо).
ПРИМЕЧАНИЕ: Если для нескольких пробирок были приготовлены одни и те же шарики, наполненные антигеном, отрегулируйте объем соответственно (для библиотеки G4 конечный объем здесь составляет 100 мкл, что дает бусины для двух пробирок).
- Подготовьте бусины (см. Таблицу материалов) для первого выбора бусин: Для каждой пробирки ресуспендируйте 10 мкл магнитных шариков биотинового связующего (4 × 105 бусин/мкл) в 990 мкл PBSA (Таблица 1) для промывки, поместите тубу на магнитную решетку на 2 мин с открытой крышкой. Осторожно удалите надосадочную жидкость. Повторите этап промывки с 1 мл PBSA.
- Подготовка дрожжевых клеток
- Измерьте наружный диаметр600 индуцированной культуры дрожжей, как описано выше.
- Гранул достаточно, чтобы покрыть в 10 раз большее разнообразие библиотеки (2000 x g, 10 мин, 4 °C). Для библиотеки G4 используйте 2,5 x 109 ячеек для центрифугирования. Удалите надосадочную жидкость и промойте клетки 10 мл PBSA.
- Снова центрифугируйте (2000 x g, 10 мин, 4 °C) и повторно суспендируйте клетки до концентрации 1,25 x 109 клеток/мл в соответствующем объеме PBSA (здесь: 2 мл) и переложите в микроцентрифужную пробирку.
ПРИМЕЧАНИЕ: Объем PBSA зависит от количества ячеек. - Центрифугируйте (2000 x g, 3 мин, 4 °C) и ресуспендируйте в 950 мкл PBSA.
- Инкубация дрожжевых клеток с антигенными гранулами
- Добавьте 50 мкл наполненных антигеном гранул в 950 мкл клеточной суспензии и инкубируйте в течение 2 ч при 4 °C при встряхивании или вращении.
- После инкубации поместите раствор клеточного антигена-шарика на магнитный штатив с открытой крышкой. Пипеткой нагнетайте жидкость с крышки в пробирку, чтобы подвергнуть всю дрожжевую культуру магнитному отбору. Подождите 2 минуты, прежде чем аккуратно отбросить несвязанные ячейки. Немедленно умыться 1 мл PBSA.
ПРИМЕЧАНИЕ: Если отбор проводится в присутствии лекарственного средства/лиганда - в данном примере А1120 - он должен присутствовать в PBSA. - После этапа промывки PBSA быстро ресуспендируйте клетки в 1 мл SD-CAA, чтобы предотвратить их высыхание.
- Добавьте 200 мл SD-CAA и выполните последовательное разведение, как описано выше. Поместите 100 μL на планшеты SD-CAA и инкубируйте в течение 2-3 дней при 30 °C для определения нового максимального разнообразия. Здесь разнообразие библиотеки G4 после первого выбора бусин составляет 1,25 x 106 ячеек.
ПРИМЕЧАНИЕ: Поскольку исходная библиотека была передискретизирована в 10 раз в этом раунде отбора, разумно предположить, что большинство клонов будут присутствовать в обогащенном пуле несколько раз после отбора. Как следствие, реальное разнообразие будет ниже, и это число следует рассматривать как максимальное значение. Обычно разнообразие уменьшается как минимум в 100 раз в течение этого первого раунда отбора. Оставшуюся культуру (200 мл) инкубировать при 30 °C с встряхиванием (180 об/мин) в течение не менее 16 ч. Протокол можно приостановить после ночной инкубации, поместив клетки при температуре 4 °C.
4. Удаление бусин и культивация перед следующим раундом отбора бусин
- Измерьте внешний диаметр600 , как описано выше. Гранулы должны иметь достаточное количество ячеек, чтобы покрыть разнообразие не менее чем в 10 раз (2000 x g, 3 мин, RT). Выбросьте надосадочную жидкость и ресуспендируйте клетки в 1 мл SD-CAA.
- Переложите в микроцентрифужную пробирку и поместите на магнитную решетку на 2 минуты с открытой крышкой. Перенесите несвязанные ячейки в новую микроцентрифужную пробирку и повторите процесс разделения.
- Разбавьте несвязанные клетки в 100 мл SD-CAA и инкубируйте при 30 °C с встряхиванием при 180 об/мин до тех пор, пока не будет достигнут внешний диаметр600 3-4.
ПРИМЕЧАНИЕ: Если наружный диаметр600 ниже 4 до удаления гранул, клетки могут быть индуцированы сразу после этого без инкубации. - При достижении наружного диаметра600 из 3-4 центрифугируют достаточное количество клеток, чтобы покрыть разнообразие не менее чем в 10 раз (2000 x g, 3 минуты, RT) и повторно суспендируют в SG-CAA до наружного диаметра600 из 1 для индукции экспрессии белка и инкубируют при 20 °C при встряхивании при 180 об/мин в течение ночи.
- Когда оставшаяся культура дрожжей SD-CAA достигнет стационарной фазы роста (OD600 выше 15), гранулируйте достаточное количество клеток, чтобы покрыть разнообразие в 50 раз (2000 x g, 3 мин, RT). Заморозьте клетки центрифугированием (2000 x g, 3 мин) с последующей ресуспензией клеточной гранулы в SD-CAA, содержащей 15% глицерина, и храните при -80 °C.
5. Второй раунд отбора бусин с 3 отрицательными и 1 положительным выбором
- Подготовьте клетки и бусины антигена, как описано выше. Приготовьте раствор из голых бусин (без добавления антигена) для отрицательных выделений.
ПРИМЕЧАНИЕ: Поскольку выполняется 3 отрицательных выбора, бусины могут быть подготовлены вместе для всех трех раундов. - После промывки ресуспендируйте гранулы антигена в 50 мкл PBSA и ресуспендируйте голые бусины в 150 мкл PBSA (если они были подготовлены для всех трех отрицательных отборов).
- Для первого отрицательного отбора добавьте 50 мкл промытых голых шариков к 950 мкл промытых клеток в PBSA и инкубируйте в течение 1,5 ч при 4 °C.
- После инкубации поместите пробирки, содержащие суспензии голых шариковых клеток, на магнитный штатив с открытой крышкой. Налейте пипеткой в пробирку любую жидкость из крышки и подождите 2 минуты. Переложите несвязанные клетки в свежую микроцентрифужную пробирку и добавьте 50 μл промытых голых шариков.
- Повторите процедуру еще для двух раундов отрицательного отбора. Тем временем подготовьте наполненные антигеном бусины для положительного отбора.
- После 3 раундов отрицательного отбора добавьте в клетки 50 мкл раствора, наполненного антигенами, и инкубируйте в течение 2 ч при 4 °C.
- Поместите клетки, содержащие наполненные антигеном шарики, на магнитный штатив с открытой крышкой и заведите в пробирку всю жидкость, находящуюся в крышке. Подождите 2 минуты, прежде чем отбросить несвязанные ячейки.
- Выполните все оставшиеся шаги, как описано для первого выбора антигенных шариков. Единственное отличие от первого положительного отбора (шаг 3.3.2) заключается в количестве этапов промывки: вместо однократной промывки PBSA, дважды промойте суспензию шариковых клеток PBSA для повышения строгости отбора.
ПРИМЕЧАНИЕ: Обычно разнообразие снова уменьшается в 10-100 раз. Максимальное разнообразие библиотеки G4 после этого второго выбора бусин составило 3,45 x 105 ячеек. Как отмечалось выше, ожидается, что фактическое разнообразие будет ниже из-за чрезмерной выборки библиотеки.
6. Отбор библиотек с помощью проточной цитометрической сортировки
- После ночной индукции поверхностной экспрессии в SG-CAA измерьте внешний диаметр600 , как описано выше. Гранулы содержат достаточное количество ячеек, чтобы покрыть в 10 раз большее разнообразие (2000 x g, 5 мин, RT). Выбросьте надосадочную жидкость.
- Повторно суспендируйте гранулу в PBSA и перенесите ее в микроцентрифужные пробирки. Используйте 3 x 107 ячеек для окрашивания для каждой трубки. Подготовьте столько пробирок, сколько требуется, в зависимости от сорта. Подготовьте одну контрольную пробирку для окрашивания без антигена.
ПРИМЕЧАНИЕ: Например, если необходимо отсортировать 30 x 106 клеток, гранулируйте 300 x 106 клеток, чтобы получить видимую клеточную гранулу и предотвратить потерю клеток на последующих этапах. Повторно суспендируйте в 5 мл PBSA и перенесите от 3 до 4 аликвот по 500 мкл каждая в микроцентрифужные пробирки. Подготовьте примерно в 3 раза больше клеток, чем должно быть отсортировано, потому что часть клеток будет потеряна в процессе окрашивания. Если отбор проводится в присутствии препарата/лиганда, готовят дополнительный контроль только с антигеном, но без препарата/лиганда. - Снова центрифугируйте (2000 x g, 5 мин, RT), повторно суспендируйте гранулу в 200 мкл PBSA, содержащего антиген (hRBP4 с 5 мкМ A1120) и инкубируйте в течение 1 ч при 4 °C.
ПРИМЕЧАНИЕ: Все шаги, начиная с этого момента, следует выполнять на льду и с холодными реагентами. Чтобы предотвратить истощение антител или антигенов, все реагенты должны быть добавлены в стехиометрическом избытке по сравнению с общим количеством белков, отображаемых дрожжами в пробирке. В противном случае может возникнуть риск ограничения связывания дрожжевых вариантов из-за ограниченной доступности антигенов и/или антител. Обычно для расчета используется 5 x 104 молекулы на дрожжевую клетку, и рекомендуется добавить по крайней мере 10-кратный стохиометрический избыток антигена и антител. Если антиген сильно ограничен, то избыток можно уменьшить до 5 раз. Более подробное объяснение, включая примеры расчетов во избежание истощения лиганда, приведено в предыдущем протоколе9. - После инкубации центрифугируйте клетки (2000 x g, 5 мин, 4 °C) и промойте 1 мл PBSA (если препарат/лиганд присутствует во время отбора - в данном случае как A1120 - он должен присутствовать в промывном буфере).
- Ресуспендируют клетки в 100 мкл холодного PBSA, содержащего антитела, для окрашивания дисплея и обнаружения связанного антигена и инкубируют в течение 30 мин при 4 °С.
- Используйте антитела против c-myc (окончательное разведение 1:100) для обнаружения отображаемых POI по всей длине. Используйте антитела против ГК (окончательное разведение 1:50) для определения общего количества отображаемых белков. Используйте антитела Penta-His (окончательное разведение 1:20) или стрептавидин (окончательное разведение 1:200) для отбора дрожжевых клеток, связывающихся с растворимым антигеном, содержащим либо 6-кратную метку HIS, либо биотин, соответственно.
- Чередуйте реагенты для обнаружения между раундами сортировки, чтобы предотвратить обогащение вариантов белка, которые связываются с вторичным реагентом.
ПРИМЕЧАНИЕ: Можно использовать различные комбинации флуорофоров, но Alexa Fluor 488 и Alexa Fluor 647 являются хорошими вариантами, поскольку они не требуют компенсации в проточной цитометрии. Чтобы предотвратить истощение антител, его следует добавлять в стехиометрическом избытке по сравнению с общим количеством дрожжевых белков в пробирке. Обычно расчеты основаны на соотношении 5 x 104 молекул на дрожжевую клетку. Более подробное объяснение приведено в предыдущих протоколах9.
- После инкубации центрифугируют ячейки (2000 x g, 5 мин, 4 °C) и добавляют 1 мл PBSA (содержащего препарат/лиганд, для выбранной hRBP4 библиотеки G4 5 мкМ A1120) в гранулу и снова центрифугируют. Удалите большую часть надосадочной жидкости и оставьте только 20-30 μл, чтобы предотвратить высыхание гранул.
- Суспендировать гранулу в холодном PBSA можно только непосредственно перед сортировкой и отсортировать дрожжевые клетки, демонстрирующие молекулы Fn3 с желаемыми свойствами (т.е. связывающиеся с hRBP4 в присутствии A1120).
ПРИМЕЧАНИЕ: Стратегия стробирования изображена на рисунке 2А. - Отсортируйте клетки непосредственно в среду SD-CAA, добавьте больше среды SD-CAA после сортировки и инкубируйте при 30 °C с встряхиванием при 180 об/мин.
ПРИМЕЧАНИЕ: В случае, если объем значительно увеличивается после сортировки, следует провести этап центрифугирования (2000 x g, 5 мин, RT) для удаления избытка жидкости оболочки, и клетки ресуспендируют в свежей среде SD-CAA. Запишите количество отсортированных клеток в новую пробирку, так как это становится новым максимальным разнообразием для следующего раунда сортировки. Как отмечалось выше, фактическое разнообразие библиотеки будет ниже из-за чрезмерной выборки библиотеки. - Повторите проточную цитометрическую сортировку в течение нескольких раундов, чтобы увидеть обогащение связывающих белковых вариантов в библиотеке дрожжей (рис. 2B).
ПРИМЕЧАНИЕ: Может быть полезно сравнить библиотеки бок о бок в аналитическом проточно-цитометрическом эксперименте. В более поздних раундах отбора снижение концентрации антигена может быть полезным для различения связывающих веществ с более низким и высоким сродством и сортировки специально для вариантов белка, которые демонстрируют более высокий сигнал связывания с мишенью при низких концентрациях (рис. 2C). В более поздних раундах отбора также есть возможность сортировки по повышенной термостабильности. С этой целью проводили случайный мутагенез библиотеки методом эпПЦР, как описано в разделе 7. Подготавливайте клетки аналогично другим раундам проточной цитометрической сортировки путем индукции, определения OD600 и промывки клеток. Поместите дрожжевые клетки в PBSA на лед на 10 минут, а затем 10 минут инкубируйте в термоамплификаторе для теплового шока при повышенной температуре. После этого снова положите их на лед на 10 минут и следуйте протоколу окрашивания. Стабилизированные варианты устойчивы к термической денатурации и, следовательно, сохраняют связывание после тепловой инкубации. Различные температуры могут быть испытаны на тепловой шок, в зависимости от начальной термостабильности белка дикого типа. Как правило, подходит температура в диапазоне от 40 до 80 °C. Однако при температуре выше 48 °C жизнеспособность дрожжевых клеток резко снижается. Следовательно, необходимо извлечь плазмиды после сортировки библиотеки дрожжей, подвергшихся тепловому удару, как описано в шаге 7.1. В этом случае клетки EBY100 должны быть свежепреобразованы с обогащенными вариантами, прежде чем перейти к следующему раунду сортировки36. Более подробный протокол повышения термостабильности можно найти в другом месте37. Если денатурация выводимого белка обратима, то этот метод не сработает. В качестве альтернативы индуцируйте экспрессию белка при 37 °C вместо 20 °C за день до сортировки и отсортируйте варианты белка, которые все еще демонстрируют эффективное полноразмерное отображение.
7. Аффинное созревание с помощью эпПЦР для введения случайных мутаций
ПРИМЕЧАНИЕ: Аффинное созревание с помощью эпПЦР может быть выполнено либо до первого раунда проточной цитометрической сортировки, либо между раундами проточной цитометрической сортировки. Для отбора библиотеки G4 с hRBP4 в присутствии A1120 аффинное созревание проводили перед первым раундом проточной цитометрической сортировки. Это также зависит от размера библиотеки после выбора шариков и сигнала связывания, который можно обнаружить с помощью проточной цитометрии. В частности, в тех случаях, когда аффинности после отбора шариков недостаточны для получения сигнала в экспериментах с проточной цитометрией (поскольку антиген быстро диссоциирует на этапах промывки), эпПЦР может генерировать улучшенные варианты, которые впоследствии могут быть обнаружены и отобраны с помощью проточной цитометрии.
- Провести экстракцию ДНК дрожжей miniprep из обогащенной библиотеки дрожжей с использованием коммерческого набора Plasmid miniprep II (см. Таблицу материалов) со следующими модификациями:
- Измерьте плотность клеток ночной культуры, разбавьте до наружного диаметра600 0,2 утром и инкубируйте при 30 °C с встряхиванием.
- Когда наружный диаметр600 достигнет 0,6, соберите 3 мл культуры, центрифугируя клетки при 600 x g в течение 2 минут.
- Добавьте в гранулу 200 μл раствора 1. Добавьте 6 μL Zymolyase. Повторно суспендируйте гранулу путем щелканья или легкого вихревания и инкубируйте при 37 °C в течение 3 часов.
- Добавьте 200 μL раствора 2 и аккуратно перемешайте. Добавьте 400 μL раствора 3 и аккуратно перемешайте.
- Центрифуга на максимальной скорости в течение 3 мин.
- Перенесите надосадочную жидкость в колонку со спин-I, центрифугируйте на максимальной скорости в течение 30 с и выбросьте проточный.
- Промойте 550 μL промывочного буфера, содержащего этанол, и центрифугируйте на максимальной скорости в течение 2 минут.
- Переложите колонку в чистую микроцентрифужную пробирку и центрифугуйте на максимальной скорости в течение 2 минут, чтобы удалить остатки промывочного буфера.
- Перенесите колонку в новую микроцентрифужную пробирку, добавьте 10 мкл воды, не содержащей нуклеаз, и вращайте в течение 1 минуты на максимальной скорости, чтобы элюировать ДНК.
- Проведите первичную ПЦР для амплификации шаблона для эпПЦР.
- Амплифицируйте ДНК методом ПЦР с использованием праймеров, отжигая на внешней стороне вставки на поверхности дрожжевого вектора отображения.
ПРИМЕЧАНИЕ: Поскольку большая часть геномной ДНК дрожжей элюируется вместе с интересующей плазмидой во время мини-подготовки дрожжей, этот этап рекомендуется для получения чистого фрагмента ДНК, который будет использоваться в качестве матрицы для эпПЦР. - Для pCTCON2 используйте праймеры fwd 5'- GGCTGGTGGAGGCGGTAGCGGAGGCGGAG
GGTCGGCTAGC и rev 5'- CTATTACAAGTCC
TCTTCAGAAATAAGCTTTTGTTCGGATCC с компонентами реакции, описанными в таблице 3 , и условиями цикла, изображенными в таблице 4. - Очистите ПЦР с помощью набора для очистки ПЦР и ДНК. Выведите в 20 мкл безнуклеазного H2O.
- Амплифицируйте ДНК методом ПЦР с использованием праймеров, отжигая на внешней стороне вставки на поверхности дрожжевого вектора отображения.
- Проведите эпПЦР для введения случайных мутаций.
- Проведите эпПЦР с аналогами нуклеотидов с указанными ингредиентами (Таблица 5) и условиями (Таблица 6).
- Запустите весь продукт epPCR на препаративном 1% агарозном геле при напряжении 120 В в течение 45 минут. Добавьте маркер и оставьте одну полосу между продуктом epPCR и лестницей. Вырежьте нужную полосу, соответствующую предполагаемому размеру вставки, очистите ее с помощью набора для экстракции гелем и измерьте концентрацию ДНК.
- Амплифицируйте рандомизированную ДНК методом ПЦР.
- Амплифицируйте 50 нг ДНК с помощью Q5-полимеразы. Установите 2 x 100 мкл ПЦР-реакции для каждой электропорации (Таблица 7) с указанными условиями (Таблица 8). Используйте те же праймеры, которые использовались для первой ПЦР.
Примечание: Для электропорации в дрожжи требуется большое количество ДНК. - Проанализируйте 5 мкл продукта ПЦР на 1% агарозном геле, прогоните при напряжении 120 В в течение 45 минут.
- Амплифицируйте 50 нг ДНК с помощью Q5-полимеразы. Установите 2 x 100 мкл ПЦР-реакции для каждой электропорации (Таблица 7) с указанными условиями (Таблица 8). Используйте те же праймеры, которые использовались для первой ПЦР.
- Очистите амплифицированную ДНК с помощью осаждения этанола.
- Бассейн 2 x 100 μл вместе. Добавьте 20 μЛ (10% от объема) стерильного фильтрованного 3 М ацетата натрия, pH 5,2.
ПРИМЕЧАНИЕ: Этот буфер может быть приготовлен заранее и храниться в течение нескольких месяцев в RT. - Добавьте 440 μл 100% этанола (или не менее 2 x объема) и инкубируйте при RT в течение 2 минут.
- Центрифугируйте при 20 000 x g в течение 5 минут при RT. Удалите надосадочную жидкость, добавьте 500 μл 70% этанола и быстро перемешайте.
- Центрифугируйте при 20 000 x g в течение 5 минут при RT. Удалите надосадочную жидкость, добавьте 500 μл 100% этанола и быстро перемешайте.
- Центрифугируйте при 20 000 x g в течение 5 минут при RT. Удалите надосадочную жидкость и дайте грануле ДНК высохнуть под ламинарным проточным колпаком, пока весь этанол не испарится.
ПРИМЕЧАНИЕ: Используйте нагревательный блок при температуре 48 °C, чтобы ускорить этот процесс. Гранула ДНК может быть не видна все время. - Растворите высушенную гранулу ДНК в 10 мкл безнуклеазного H2O.
ПРИМЕЧАНИЕ: ДНК должна быть растворена в как можно меньшем объеме. Не измеряйте концентрацию ДНК.
- Бассейн 2 x 100 μл вместе. Добавьте 20 μЛ (10% от объема) стерильного фильтрованного 3 М ацетата натрия, pH 5,2.
| Объем [μл] | Конечная концентрация | |
| 5x усилитель Q5 | 10 | 1x |
| 5x буфер Q5 | 10 | 1x |
| Грунтовка fwd 10 μM | 2.5 | 0,5 мкМ |
| Обороты грунтовки 10 μМ | 2.5 | 0,5 мкМ |
| dNTP 10 мМ | 1 | 200 мкМ |
| Полимераза Q5 | 0.5 | 20 Ед/мл |
| ДНК из дрожжевого miniprep | 10 | |
| Безнуклеаза H2O | 13.5 |
Таблица 3: Условия проведения ПЦР1-го этапа для амплификации генов POI из изолированного минипрепа дрожжей.
| Шаг | Температура | Время |
| Первичная денатурация | 98 °C | 30 с |
| 25 циклов | 98 °C | 10 с |
| 72 °C | 30 с | |
| 72 °C | 30 с | |
| Окончательное продление | 72 °C | 2 мин |
| Держать | 4 °C |
Таблица 4: Условия циклирования для1-го этапа ПЦР для амплификации генов POI из изолированного минипрепа дрожжей.
| Объем [μл] | Конечная концентрация | |
| Безнуклеаза H2O | до 50 | |
| 10x буфер Thermopol | 5 | 1x |
| Primer_fwd (10 мкМ) | 2.5 | 0,5 мкМ |
| Primer_rev (10 мкМ) | 2.5 | 0,5 мкМ |
| dNTP (10 мМ) | 1 | 200 мкМ |
| 8-оксо-дГТФ (100 мкМ) | 1 | 2 мкМ |
| dPTP (100 мкМ) | 1 | 2 мкМ |
| Продукт ПЦР из 1-й ПЦР | ХХ | 50 нг |
| Taq ДНК-полимераза | 0.5 | 0,05 ед/μл |
Таблица 5: Условия проведения эпПЦР, которая проводится после амплификации ДНК POI с помощью ПЦР1-го этапа.
| Шаг | Температура | Время |
| Первичная денатурация | 94 °C | 30 с |
| 15 циклов | 94 °C | 45 с |
| 60 °C | 30 с | |
| 72 °C | 1 мин | |
| Окончательное продление | 72 °C | 10 мин |
| Держать | 4 °C |
Таблица 6: Условия цикличности для эпПЦР.
| Объем [μл] | Конечная концентрация | |
| 5x усилитель Q5 | 20 | 1x |
| 5x буфер Q5 | 20 | 1x |
| Грунтовка fwd 10 μM | 5 | 0,5 мкМ |
| Обороты грунтовки 10 μМ | 5 | 0,5 мкМ |
| dNTP 10 мМ | 1 | 200 мкМ |
| Полимераза Q5 | 1 | 20 Ед/мл |
| 50 нг ДНК | ХХ | |
| ддН20 | до 100 |
Таблица 7: Условия проведения2-го этапа ПЦР для амплификации продукта эпПЦР перед электропорацией клеток EBY100.
| Шаг | Температура | Время |
| Первичная денатурация | 98 °C | 30 с |
| 25 циклов | 98 °C | 10 с |
| 72 °C | 30 с | |
| 72 °C | 30 с | |
| Окончательное продление | 72 °C | 2 мин |
| Держать | 4 °C |
Таблица 8: Условия цикличности для2-го этапа ПЦР для амплификации продукта epPCR.
8. Линеаризация дрожжевого вектора для электропорации
- Линеаризовать вектор поверхностного отображения дрожжей pCTCON2 (плазмида Addgene #41843).
- Выполните крупномасштабный дайджест и используйте этот переваренный линеаризованный вектор для всех последующих электропораций.
- Расщепите 2 x 200 мкг плазмидной ДНК pCTCON2 с Sal I в течение 24 ч при 37 °C для начальной линеаризации (Таблица 9).
- Варить с NheI и BamHI в течение 24 часов при 37 °C для удаления вкладыша (Таблица 10).
- Наконец, добавьте по 5 мкл каждого фермента в пробирку (Sal I, NheI, BamHI) и снова инкубируйте в течение 24 ч при 37 °C.
- Запустите переваренный вектор на препаративный агарозный гель. Вырежьте ленты и очистите с помощью набора для экстракции геля. Разбавьте вектор в 20 μл, измерьте концентрацию ДНК и отрегулируйте ее примерно до 2 μг/μл.
ПРИМЕЧАНИЕ: Расщепление плазмиды в течение длительного времени обеспечивает достаточное расщепление вставки и приводит к уменьшению фона от вектора после электропорации.
| ДНК | 200 μг |
| 10x CutSmartBuffer | 50 μл |
| Саль I-HF (NEB) | 30 μЛ (60 U) |
| Н2О | До 500 μл |
Таблица 9: Условия для первого этапа крупномасштабного расщепления вектора отображения поверхности дрожжей pCTCON2.
| pCTCON2 (Сал я переварил) | 500 мкл |
| 10x CutSmartBuffer | 37,5 мкл |
| NHEI-HF (NEB) | 15 μЛ (30 U) |
| БамHI-КВ (НЭБ) | 15 μЛ (30 U) |
| Н2О | до 875 μл |
Таблица 10: Условия для второго этапа крупномасштабного расщепления вектора отображения поверхности дрожжей pCTCON2.
9. Электропорация EBY100 рандомизированной ДНК и линеаризованным вектором
- Подготовьте дрожжевые клетки EBY100 (ATCC, см. Таблицу материалов), вычеркнув их из глицеринового запаса за 3 дня до электропорации. Поскольку нетрансформированные клетки EBY100 не содержат вектора отображения поверхности дрожжей, используйте полнофункциональные YPD-планшеты (Таблица 1). Инкубировать при 30 °C.
- За день до электропорации инокулируйте одну колонию EBY100 в 30 мл свежеприготовленной среды YPD (Таблица 1). Выдерживать в течение ночи при температуре 30 °C и встряхивать при 180 об/мин.
ПРИМЕЧАНИЕ: Среда YPD не должна быть старше 1 месяца. - На следующее утро разбавьте ночную культуру EBY100 до наружного диаметра600 0,2 в 100 мл среды YPD и инкубируйте при 30 °C при встряхивании при 180 об/мин.
ПРИМЕЧАНИЕ: Объем культуры 50 мл достаточен для 2 электропорации. Как правило, 100 мл подготавливают для того, чтобы иметь достаточное количество клеток для контроля (только клетки и клетки плюс вектор). - При достижении наружного диаметра600 1,3-1,5 гранулируйте клетки в пробирках объемом 50 мл при температуре 2000 x g в течение 3 мин и удалите надосадочную жидкость.
- Ресуспендируйте гранулу в 25 мл 100 мл стерильного фильтрованного ацетата лития (см. Таблицу материалов). Добавьте свежеприготовленный стерильный фильтрованный DTT (1 М) (см. Таблицу материалов) до конечной концентрации 10 мМ.
ПРИМЕЧАНИЕ: Объем ацетата лития должен составлять половину исходного объема YPD. При необходимости увеличьте или увеличьте масштаб. Ацетат лития (100 мМ) можно приготовить заранее и хранить в течение нескольких месяцев в RT, но запас 1 М DTT следует готовить прямо перед использованием и хранить на льду. - Инкубируйте клетки при температуре 30 °C при встряхивании в течение 10 минут.
- Центрифугируйте при 2000 x g в течение 3 минут, выбросьте надосадочную жидкость и поместите клетки на лед.
ПРИМЕЧАНИЕ: Все последующие шаги следует выполнять на льду и с охлажденными реагентами, кюветами и микроцентрифужными пробирками. - Ресуспендировать гранулу в 25 мл холодного стерильного H2O. Центрифуга при 2000 х г в течение 3 мин.
ПРИМЕЧАНИЕ: Используемый объем H2O должен составлять половину от исходного объема YPD. - Ресуспендируйте в 350 мкл стерильного H2O.
ПРИМЕЧАНИЕ: Общий объем после ресуспензии должен составлять 500 μл. При необходимости добавьте еще H2O. - Подготовьте библиотеку ДНК параллельно: поместите микроцентрифужные пробирки, содержащие очищенные от этанола вставки, на лед и добавьте 4 мкг линеаризованного вектора pCTCON2.
- Добавьте 250 μл клеток в ДНК и аккуратно перемешайте, срезая. Переложите в предварительно охлажденную 2 мм электропорационную кювету и держите на льду до начала электропорации.
ПРИМЕЧАНИЕ: Добавьте контрольные элементы (клетки только без ДНК и клетки с линеаризованным вектором только без вставки) для определения успешности электропорации. - Электропорация с использованием электропорационных кювет диаметром 2 мм (см. Таблицу материалов) при следующих условиях: протокол прямоугольной волны, одиночный импульс, 500 В, длительность импульса 15 мс.
ПРИМЕЧАНИЕ: Типичные отклонения составляют 3-9%. Можно использовать и другие электропораторы, но указанные условия оптимизированы для инструмента, используемого в данном исследовании. - Спасите клетки с помощью 1 мл предварительно подогретого YPD, переложите в предварительно нагретую микроцентрифужную пробирку и инкубируйте без встряхивания в течение 1 ч при 30 °C.
ПРИМЕЧАНИЕ: Поместите нагревательный блок рядом с электропоратором, чтобы обеспечить надлежащую температуру восстановительных трубок. - Планшет 100 мкл 10-3, 10-4, 10-5 и 10-6 разбавляет клетки на планшетах SD-CAA (см. таблицу 1) и позволяет выращивать трансформанты при 30 °C.
Примечание: Теоретическое разнообразие библиотек получается путем обратного вычисления количества трансформантов до исходного объема культуры, в данном случае 1 мл. Как правило, этот протокол трансформации дает примерно 107-10 8 трансформированных клеток. - Гранулируйте оставшиеся клетки при 2000 x g в течение 3 мин, повторно суспендируйте в 200 мл SD-CAA и инкубируйте при 30 °C с встряхиванием при 180 об/мин в течение ночи. Трансформированные клетки, содержащие рандомизированную библиотеку epPCR, могут быть либо пропущены в SD-CAA и индуцированы для поверхностной экспрессии в SG-CAA на следующий день, либо сохранены при температуре 4 °C до дальнейшего использования. Заморозьте запасы глицерина библиотеки после эпПЦР после определения разнообразия. Поскольку на следующий день часть клеток все еще будет представлять собой нетрансформированные клетки, которые были перенесены в процедуре трансформации без получения плазмиды, рекомендуется для компенсации этого эффекта на первом этапе пассирования пройти не менее 20-кратного разнообразия библиотеки.
10. Секвенирование библиотек дрожжей после нескольких раундов отбора
- Извлеките ДНК дрожжей, выполнив минипреп дрожжей, как описано выше.
- Электропорировать коммерческие электрокомпетентные клетки E. coli 10-beta (см. Таблицу материалов) с выделенной ДНК.
- Поместите кюветы для электропорации (1 мм, см. Таблицу материалов) и микроцентрифужные пробирки на лед. Предварительно нагрейте микроцентрифужные пробирки с 950 мкл среды роста при 37 °C для восстановления клеток после электропорации.
- Разморозьте ячейки на льду и перемешайте, осторожно переворачивая трубку. Перенесите 50 μL ячеек в охлаждаемую микроцентрифужную пробирку. Добавьте 1 мкл ДНК дрожжевого минипрепа.
- Осторожно переложите смесь клеток и ДНК в охлажденную электропорационную кювету.
ПРИМЕЧАНИЕ: Избегайте образования пузырьков и следите за тем, чтобы ячейки оседали на дне кюветы. - Электропорация с помощью устройства электропорации с использованием следующих условий: 2,0 кВ; 200 Ω; 25 мкФ.
ПРИМЕЧАНИЕ: Типичные временные константы составляют 4,8-5,1 мс. Можно использовать и другие электропораторы, но указанные условия оптимизированы для инструмента, используемого в данном исследовании. - Немедленно добавьте в кюветы 950 мкл предварительно подогретой при температуре 37 °C среды (см. Таблицу материалов), аккуратно перемешайте и переложите обратно в теплые микроцентрифужные пробирки.
ПРИМЕЧАНИЕ: Поместите нагревательный блок рядом с электропоратором, чтобы обеспечить надлежащую температуру восстановительных трубок. - Инкубировать в течение 40 минут при температуре 37 °C и 300 об/мин. Планшет 10 μл, 100 μл и остальные клетки (после центрифугирования) на предварительно нагретый селективный планшет LB (см. Таблицу 1). Инкубируйте планшеты в течение ночи при температуре 37 °C.
ПРИМЕЧАНИЕ: Для вектора pCTCON2 для выбора антибиотика используется ампициллин. - Приготовьте копировальную пластину с LB агаром и необходимым антибиотиком (здесь используется ампициллин): пипеткой 200 мкл LB агара с концентрацией ампициллина 100 мкг/мл в каждой лунке 96-луночного планшета и дайте ему застыть.
- Приготовьте планшет для секвенирования на 96 лунок с 50 мкл LB и 100 мкг/мл ампициллина на лунку.
- Выберите одну колонию из пластин и сначала привите копировальную пластину и ту же лунку секвенционной пластины.
ПРИМЕЧАНИЕ: Копировальная пластина гарантирует, что желаемые плазмиды могут быть легко получены путем инокуляции жидкой культуры и выполнения мини-подготовки. - Инкубируйте копировальную пластину в течение ночи при температуре 37 °C, а затем храните при 4 °C.
- Инкубируйте секвенирующую пластину в течение 3-4 ч при 37 °C при встряхивании (180 об/мин), накрытой пластиковой или алюминиевой фольгой.
- Запечатайте планшет для секвенирования и отправьте его на секвенирование вместе с 500 μL 10 μM праймера для секвенирования pCTCON2 (5'-CGTTTGTCAGTCAGTAATTGCGGTTCTC).
- Для анализа последовательностей следует транслировать полученные последовательности ДНК в аминокислотные последовательности с помощью подходящей программы выбора, например, EMBOSS Transeq38. Затем используйте метод множественного выравнивания последовательностей (MUSCLE)39 или аналогичный инструмент для выравнивания вариантных последовательностей друг относительно друга и с родительским белком дикого типа.
ПРИМЕЧАНИЕ: Выполняя секвенирование библиотек различных раундов отбора (например, после более раннего раунда отбора, а также после дополнительных раундов отбора с более низкими концентрациями антигена для обогащения высокоаффинных вариантов) можно идентифицировать различные варианты. Это в значительной степени зависит от строгости стробов, применяемых во время сортировки, и, следовательно, от разнообразия библиотек.
Результаты
Библиотека G4 была выбрана против антигена hRBP4, связанного с низкомолекулярным препаратом A1120. Окрашивание библиотек для проточной цитометрической сортировки проводили, как описано в методе 6, а применяемая стратегия стробирования показана на рисунке 2А. Первоначальный вентиль включал все клетки на основе морфологии клеток, а второй вентиль (гистограмма FSC-ширины) демонстрировал строгую стратегию стробирования, которая применялась для выбора отдельных клеток и удаления клеточных агрегатов. Третий и последний гейт показал отображение вариантов белка (ось x) в сравнении с связыванием антигена (ось y). Дрожжевые клетки, показывающие как сигналы отображения, так и сигналы связывания, были отсортированы. Важно отметить, что сортировочный гейт был настроен строгим образом, чтобы обогатить связывающие домены высоким сигналом связывания и, следовательно, высоким сродством. Этот строгий отбор позволил получить обогащение дрожжевыми клетками, которые специфически связываются с целевым антигеном на протяжении всей селекционной кампании (рис. 2B). В последующих циклах проточной цитометрической сортировки концентрация антигена была снижена в 10 раз (со 100 нМ до 10 нМ). Таким образом, общий сигнал связывания был снижен, и только связующие вещества с высоким сродством по-прежнему обнаруживались и сортировались (рис. 2C).
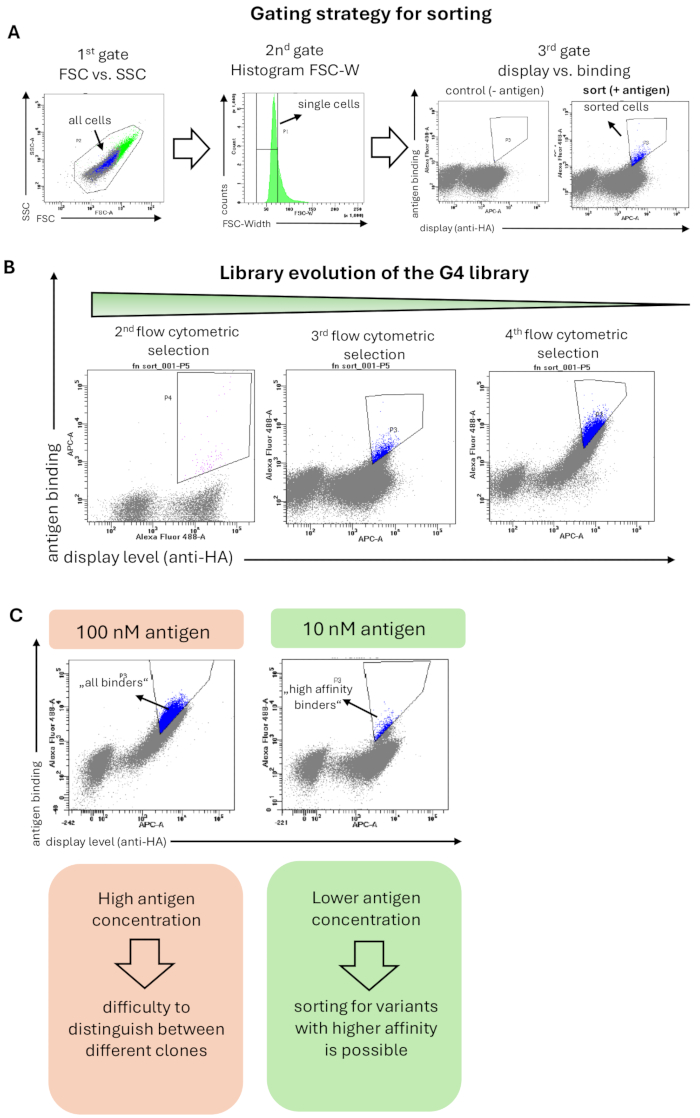
Рисунок 2: Репрезентативные результаты выбора на поверхности дрожжей библиотеки G4 на основе Fn3 для связывания с антигеном (hRBP4 в присутствии A1120). (A) Общая стратегия гейтирования для сортировки дрожжевых библиотек. Первый вентиль (FSC против SSC) заключается в выборе всех дрожжевых клеток и исключении событий рассеяния; второй вентиль (гистограмма FSC-W) направлен на удаление клеточных агрегатов и выбор только одиночных дрожжевых клеток. Третий вентиль отображает уровень отображения поверхности (обнаружение HA- или c-myc-метки) в зависимости от связывания с антигеном (в данном случае hRBP4 в присутствии 5 мкМ A1120, детектируемого антителом против His). Библиотека была дополнительно окрашена только вторичными антителами (без антигена), где связывание с антигеном не ожидается. Отсортированные ячейки подсвечиваются синим цветом. (B) Эволюция библиотеки G4 в течение 3 раундов проточной цитометрической сортировки. Обогащение популяции связывания можно наблюдать при каждом раунде отбора. (C) Использование более низких концентраций антигена позволяет выбирать варианты белка с более высоким сродством к целевому антигену. При снижении концентрации антигена (в данном случае hRBP4) в 10 раз появляются различные диагонали, свидетельствующие о наличии клонов с более высоким (отсортированные клетки, синий) или меньшим сродством. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Обсуждение
Отображение поверхности дрожжей превратилось в один из ключевых методов, используемых в белковой инженерии. Хотя он обычно используется для создания аффинности 1,18,40,41, экспрессии/стабильности 24,27,42,43 и активности28,44, другие применения, такие как картирование эпитопов45,46 или характеристика отдельных мутантов на поверхности дрожжевых клеток9 также возможны. В этом протоколе мы описываем основные этапы для начала кампании по выбору поверхностного отображения дрожжей, включая отбор с помощью магнитных гранул и проточную цитометрическую сортировку, а также диверсификацию библиотеки дрожжей с помощью эпПЦР для аффинного созревания.
Одним из основных требований к выбору обычных дрожжевых поверхностей является наличие растворимого белка достаточного качества. Начало с хорошо свернутого белка-мишени с высокой чистотой и определенным состоянием олигомеризации (т.е. мономерный белок должен присутствовать только в виде мономера) обеспечивает наивысший процент успеха при выборе варианта белка, связывающегося с антигеном-мишенью с высоким сродством. Альтернативой трудноэкспрессируемым белкам-мишеням является клеточный отбор, который представляет собой разумную стратегию для обхода этого ограничения. Тем не менее, отображение поверхности дрожжей имеет множество преимуществ, таких как возможность характеристики полученных вариантов белка непосредственно на поверхности дрожжей без необходимости выполнения трудоемкого и трудоемкого клонирования, экспрессия в растворимом формате и очистка белка. Как аффинность, так и стабильность вариантов могут быть проанализированы непосредственно на поверхности дрожжей9.
В этом протоколе мы показываем, как библиотека вариантов белка G4, а точнее10-го домена типа III человеческого фибронектина, была выбрана для связывания с антигеном hRBP4 в присутствии малой молекулы A1120. Комбинация отбора гранул и проточной цитометрической сортировки привела к обогащению вариантов, которые показали повышенное связывание с целевым антигеном на протяжении раундов отбора (рис. 2B). Мы показали, что использование более низких концентраций антигена позволяет выбирать варианты высокоаффинных белков (рис. 2C). Как правило, аффинности, которые могут быть достигнуты с помощью выбора дрожжевого дисплея, находятся в наномолярном или даже пикомолярном диапазоне18. Конечное сродство зависит от целевого антигена, количества раундов отбора и созревания аффинности, используемого связывающего каркаса и применяемой стратегии гейтирования. Характеристика отдельных вариантов белка не рассматривается в этом протоколе, но подробно объясняется в нашей предыдущей работе9. Хотя дрожжевой дисплей первоначально использовался для конструирования фрагментов антител, таких как scFvs 1,40, этот метод широко использовался и для белков, не основанных на антителах.
Подводя итог, можно сказать, что отображение поверхности дрожжей является мощным инструментом белковой инженерии, который позволяет создавать варианты белка с новыми или улучшенными свойствами, такими как связывание практически с любым белком-мишенью и/или повышенная стабильность.
Раскрытие информации
M.W.T. получает финансирование от компании Miltenyi Biotec. Все авторы являются изобретателями патентных заявок на технологии и сконструированные белки, которые были разработаны с использованием дисплея поверхности дрожжей.
Благодарности
Эта работа была поддержана Австрийским научным фондом (FWF Project W1224 - Докторская программа по биомолекулярной технологии белков - BioToP и FWF Project ESP 465-B), Федеральным министерством цифровых и экономических дел Австрии, Национальным фондом исследований, технологий и развития Австрии и Исследовательской ассоциацией Кристиана Доплера (Christian Doppler Laboratory for Next Generation CAR T-клеток), а также частными пожертвованиями в Детский научно-исследовательский институт рака Святой Анны (Вена, Австрия). Э.С. является стипендиатом DOC Австрийской академии наук в Научно-исследовательском институте детского рака Святой Анны. Фигуры создавались с BioRender.com.
Материалы
| Name | Company | Catalog Number | Comments |
| 10-beta electrocompetent E. coli | NEB | C3020K | |
| Agar-Agar, Kobe I | Carl Roth | 5210.5 | |
| Ampicillin sodium salt | Carl Roth | K029.2 | |
| Anti-c-myc antibody, clone 9E10, AF488 | Invitrogen | MA1-980-A488 (Thermo Fisher) | |
| Anti-c-myc antibody, clone 9E10, AF647 | Invitrogen | MA1-980-A647 (Thermo Fisher) | |
| Anti-HA antibody, clone 16B12, AF488 | BioLegend | 901509 (Biozym) | |
| Anti-HA antibody, clone 16B12, AF647 | BioLegend | 682404 (Biozym) | |
| BamHI-HF | NEB | R3136S | |
| Bovine serum albumin, cold ethanol fraction | Sigma-Aldrich | A4503 | |
| Citric acid monohydrate | Sigma-Aldrich | C1909 | |
| D-Galactose | Carl Roth | 4987.2 | |
| D-Glucose | Sigma-Aldrich | G8270 | |
| Difco yeast nitrogen base | Becton Dickinson (BD) | 291940 | |
| Di-Sodium hydrogen phosphate heptahydrate | Carl Roth | X987.3 | |
| DL-Dithiothreitol | Sigma-Aldrich | D0632 | |
| D-Sorbitol | Carl Roth | 6213.1 | |
| Dulbecco’s phosphate buffered saline (10x) | Thermo Scientific | 14190169 | |
| Dynabeads Biotin Binder | Invitrogen | 11047 (Fisher Scientific) | |
| DynaMag-2 Magnet | Thermo Fisher | 12321D | |
| EBY100 | ATCC | MYA-4941 | |
| Electroporation cuvette 1 mm (for E.coli) | VWR | 732-1135 | |
| Electroporation cuvette 2 mm (for yeast) | VWR | 732-1136 | |
| Ethanol absolute | MERCK | 1070172511 | |
| GeneMorph II Random Mutagenesis Kit | Agilent Technologies | 200550 | |
| Gibco Bacto Casamino Acids | Becton Dickinson (BD) | 223120 | |
| Glycerol | AppliChem | 131339.1211 | |
| LE agarose | Biozym | 840004 | |
| Lithium acetate dihydrate | Sigma-Aldrich | L4158 | |
| Monarch DNA Gel Extraction Kit | NEB | T1020S | |
| Monarch PCR & DNA Cleanup Kit | NEB | T1030S | |
| Multifuge 1S-R | Heraeus | ||
| NheI-HF | NEB | R3131S | |
| Outgrowth medium | NEB | B9035S | |
| pCTCON2 | Addgene | #41843 | |
| Penicillin G sodium salt | Sigma-Aldrich | P3032 | |
| Penta-His antibody, AF488 | Qiagen | 35310 | |
| Penta-His antibody, AF647 | Qiagen | 35370 | |
| Peptone ex casein tryptically digested | Carl Roth | 8986.3 | |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491S | |
| SaII-HF | NEB | R3138S | |
| Sodium acetate | Sigma-Aldrich | S8750-1KG | |
| Sodium chloride | Carl Roth | 3957.2 | |
| Sodium dihydrogen phosphate monohydrate | Carl Roth | K300.2 | |
| Steritop threaded bottle top filter | MERCK | S2GPT01RE | |
| Streptavidin, AF488 | Invitrogen | S32354 (Thermo Fisher) | |
| Streptavidin, AF647 | Invitrogen | S32357 (Thermo Fisher) | |
| Streptomycin sulfate | Sigma-Aldrich | S6501 | |
| Tri-Sodium citrate dihydrate | Carl Roth | 4088.1 | |
| UV-Vis spectrophotometer | Agilent | 8453 | |
| Yeast extract, micro-granulated | Carl Roth | 2904.4 | |
| Zymoprep Yeast Plasmid Miniprep II | Zymo Research | D2004 |
Ссылки
- Boder, E. T., Wittrup, K. D. Yeast surface display for screening combinatorial polypeptide libraries. Nat Biotechnol. 15 (6), 553-557 (1997).
- Scott, J. K., Smith, G. P. Searching for peptide ligands with an epitope library. Science. 249 (4967), 386-390 (1990).
- Smith, G. P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 228 (4705), 1315-1317 (1985).
- Schaffitzel, C., Hanes, J., Jermutus, L., Pluckthun, A. Ribosome display: an in vitro method for selection and evolution of antibodies from libraries. J Immunol Methods. 231 (1-2), 119-135 (1999).
- Ho, M., Nagata, S., Pastan, I. Isolation of anti-CD22 Fv with high affinity by Fv display on human cells. Proc Natl Acad Sci U S A. 103 (25), 9637-9642 (2006).
- Wagner, A., et al. Identification of activating mutations in the transmembrane and extracellular domains of EGFR. Biochemistry. 61 (19), 2049-2062 (2022).
- Wagner, A., et al. PhosphoFlowSeq - A high-throughput kinase activity assay for screening drug resistance mutations in EGFR. J Mol Biol. 433 (22), 167210(2021).
- Cherf, G. M., Cochran, J. R. Applications of yeast surface display for protein engineering. Methods Mol Biol. 1319, 155-175 (2015).
- Zajc, C. U., Teufl, M., Traxlmayr, M. W. Affinity and stability analysis of yeast displayed proteins. Methods Mol Biol. 2491, 155-173 (2022).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr Opin Struct Biol. 17 (4), 467-473 (2007).
- Orr, B. A., Carr, L. M., Wittrup, K. D., Roy, E. J., Kranz, D. M. Rapid method for measuring ScFv thermal stability by yeast surface display. Biotechnol Prog. 19 (2), 631-638 (2003).
- Teufl, M., Zajc, C. U., Traxlmayr, M. W. Engineering strategies to overcome the stability-function trade-off in proteins. ACS Synth Biol. 11 (3), 1030-1039 (2022).
- Wozniak-Knopp, G., et al. Introducing antigen-binding sites in structural loops of immunoglobulin constant domains: Fc fragments with engineered HER2/neu-binding sites and antibody properties. Protein Eng Des Sel. 23 (4), 289-297 (2010).
- Traxlmayr, M. W., et al. Directed evolution of stabilized IgG1-Fc scaffolds by application of strong heat shock to libraries displayed on yeast. Biochim Biophys Acta. 1824 (4), 542-549 (2012).
- Traxlmayr, M. W., et al. Directed evolution of Her2/neu-binding IgG1-Fc for improved stability and resistance to aggregation by using yeast surface display. Protein Eng Des Sel. 26 (4), 255-265 (2013).
- Hasenhindl, C., et al. Stability assessment on a library scale: a rapid method for the evaluation of the commutability and insertion of residues in C-terminal loops of the CH3 domains of IgG1-Fc. Protein Eng Des Sel. 26 (10), 675-682 (2013).
- Koide, A., Wojcik, J., Gilbreth, R. N., Hoey, R. J., Koide, S. Teaching an old scaffold new tricks: monobodies constructed using alternative surfaces of the FN3 scaffold. J Mol Biol. 415 (2), 393-405 (2012).
- Hackel, B. J., Kapila, A., Wittrup, K. D. Picomolar affinity fibronectin domains engineered utilizing loop length diversity, recursive mutagenesis, and loop shuffling. J Mol Biol. 381 (5), 1238-1252 (2008).
- Traxlmayr, M. W., et al. Strong enrichment of aromatic residues in binding sites from a charge-neutralized hyperthermostable Sso7d scaffold library. J Biol Chem. 291 (43), 22496-22508 (2016).
- Zajc, C. U., et al. A conformation-specific ON-switch for controlling CAR T cells with an orally available drug. Proc Natl Acad Sci U S A. 117 (26), 14926-14935 (2020).
- Moore, S. J., Cochran, J. R. Engineering knottins as novel binding agents. Methods Enzymol. 503, 223-251 (2012).
- Hackel, B. J., Ackerman, M. E., Howland, S. W., Wittrup, K. D. Stability and CDR composition biases enrich binder functionality landscapes. J Mol Biol. 401 (1), 84-96 (2010).
- Hackel, B. J., Wittrup, K. D. The full amino acid repertoire is superior to serine/tyrosine for selection of high affinity immunoglobulin G binders from the fibronectin scaffold. Protein Eng Des Sel. 23 (4), 211-219 (2010).
- Laurent, E., et al. Directed evolution of stabilized monomeric CD19 for monovalent CAR interaction studies and monitoring of CAR-T cell patients. ACS Synth Biol. 10 (5), 1184-1198 (2021).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng Des Sel. 28 (10), 339-350 (2015).
- Cochran, J. R., Kim, Y. S., Lippow, S. M., Rao, B., Wittrup, K. D. Improved mutants from directed evolution are biased to orthologous substitutions. Protein Eng Des Sel. 19 (6), 245-253 (2006).
- Kieke, M. C., et al. High affinity T cell receptors from yeast display libraries block T cell activation by superantigens. J Mol Biol. 307 (5), 1305-1315 (2001).
- Lipovsek, D., et al. Selection of horseradish peroxidase variants with enhanced enantioselectivity by yeast surface display. Chem Biol. 14 (10), 1176-1185 (2007).
- Fushimi, T., et al. Mutant firefly luciferases with improved specific activity and dATP discrimination constructed by yeast cell surface engineering. Appl Microbiol Biotechnol. 97 (9), 4003-4011 (2013).
- Lam, S. S., et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods. 12 (1), 51-54 (2015).
- Park, S. W., Lee, D. S., Kim, Y. S. Engineering a U-box of E3 ligase E4B through yeast surface display-based functional screening generates a variant with enhanced ubiquitin ligase activity. Biochem Biophys Res Commun. 612, 147-153 (2022).
- Schutz, M., et al. Directed evolution of G protein-coupled receptors in yeast for higher functional production in eukaryotic expression hosts. Sci Rep. 6, 21508(2016).
- Angelini, A., et al. Protein engineering and selection using yeast surface display. Methods Mol Biol. 1319, 3-36 (2015).
- Chen, T. F., de Picciotto, S., Hackel, B. J., Wittrup, K. D. Engineering fibronectin-based binding proteins by yeast surface display. Methods Enzymol. 523, 303-326 (2013).
- Shusta, E. V., Holler, P. D., Kieke, M. C., Kranz, D. M., Wittrup, K. D. Directed evolution of a stable scaffold for T-cell receptor engineering. Nat Biotechnol. 18 (7), 754-759 (2000).
- Traxlmayr, M. W., Obinger, C. Directed evolution of proteins for increased stability and expression using yeast display. Arch Biochem Biophys. 526 (2), 174-180 (2012).
- Traxlmayr, M. W., Shusta, E. V. Directed evolution of protein thermal stability using yeast surface display. Methods Mol Biol. 1575, 45-65 (2017).
- Madeira, F., et al. The EMBL-EBI Job Dispatcher sequence analysis tools framework in 2024. Nucleic Acids Res. 52 (W1), W521-W525 (2024).
- Edgar, R. C. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Res. 32 (5), 1792-1797 (2004).
- Chao, G., et al. Isolating and engineering human antibodies using yeast surface display. Nat Protoc. 1 (2), 755-768 (2006).
- VanAntwerp, J. J., Wittrup, K. D. Fine affinity discrimination by yeast surface display and flow cytometry. Biotechnol Prog. 16 (1), 31-37 (2000).
- McConnell, A. D., et al. A general approach to antibody thermostabilization. MAbs. 6 (5), 1274-1282 (2014).
- Pavoor, T. V., Wheasler, J. A., Kamat, V., Shusta, E. V. An enhanced approach for engineering thermally stable proteins using yeast display. Protein Eng Des Sel. 25 (10), 625-630 (2012).
- Chen, I., Dorr, B. M., Liu, D. R. A general strategy for the evolution of bond-forming enzymes using yeast display. Proc Natl Acad Sci U S A. 108 (28), 11399-11404 (2011).
- Traxlmayr, M. W., et al. Construction of a stability landscape of the CH3 domain of human IgG1 by combining directed evolution with high throughput sequencing. J Mol Biol. 423 (3), 397-412 (2012).
- Chao, G., Cochran, J. R., Wittrup, K. D. Fine epitope mapping of anti-epidermal growth factor receptor antibodies through random mutagenesis and yeast surface display. J Mol Biol. 342 (2), 539-550 (2004).
- Stern, L. A., et al. Cellular-based selections aid yeast-display discovery of genuine cell-binding ligands: Targeting oncology vascular biomarker CD276. ACS Comb Sci. 21 (3), 207-222 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены