このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
エメラルドアッシュボーラーの遺伝子発現の解析( Agrilus planipennis)を使用して定量的リアルタイム- PCR
要約
定量リアルタイムPCR(定量RT - PCR)が異なる昆虫の組織と発達段階にmRNAレベルを診断する有効なツールです。このレポートでは、異なる幼虫の組織や侵襲的な昆虫種の発達段階で把握するmRNAのレベルまで定量RT - PCRの使用、エメラルド灰ボーラーを示す。
要約
エメラルド灰のボーラー(EAB、
プロトコル
別のステップで完全なプロトコルは、図1のフローチャートに示されている。解剖を構成する個々のステップは、RNA抽出、ファーストストランドcDNA合成や定量RT - PCRは以下に詳述する。
I.幼虫の解剖
- この手順の開始時、代わりにエメラルド灰ボーラー、またはEAB、湿ったティッシュペーパーで幼虫の前に解剖が行われるまで。新しく調製した1Xリン酸塩は解剖を通してバッファの寒さを保つために氷の上で、またはPBS、生理食塩水バッファリングしてください。
- 開始するには、微細な解剖ピンと解剖板にEAB幼虫を修正。その後、解剖の板に冷1X PBSを加える。
- 最初に、解剖鋏の罰金ペアで幼虫を斬る。その後、幼虫の最後の腹部のセグメントを削除します。
- 鉗子の時計職人のペアを使用して、幼虫の死骸からキューティクルを持ち上げます。その後、はさみを使用して、枝肉の長さに沿って切開を行います。切開がなされると同時に破裂腸をしないように注意してください。
- 慎重にそのような脂肪体や結合組織などの他の組織から中腸組織を分離する。その後、fatbodyのコンタミネーションがないことを保証するために新鮮な1 × PBS緩衝液中の組織をすすいでください。 1.5mlのエッペンドルフチューブにあらかじめ冷却Trizol試薬を500μLに分離された腸を転送します。
- 次に、鉗子を用いて脂肪体の組織を分離し、チルドTrizol試薬500μLを含む1.5mlのエッペンドルフチューブに移す。
- 最後に、組織の整合性を傷つけないように注意しながら、任意の付着組織をオフ解体によって幼虫の死骸から表皮組織を分離する。新鮮なPBSバッファーで分離された表皮の組織をリンスし、チルドTrizol試薬500μLを1.5 mLのエッペンドルフチューブに移す。
- 孤立した脂肪体、表皮と中腸組織を-80℃で保存できますRNA抽出までのC。
- RNA抽出のための準備として、発達段階に応じて様々なEABのサンプルを並べ替える。これらの段階は、1次、2次、第3、および4次齢期、prepupaeと大人が含まれています。
II。 RNA抽出(TRIzol試薬を用いての一人当たりの製造業者のプロトコルとして)
均質化
- RNAの抽出を開始するには、まずトリゾール含む1.5mlのエッペンドルフチューブに組織を均質化するプラスチック製乳棒を使用してください。均質化した後、Trizol試薬でそれぞれサンプルTO1 mLの合計量をもたらす。
- 発達段階の場合は、乳鉢と乳棒で液体窒素で全体の動物をホモジナイズする。その後トリゾールの1mLにホモジネートのアリコート(50μg)を追加。
相分離:
- 室温で15分間1 mLのトリゾールでサンプルチューブをインキュベートする。
- インキュベーション後、各チューブにクロロホルムの200μLを加える。すぐにクロロホルムを加えた後、約15秒間激しく振らない。その後、さらに2〜3分間室温でチューブをインキュベートする。
- 次に、2℃で15分間12,000 xgでサンプルを遠心遠心分離後、RNAは水相になります。水相の体積は600μlの、または、総トリゾール容積の60%にする必要があります。
RNA沈殿
- 慎重にのみ水相を除去。水相以下の物質の存在は、抽出されたRNAのコンタミネーションの原因となります。その後、適切に標識された1.5mlのエッペンドルフチューブに水相を移す。
- 水相からRNAを沈殿させ、各チューブにイソプロピルアルコール0.5 mLを混合する。室温で10分間サンプルをインキュベートする。インキュベーション後、2℃で10分間、12,000 xgでチューブを遠心
RNAウォッシュ
- 遠心分離に続いて、チューブから上清を捨てる。 RNAは、チューブの底に形成されたゲル状のペレットに存在しています。トリゾールの1mLあたり75%エタノールの少なくとも1 mLを加え、75%エタノールでペレットを洗浄します。ボルテックスにより混和する。その後、2℃で5分間7500Xgでチューブを遠心
RNA溶出
- チューブから上清を捨てる。 1分間の小さなテーブルの遠心機で再びチューブを遠心してください。慎重にピペットでチューブから余分な上清を取り除きます。管が開いたままにしておくと5〜10分間ペレットの空気は乾燥させて。これは大幅にその溶解度が低下すると上のペレットを乾燥しないように注意してください。
- 空気乾燥した後、でペレット懸濁しますジエチル 、またはDEPC処理水を。ペレットを再懸濁するために使用される水の量は、ペレットの大きさに依存します。 SMのためのDEPC処理水50μLを使用大きなペレットのすべてのペレットまたは100μL。ペレットが完全に上下に数回ピペットの先端を移動することによって、DEPC処理水に溶けてみましょう。
- ペレットが溶解した後、10分間55℃でサンプルを置きます。
- その後、光度計分光光度計(RNAサンプルを2μL)を使用して、各RNAサンプルの濃度を測定する。
- -80 RNAサンプルを格納℃でさらに使用するまで。
III。ファーストストランドcDNA合成(Invitrogen社のSuperScriptファーストストランド合成キットを使用してあたりの製造業者のプロトコルとして。)
- 各RNAサンプルについてPCRチューブに以下を追加します。
RNA Xμlを 10mMのdNTPミックスM 1μlの オリゴ(dT)(0.5μg/μLの) 1μL DEPCは水を扱わ (8 - x)を添加 総量: 10μlの 注:xはcDNA合成に使用するRNAの量です。 RNAの濃度に応じて、RNAの2-3μgの各反応に使用され、各反応の合計量は10μlにする必要があります。 - 4分間で65サーマルサイクラーで上記の反応ミックス° Cを置きます。
- theromocylerからチューブを外して、少なくとも2分間、すぐに氷上に置きます。
- 以下を追加することによって、最初の鎖のマスターミックスを調製する:
10X RTバッファー 2μlの 25mMのMgCl 2の 4μlの 0.1M DTT 2μlの RNaseの出 1μL - 、RNAサンプルを含む各チューブにマスターミックスの9μlを加えペッティングにより混合し、混合物を手短に遠心します。
- クラーに戻ってチューブをセットし、42℃で2分間インキュベート
- 一時停止クラーと各チューブにSuperscript II逆転写酵素酵素(Invitrogen社)を1μl加える。この手順は素早く行う必要があります。
- 1.5時間42℃でサンプルをインキュベートする。
- 70反応℃で15分間を終了します。
- クラーからチューブを取り出して、氷の上に置きます。
- 各チューブにRNase H 1μlを加え、37℃でサーマルサイクラーに戻して置きます℃で20分間。
- クラーからサンプルを取った後、各チューブにヌクレアーゼフリー水20μlを加える。各サンプルの体積は、この段階で倍になります。
- サンプル2μlを用いて各サンプルの濃度を確認してください。定量RT - PCRで使用するために20ng/μlの濃度を作るために、各試料のアリコートを取る。 20ng/μlの濃度にサンプルを補うために水の量は、合成されたcDNAの初期濃度に依存します。
IV。定量RT - PCR:
リファレンス遺伝子のプライマーの設計と測定
- 60 ° Cと〜100ベーシスポイントの製品サイズの融解温度(Tm)と設計プライマー。
- AP - PERI1は、この実験の目的の遺伝子として使用されます。リファレンス遺伝子や内部統制は、データの後の分析と正規化のために必要とされる。リボソームタンパク質(AP - RP1)がこの実験のためのリファレンス遺伝子として使用されます。
- ご参照の遺伝子を含む使用されるプライマーの5uMワーキングストックを準備する。
標準の調製
- 標準がなされるべき使用されているプライマーの効率を計算するために。
- テストされるさまざまなサンプルからのcDNAのミックスをしてください。
- グラフ内の各目的のポイントの5倍希釈系列を作る。四希釈は十分なはずですが、5を強くお勧めします。
- ボリュームが使用されているポイント数とどのように多くの技術的複製によって異なります。十分なリファレンス遺伝子を含むテストされる各遺伝子のための標準を作るためにあるはずです。
プレートテンプレート
- 定量RT - PCRプレートの各ウェルのサンプル名を示すテンプレートを設計します。各遺伝子の基準と少なくとも二つの技術は、サンプルあたりの複製を含めることを忘れないでください。
サイクリングのパラメータを設定する
- に定量RT - PCRマシンの電源を入れ、サイクリングパラメータを入力します。次のようにこの実験のためのサイクリングパラメータは、次のとおりです。
ステップ1:- 1サイクル:95℃で3分間
- 40サイクル:95℃15秒、60℃で30秒続く
(オプション:融解曲線を決定するためには、次の手順を含む)
- 1サイクル:95℃1分
- 1サイクル:55℃1分
- 81サイクル:55℃30秒
CFX96(BioRad社)マシン用に設定されたプレート
- 別のチューブ内の各遺伝子(リファレンス遺伝子を含む)のためのマスターミックスを調製する。各ウェルについて、マスターミックスは、構成されています
ヌクレアーゼフリー水 2μlの 2.5 X SYBRグリーン 4μlの フォワードプライマー 1μL プライマーリバース 1μL 合計ボリューム μlの8 - 準備中にマスターミックスは、ピペッティングエラーのアカウント1-2余分な反応が含まれています。
- 以前に設計されたテンプレートに従って、各ウェルにcDNAを2μL(20 ng /μLの)追加。その後、反応あたり10μLの合計量で、その結果、各ウェルにマスターミックスの8μLを加える。
- サンプルがよく混合されていることを確認するまでピペットとダウン(2 - 3X)。液体が先端にとどまるいないことを確認してください。クロスコンタミネーションを避けるために、各サンプルのための新しいヒントを使用してください。
- プレート全体をセットアップしたら、光学テープでプレートをカバーしています。グリースの存在は読書に影響を与えることができるようにテープに触れないようにしてください。その後、ウェル内のすべての製品は、プレートの底にあることを保証するために1分間500rpmでプレートを遠心する。
- 遠心分離に続いて、プレートの底には氷や水がないことを確認してください。最後に、定量RT - PCRマシンにプレートを配置し、PCRプログラムを実行します。
実験のV.模式図

図1:遺伝子発現研究のためのステップの順序を示すフローチャート。
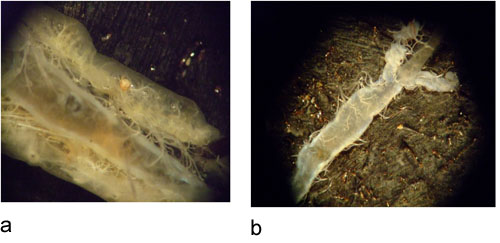
図2:中央に中腸を示す)幼虫のEABの解剖。幼虫のEABのB)孤立の中腸

図3:)キューティクル(銅)、中腸(MG)と脂肪体(FB)を含むさまざまな幼虫組織におけるperitrophin遺伝子(AP - PERI1)の相対式の値を(回転数)を意味します。 EAB特定のリボソームタンパク質は、(ここに名前、AP - RP1)目的の遺伝子、AP - PERI1ために得られたデータを正規化するための内部コントロールとして使用した。幼虫の組織中のAP - PERI1のB)相対倍に変化。キューティクル組織は、少なくとも発現を示し、したがって、校正器(1X)のサンプル(Pfaffl、2001)とした。

図4:)幼虫の齢期(第1、第2、第3および第4)、prepupae(PP)および成人()を含むEABの異なる発達段階におけるperitrophin遺伝子(AP - PERI1)の相対式の値を(回転数)を意味します。 EAB特定のリボソームタンパク質(AP - RP1)が目的の遺伝子、AP - PERI1ために得られたデータを正規化するための内部コントロールとして使用した。 B)様々な発達段階におけるAP - PERI1の相対倍の変化が。 PPのサンプルは、mRNAレベルの最低水準を示したため、キャリブレーター(1Xサンプル)とした。
VI。結論
定量リアルタイムPCR(定量RT - PCR)が異なる昆虫の組織と発達段階にmRNAレベルを診断する有効なツールです。さらに、定量RT - PCRは、主にマイクロアレイと新世代のRNA - seqのようなハイスループット遺伝子発現解析から生成されたデータを検証するために重要なツールとなっています。
ディスカッション
閾値サイクル(Ct値)の値は、定量RT - PCRプレートに各サンプルについて得られる。標準曲線を作成するために、各希釈液について得られたCt値は、その濃度の対数に対してプロットした。実験サンプルのCt値は、この希釈系列の標準曲線にプロットした。目標量は、それぞれの実験、すなわち組織や発生過程での発現用に生成された別々の標準曲線から計算した。相対式の値は、(回転)し?...
開示事項
謝辞
我々は、実験のセットアップでルルドデルタArruetaアンテケラ(昆虫学科、オハイオ州立大学/ OARDC、ウースター、オハイオ州)が提供するヘルプを認めます。我々は、EAB幼虫のサンプルを送るための博士テレーズポーランドを(USDA、森林サービス、NRS)感謝。顕微鏡のセットアップが認識されているとDr。ルイスカナスとNuris Mアコスタが提供するのに役立ちます。このプロジェクトの資金は、オハイオ農業研究開発センター、オハイオ州立大学に充当州と連邦資金によって提供されていました。
参考文献
- Lehane, M. J. Peritrophic matrix structure and function. Annu Rev Entomol. 42, 525-550 (1997).
- Mittapalli, O., Sardesai, N., Shukle, R. H. cDNA cloning and transcriptional expression of a peritrophin-like gene in the Hessian fly, Mayetiola destructor [Say]. Arc of Insect Bio. 64, 19-29 (2007).
- Pauchet, Y., Wilkinson, P., van Munster, M., Augustin, S., Pauron, D., ffrench-Constant, R. H. Pyrosequencing of the midgut transcriptome of the poplar leaf beetle Chrysomela tremulae reveals new gene families in Coleoptera. Ins Bio and Mol. 39, 403-413 (2009).
- Pfaffl, W. M. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 29, 2002-2007 (2001).
- Tellam, R. L., Lehane, M. J., Billingsley, P. F. . Biology of the Insect Midgut. , 86-114 (1996).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved