Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Biology
Fag-medieret Levering af målrettet Srna Konstruerer til Knock Down genekspression i
We describe a method to knock down gene expression in a growing population of E. coli cells using sequence-targeted sRNA expression cassettes delivered by an M13 phagemid vector.
RNA-medieret knockdowns er almindeligt anvendt til at kontrollere genekspression. Denne alsidige familie af teknikker gør brug af korte RNA (Srna), der kan syntetiseres med en hvilken som helst sekvens og designet til at supplere helst gen målrettet til nedregulering. Fordi Srna konstruktioner kan indføres på mange celletyper direkte eller ved anvendelse af en række vektorer, kan genekspression undertrykkes i levende celler uden besværlige genetisk modifikation. Den mest almindelige RNA knockdown teknologi, RNA-interferens (RNAi), gør brug af det endogene RNA-induceret silencing kompleks (RISC) til at mediere sekvens genkendelse og spaltning af mål-mRNA'et. Anvendelser af denne teknik er derfor begrænset til RISC-udtrykkende organismer, primært eukaryoter. For nylig har en ny generation af RNA bioteknologer udviklet alternative mekanismer til styring af genekspression gennem RNA, og så gjort mulige RNA-medieret gen knockdowns i bakterier. Her beskriver vi en metode til lyddæmpning gen expression i E. coli, som funktionelt ligner RNAi. I dette system en syntetisk phagmid er designet til at udtrykke Srna, som kan designet til at målrette helst sekvens. Ekspressionskonstruktionen afgives til en population af E. coli-celler med ikke-lytisk M13-fag, hvorefter det er i stand til stabilt at replicere et plasmid. Antisense anerkendelse og lyddæmpning af mål-mRNA'et medieres af Hfq proteinet, endogene for E. coli. Denne protokol omfatter metoder til at designe antisense Srna, konstruere fagmidvektoren, emballering fagmidet i M13 bakteriofag, forbereder en levende celle population for infektion, og udfører infektionen selv. Den fluorescerende protein mKate2 og antibiotikaresistensgenet chloramphenicolacetyltransferase (CAT) er målrettet til at generere repræsentative data og kvantificere knockdown effektivitet.
RNA-medieret gen knockdowns frem i to etaper. Først et RNA-molekyle indført i en cellelinie eller organisme af undersøgelsen. For det andet, endogene RNA-bindende proteiner lette RNA-target anerkendelse og producere den lyddæmpende effekt. Alle RNA knockdown teknologier drage fordel af den tilpasses karakteren af syntetiske sRNA'er, som let kan fremstilles til at matche et specifikt mål af interesse. Men de molekylære detaljer RNA optagelse og lyddæmpning varierer meget i modelsystem, begrænse, hvor og hvordan RNA knockdowns kan anvendes.
I nematoder, dobbeltstrenget RNA (dsRNA) molekyler kan indføres direkte i medierne eller ved at fodre ormene med en befolkning på dsRNA-udtrykkende E. coli-celler 1,2. I Drosophila, kan RNAi opnås ved mikroinjektion embryoner med dsRNA 3, eller gennemføres i cellelinier ved blot at tilføje dsRNA til dyrkningsmediet 4. I mammale cellelinjer,syntetiske små interfererende RNA'er (siRNA'er) kan leveres til levende celler ved elektroporering 1,2,5, pakket i liposomer 3,6, eller udtrykt fra DNA plasmidvektorer 4,7. Når RNA-species når cytosolen, RNAi pathway afhængig af RISC-komplekset til at behandle dsRNA, lette antisense genkendelsen af målet, og katalysere translationel undertrykkelse, mRNA nedbrydning eller heterochromatin formation, afhængig af værten.
På grund af disse krav, kan klassisk RNAi kun udføres i organismer, der optager eksogen RNA effektivt og udtrykker RISC eller en RISC-lignende aktivitet. Især dette udelukker model bakterien E. coli, som mangler RNAi pathway. Men de seneste fremskridt i syntetisk biologi give værktøjer til at løse både levering problemet og lyddæmpning problem.
I denne protokol, er Srna konstruktioner udtrykt i E. coli fra en DNA-vektor leveret til living celler under anvendelse af M13 phagmid / hjælper-system. En fagmid er enhver plasmid med en fag-afledt f1-replikationsstartsted. En hjælpe-plasmid, i dette tilfælde M13KO, bærer alle maskiner kræves for at producere viruspartikler, men er ikke selv kompetent til replikation og pakning. Når en phagemid og hjælper plasmid er co-transformeret, er alene fagmidet replikeres på f1 oprindelse, pakket og udskilles. Den vektoriseret phagmid er derefter kompetent til at inficere levende E. coli via F pilus.
I dette system er den lyddæmpende virkning, som brugerdefinerede Srna kassetter kombinerer et stillads sekvens med en mål-bindende sekvens. Target-bindende sekvens er 24 basepar antisense til et mRNA target, typisk ved ribosom-bindingssted (RBS). Stilladset sekvens, udviklet af Na og kolleger 8, indeholder en Hfq-bindende motiv ekstraheret fra MICC, en lille regulerende RNA endogent for E. coli. Den Hfq protein stimulerer RNA-RNA binding eng og mRNA nedbrydning, der betjener en rolle i dette system svarer til RISC i RNAi. Figur 1 viser en komplet ordning for fag-medieret Srna knockdowns, herunder Srna kassette struktur, phagemid vectorization og lyddæmpning mekanisme.
Som en fremgangsmåde til at modulere genekspression i E. coli, Srna nedregulering er enkel, hurtig og alsidig. Den målrettede E. coli ikke belastes udover formerings fagmidet og udtrykker Srna. Dette kan være relevant i forbindelse med syntetisk biologi eller grundforskning hvor udtrykket af større heterologe konstruktioner kan belaste cellulære ressourcer 9. Fagmider med nye mål kan fremstilles med en enkelt PCR og høstet en dag efter fagmid transformation. Endelig kan næsten enhver mRNA målrettes. Den Srna forordning kassette (på en standard plasmid) har vist sig at arbejde på en række forskellige mål i stofskiftet med typiske undertrykkelse niveauer> 90% 8.
10. Først en emballeret phagemid introduceret til en batch kultur af E. coli-celler og bruges til at lukke munden ekspressionen af det fluorescerende protein mKate2. Efterfølgende fluorescensændringer overvåges i realtid. For det andet, banker ned CAT-genet er vist at reducere fænotypisk chloramphenicolresistens på agarplader. I begge tilfælde fagmidet selv bærer en GFP markering, tillader infektion, der skal måles uafhængigt af knockdown effektivitet.
1. Design og konstruktion af fagmidvektorer Bearing Srna Silencing Kassetter
- De Novo Design af Srna Lyddæmpningsplader kassetter 8
- Identificere hele sekvensen af mRNA'et, der skal til tavshed anvendelse af en DNA-sekvens database. For at generere målsekvensen, bemærk første 24 bp af den kodende sekvens, fra stilling +1 til +24 begyndende med startkodonen (f.eks ATG).
Bemærk: Silencing er mindre effektive, når andre websteder eller segmenter af mRNA er målrettet 8. - Tag den omvendte komplement af målsekvensen til frembringelse GUIDE sekvens for Srna kassetten. Se tabel 1 for eksempler på TARGET og GUIDE sekvenser for chloramphenicolacetyltransferase (CAT).
- At designe 292 bp komplet Srna ekspressionskassette, arrangere pR-promotoren, styresekvens, Hfq proteinbindingsdomæne og T1 / TE transkriptionelle terminatorsekvenser i serie (tabel 2).
- Tilføje yderligere kloningssites af valg for at lette kloning af Srna kassetten i målvektoren.
- Opnå den fuldstændige Srna kassetten gennem kommerciel gensyntese eller en lignende fremgangsmåde og klone den ind i enhver fagmidvektor med en funktionel f1 replikationsstartsted 11. Se yderligere oplysninger til den fuldstændige sekvens af det endelige fagmidvektor.
- Identificere hele sekvensen af mRNA'et, der skal til tavshed anvendelse af en DNA-sekvens database. For at generere målsekvensen, bemærk første 24 bp af den kodende sekvens, fra stilling +1 til +24 begyndende med startkodonen (f.eks ATG).
- Ændring af Sequence Målrettet af en eksisterende Srna Expression Kassette hjælp PCR-baseret mutagenese 12
- Identificer 24 bp GUIDE sekvens i en eksisterende Srna ekspressionskassette. Bemærk: Den kommenteret pAB.001 plasmid, der anvendes i dette arbejde, er tilgængelig som en supplerende sekvens fil.
- Design fremadrettede og reverse primere med korte regioner af homologi med eksisterende Srna kassetten flankerer den nye 24 bp GUIDE sekvens. Få primerne gennem kommercielle oligonucleotidsyntese.
Bemærk: Primer design for site-directed mutagenese er depicted i figur 2. Exact primer template-sekvenser er tilvejebragt i tabel 3. - Forbered en 5 ml kultur af E. coli bærende skabelonen Srna ekspression phagemid. Grow cellerne natten over ved 37 ° C under omrystning, i LB-medium med passende antibiotika.
- Uddrag og oprense skabelonen Srna ekspression fagmid fra 5 ml bakteriekultur under anvendelse af et DNA miniprep kit eller lignende metode 12.
- Forbered to PCR-reaktioner ved anvendelse af skabelonen Srna ekspression phagemid og high fidelity polymerase, et med den forreste og et med den bagudrettede primer (tabel 4). Bruge PCR-betingelser som anbefalet af polymerase leverandøren (tabel 5). Forøg skabelonen fusions 10-50x højere end en standard reaktion at tage højde for det forhold, at den enkelt primer reaktionen ikke producerer eksponentiel amplifikation.
- Kombinere de to ovennævnte PCR-reaktioner i et mikrocentrifugerør. Anneal produkterne ved opvarmning til 98 ° C i et kogende vandbad. Umiddelbart efter placering af mikrocentrifugerør i vandbadet, fjern varmekilde og tillader badet til langsomt at vende tilbage til stuetemperatur i løbet 1-2 timer.
- For at eliminere ikke-muterede skabelon Srna udtryk phagemid, tilsættes 1 pi. Dpnl restriktionsenzym til blandingen, og der inkuberes ved 37 ° C i 1 time, eller den tid, som anbefales af producenten til fuldstændig fordøjelse.
Bemærk: Dpnl fordøjer kun methylerede målsteder, som er til stede på værten-replikerede fagmider men ikke PCR-produkter. - Transform købt eller forberedt 13 kemisk kompetent E. coli med 1-5 pi af det annealede PCR-produkt. Isolere enkelte kolonier af den transformerede stamme ved selektiv udpladning på LB-agarplader indeholdende passende antibiotika.
- For at verificere inkorporeringen af den korrekte GUIDE sekvens, screene de resulterende kolonier ved koloni-PCR. Under anvendelse af en 200 pi pipettespids, collect en lille mængde af celler fra en enkelt transformeret koloni. Mark og bevare den oprindelige koloni til downstream brug efter kontrol.
- Tilføj de indsamlede celler til 50 pi nuclease-frit vand i et mikrocentrifugerør. Bland ved at pipettere op og ned.
- Ved hjælp af en bench-top thermocycler eller et kogende vandbad, lysere cellerne ved opvarmning til 95 ° C i 2 min.
- PCR-amplifikation fagmidet region anvendelse af 1 pi varme-lyseret celler som en DNA-template. PCR-betingelser og thermocycler protokoller findes i tabel 6 og 7. Se den supplerende pAB.001 sekvens fil til verifikation primersekvenserne.
- Sekventere PCR-produktet for at verificere inkorporering af den korrekte styresekvens.
- Inokulere en 5 ml kultur af E. coli klon bærer sekvensverificeret Srna ekspression phagemid. Grow cellerne natten over ved 37 ° C under omrystning, i selektivt LB-medier.
- Forbered en glycerolstammeaf sekvensen-verificeret klon. Med 750 pi af overnatskulturen til 250 pi 60% glycerol i et skruelåg kryo rør.
- Opbevar glycerolstamme ved -80 ° C uendeligt. Resten af overnatskulturen kan anvendes som en kilde til den Srna ekspression fagmid i trin 2.
2. Produktion og Høst af M13-pakkede phagemid Stocks
- Forbered en 5 ml kultur af E. coli der bærer Srna ekspression phagemid. Grow cellerne natten over ved 37 ° C under omrystning, i LB-medium med passende antibiotika. Bemærk: Den Srna udtryk phagemid kan fås gennem de novo kloning som beskrevet i trin 1.1.5, eller modificeret fra en eksisterende phagemid og høstet i trin 1.2.16.
- På lignende måde fremstilles en 5 ml kultur af E. coli der bærer M13KO7 hjælper-plasmidet. Grow cellerne natten over ved 37 ° C under omrystning, i selektivt LB-medier.
- Uddrag og rense Srna expression phagmid og hjælpe-plasmid under anvendelse af et DNA-ekstraktion kit eller lignende metode 12.
- Samtidig transformation købt eller forberedt 13 kemisk kompetente E. coli med 1 pi hver af Srna udtryk phagemid og hjælper plasmid. Vælg for cotransformanterne ved udpladning på LB agar med selektive antibiotika til begge konstruktioner.
- Forbered en 10 ml kultur fra en enkelt koloni af den co-transformeret stammen i LB med selektive antibiotika. Der inkuberes ved 37 ° C under omrystning i 8-12 timer eller natten over.
- Centrifugeres kulturen ved 3.300 xg i 10 min. Opsaml supernatanten og filtreres gennem et 0,2 um filter. Forsigtig: I tilfælde af medier spild, rengøre området med fortyndet blegemiddel (0,5%) til at ødelægge infektiøse fagpartikler.
- den emballerede phagemid filtrat ved 4 ° C opbevares. Bemærk: Prøverne kan opretholdes i dage til uger uden tab af aktivitet.
3. Fremstilling af F + targetceller for Silencing
- Undersøg, om de celler, der skal målrettet til tavshed udtrykker F pilus 14. Hvis F pilus er allerede til stede, fortsæt til trin 4.
Bemærk: Fælles laboratoriestammer af E. coli betegnet som F + eller F 'til at indikere tilstedeværelsen af F pilus i deres genom eller på et plasmid. - Opnå en F + stamme af E. coli, såsom TOP10F '.
Bemærk: Sørg for, at målet stammen bærer en unik modstand markør for at være adskilt fra F-plasmidet donor efter konjugering. - At introducere F pilus ved konjugation med en F + stamme, forberede 5 ml kulturer af både mål-stamme og F-plasmid donor 14. Grow cellerne natten over ved 37 ° C under omrystning, i LB-medium med passende antibiotika.
- Den følgende dag, fortyndes begge stammer 1: 100 i 5 ml selektive LB og fortsætte dyrkning ved 37 ° C under omrystning.
- Bestem vækstfasen af cellerne ved at måle optical densitet af kulturen ved 600 nm (OD600) ved anvendelse af en benchtop spektrofotometer. Dyrke cellerne i ca. 2 timer, indtil en OD600 på 0,3 er opnået, hvilket indikerer log-vækstfase 15.
- Forbered 3 konjugeringsreaktioner i mikrocentrifugerør: 0,5 ml F-plasmid donor + 0,5 ml target-stamme, 0,5 ml F-plasmid donor + 0,5 ml LB-medium (negativ kontrol) og 0,5 ml target stamme + 0,5 ml LB-medium (negativ kontrol). Tillad konjugeringen forløbe i 2 timer ved 37 ° C under omrystning.
- Plade 100 pi af hver konjugeringsreaktionen på selektiv LB-agar med antibiotika er specifikke for F-plasmidet (typisk tetracyclin) og target-stamme. Plate de negative kontrolreaktioner for at bekræfte, at hverken donor eller recipient stamme udtrykke begge antibiotikaresistenser.
4. Infektion med Pakkede Fagmider til Lyddæmpning
- Inokulere en enkelt koloni af F + målceller i en 5 ml kultur af LB medien med passende antibiotika. Inkuber natten over ved 37 ° C under omrystning.
- Den følgende dag, fortyndes F + målceller 1: 100 i 5 ml selektive LB-medier og fortsat dyrkning ved 37 ° C under omrystning.
- Bestem vækstfasen af cellerne ved at måle den optiske densitet af kulturen ved 600 nm (OD600) ved anvendelse af en benchtop spektrofotometer. Dyrke cellerne i ca. 2 timer, indtil en OD600 på 0,3 er opnået, hvilket indikerer log-vækstfase 15. Bemærk: Ekspression af F pilus og infektion effektivitet er højest i log-fase.
- Tilsæt M13-emballerede fagmider (fra trin 2.6) til målcellerne i et volumenforhold på 1: 100 for at opnå næsten 99% infektion af målpopulationen. Tillad infektionen forløbe ved 37 ° C under omrystning i 30-60 min.
- Analysere de Srna-dæmpende fænotype ifølge den foretrukne metode.
Bemærk: For et fluorescerende protein mål, kan den lyddæmpende effekt kvantificeresdirekte ved fluorometri 10. Alternativt kan fænotypiske analyser anvendes til at observere fænotypiske konsekvenser af gen knockdown 8. - Forbered en glycerol lager for Srna-udtrykkende phagemid vært efter trin 1.2.14-1.2.16. Bemærk: fagmidet vil udbrede sig i værtsstammen på ubestemt tid og kan opretholdes med antibiotika ligner en konventionel plasmid.
Silencing af mKate2 Fluorescens i Liquid Media
Figur 1 viser den ordning for Srna-medierede knockdowns beskrevet i dette arbejde, herunder Srna kassette design, phagemid vectorization, og lyddæmpning mekanisme. Efter protokol 1.2 blev Srna lyddæmpende kassette af plasmid pAB.001 ændret til at målrette mKate. Den Srna kassetten blev syntetiseret og klonet ind phagemid Litmus28i_J23115-B0032-GFP, en gave fra Monica Ortiz og Drew Endy 11. Dette phagemid bærer markører for GFP udtryk og kanamycin resistens, så succesrige infektioner, der skal spores. Pakkede fagemid lagre udarbejdet følgende protokol 2.
Et derivat af E. coli K12 MG1655 bærer en konstitutivt udtrykt, kromosomalt integreret mKate2 markør blev forberedt til fag infektion ved konjugation med en F-plasmid donorstamme følgende protokol 3. Cellerne blev dyrket til midt log-fase og fagmidet indført følgende protokol 4. Efter fagmid infektion blev 200 pi kulturer overført til en fluorescenspladelæser og fluorescens blev monitoreret kontinuerligt i 24 timer.
Figur 3 viser virkningen af Srna-medieret inaktivering på mKate2 ekspression. Stammen inficeret med et anti-mKate2 fagmid viste ingen påviselig mKate2 fluorescens over baggrund. I modsætning hertil denne stamme gjorde udtrykke GFP markering, som angiver en vellykket optagelse af fagmidet. Uinficerede kontrolceller produceret mKate2 fluorescens, men ikke GFP. En yderligere kontrol, hvori det målrettende domæne anti-mKate2 blev erstattet med en sekvens målretning CAT, havde ingen virkning på mKate2 fluorescens.
Silencing af chloramphenicolresistens på agarplader
indhold "fo: holde-together.within-side =" 1 "> Efter protokol 1.1, en Srna silencing kassette blev produceret til at målrette CAT Et derivat af E. coli K12 MG1655 bærer en konstitutivt udtrykt, blev kromosomalt integreret CAT markør forberedt. faginfektion ved konjugation med en F-plasmid donorstamme følgende protokol 3. cellerne blev dyrket til midt log-fase og fagmidet indført følgende protokol 4. efter 1 times inkubation ved 37 ° C blev inficerede celler serielt fortyndet og udpladet ved en vifte af chloramphenicol-koncentrationer. Plader blev inkuberet natten over, og andelen af resistente celler ved hver chloramphenicol blev bestemt ved at tælle kolonidannende enheder (CFU'er) den næste dag.Figur 4 viser virkningen af Srna-medieret inaktivering på chloramphenicol resistens fænotype. Uinficerede celler eller celler inficeret med fagmid målretning mKate2, var resistente over forchloramphenicol på alle testede koncentrationer. I modsætning hertil celler inficeret med fagmid målretning CAT viste nedsat overlevelse ved lave koncentrationer chloramphenicol, og næsten 99% drab ved højere koncentrationer. Tilsætningen af ampicillin, for at vælge kun bakterier, som bærer fagmidet, reducerede chloramphenicol overlevelse til ikke-detekterbare niveauer. Dette er i overensstemmelse med tidligere arbejde viser, at flugt fra fag-infektion er en almindelig vej at flygte lyddæmpning 10.
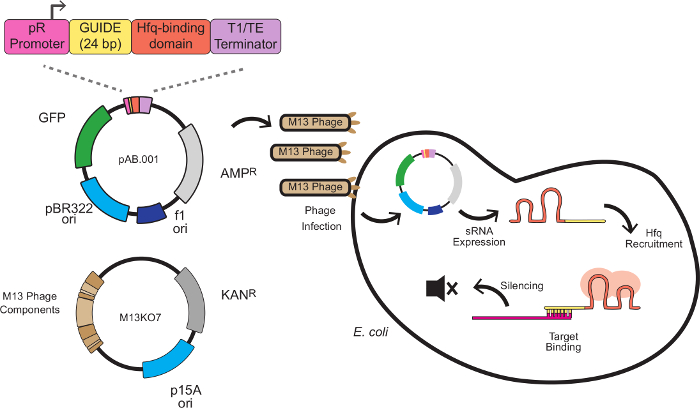
Figur 1: gendæmpning i E. coli med Srna ekspressionskassetter Leveres af M13-fag The Srna kassette består af 4 moduler:. PR promoteren (en konstitutiv promotor er afledt af bakteriofag lambda), en 24 bp målretningsdomæne, en Hfq bindingsdomæne ekstraheret fra MICC og en transkriptionsterminator 8 E. coli, der udtrykker F pilus, hvor Srna ekspression begynder. Den Srna rekrutterer så Hfq protein (afbildet i rødt) og binder en antisense mRNA mål nær ribosombindingsstedet, hvilket resulterer i translationel undertrykkelse og mRNA nedbrydning. Klik her for at se en større version af dette tal.
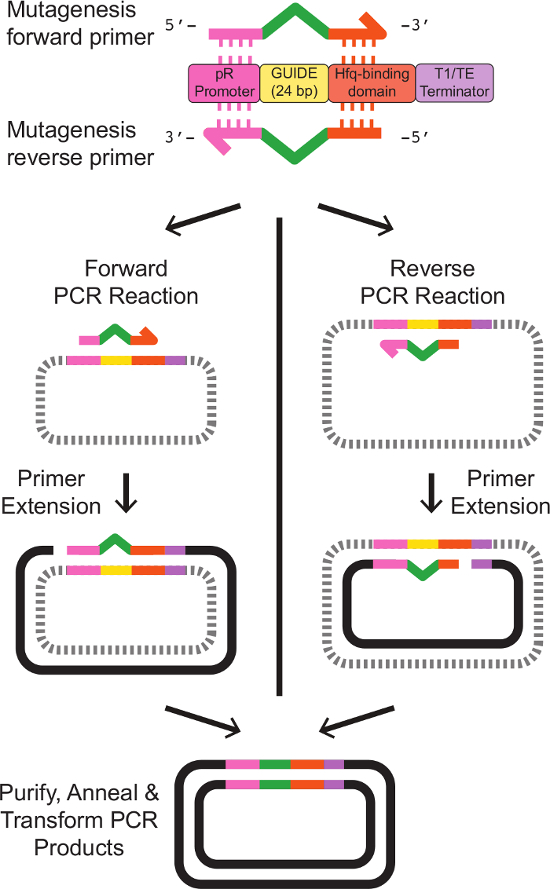
Figur 2:. Primer Design og stedorienteret mutagenese af Srna målstedet To primere er udformet med delvis homologi med en eksisterende Srna kassette. Den fremadrettede primer indeholder 20 bp homolog med pR-promotoren i 5'-enden, efterfulgt af24 bp repræsenterer det nye styresekvens, derefter 18 bp homolog med Hfq-bindende domæne ved 3 'enden. Den reverse primer er den nøjagtige omvendte komplement af den fremadrettede primer, med regioner af homologi med eksisterende Srna kassetten flankerer reverse komplement af den nye styresekvens. Nøjagtige primersekvenser er angivet i tabel 3. Separate single-primer PCR-reaktioner med den forreste og reverse primere producerer lineære, enkeltstrenget DNA med den ønskede modificerede sekvens. Annealing fremad og bagud reaktionsprodukter efter oprydning som beskrevet i protokollen, resulterer i dobbeltstrenget plasmid-DNA, der bærer det ønskede modificerede Srna kassette. Klik her for at se en større version af dette tal.
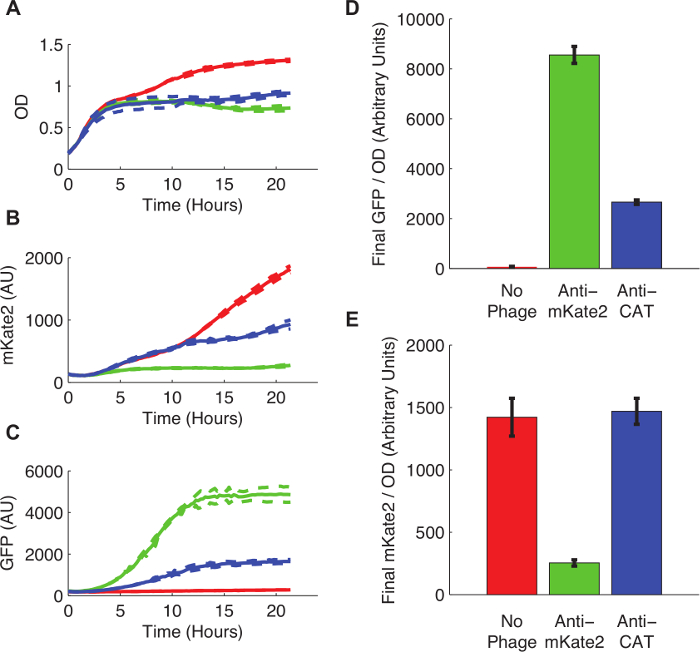
Figur 3: Knockdown af et kromosomalt integreret mKate2 Fluorescerende Reporter. E. coli MG1655 K12 udtrykker mKate2 var enten ubehandlet (røde linjer og barer), inficeret med en anti-mKate2 phagemid (grønne linjer og barer), eller inficeret med en kontrol phagemid målrettet CAT (blå linjer og barer). (A) Ubehandlet E. coli voksede til højere mætning tætheder, hvilket indikerer et metabolisk omkostning til faginfektion. Stiplede linjer angiver standardafvigelser af 3 replikater. (B) Den mKate2 signal blev reduceret til nær baggrundsniveauer i anti-mKate2 behandlet stamme, men ikke i kontrolstammer. (C) GFP-fluorescens, også båret af fagmidet, var påviselig kun i fagmid-behandlede kontroller. (D, E) Afsluttende fluorescensaflæsninger efter 24 timers vækst blev normaliseret til OD. GFP-signal, der angiver phagemid infektion, var fraværende i ubehandlede kontroller, men også væsentligt reduceret efter anti-CAT phagemid behandling. Dette kan indikere ikke-tilsigtede virkninger fagmidet. Den mKate2 signal blev reduceret med anti-mKate2 fagmid behandling sammenlignet med ubehandlede kontroller. CAT-målrettet kontrol phagemid viste ingen effekt på mKate2 fluorescens. Fejl søjler repræsenterer standardafvigelse på 3 gentagelser. Klik her for at se en større version af dette tal.
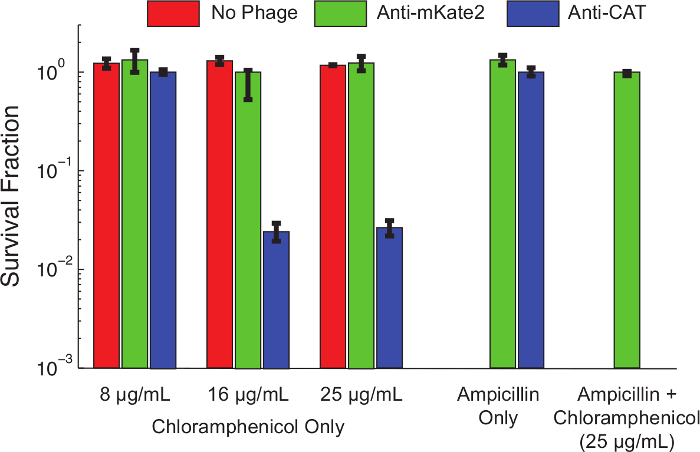
Figur 4: Knockdown af CAT Gendanner Chloramphenicol Følsomhed over for en genetisk Resistent Befolkning E.. coli MG1655 K12 udtrykker en kromosomalt integreret CAT-gen blev efterladt ubehandlet (røde søjler), behandlet med en kontrol phagemid målrettet mKate2 (grønne søjler), eller behandlet med en phagemid udtrykker en anti-CAT Srna (blå søjler). Efter 1 time for infektion, levedygtighed på den angivne myreibiotics blev vurderet ved serielle fortyndinger og plating. Stammer behandlet med anti-CAT fagmid blev signifikant dræbt (> 90%) ved chloramphenicol ved højere koncentrationer, mens kontrol behandlinger var upåvirket. Tilføjelse ampicillin til dyrkningspladerne positivt selekterer for fagmid infektion og eliminerer ikke-inficerede celler. Under disse betingelser blev der ikke chloramphenicolresistente kolonier observeret efter anti-CAT behandling. Dette indikerer, at de fleste overlevende repræsenterer et svigt af infektion, snarere end en fejl i nedregulering. Fejl søjler repræsenterer standardafvigelsen af 3 gentagelser. Klik her for at se en større version af dette tal.
| CAT målsekvensen | 5 '- ATGGAGAAAAAAATCACTGGATAT - 3' |
| CAT styresekvens | 5 '- ATATCCAGTGATTTTTTTCTCCAT - 3' |
Tabel 1:. Et eksempel TARGET og GUIDE Sekvens for CAT Gene Bemærk den omvendte komplement forhold.
| pR promotor | TAACACCGTGCGTGTTGACTATTTTACCTCTGGCGGTGATAATGGTTGC | ||||
| styresekvens | ATATCCAGTGATTTTTTTCTCCAT | ||||
| Hfq bindende domæne | TTTCTGTTGGGCCATTGCATTGCCACTGATTTTCCAACATATAAAAAGACAAGCCCGAACAGTCGTCCGGGCTTTTTT TCTCGAG | ||||
| T1 / TE terminator | CTCGAGCCAGGCATCAAATAAAACGAAAGGCTCAGTCGAAAGACTGGGCCTTTCGTTTTATCTGTTTTTGTCGGTGAA CGCTCTCTACTAGAGTCACACTGGCTCACCTTCGGGTGGGCCTTTCTGCGTTTATA | ||||
Tabel 2:. Sekvens Komponenter i Srna Cassette Hver sekvens er skrevet 5'-3 '. Den komplette kassette er sammenkædningen af disse 4 elementer i orden og omfatter 292 bp.
| Forward Primer | 5 '- CTGGCGGTGATAATGGTTGC [GUIDE] TTTCTGTTGGGCCATTGC - 3' |
| reverse primer | 5 '- GCAATGGCCCAACAGAAA [TARGET] GCAACCATTATCACCGCCAG - 3' |
Tabel 3: Primer Design at ændre en eksisterende føringselementer Den fremadrettede primer omfatter de sidste 20 bp af pR-promotoren, den nye GUIDE sekvens og de første 18 bp af Hfq-bindende domæne.. Målsekvensen er den nøjagtige omvendte komplement af GUIDE sekvens. Den reverse primer er den nøjagtige omvendte komplement af den fremadrettede primer.
Tabel 4: Foreslået Betingelser for Single-Primer Mutagen PCR.
| Trin | Temp | Tid |
| indledende denaturering | 98 ° C | 30 sek |
| 30 cykler | 98 ° C | 10 sek |
| 55 ° C | 30 sek | |
| 72 ° C | 120 sek | |
| Final Extension | 72 ° C | 300 sek |
| Opbevaring | 10 ° C |
Tabel 5: Foreslået Thermocycler Protokol til Single-Primer Mutagen PCR.
| Komponent | Bind |
| template-DNA | 1 pi |
| 10 pM Forward Primer | 0,5 pi |
| 10 uM revers primer | 0,5 pi |
| Taq 2X Master Mix | 25 pi |
| Nuclease-free water | 23 pi |
| totalvolumen | 50 pi |
Tabel 6: Foreslåede Betingelser for Sequence Verification PCR.
| Trin | Temp | Tid |
| indledende denaturering | 95 ° C | 30 sek |
| 30 cykler | 95 ° C | 30 sek |
| 55 ° C | 30 sek | |
| 68 ° C | 30 sek | |
| Final Extension | 68 ° C | 300 sek |
| Opbevaring | 10 ° C |
Tabel 7: Foreslået Thermocycler Protocol for sekvensverificering PCR.
Den foreliggende fremgangsmåde opnåede 80% reduktion i mKate fluorescensniveauer sammenlignet med ikke-målrettede kontroller. Dette er i overensstemmelse med andre RNA knockdown metoder, når fuldstændige lyddæmpning ikke overholdes, og 50-90% effektivitet er typisk 16,17. På den fænotypiske niveau, CAT målrettet knockdowns kunne væsentligt dæmpe chloramphenicol resistens, og fjerne det under visse betingelser.
Knockdown fænotype var detekterbar på populationsniveau efter kun få timer efter infektion (figur 3B). Dette illustrerer en vigtig egenskab af fag-baserede levering: en høj knockdown frekvens kan fås direkte i batch kultur uden forudgående genetisk modifikation. I modsætning til konventionelle genetiske modifikationer ved anvendelse af plasmid transformation eller genomisk integration, går faginfektion ikke kræve, at en population re-vokset fra en enkelt isoleret koloni. Dette tillader virkningerne af faginfektion at være udfored i populationer med komplekse rumlige dynamik 11, med præ-eksisterende rumlige strukturer som biofilm 18, eller i genetisk blandede naturlige populationer 19.
Et kritisk trin i denne metode er fremstilling af emballeret fagmid ved høj titer. Den metaboliske byrde forbundet med produktion fagpartikel kan medføre høje mutation eller plasmidtab i fagmidet produktionsstamme. Det anbefales, at fagmidet produktionsstamme dyrkes direkte fra en enkelt cotransformeret koloni og ikke kølet, frosset eller sub-dyrket før fag høst. En lav effektivitet i co-transformation kan også ses ved indførelsen fagmidet og hjælper plasmid til E. coli samtidigt. I dette tilfælde kan højere effektivitet opnås ved at transformere hjælpe-plasmid først, derefter fremstille kompetente celler, der bærer hjælpe-plasmid til efterfølgende transformation med fagmidet.
Phagemid infektion eller Srna udtryk pålægger også en påviselig metabolisk byrde på målcellerne, og kan resultere i nogle fænotypisk forstyrrelse. For eksempel blev et fald i mKate2 observerede fluorescens, selv når celler blev inficeret med en fagmid målretning CAT (figur 3). Infektion med M13 menes ikke at udløse systemiske stressreaktioner i E. coli 20, men kan ændre transskriptionelle mønstre indirekte. Alternativt kan GFP eller ampicillin resistens markører opført på fagmidet konkurrerer om cellulære ressourcer, reducere mKate2 udtryk og vækst 9. Endelig kan Srna kassette selv globalt ændrer genekspressionsprofiler ved titrering af Hfq proteinet, eller gennem off-target mRNA silencing. Forbi mål effekter er almindelige i in vivo-RNAi målrettet 21-23, men de har endnu ikke systematisk undersøgt for dette system.
En begrænsning ved denne metode er, at infektionen effvitet er mindre end 100%, så nogle ikke-inficerede bakterier at fortsætte i befolkningen. Resultaterne af dette arbejde og tidligere arbejde 10 tyder på, at inficerede celler udgør 1-10% af den endelige befolkning, og er ansvarlige for de fleste af de nonsilenced fænotyper observerede. En række veje til M13-resistens er kendte, med de mest almindelige er mutations tab af pilus-ekspression 24. I lyset af disse begrænsninger, bør kontrol bruges til at bekræfte høje smittespredningen og knockdown effektivitet.
En anden potentiel begrænsning for nogle anvendelser er lejlighedsvis overførsel af kontaminerende hjælperfag. Selvom M13K07 indeholder et muteret emballage signal, kan det pakkes i fag-capsidet ved lav frekvens og overført til inficerede populationer, hvilket resulterer i celler kompetente til fag produktion og den fortsatte spredning af fag ud over den oprindelige infektion begivenhed 25. Modifikationer af hjælperfag har vist sig effektiveat reducere uspecifik emballage, men nogle gange på bekostning af reduceret fag produktion 26.
Udviklet bakteriofager er blevet et uundværligt værktøj for E. coli syntetisk biologi, så hurtig levering af nye gener til en voksende befolkning. Nyligt arbejde har produceret intercellulære kommunikation kredsløb 11 eller udtrykt transkriptionsfaktorer til at undertrykke antibiotikaresistens veje 27. Protokollen præsenteres her føjer til en voksende samling af værktøjer, der giver kontrol af bakteriel fysiologi gennem programmerede RNA. CRISPR-Cas nukleaser, når muteret for at eliminere nukleaseaktivitet, har vist sig at undertrykke transkription ved RNA-styrede genmål 17,28. I modsætning hertil Srna nedregulering virker på det translationelle niveau og kræver ikke eksogent protein ekspression. Næste generation af bioteknologi kan forene transkriptionel og translationel kontrol med fag-medieret levering at programmere komplekse phenotyper i realtid.
The authors have nothing to disclose.
Finansieringen af dette arbejde blev leveret af Fondation Bettencourt Schueller til støtte for Paris Bettencourt iGEM hold. Vi takker INSERM U1001 forskningsenheden og Chantal Lotton til teknisk bistand og rådgivning. Phagemid Litmus28i_J23115-B0032-GFP blev leveret af Monica Ortiz og Drew Endy fra Stanford.
| Name | Company | Catalog Number | Comments |
| Plasmid Miniprep Kit | Qiagen | 27104 | |
| DpnI Enzyme | NEB | R0176S | |
| Phusion High Fidelity Polymerase | NEB | M0530S | |
| Taq 2x Master Mix | NEB | M0270L | |
| M13KO7 Helper Phage | NEB | N0315S | |
| DH5α Competent Cells | Life Technologies | 18265-017 | |
| TOP10F' Cells | Life Technologies | C3030-03 | |
| LB Broth | Sigma | L3022-250G | |
| Ampicillin | Sigma | A9393-5G | |
| Kanamycin | Sigma | 60615-5G | |
| Chloramphenicol | Sigma | C0378-5G | |
| Tetracycline | Sigma | 87128-25G |
- Ohkumo, T., Masutani, C., Eki, T., Hanaoka, F. Use of RNAi in C. elegans. RNAi. , 129-137 (2008).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Iordanou, E., Chandran, R. R., Blackstone, N., Jiang, L. RNAi interference by dsRNA injection into Drosophila embryos. J Vis Exp. (50), e2477 (2011).
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2 (9), 2245-2264 (2007).
- Tsong, T. Y. Electroporation of cell membranes. Biophys J. 60 (2), 297-306 (1991).
- Kim, W. J., Chang, C. -. W., Lee, M., Kim, S. W. Efficient siRNA delivery using water soluble lipopolymer for anti-angiogenic gene therapy. J Control Release. 118 (3), 357-363 (2007).
- Shi, Y. Mammalian RNAi for the masses. Trends Genet. 19 (1), 9-12 (2003).
- Na, D., Yoo, S. M., Chung, H., Park, H., Park, J. H., Lee, S. Y. Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol. 31 (2), 170-174 (2013).
- Ceroni, F., Algar, R., Stan, G. -. B., Ellis, T. Quantifying cellular capacity identifies gene expression designs with reduced burden. Nat Methods. 12 (5), 415-418 (2015).
- Libis, V. K., Bernheim, A. G., et al. Silencing of Antibiotic Resistance in E. coli with Engineered Phage Bearing Small Regulatory RNAs. ACS Synth Biol. 3 (12), 1003-1006 (2014).
- Ortiz, M. E., Endy, D. Engineered cell-cell communication via DNA messaging. J Biol Eng. 6 (1), 16 (2012).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnol. 9 (1), 61 (2009).
- Chung, C. T., Miller, R. H. Preparation and storage of competent Escherichia coli cells. Recombinant DNA Part I. , 621-627 (1993).
- Phornphisutthimas, S., Thamchaipenet, A., Panijpan, B. Conjugation in Escherichia coli. Biochem Mol Biol Educ. 35 (6), 440-445 (2007).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli Physiology in Luria-Bertani Broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Mittal, V. Improving the efficiency of RNA interference in mammals. Nat Rev Genet. 5 (5), 355-365 (2004).
- Qi, L. S., Larson, M. H., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Lu, T. K., Collins, J. J. Dispersing biofilms with engineered enzymatic bacteriophage. Proc Natl Acad Sci USA. 104 (27), 11197-11202 (2007).
- Yosef, I., Manor, M., Kiro, R., Qimron, U. Temperate and lytic bacteriophages programmed to sensitize and kill antibiotic-resistant bacteria. Proc Natl Acad Sci USA. 112 (23), 7267-7272 (2015).
- Karlsson, F., Malmborg-Hager, A. -. C., Albrekt, A. -. S., Borrebaeck, C. A. K. Genome-wide comparison of phage M13-infected vs. uninfected Escherichia coli. Can J Microbiol. 51 (1), 29-35 (2005).
- Senthil-Kumar, M., Mysore, K. S. Caveat of RNAi in plants: the off-target effect. Methods in molecular biology. 744, 13-25 (2011).
- Jackson, A. L., Linsley, P. S. Recognizing and avoiding siRNA off-target effects for target identification and therapeutic application. Nat Rev Drug Discov. 9 (1), 57-67 (2010).
- Cho, S. W., Kim, S., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Hagens, S., Blasi, U. Genetically modified filamentous phage as bactericidal agents: a pilot study. Lett Appl Microbiol. 37 (4), 318-323 (2003).
- Kasman, L. M., Kasman, A., Westwater, C., Dolan, J., Schmidt, M. G., Norris, J. S. Overcoming the phage replication threshold: a mathematical model with implications for phage therapy. J Virol. 76 (11), 5557-5564 (2002).
- Chasteen, L., Ayriss, J., Pavlik, P., Bradbury, A. R. M. Eliminating helper phage from phage display. Nucleic Acids Res. 34 (21), e145 (2006).
- Lu, T. K., Collins, J. J. Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy. Proc Natl Acad Sci USA. 106 (12), 4629-4634 (2009).
- Bikard, D., Jiang, W., Samai, P., Hochschild, A., Zhang, F., Marraffini, L. A. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. Nucleic Acids Res. 41 (15), 7429-7437 (2013).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved