Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Biology
Fag-mediert Levering av målrettet Srna Konstruerer å slå ned Gene Expression in
We describe a method to knock down gene expression in a growing population of E. coli cells using sequence-targeted sRNA expression cassettes delivered by an M13 phagemid vector.
RNA-mediert knock-out er mye brukt til å kontrollere genekspresjon. Denne allsidige familien av teknikker som gjør bruk av korte RNA (Srna) som kan syntetiseres med en hvilken som helst sekvens og er utformet for å utfylle eventuelle gen målrettet for demping. Fordi Srna-konstruksjonene kan innføres i mange celletyper, enten direkte eller ved hjelp av en rekke forskjellige vektorer, kan genekspresjon bli undertrykt i levende celler, uten arbeidskrevende genetisk modifisering. Den vanligste RNA knockdown teknologi, RNA interferens (RNAi), gjør bruk av det endogene RNA-indusert stanse kompleks (RISC) for å formidle sekvens gjenkjenning og spalting av mål-mRNA. Anvendelser av denne teknikken er derfor begrenset til RISC-uttrykker organismer, primært eukaryoter. Nylig har en ny generasjon av RNA biotechnologists utviklet alternative mekanismer for å kontrollere genekspresjon gjennom RNA, og så gjort mulige RNA-mediert genet knockdowns i bakterier. Her beskriver vi en metode for å kneble genet expressjon i E. coli som funksjonelt ligner RNAi. I dette system et syntetisk fagemid er utformet for å uttrykke Srna, som kan designet for å målrette en hvilken som helst rekkefølge. Uttrykket konstruksjon blir levert til en populasjon av E. coli-celler med ikke-lytisk M13-fag, hvoretter den er i stand til å stabilt replikerer som et plasmid. Antisense anerkjennelse og stanse av målet mRNA er mediert av Hfq protein, endogen til E. coli. Denne protokollen omfatter metoder for å designe den anti Srna, konstruere phagemidvektor, emballasje fagmidet inn M13 bakteriofag, forbereder en levende celle befolkning for infeksjon, og utfører infeksjonen selv. Den fluorescerende protein mKate2 og antibiotikaresistens genet kloramfenikol acetyltransferase (CAT) er målrettet for å generere representative data og å kvantifisere knockdown effektivitet.
RNA-mediert genet knockdowns fortsette i to etapper. Først blir et RNA-molekyl innføres i en cellelinje eller organisme for undersøkelsen. For det andre endogene RNA-bindende proteiner lette RNA-målet anerkjennelse og produsere lyddemping effekt. Alle RNA knockdown teknologier dra nytte av den tilpasses naturen av syntetiske sRNAs, som lett kan produseres for å matche et bestemt mål av interesse. Men de molekylære detaljene i RNA opptak og lyddemping varierer mye på tvers av modellsystemet, begrensende hvor og hvordan RNA knockdowns kan brukes.
I nematoder, dobbel-strandet RNA (dsrna) molekyler kan innføres direkte i media eller ved å mate ormer med en befolkning på dsRNA-uttrykke E. coli-celler 1,2. I Drosophila, kan RNAi oppnås ved microinjecting embryoer med dsRNA 3, eller implementert i cellelinjer ved å legge dsRNA til kulturmediet 4. I pattedyrcellelinjer,syntetiske små interfererende RNA (sirnas) kan leveres til levende celler ved elektroporering 1,2,5, pakket i liposomer 3,6, eller uttrykt fra DNA plasmidvektorer 4,7. Når RNA arter når cytosol, baserer RNAi pathway på RISC-komplekset for å behandle dsRNA, lette antisense gjenkjennelse av målet, og katalysere translasjons-undertrykkelse, mRNA degradering, eller heterochromatin formasjon, avhengig av verten.
På grunn av disse kravene, kan klassisk RNAi kun utføres i organismer som tar opp eksogene RNA effektivt og uttrykker RISC eller en RISC-lignende aktivitet. Spesielt, utelukker denne modellen bakterien E. coli, som mangler den RNAi pathway. Men nyere fremskritt innen syntetisk biologi gir deg verktøyene for å løse både leveranseproblem og lyddemping problem.
I denne protokollen, Srna konstruerer uttrykt i E. coli fra en DNA-vektor levert til litreråvare celler ved hjelp av M13-fagmid / hjelpesystemet. En fagmid er enhver plasmid med et fag-avledet f1 replikasjons. En hjelper plasmid, i dette tilfellet M13KO, bærer alle maskiner som kreves for å fremstille viruspartikler, men er ikke i seg selv kompetent for replikasjon og pakking. Når en fagmid og hjelper plasmid er co-transformert, er fagmidet alene replikert på f1 opprinnelse, pakket og utskilt. Den vektorisert fagmid er så kompetent til å infisere levende E. coli via F pilus.
I dette system blir den stanse effekten som produseres av tilpassede Srna kassetter som kombinerer et stillas sekvens med en målbindende sekvens. Den målbindende sekvens er 24 basepar antisens til en mRNA-mål, typisk ved ribosom-bindingssetet (RBS). Stillaset sekvens, utviklet av Na og kolleger 8, inneholder en Hfq-bindende motiv hentet fra MICC, en liten regulatoriske RNA endogen til E. coli. Den Hfq protein stimulerer RNA-RNA binding og mRNA degradering, tjener en rolle i dette system som ligner på RISC i RNAi. Figur 1 viser et komplett skjema for fag-mediert Srna knock-out, herunder Srna kassettkonstruksjonen, fagmid vektorisering, og tie mekanisme.
Som en metode til å modulere genekspresjon i E. coli, Srna stanse er enkel, rask og allsidig. Den målrettede E. coli er ikke tynget utover spre fagmidet og uttrykker Srna. Dette kan være aktuelt i forbindelse med syntetisk biologi eller grunnforskning der uttrykk for større heterologe konstruksjoner kan stamme cellulære ressurser 9. Fagmider med nye mål kan fremstilles med en enkelt PCR og høstet en dag etter fagemid transformasjon. Endelig kan nesten enhver mRNA være målrettet. Den Srna regulering kassett (på en standard plasmid) har vist seg å fungere på en rekke mål i stoffskiftet med typiske undertrykkelse nivåer> 90% 8.
10. For det første er en pakket fagemid innført i en satsvis kultur av E. coli-celler og brukes til å slå ekspresjon av det fluorescerende protein mKate2. Påfølgende fluorescens-forandringer blir målt i sann tid. For det andre, banket ned CAT genet er vist å redusere fenotypisk kloramfenikol motstand på agarskåler. I begge tilfeller fagmidet selv bærer en GFP markør, slik at infeksjonsrate som skal måles, uavhengig av knockdown effektivitet.
1. Design og konstruksjon av fagmidekspresjonsvektoren vektorer Peiling Srna lyddemping Kassetter
- De Novo Design av Srna Lyddempningsplater Kassetter 8
- Identifisere den fullstendige sekvensen av mRNA for å slås ved hjelp av en DNA-sekvens-database. For å generere TARGET sekvensen kan du første 24 bp av kodende sekvens, fra posisjon 1-24 begynner med startkodonet (f.eks ATG).
Merk: Dempe er mindre effektiv når andre nettsteder eller deler av mRNA er målrettet 8. - Ta det motsatte komplementet av targetsekvensen for å produsere den GUIDE sekvensen for Srna kassetten. Se tabell 1 for eksempler på målet og GUIDE sekvenser for kloramfenikol acetyltransferase (CAT).
- Å designe den 292 bp komplett Srna uttrykk kassett, ordne pR promoteren GUIDE sekvens, Hfq proteinbinding domene og T1 / TE transkripsjonsterminatorsekvenser i serien (tabell 2).
- Legg til flere kloningsseter av valget for å lette kloning av Srna kassetten inn i målet vektor.
- Skaff komplett Srna kassetten gjennom kommersiell genet syntese eller en lignende metode og klone den inn i noen phagemidvektor med en funksjonell f1 replika 11. Se Hjelpemiddel Informasjon for den komplette sekvens av det endelige fagemid vektor.
- Identifisere den fullstendige sekvensen av mRNA for å slås ved hjelp av en DNA-sekvens-database. For å generere TARGET sekvensen kan du første 24 bp av kodende sekvens, fra posisjon 1-24 begynner med startkodonet (f.eks ATG).
- Endring av Sequence Målrettet av en eksisterende Srna uttrykk kassett ved hjelp av PCR-basert nettsted mutagenese 12
- Identifiser 24 bp GUIDE sekvens i en eksisterende Srna uttrykk kassett. Merk: kommentert pAB.001 plasmid, som brukes i dette arbeidet, er tilgjengelig som en supplerende sekvens fil.
- Design forover og bakover primere med korte regioner av homologi til eksisterende Srna kassetten flankerer den nye 24 bp GUIDE sekvens. Skaff primere gjennom kommersielle oligonukleotid-syntese.
Merk: Primer design for seterettet mutagenese er depicted i figur 2. Med den nøyaktige primer-templat-sekvenser er gitt i tabell 3. - Forbered en 5 ml kultur av E. coli bærer malen Srna uttrykk fagmidekspresjonsvektoren. Dyrke cellene over natten ved 37 ° C med risting, i LB-medium med egnede antibiotika.
- Ekstrahere og rense malen Srna uttrykket fagmid fra 5 ml bakteriekultur ved anvendelse av en DNA-miniprep-kit eller lignende metode 12.
- Forbered to PCR reaksjoner etter mønster Srna uttrykk fagmid og high fidelity polymerase, ett med forover og ett med revers primer (tabell 4). Bruke PCR-betingelsene som er anbefalt av leverandøren polymerase (tabell 5). Øke malen konsentrasjon til 10-50x høyere enn en standard reaksjon for å ta hensyn til det faktum at den eneste primeren reaksjonen ikke produserer eksponensiell amplifikasjon.
- Kombinere de to ovennevnte PCR-reaksjonene i et mikrosentrifugerør. Anneal produktene ved oppvarming til 98 ° C i et kokende vannbad. Umiddelbart etter å ha plassert mikrosentrifugerør i vannbadet, fjern varmekilden og la badet til sakte vende tilbake til romtemperatur i løpet av 1-2 timer.
- For å eliminere ikke-muterte malen Srna uttrykk fagmidekspresjonsvektoren, tilsett 1 mL. DpnI restriksjonsenzym til blandingen og inkuberes ved 37 ° C i 1 time, eller den tid som anbefales av produsenten for fullstendig nedbrytning.
Merk: DpnI fordøyer bare denaturert målse, som er til stede på verts replikert fagmider men ikke PCR-produkter. - Transform kjøpt eller forberedt 13 kjemisk kompetente E. coli med 1-5 ul av herdet PCR-produktet. Isolere enkelte kolonier av den transformerte stammen ved selektiv plettering på LB-agarplater inneholdende egnede antibiotika.
- For å verifisere inkorporering av riktig GUIDE sekvens, screene de resulterende kolonier ved koloni PCR. Ved hjelp av en 200 mL pipette, collect en liten mengde av celler fra en enkelt transformert koloni. Mark og bevare den opprinnelige kolonien for nedstrøms bruk etter verifisering.
- Tilsett oppsamlede celler til 50 ul av nuklease-fritt vann i et mikrosentrifugerør. Bland ved å pipettere opp og ned.
- Ved hjelp av en benk-topp thermocycler eller et kokende vannbad, lysere cellene ved oppvarming til 95 ° C i 2 min.
- PCR-amplifisere den fagmid region ved hjelp av 1 ul varme lyserte celler som et DNA-templat. PCR-betingelser og termo protokoller er gitt i tabellene 6 og 7. Se den supplerende pAB.001 sekvens fil for verifisering primer sekvenser.
- Sekvensere PCR-produktet for å kontrollere inkorporering av den korrekte GUIDE sekvens.
- Inokulere en 5 ml kultur av E. coli klone bærer sekvensen-verifisert Srna uttrykk fagmidekspresjonsvektoren. Dyrke cellene over natten ved 37 ° C med risting, i selektive LB-medium.
- Forbered en glyserol lagerav sekvensen-verifisert klone. Legg 750 ul av over natten-kulturen til 250 ul av 60% glycerol i en skrukork cryo rør.
- Oppbevar glyserol lager ved -80 ° C på ubestemt tid. Resten av over natten-kulturen kan brukes som en kilde for den Srna ekspresjon fagmid i trinn to.
2. Produksjon og Harvest of M13-pakket fagmidekspresjonsvektoren Stocks
- Forbered en 5 ml kultur av E. coli bærer Srna uttrykk fagmidekspresjonsvektoren. Dyrke cellene over natten ved 37 ° C med risting, i LB-medium med egnede antibiotika. Merk: Srna, Uttrykket fagemid kan oppnås gjennom de novo kloning som beskrevet i trinn 1.1.5, eller modifisert fra en eksisterende fagmid og høstet i trinn 1.2.16.
- Tilsvar forberede en 5 ml kultur av E. coli bærer M13KO7 helper plasmid. Dyrke cellene over natten ved 37 ° C med risting, i selektive LB-medium.
- Pakk og rense Srna expression fagmid og helper plasmid ved hjelp av en DNA-ekstraksjon kit eller lignende metode 12.
- Cotransform kjøpt eller forberedt 13 kjemisk kompetente E. coli med 1 mL hver av Srna uttrykk fagmid og helper plasmid. Velg for cotransformants ved utsåing på LB agar med selektive antibiotika for begge konstruksjoner.
- Forbered en 10 ml kultur fra en enkelt koloni av kotransformert belastningen i LB med selektive antibiotika. Inkuber ved 37 ° C med risting i 8-12 timer eller over natten.
- Sentrifuger kulturen på 3300 xg i 10 min. Samle supernatanten og filtrer gjennom et 0,2 um filter. Forsiktig: Ved media søl, rengjøre området med fortynnet blekemiddel (0,5%) for å ødelegge smitte fagpartikler.
- Oppbevar det innpakkede fagmidekspresjonsvektoren filtratet ved 4 ° C. Merk: Prøvene kan opprettholdes i flere dager til uker uten tap av aktivitet.
3. Utarbeidelse av F + Target Cells for Dempe
- Finn ut om cellene til å være målrettet for å kneble uttrykke F pilus 14. Hvis F pilus er allerede til stede, går du videre til trinn 4.
Merk: Vanlige laboratoriestammer av E. coli er merket som + F eller F 'for å indikere nærværet av F-pilus i deres genom eller på et plasmid. - Skaff en F + stamme av E. coli som TOP10F '.
Merk: Sørg for at målet belastning bærer en unik motstand markør for å bli skilt fra F-plasmid donor etter konjugering. - Å introdusere F pilus ved konjugering med en F + belastning, forberede 5 ml kulturer av både målet belastning og F-plasmid donor 14. Dyrke cellene over natten ved 37 ° C med risting, i LB-medium med egnede antibiotika.
- Den følgende dag, fortynne begge stammer 1: 100 i 5 ml LB og selektiv fortsette dyrkning ved 37 ° C med risting.
- Bestemme vekstfasen av cellene ved å måle optical tetthet av kulturen ved 600 nm (OD 600) ved bruk av en benkeplaten spektrofotometer. Culture cellene i omtrent 2 timer inntil en OD 600 på 0,3 er oppnådd, noe som viser log-fase vekst 15.
- Forbered tre konjugeringsreaksjoner i Mikrosentrifugerør: 0,5 ml F-plasmid donor + 0,5 ml mål belastning, 0,5 ml F-plasmid donor + 0,5 ml LB media (negativ kontroll) og 0,5 ml mål belastning + 0,5 ml LB media (negativ kontroll). Tillat konjugering til å fortsette i 2 timer ved 37 ° C med risting.
- Plate 100 mL av hver konjugeringsreaksjon på selektiv LB agar med antibiotika som er spesifikke for F-plasmid (typisk tetracyclin) og målet belastning. Plate de negative kontroll reaksjoner for å bekrefte at verken donor eller mottaker belastning uttrykke både antibiotika motstander.
4. Infeksjon med Pakket Fagemider for Dempe
- Inokulere en enkelt koloni av F + målceller i en 5 ml kultur av LB medien med passende antibiotika. Inkuber over natten ved 37 ° C med risting.
- Den følgende dag, fortynnes F + målcellene 1: 100 i 5 ml LB-selektive media og fortsette til kultur ved 37 ° C med risting.
- Bestemme vekstfasen av cellene ved å måle den optiske tetthet av kulturen ved 600 nm (OD 600) ved bruk av en benkeplaten spektrofotometer. Culture cellene i omtrent 2 timer inntil en OD 600 på 0,3 er oppnådd, noe som viser log-fase vekst 15. Merk: Uttrykk for F pilus og infeksjon effektivitet er høyest i log fase.
- Tilsett M13-pakkede fagmider (fra trinn 2.6) til målcellene i et volumforhold på 1: 100 for å oppnå nesten 99% infeksjon av målgruppen. Tillat infeksjonen forløpe ved 37 ° C med risting i 30-60 min.
- Analysere de Srna-støydempere fenotype i henhold til metoden for valg.
Merk: For en fluoriserende protein mål kan stanse effekt kvantifiseresdirekte av fluorometri 10. Alternativt kan fenotypiske analyser brukes til å observere de fenotypiske konsekvensene av genet knockdown åtte. - Forbered en glyserol lager for Srna-uttrykke fagmidekspresjonsvektoren vert følgende trinn 1.2.14-1.2.16. Merk: fagmidet vil forplante seg i vertsstammen på ubestemt tid, og kan opprettholdes med antibiotika som ligner på en konvensjonell plasmid.
Stanse av mKate2 fluorescens i flytende medier
Figur 1 viser ordningen for Srna-medierte knockdowns som er beskrevet i dette arbeidet, blant annet Srna kassett design, fagmid vektorisering, og stillemekanisme. Følgende protokoll 1.2 ble Srna stanse kassetten fra plasmid pAB.001 endret for å målrette mKate. Den Srna kassett syntetisert og klonet inn fagmidekspresjonsvektoren Litmus28i_J23115-B0032-GFP, en gave fra Monica Ortiz og Drew Endy 11. Dette fagmid bærer markører for GFP uttrykk og kanamycin resistens, slik at vellykkede infeksjoner spores. Pakket fagmidpartikler aksjer ble utarbeidet følgende protokoll 2.
Et derivat av E. coli K12 MG1655 bærer en konstitutivt uttrykt, kromosomer integrert mKate2 markør ble forberedt for fag infeksjon ved konjugering med en F-plasmid donorstamme som følgende protokoll 3. Cellene ble dyrket til middels logaritmisk fase og fagmidet innført følgende protokoll 4. Etter fagmid infeksjon, ble 200 ul kulturene overført til en fluorescens plateleser og fluorescens ble overvåket kontinuerlig i 24 timer.
Figur 3 viser effekten av Srna-mediert stanse på mKate2 uttrykk. Belastningen infisert med en anti-mKate2 fagmidekspresjonsvektoren viste ingen påvisbare mKate2 fluorescens enn bakgrunnen. I motsetning til dette, denne stammen gjorde uttrykker GFP markør, noe som indikerer vellykket opptak av fagmidet. Uinfiserte kontrollceller produsert mKate2 fluorescens men ikke GFP. En ytterligere kontroll, hvor den anti-mKate2 rettet mot domenet ble erstattet med en sekvens rettet mot CAT, hadde ingen effekt på mKate2 fluorescens.
Dempe av Kloramfenikol Resistance på agarskåler
innhold "fo: keep-together.within-page =" 1 "> Etter protokoll 1.1, en Srna stanse kassett ble produsert for å målrette CAT Et derivat av E. coli K12 MG1655 bærer en konstitutivt uttrykt, kromosomer integrert CAT markør var forberedt på. fag-infeksjon ved konjugering med et F-plasmid donorstamme som følgende protokoll 3. cellene ble dyrket til middels logaritmisk fase og fagmidet innført følgende protokoll 4. etter 1 time med inkubering ved 37 ° C, ble infiserte celler seriefortynnet og plettert ved en spekter av kloramfenikol konsentrasjoner. Platene ble inkubert over natten, og andelen av resistente celler ved hvert kloramfenikol-konsentrasjon ble bestemt ved telling av kolonidannende enheter (CFU) den neste dag.Figur 4 viser effekten av Srna-mediert stanse på kloramfenikol motstand fenotype. Ikke-infiserte celler eller celler infisert med fagmid målretting mKate2, var resistente overforkloramfenikol ved alle konsentrasjoner som ble testet. I kontrast, celler infisert med fagmid rettet mot CAT viste redusert overlevelse ved lave konsentrasjoner kloramfenikol, og nesten 99% drept ved høyere konsentrasjoner. Tilsetningen av ampicillin, for å velge kun bakterier som bærer fagmidet, redusert kloramfenikol overlevelse til ikke-detekterbare nivåer. Dette er i tråd med tidligere arbeider som viser at flukt fra fag infeksjon er en vanlig rute for å unnslippe silencing 10.
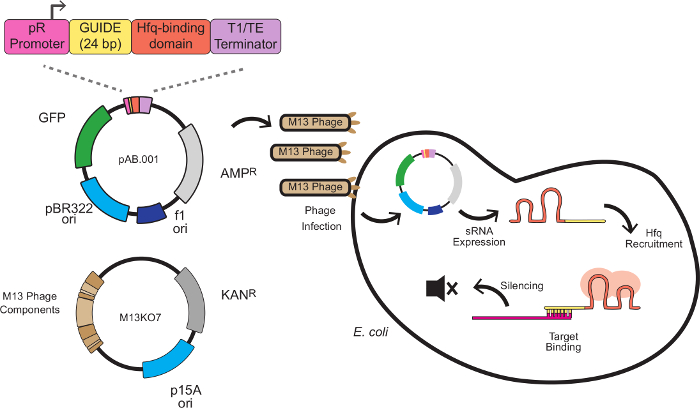
Figur 1: genet Slå i E. coli med Srna Expression Kassetter Levert av M13 Fag Den Srna Kassetten består av 4 moduler:. PR promoter (et konstituerende promoter avledet fra bakteriofag lambda), en 24 bp rettet mot domene, en Hfq bindende domene hentet fra MICC og en transkripsjonen terminator 8 E. coli uttrykker F pilus, hvor Srna uttrykk begynner. Den Srna da rekrutterer Hfq protein (avbildet i rødt) og binder en antisense mRNA mål nær ribosombindingssete, noe som resulterer i translasjonell undertrykkelse og mRNA degradering. Klikk her for å se en større versjon av dette tallet.
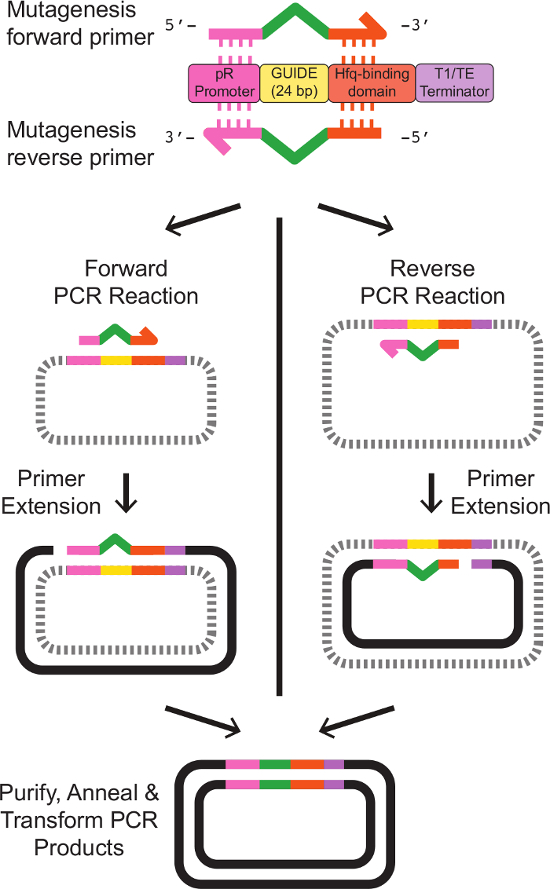
Figur 2:. Primer Design og seterettet mutagenese av Srna Target nettstedet To primere er designet med delvis homologi til en eksisterende Srna kassett. Foroverprimeren inneholder 20 bp homolog med pR promoteren i 5'-enden, etterfulgt av24 bp som representerer den nye GUIDE sekvens, deretter 18 bp homolog med Hfq-bindende domene ved 3'-enden. Det motsatte primer er nøyaktig motsatt komplementet av fremover primer, med regioner med homologi til eksisterende Srna kassett flankerer revers komplement av den nye GUIDE sekvens. Eksakte primersekvenser er gitt i tabell 3. Separate enkelt-primer PCR-reaksjoner med forover og revers primere produserer de lineære, enkeltrådet DNA med den ønskede modifiserte sekvens. Gløde forover og bakover reaksjonsprodukter, etter opprydding som beskrevet i protokollen, resulterer i dobbelt-trådet plasmid DNA bærer den ønskede modifiserte Srna kassett. Klikk her for å se en større versjon av dette tallet.
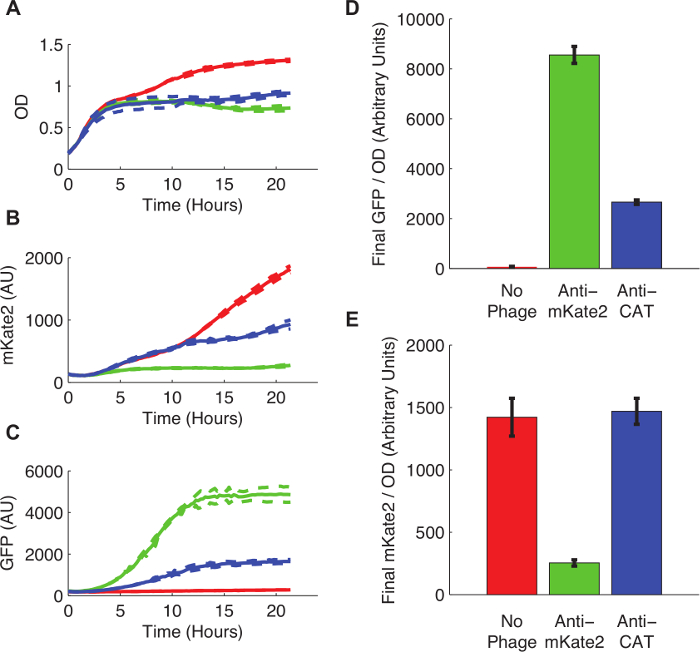
Figur 3: Knockdown av en kromosomer Integrert mKate2 Fluorescent Reporter. E. coli MG1655 K12 uttrykker mKate2 var enten ubehandlet (røde linjer og barer), infisert med en anti-mKate2 fagmidekspresjonsvektoren (grønne linjer og barer), eller infisert med en kontroll fagmidekspresjonsvektoren rettet mot CAT (blå linjer og barer). (A) Ubehandlet E. coli vokste til høyere metningstetthet, noe som indikerer en metabolsk kostnad for fag infeksjon. Stiplede linjer viser standardavvik av 3 gjentak. (B) Den mKate2 signal ble redusert til nær bakgrunnsnivået i anti-mKate2 behandlede stamme, men ikke i kontrollstammer. (C) GFP fluorescens, også båret av fagmidet, påvises bare i fagmidpartikler-behandlede kontroller. (D, E) Endelige fluorescens målinger etter 24 timers vekst ble normalisert til OD. GFP signal, som indikerer fagemid infeksjon, var fraværende i ubehandlede kontroller, men også vesentlig redusert som følge av anti-CAT phagemid behandling. Dette kan tyde på off-target effekter av fagmidet. Den mKate2 signal ble redusert ved hjelp av anti-mKate2 fagmid behandling sammenlignet med ubehandlede kontroller. CAT-målrettet kontroll fagmidekspresjonsvektoren viste ingen effekt på mKate2 fluorescens. Feilfelt representerer standardavvik av 3 replikater. Klikk her for å se en større versjon av dette tallet.
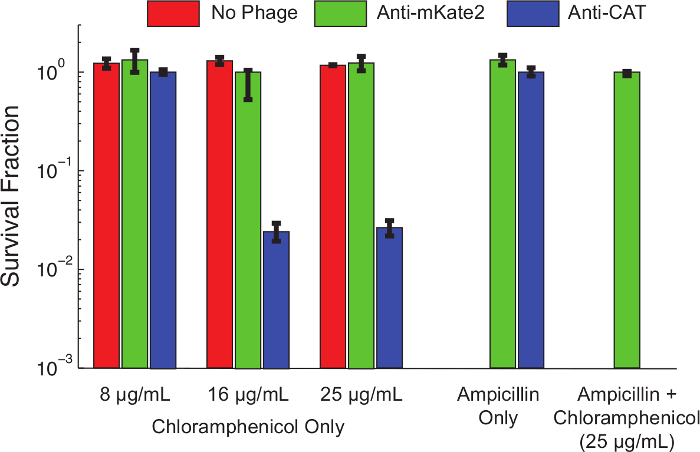
Figur 4: knockdown av CAT Gjenoppretter Kloramfenikol Følsomhet for en genetisk Resistent Befolkning E.. coli MG1655 K12 uttrykker en kromosomer integrert CAT genet ble etterlatt ubehandlet (røde søyler), behandlet med en kontroll fagmidekspresjonsvektoren målretting mKate2 (grønne søyler) eller behandlet med en fagmidekspresjonsvektoren uttrykker en anti-CAT Srna (blå søyler). Etter en time for infeksjon, levedyktighet på den indikerte mauribiotics ble bestemt ved seriefortynning og platekledning. Stammer som ble behandlet med anti-CAT fagmid ble signifikant drept (> 90%) av kloramfenikol ved høyere konsentrasjoner, mens kontrollbehandlinger var upåvirket. Legge ampicillin til kulturplater positivt velger for fagmid infeksjon og fjerner infiserte celler. Under disse betingelsene ble det ikke kloramfenikol-resistente kolonier ble observert etter anti-CAT behandling. Dette indikerer at de fleste overlevende representerer en svikt av infeksjon, snarere enn en svikt av lyddemping. Feilfelt representerer standardavviket av 3 replikater. Klikk her for å se en større versjon av dette tallet.
| CAT TARGET sekvens | 5 '- ATGGAGAAAAAAATCACTGGATAT - 3' |
| CAT GUIDE sekvens | 5 '- ATATCCAGTGATTTTTTTCTCCAT - 3' |
Tabell 1:. Et eksempel TARGET og GUIDE rekkefølge for CAT Gene Merk omvendt komplement forholdet.
| pR promoter | TAACACCGTGCGTGTTGACTATTTTACCTCTGGCGGTGATAATGGTTGC | ||||
| GUIDE sekvens | ATATCCAGTGATTTTTTTCTCCAT | ||||
| Hfq bindende domene | TTTCTGTTGGGCCATTGCATTGCCACTGATTTTCCAACATATAAAAAGACAAGCCCGAACAGTCGTCCGGGCTTTTTT TCTCGAG | ||||
| T1 / TE terminator | CTCGAGCCAGGCATCAAATAAAACGAAAGGCTCAGTCGAAAGACTGGGCCTTTCGTTTTATCTGTTTTTGTCGGTGAA CGCTCTCTACTAGAGTCACACTGGCTCACCTTCGGGTGGGCCTTTCTGCGTTTATA | ||||
Tabell 2:. Sequence Komponenter av Srna Cassette Hver sekvens er skrevet 5'-3 '. Den komplette kassett er sammensetning av disse 4 elementene i orden og består av 292 bp.
| Forward Primer | 5 '- CTGGCGGTGATAATGGTTGC [GUIDE] TTTCTGTTGGGCCATTGC - 3' |
| omvendt Primer | 5 '- GCAATGGCCCAACAGAAA [TARGET] GCAACCATTATCACCGCCAG - 3' |
Tabell 3: Primer Design Alter Eksisterende GUIDE Elements Termin primer inneholder siste 20 bp av PR arrangøren, den nye GUIDE sekvens, og den første 18 bp av Hfq-bindende domene.. Målsekvensen er nøyaktig motsatt komplementet av GUIDE sekvens. Den reverse primeren er nøyaktig omvendte komplement av den fremre primer.
Tabell 4: Forslag Vilkår for Single-Primer Mutagent PCR.
| Skritt | Temp | Tid |
| initial denaturering | 98 ° C | 30 sek |
| 30 sykluser | 98 ° C | 10 sek |
| 55 ° C | 30 sek | |
| 72 ° C | 120 sek | |
| endelig Extension | 72 ° C | 300 sek |
| Lagring | 10 ° C |
Tabell 5: Forslag Thermocycler Protokoll for Single-Primer Mutagent PCR.
| Komponent | Volum |
| mal DNA | 1 mL |
| 10 mm Forward Primer | 0,5 ul |
| 10 mm Reverse Primer | 0,5 ul |
| Taq 2X Master Mix | 25 pl |
| Nukleasefritt water | 23 mL |
| totalt volum | 50 pl |
Tabell 6: Forslag Vilkår for Sequence Verification PCR.
| Skritt | Temp | Tid |
| initial denaturering | 95 ° C | 30 sek |
| 30 sykluser | 95 ° C | 30 sek |
| 55 ° C | 30 sek | |
| 68 ° C | 30 sek | |
| endelig Extension | 68 ° C | 300 sek |
| Lagring | 10 ° C |
Tabell 7: Forslag Thermocycler Proprotokollen for Sequence Verification PCR.
Den foreliggende fremgangsmåte oppnås 80% reduksjon i mKate fluorescens-nivåer sammenlignet med vilkårlige kontroller. Dette er i tråd med andre RNA knockdown metoder, hvor fullstendig taushet ikke er observert, og 50-90% effektivitet er typisk 16,17. På den fenotypiske nivå, CAT-målrettet knockdowns var i stand til betydelig dempe kloramfenikol motstand, og eliminere det under visse forhold.
Knockdown fenotype var påviselig på befolkningsnivå etter bare noen få timer etter infeksjon (figur 3B). Dette illustrerer et viktig trekk av fag basert levering: en høy knockdown frekvens kan oppnås direkte i batch-kultur uten forutgående genetisk modifisering. I motsetning til konvensjonelle genetiske modifikasjoner ved hjelp av plasmid transformasjon eller genomisk integrasjon, betyr fag-infeksjon ikke krever at en populasjon på nytt dyrket fra en enkelt isolert koloni. Dette tillater effekten av fag-infeksjon for å være Explored i populasjoner med komplekse romlige dynamikk 11, med pre-eksisterende romlige strukturer som biofilm 18, eller i genetisk blandet naturlige populasjoner 19.
Et kritisk trinn i denne fremgangsmåte er fremstilling av pakkede fagmid ved høy titer. Den metabolske byrden forbundet med fag partikkelproduksjon kan føre til høye priser av mutasjon eller plasmid tap i fagmidekspresjonsvektoren produksjon belastning. Det anbefales at fagmidet produksjonen stammen dyrkes direkte fra en enkelt koloni kotransformert og ikke kjøling, frosset eller sub-dyrket før fag innhøsting. En lav effektivitet av co-transformasjon kan også observeres ved innføring fagmidet og hjelper plasmid til E. coli samtidig. I dette tilfellet, kan høyere virkningsgrader oppnås ved å transformere hjelperen plasmidet først, og deretter fremstilling av kompetente celler som bærer hjelpe plasmid for påfølgende transformasjon med fagmidet.
Phagemid infeksjon eller Srna uttrykk pålegger også en påviselig metabolsk belastning på målcellene, og kan resultere i noen fenotypisk forstyrrelse. For eksempel ble en reduksjon i mKate2 fluorescens observert selv når cellene ble infisert med en fagemid rettet mot CAT (figur 3). Infeksjon med M13 er ikke tenkt å utløse systemiske stressresponser i E. coli 20, men kan forandre transkripsjonsmønstre indirekte. Alternativt kan GFP eller ampicillin resistensmarkører inkludert på fagmidet konkurrere om mobilnettet ressurser, redusere mKate2 uttrykk og vekst 9. Endelig kan Srna kassetten selv globalt endre genuttrykk profiler ved å titrere Hfq protein, eller gjennom off-target mRNA stanse. Utenfor mål effekter er vanlige i in vivo RNAi målgruppe 21-23, men de har ennå ikke systematisk undersøkt for dette systemet.
En begrensning med denne metoden er at infeksjonen effeffektivitet er mindre enn 100%, slik at noen ikke-infiserte bakterier til å vedvare i befolkningen. Resultatene av dette arbeidet og tidligere arbeid 10 tyder på at noninfected celler representerer 1-10% av den endelige befolkningen, og er ansvarlig for de fleste av nonsilenced fenotyper observert. En rekke ruter til M13-motstand er kjent, og de vanligste er mutasjons tap av pilus uttrykk 24. I lys av disse begrensningene, bør kontrollene brukes til å bekrefte høye infeksjonsrater og knockdown effektivitet.
En annen potensiell begrensning for enkelte programmer er sporadisk overføring av forurensende hjelpefagen. Selv om M13K07 inneholder en mutert emballasje signal, kan det være pakket inn i fag kapsid ved lav frekvens og overføres til infiserte populasjoner, noe som resulterer i cellene kompetente for fag-produksjon, og det fortsatte spredning av fag utover den første infeksjonen arrangementet 25. Modifikasjoner av hjelpefagen har vist seg effektiveved å redusere ikke-spesifikk emballasje, men noen ganger på bekostning av redusert fag-produksjon 26.
Konstruert bakteriofag har blitt et uunnværlig verktøy for E. coli syntetisk biologi, og gir rask levering av nye gener til en voksende befolkning. Nyere arbeider har produsert interkommunikasjonskretser 11 eller uttrykt transkripsjonsfaktorer for å undertrykke antibiotikaresistens trasé 27. Protokollen presenteres her legger til en voksende samling av verktøy som tillater kontroll av bakteriell fysiologi gjennom programmerte RNA. CRISPR-Cas nukleaser, når mutert til å eliminere nukleaseaktiviteten, har vist seg å undertrykke transkripsjon ved RNA-guidet genet målene 17,28. I motsetning til dette, virker Srna stanse på det translasjonelle nivå og krever ikke eksogene proteinekspresjon. Neste generasjons bioteknologi kan forene transkripsjonen og translasjonell kontroll med fag-mediert levering å programmere komplekse phenotyper i sanntid.
The authors have nothing to disclose.
Midler til dette arbeidet ble gitt av Fondation Bettencourt Schueller til støtte for Paris Bettencourt iGEM team. Vi takker INSERM U1001 forskningsenheten og Chantal Lotton for teknisk hjelp og råd. Fagmidekspresjonsvektoren Litmus28i_J23115-B0032-GFP ble gitt av Monica Ortiz og Drew Endy av Stanford.
| Name | Company | Catalog Number | Comments |
| Plasmid Miniprep Kit | Qiagen | 27104 | |
| DpnI Enzyme | NEB | R0176S | |
| Phusion High Fidelity Polymerase | NEB | M0530S | |
| Taq 2x Master Mix | NEB | M0270L | |
| M13KO7 Helper Phage | NEB | N0315S | |
| DH5α Competent Cells | Life Technologies | 18265-017 | |
| TOP10F' Cells | Life Technologies | C3030-03 | |
| LB Broth | Sigma | L3022-250G | |
| Ampicillin | Sigma | A9393-5G | |
| Kanamycin | Sigma | 60615-5G | |
| Chloramphenicol | Sigma | C0378-5G | |
| Tetracycline | Sigma | 87128-25G |
- Ohkumo, T., Masutani, C., Eki, T., Hanaoka, F. Use of RNAi in C. elegans. RNAi. , 129-137 (2008).
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391 (6669), 806-811 (1998).
- Iordanou, E., Chandran, R. R., Blackstone, N., Jiang, L. RNAi interference by dsRNA injection into Drosophila embryos. J Vis Exp. (50), e2477 (2011).
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2 (9), 2245-2264 (2007).
- Tsong, T. Y. Electroporation of cell membranes. Biophys J. 60 (2), 297-306 (1991).
- Kim, W. J., Chang, C. -. W., Lee, M., Kim, S. W. Efficient siRNA delivery using water soluble lipopolymer for anti-angiogenic gene therapy. J Control Release. 118 (3), 357-363 (2007).
- Shi, Y. Mammalian RNAi for the masses. Trends Genet. 19 (1), 9-12 (2003).
- Na, D., Yoo, S. M., Chung, H., Park, H., Park, J. H., Lee, S. Y. Metabolic engineering of Escherichia coli using synthetic small regulatory RNAs. Nat Biotechnol. 31 (2), 170-174 (2013).
- Ceroni, F., Algar, R., Stan, G. -. B., Ellis, T. Quantifying cellular capacity identifies gene expression designs with reduced burden. Nat Methods. 12 (5), 415-418 (2015).
- Libis, V. K., Bernheim, A. G., et al. Silencing of Antibiotic Resistance in E. coli with Engineered Phage Bearing Small Regulatory RNAs. ACS Synth Biol. 3 (12), 1003-1006 (2014).
- Ortiz, M. E., Endy, D. Engineered cell-cell communication via DNA messaging. J Biol Eng. 6 (1), 16 (2012).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnol. 9 (1), 61 (2009).
- Chung, C. T., Miller, R. H. Preparation and storage of competent Escherichia coli cells. Recombinant DNA Part I. , 621-627 (1993).
- Phornphisutthimas, S., Thamchaipenet, A., Panijpan, B. Conjugation in Escherichia coli. Biochem Mol Biol Educ. 35 (6), 440-445 (2007).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli Physiology in Luria-Bertani Broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Mittal, V. Improving the efficiency of RNA interference in mammals. Nat Rev Genet. 5 (5), 355-365 (2004).
- Qi, L. S., Larson, M. H., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Lu, T. K., Collins, J. J. Dispersing biofilms with engineered enzymatic bacteriophage. Proc Natl Acad Sci USA. 104 (27), 11197-11202 (2007).
- Yosef, I., Manor, M., Kiro, R., Qimron, U. Temperate and lytic bacteriophages programmed to sensitize and kill antibiotic-resistant bacteria. Proc Natl Acad Sci USA. 112 (23), 7267-7272 (2015).
- Karlsson, F., Malmborg-Hager, A. -. C., Albrekt, A. -. S., Borrebaeck, C. A. K. Genome-wide comparison of phage M13-infected vs. uninfected Escherichia coli. Can J Microbiol. 51 (1), 29-35 (2005).
- Senthil-Kumar, M., Mysore, K. S. Caveat of RNAi in plants: the off-target effect. Methods in molecular biology. 744, 13-25 (2011).
- Jackson, A. L., Linsley, P. S. Recognizing and avoiding siRNA off-target effects for target identification and therapeutic application. Nat Rev Drug Discov. 9 (1), 57-67 (2010).
- Cho, S. W., Kim, S., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Hagens, S., Blasi, U. Genetically modified filamentous phage as bactericidal agents: a pilot study. Lett Appl Microbiol. 37 (4), 318-323 (2003).
- Kasman, L. M., Kasman, A., Westwater, C., Dolan, J., Schmidt, M. G., Norris, J. S. Overcoming the phage replication threshold: a mathematical model with implications for phage therapy. J Virol. 76 (11), 5557-5564 (2002).
- Chasteen, L., Ayriss, J., Pavlik, P., Bradbury, A. R. M. Eliminating helper phage from phage display. Nucleic Acids Res. 34 (21), e145 (2006).
- Lu, T. K., Collins, J. J. Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy. Proc Natl Acad Sci USA. 106 (12), 4629-4634 (2009).
- Bikard, D., Jiang, W., Samai, P., Hochschild, A., Zhang, F., Marraffini, L. A. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. Nucleic Acids Res. 41 (15), 7429-7437 (2013).
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved