3 A-seq2 的末端测序库的研制
摘要
本协议描述了一种映射体3的端处理站点的方法。
摘要
过去十年的研究揭示了体裂解和多反应的复杂和动态变化。基因与 3 ' 未翻译的区域 (UTRs) 在分化的细胞中产生, 而增殖细胞优先表达的抄本与短 3 ' UTRs。我们描述了 A-seq 协议, 现在在其第二个版本, 这是开发地图多网站的全基因组和研究的规则体3的最终处理。并且这个当前协议利用 polyadenylate (聚 (A)) 尾巴在多数哺乳动物基因的生物期间增加, 丰富为充分地被处理的基因。一个 DNA 适配器与 deoxyuracil 在其第四位置允许精确处理 mRNA 3 的末端片段排序。不包括细胞培养和过夜结扎, 该协议需要大约8小时的动手时间。同时, 还提供了一个易于使用的软件包, 用于分析导出的测序数据。A-seq2 和相关的分析软件提供了一个有效的和可靠的解决方案, 体 3 ' 两端在广泛的条件, 从 106或更少的细胞。
引言
mrna 3 末端的捕获和测序可以研究 mrna 的处理和基因表达的定量。由于其聚 (A) 尾, 真核基因可有效纯化的总细胞裂解与珠固定寡苷 (dT) 分子, 这也可以是主要的 cDNA 合成。但是, 这种方法有两个缺点。首先, A 的延伸是内部的抄本也可以是主要的 cDNA 合成, 造成杂散 (a) 网站。第二, 同质聚 (A) 拉伸对测序有特定的挑战, 除了不提供信息以记录身份。已经提出了各种方法来绕过这些限制, 如通过聚 (A) 尾的反向转录, 其次是核糖核酸 H 消化 (3 p-seq 1), 使用自定义的测序底漆结束于 20 Ts (2 p-seq 2), 预选RNA 片段与聚 (a) 超过50核苷酸的尾巴与 CU5T45底漆跟随核糖核酸 H 消化 (3 ' 读3), 以及在发夹 (a seq 4) 中含有 3 "适配器" 的寡糖-dT 底漆的使用。
最近开发的 A-seq2 方法5的目的是通过聚 (A) 绕过测序, 同时使适配器 self-ligation 产生的聚比例最小化, 特别是当适配器的摩尔浓度超过了插入浓度。当两个适配器都与 A-seq2 中的相同类型的核苷酸端点相连时, 就可以消除这个问题, 在这种情况下, 3 "适配器与 RNA 片段的 5" 端相连, 而 5 "适配器" 则与基因在反向转录后的5端相连。该方法比我们先前提出的 seq-in 更方便, 它的测序是在 5 ' 至-3 ' 方向, 从而需要精确控制的 RNA 碎片-, 同时保持高精度的聚 (A) 站点识别。大约80% 的测序读数在典型的样本中具有独特的基因组, 并导致2万多个 (A) 站点簇的识别, 其中超过70% 是与注释的 3 "UTRs" 重叠的。
简言之, A-seq2 协议开始于 mRNA 分裂和结扎的反向互补 3 ' 适配器到 5 ' 端的 RNA 片段。聚 (a) 包含 rna 然后反向转录与25核苷酸 (nt) 长寡糖 (dT) 底漆, 其中包含一个锚核苷酸在 3 ' 年底, 杜在位置4和生物素在 5 ' 年底, 允许结合的 cDNA 磁性亲和珠。大部分的引物, 包括生物素, 是从在杜分裂的用户酶组合, 以嘧啶 dna glycosylase (UDG) 和 dna glycosylase 裂解切 VIII。这个反应留下完整的两端结扎的 5 ' 适配器, 和三 Ts 留下后, 分裂仍然是标记的位置, 聚 (a) 尾。由于 5 "和 3" 适配器都通过结扎连接到收件人5端, 因此不生成适配器聚。在读取开始时引入的四核苷酸随机市场分析允许对最先进的测序仪器进行簇解析, 也可以作为唯一的分子识别器 (UMI) 用于检测和去除 PCR 扩增伪影。UMI 的大小可进一步增加, 如其他研究6所示。该协议生成的读取是反向互补的 mRNA 3 的两端, 所有开始的随机聚丙烯, 其次是 3 ts. 对3诊断 Ts 的读取的处理在其5的末尾开始与 PCR 扩增伪影的修正利用 UMIs, 去除3的适配器序列, 并反向互补。可能源自于内部 A 丰富站点的寡聚 (dT) 启动的读取也被计算出来并被丢弃。杂散站点通常缺少 18 良好和保守的聚 ( A ) 信号中的一个 , 应该位于明显的站点7的上游 21 核苷酸。
该协议需要大约8小时的动手时间, 不计算细胞培养和过夜结扎。关联的读取分析软件可实现高度精确的多 (a) 站点标识。从聚 (A) 网站集群创建的基础上进一步突出的4样本在这篇手稿 (两个生物复制控制 siRNA 和硅 HNRNPC 处理细胞) 84% 重叠与注释基因, 和这些, 75% 重叠与 3 ' UTR, 和86% 与任何一个3 ' UTR 或终端外显子。在复制样本中, 3 ' 端的皮尔逊相关系数为 0.92, 通常用该方法获得0.9 以上的值。因此, A-seq2 是一种方便的方法, 可以获得非常重现的结果。
研究方案
1. 细胞生长和 mRNA 分离
- 根据您在6孔板上的实验设计, 在6汇合处对 1 x 10 80% 单元格中的单元格进行扩展.
- 去除培养基, 用磷酸盐缓冲盐水冲洗细胞一次。通过从 mRNA 隔离试剂盒中加入1毫升的裂解缓冲液直接溶解板上的细胞。将粘性的裂解液转化为15毫升的塑料管, 用1毫升吸管尖端。使用橡皮刮刀将电池材料从板材表面完全分离.
- 剪切含粘性 DNA 的裂解物, 用1毫升注射器附着在23克皮下注射针头上, 由柱塞向上和向下运动, 直到裂解物不再粘性。将注射器针指向底部的中心, 以避免将裂解液从试管中弹出.
- 使用注射器将裂解液转化为1.5 毫升的管子。旋转5分钟在 2万 x g 和4和 #176; C 移除碎片。在整个协议中使用 DNA 低绑定1.5 毫升的小瓶.
- 当离心机运行时, 洗涤300和 #181; 悬浮寡糖 (dT) 25 磁性齿条上有500和 #181 的磁珠; 溶解缓冲的 l。将管子在机架上搅拌2-3 次。解决方案清除后, 删除缓冲区。从步骤1.4 中收集清上清液, 并加入珠子。重和放置在旋转车轮上的管10分钟
- 将管放置在磁性机架上。2分钟后取出透明液体, 从 mRNA 隔离套件中添加0.8 毫升缓冲 A。把管子按180和 #176; 在机架上的度数, 2-3 次。重复此洗涤步骤再次与缓冲区 A.
- 步骤1.6 中所述, 用0.8 毫升缓冲 B 洗涤珠子2次.
- 要从珠子洗的绑定 mRNA, 添加33和 #181; L H 2 O 和重的珠子。热到75和 #176; C 在加热块上为5分钟。立即旋转管1秒, 并把它们放在磁性架上。把上清液转移到新的管子。样品可以存储在-80 和 #176; C, 直到进一步使用.
- 添加66和 #181; l 碱性水解缓冲到33和 #181; l mRNA (步骤 1.8), 混合和热为恰好5分钟在95和 #176; C 在加热块上。立即冷却冰管.
- 将 rna 与 rna 清除工具包隔离.
注: 确认音量;应该是100和 #181; 我。- 添加350和 #181; 从工具包和 250 #181 RLT 缓冲区; l 乙醇。三十年代在室温 (RT) 上加载到柱上并旋转 8000 x g。用500和 #181 冲洗, 从试剂盒中洗涤。用500和 #181 冲洗, 80% 乙醇。旋转5分钟, 在 2万 x g 干燥的列。添加36和 #181; L 2 O 到列, 并在 2万 x g 处旋转1分钟. 丢弃该列并保存洗.
2. 5 和 #39; 终止磷酸化和 dnasei 处理
- 添加5和 #181; 核苷酸激酶缓冲, 5 和 #181; l 10 毫米 ATP, 1 和 #181; l 核糖核酸抑制剂, 1 和 #181; l dnasei 和2和 #181; l 核苷酸激酶样品和孵育在37和 #176; C 为 30 min. 可选地在整个协议中准备主反应混合, 方法是将每个组件的1.1 卷 x n (n = 样本数) 混合在一起.
- 更改缓冲区并移除自旋列上的 ATP, 以防止在下一步骤中添加聚 (a).
- Prespin 在 735 x g 处旋转列1分钟, 将列转换为新的1.5 毫升小瓶, 并将激酶反应加载到柱上。旋转列2分钟, 在 735 x g. 丢弃的列, 并把收集的反应在冰或存储在-80 和 #176 的管; C.
3。阻塞3和 #39; 以虫草三磷酸盐结束
注意: 必须阻止3和 #39; 在随后的结扎反应中, 要避免 RNA 片段的 concatemerization. 3 和 #39; 尚未被 a 阻塞的端点 (循环) 磷酸盐水解后, 加入了3和 #39; dATP (虫草三磷酸酯) 链终止核苷酸的帮助下聚 (a) 聚合酶。在这里, 酵母聚 (A) 聚合酶 (yPAP), 这是表达和纯化的描述, 在 8 中使用的浓度为0.5 毫克/毫升。酵母或 大肠杆菌 PAP 都有几乎相同的活动, 增加3和 #39;d atp 和可以购买商业 (见材料表).
- 添加13.5 和 #181; l 5x 浓缩聚 (a) 聚合酶反应缓冲器, 2 和 #181; 10 mM 3 和 #39; dATP, 1 和 #181; l 核糖核酸抑制剂和1和 #181; 聚 (a) 聚合酶对从台阶2.2.1 的反应。混合和旋转 1 s. 孵育在37和 #176; C 30 min. 添加32.5 和 #181; 每个反应都有 2 O。纯化 RNA 的步骤1.10.1。洗14和 #181 的 RNA; L H 2 O.
4。结扎的反向3和 #39; 适配器到5和 #39; RNA 片段的结尾
- 将反应放在真空浓缩器中10分钟, 以减少音量到6和 #181; l. 增加3和 #181; l 10x T4 RNA 结扎缓冲, 3 和 #181; l 10 毫米 ATP, 15 和 #181; 我 #173-8000, 1, #181; l 核糖核酸抑制剂, 1 和 #181; 0.1 mM 反向互补3和 #39; 适配器和 #34; revRA3 和 #34; (见材料表) 和1和 #181; 高浓度 RNA 连接 1, 混合.
- 在24和 #176 上孵育反应; 在 1000 rpm 间歇混合的加热混频器上, C 为16小时。添加70和 #181; 每一个反应和混合的 L H 2 O。纯化 RNA 的步骤1.10.1。洗14和 #181 的 RNA; L H 2 o. 在这一点上, 样品可以存储在-80 和 #176; C.
5。反向转录 (RT)
- 将溶放在真空浓缩器中3分钟, 以将体积减小到11和 #181; l. 转移反应到200和 #181; l PCR 管。添加1和 #181; 0.05 mM RT 底漆和 #34; Bio-dU-dT25 和 #34;热5分钟, 在70和 #176; C 在 PCR 循环和离开 RT 为5分钟.
- 添加1和 #181; l 10 mM dNTPs、4和 #181; l 5x 反转录酶缓冲器、1和 #181; l 0.1 M、1和 #181; l 核糖核酸抑制剂, 1 和 #181; l 反转录酶。混合和加热反应10分钟到55和 #176; c 和10分钟到80和 #176; c 在 PCR 循环。保持在冰上或在-80 和 #176; C 用于更长的存储.
6。消化与嘧啶 DNA Glycosylase 酶混合
- 吸管100和 #181; 亲和珠成1.5 毫升的小瓶, 重在800和 #181; 生物素结合缓冲和放置在一个磁性的架子上。倒置管2-3 次。清除时删除缓冲区。重复洗涤步骤。重200和 #181 中的珠子; 生物素结合缓冲.
- 添加反向转录反应的珠子解决方案和孵化20分钟, 在4和 #176; C 在一个旋转的车轮。洗珠2x 与生物素结合的缓冲, 在步骤6.1 和2x 与十缓冲在一个磁性机架上。重50、#181; l 十缓冲, 添加2和 #181; 嘧啶 DNA glycosylase 酶混合, 孵育 1 h 在37和 #176; C 在间歇混合的混合器中.
- 添加50和 #181; l h 2 O、11和 #181; 核糖核酸 h 缓冲器和1和 #181; 我核糖核酸的反应孵育在37和 #176; C 为20分钟放置在磁性机架上的管, 并将含有该裂解 cdna 的液体转移到一个新的管
- 纯化裂解 cdna.
- 添加550和 #181; 从 PCR 纯化试剂盒中的缓冲器 PB 到裂解反应。添加10和 #181; L 3 米醋酸钠, ph 5.2, 降低 ph 值。装载反应在最小洗脱自旋柱和旋转在 1.7万 x g 为 1 min.
- 添加750和 #181; 将 PE 缓冲到列, 并在 1.7万 x g 处旋转1分钟以丢弃流。旋转的列在 1.7万 x g 为1分钟干。将柱形转换为1.5 毫升的小瓶, 添加16和 #181; l H 2 O, 在 1.7万 x g 处旋转1分钟, 将反应放置在真空浓缩器中8分钟, 集中到7和 #181 的体积; l.
7。结扎5和 #39; 适配器到5和 #39; cdna 末端
- 对孤立的 cdna, 添加3和 #181; l 10x T4 RNA 连接1缓冲, 3 和 #181; l 10 毫米 ATP, 15 和 #181; l #173;-8000, 1 和 #181; l 50 和 #181; M 和 #34; revDA5 和 #34; 寡聚, 1 和 #181; 高浓度 T4 RNA 连接1。孵育在24和 #176; C 为 20 h. 添加70和 #181; 每一个反应的 L h 2 O。样品可以存储在-20 和 #176; C 在这一点上.
8。先导 pcr、扩增文库和大小选择
- 在先导反应中, 确定 pcr 周期的最佳数目, 以在指数阶段内达到库的放大倍数.
- 吸管25和 #181; L DNA 聚合酶混合, 20 和 #181; 结扎反应, 2 和 #181; l H 2 O、1.5 和 #181; l 10 和 #181; m 正向 pcr 引物 (RP1) 和1.5 和 #181; l 10 和 #181; m. 反向 pcr 索引底漆为200和 #181; l pcr 管.
- 使用以下程序运行循环: 3 分钟95和 #176; c, 其次20周期二十年代98和 #176; c, 二十年代67和 #176; c 和三十年代72和 #176; c. 收集7和 #181; L 等分后 6, 8, 10, 12, 14, 16 和18的周期直接从循环。添加1和 #181; L 10x 负载缓冲器 (50% 甘油, 0.05% 二甲苯蓝)。注: 如果在合并条码时使用复用, 请遵循供应商的建议.
- 将2% 琼脂糖凝胶中的小插槽中的产品分别放在1x 的缓冲液中, 其中含有1:10,00 稀释的荧光绿色染料。
- 在2% 琼脂糖凝胶上加载等分, 并将凝胶以100伏的电压运行15分钟. 可视化 PCR 产品在凝胶文献系统上的迁移.
- 使用在先导反应中的 large-scale PCR 反应开始时的周期数, 两倍于用于先导反应的卷 ( 图 2 ).
- 用于 large-scale pcr 反应, 首先用 pcr 纯化试剂盒浓缩和淡化反应, 然后在1x 的缓冲液中将产品分别放在2% 琼脂糖凝胶的宽槽上.
- 剪切含有 200-350 nt DNA 产品的凝胶切片。融化的凝胶在 chaotropic 缓冲在 RT 30 分钟。用凝胶萃取试剂盒从凝胶切片中提取 DNA。不要加热到50和 #176; C, 以防止在具有丰富 DNA 的绑定中出现偏倚 9 .
- 提交以进行排序.
注意: 通常, 50 周期 single-read (SR50) 是足够的 (参见, 对于 例如 , https://www.illumina.com/technology/next-generation-sequencing.html).
9。数据处理
注意: 生成的排序数据 (以 fastq 格式) 使用 gitlab 存储库 (https://git.scicore.unibas.ch/zavolan_public/A-seq2-processing) 中提供的软件进行处理。分析包括四主要步骤: (1) 下载 git 存储库, (2) 安装虚拟环境, (3) 在配置文件中设置特定参数; (4) 通过和 #8216 启动分析; snakemake 和 #8217; 10 . 在步骤4中完成的整个分析只需要一个命令。可以在 gitlab 存储库的自述文件中找到分析的详细 step-by 步骤说明, 下面提供了简短说明。所有单独处理步骤由公开地可利用的工具的施行完成, 无论是从外部来源或准备 in-house。计算管道依赖于 anaconda-based 的 11 python 3 虚拟环境, snakemake 软件包可用 10 。它在具有 Unix 操作系统的计算机上运行, 并在 Linux 环境中进行了测试, 安装了 CentOS 6.5 操作系统和可用的 40 GB RAM。软件依赖项在虚拟环境中自动控制。下列可公开使用的软件工具是必需的, 从而与环境一起安装: snakemake (v3.9.1) 10 , fastx 工具箱 (v0.0.14) 12 , 星形 (v2.5 2 a) 13, cutadapt (v1.12) 14 , samtools (v1.3.1) 14 , 15 , bedtools (v2.26.0) 16 , 17 .
- 从读取到基因的数据预处理
注意: 顺序深度可能因运行而异, 根据仪器的不同, 一个样本中的数据可以在多个序列文件上分割。如果是这种情况, 请将对应于一个示例的文件连接到单个输入文件中, 并在下面的步骤中使用。- 将文件从 fastq 转换为 fasta 格式.
- 提取具有正确结构的读取 (3 thymidines 在读取位置5、6和7处)
注: 根据上述实验协议正确准备的读取应具有结构 (从5和 #39; 结束): 4-核苷酸条码-3 thymidines-反向补充的成绩单3和 #39; 结束. - 将有关起始聚丙烯的信息存储在序列的说明行中.
注意: 聚丙烯是一种独特的分子标识符 (UMI), 它便于以后在分析中对放大工件进行校正. - 从读取和 #39 中删除前七核苷酸; 5 #39; 结束.
- 通过只保留同一插入序列和 UMI 的读取的一个副本来更正放大工件.
- 移除3和 #39 的部分; 与适配器序列匹配的端, 然后反向补充序列。只继续进行具有最小长度 (默认值:15 nt) 的读取.
注: 根据原 mRNA 片段的长度和测序周期数, 3 和 #39; 读的结尾可能包含3和 #39; 适配器的一部分, 在这一步中将被删除.
- 提取满足下列条件的所有读取: 最大2未知核苷酸 (和 #39; N 和 #39;), 最大80% 为, 最后一个核苷酸为非。这些读数被认为是在分析中使用的足够的质量.
- 使用一个处理拼接读取的工具将读取结果映射到基因组, 并以 BAM 格式生成输出文件.
- 如果使用星号, 则创建一个文件, 其中带有要映射其读取的基因组索引。对于人类基因组, 这一步需要 35 GB 内存 (RAM).
- 将读取映射到基因组.
注: (特定于星号的音符) 软剪裁被禁用, 以强制3和 #39 的映射; 每读结束, 因为这是核苷酸在部位的上游 .
- 将 BAM 转换为床文件。如果读取映射到多个位置, 则只保留编辑距离最低的那些.
注意: 在特定位置的读取映射的副本号用作分数。读取映射到多个位置的分数在每个位置都有一个加权等于 1/个读取映射的位置数. - 折叠读取可能会因排序错误而异。如果两个不同的读取映射到相同的位置 (映射的起始和结束位置相同), 并且它们共享相同的 UMI, 则将它们视为 PCR 重复项并只保留一个.
- 推断所有单独的体3和 #39; 结束处理站点.
注: 个人阅读为3和 #39 提供证据; 结束时, 它的最后四核苷酸映射到基因组没有错误。3和 #39 的位置; 读取映射的末尾存储为解理站点. - 检测3和 #39; 可能源自内部启动的结束站点。将站点定义为内部启动工件当基因组中的部位的 10 nt 下游满足下列条件之一时 : 包含六个以上 , 连续包含六 , 或以下列体开头 : AAAA 、 AGAA 、 AAGA 、 AAAG.
- 生成单个3和 #39 的表; 以床格式结束处理站点.
- 标识独立调节的聚类 (A) 站点群集.
注意: 此处介绍的步骤遵循以前发布 5 中介绍的过程。- 首先收集单个3和 #39; 在所有研究样本中获得的结束处理站点.
- 注释已知的聚 (A) 信号 7 在每个单独的3和 #39 的区域中有-60 到 +10 核苷酸; 结束处理站点.
- 标识在每个示例的背景上方表示的聚 (A) 站点, 如下所示。
- 根据当前示例中的原始表达式对站点进行排序。从上到下遍历站点列表, 将排名较低的站点与较高排名的网站关联, 如果它们位于高级站点的预定义距离 (默认值:25 nt 向上或下游) 中.
注: 与高级网站关联的所有低级站点都定义了一个群集, 其表达式是记录所有这些站点的读取数. - 按表达式对这些群集进行排序, 并遍历从最高到最低表达式的群集列表, 确定表达式阈值 c , 在该列中, 带有带注释的聚 (A) 信号的簇的百分比低于预定义阈值 (默认值: 90%).
- 丢弃位于截止点下面的任何群集中的站点.
- 根据当前示例中的原始表达式对站点进行排序。从上到下遍历站点列表, 将排名较低的站点与较高排名的网站关联, 如果它们位于高级站点的预定义距离 (默认值:25 nt 向上或下游) 中.
- 群集紧密间隔的3和 #39; 通过示例获得的结束站点.
注: 排序3和 #39; 结束处理站点首先由支持的样品的数量, 然后由规范化的读计数的总和 (读每百万 (RPM)) 在样品。从上到下遍历列表, 将级别较低的站点与级别较高的站点进行关联时, 它们与较高秩站点的距离不大于预定义的限制 (默认值:12 nt)。当任何构成3和 #39; 结束站点与带注释的聚 (a) 信号重叠或直接在下游有一个聚 (a) 信号时, 相应的簇被标记为进一步检查以检测内部启动. - 合并聚合 (A) 站点群集.
注: 当群集被标记为假定的内部启动候选时, 如果两个簇共享它们的聚 (a) 信号或保留在群集中的最下游的站点有一个聚 (a) 信号位于最小上游距离 (默认值:15 nt)。最后, 如果: (i) 它们共享相同的聚 (A) 信号, 或者 (ii) 所产生的簇的跨度不超过最大值 (默认值:25 nt), 则会合并紧密间隔的簇. - 将群集存储在床文件格式中, 并将所有3和 #39 中的总规范化读取计数用于每个群集中的结束站点.
结果
从培养细胞中分离出含有聚 (A) 的 RNA, 通过碱性水解和基因, 用寡核苷酸 (dT) 引物进行反转录。所产生的 cDNA 固定在亲和珠上, 杜在嘧啶特异性切除反应中被劈开, 适配器被结扎到 5 ' 和 3 ' 两端的切割片段和插入被测序。图 1描述了实验的图形化轮廓。
对于 HeLa 和 HEK293 细胞, 106细胞足以在程序结束时为绝大多数蛋白质编码基因识别聚 (A) 点。然而, 对于其他细胞类型或组织, 可能有必要测试的饱和度确定的聚 (A) 点的数量, 在实验中使用的细胞增加。在图 2中显示了 PCR 试验步骤的代表性结果和样品的 DNA 片段分析。
图 3显示了计算分析的预处理步骤, 从从排序器获得的 fastq 文件开始, 到已准备好映射到基因组的质量检查、适配器修剪的读取结束。图 4显示了从读取到相应基因组的映射开始的分析步骤, 并以特定示例中标识的 mRNA 3 的结束处理站点的目录结束。当分析多个样本时, 将执行其他步骤, 以匹配在单个样本中找到的3个最终处理站点, 并在样本间报告它们的丰度。这些步骤如图 5所示。
因此, 在对样本进行排序后, 通过可用的处理管道对生成的序列读取文件 (以 fastq 格式) 进行分析是很简单的。在将有关示例的信息添加到配置文件之后, 管线的执行将导致两种主要类型的输出文件: 1) 床-文件与所有 3 ' 结束处理站点在各自的样品被辨认 (例如sample1.3pSites noIP), 和 2) 一个床文件与所有聚 (a) 站点群 (clusters.merged.bed) 横跨所有样品研究。输出还包括每个样本的所有读取的基因组坐标 (例如"sample1"。STAR_out/对齐. sortedByCoord. bam "), 稍后可以在像 IGV16这样的基因组浏览器中查看。对读取剖面的目视检查通常提供了第一次瞥见在基因组中聚 (a) 位点的分布以及在研究中所进行的特定摄动所发生的变化。例如, 在图 6中显示了特定基因对 HNRNPC 蛋白的击倒反应。
还提供了这些全基因组分布的摘要 (表 1)。具体地说, "计数/annotation_overlap" 目录中的输出文件包含与特定注释特征重叠的站点的分数 (从作为输入提供的 gtf 文件中; 注释为: 3 "UTR、终端外显子、子外显子、内含子、基因)。最后, 对于每个示例, 也保存了单个处理步骤的结果 (例如 "sample1.summary.tsv")。这包括数量: 原始的读在每个样品, 读有预期的结构 5 ' 末端, 读保留在折叠的完全 PCR 重复之后, 高质量读根据在步骤9.2 定义的标准, 读那地图独特地对基因组(在折叠那些由于排序错误而导致的结果之后, 请参见步骤 9.5), multi-mapping 读取 (在折叠导致顺序错误的结果之后, 请参见步骤 9.5), 在每个示例中原始的 (非群集的) 3 ' 结束处理站点, 原始的 3 ' 结束处理站点没有潜在的内部启动候选, 独特的 3 ' 结束处理网站从所有样品没有内部启动候选者, 和最后一套聚 (A) 网站集群。
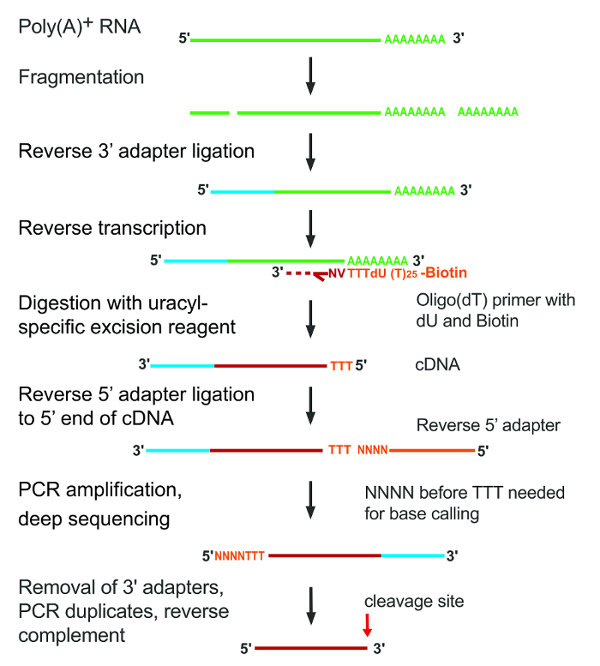
图 1: A-seq2 协议的主要步骤.图的左侧显示了各个步骤。插入 RNA 片段被描述作为绿色线转动红色为 cDNA 在反向转录以后;适配器以浅蓝色或橙色着色。请单击此处查看此图的较大版本.
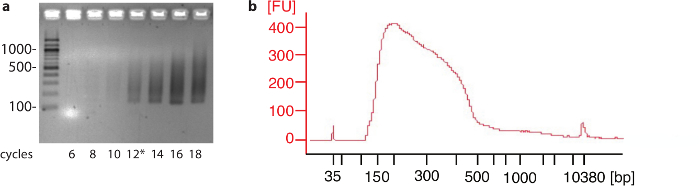
图 2: PCR 试验和最终产品简介(a) 等分从 PCR 反应收集在不同的周期和分离的2% 琼脂糖凝胶。数字到左边表明大小在核苷酸各自带在脱氧核糖核酸梯子。在本实验中, 选择了12循环 (*) 进行大规模 PCR 反应。(b) 样本大小选择运行在片段大小分析器上, 它揭示了平均大小约280核苷酸。左边的数字表示相对的信号强度。请单击此处查看此图的较大版本.
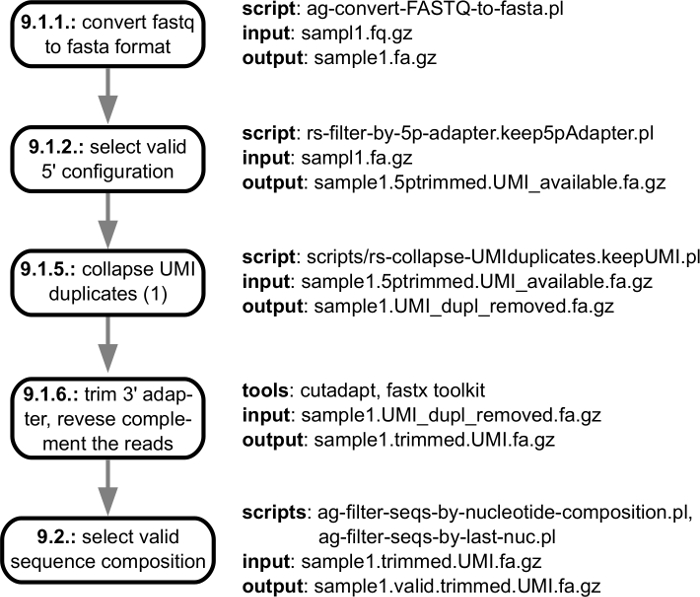
图 3: 顺序读取的预处理大纲.处理由测序仪器相关软件生成的 fastq 文件, 以确定将映射到相应基因组的高质量读取。该图显示了管道中各个步骤的输入/输出规范, 并链接到了 "数据处理" 一节中描述的协议的各个步骤。请单击此处查看此图的较大版本.
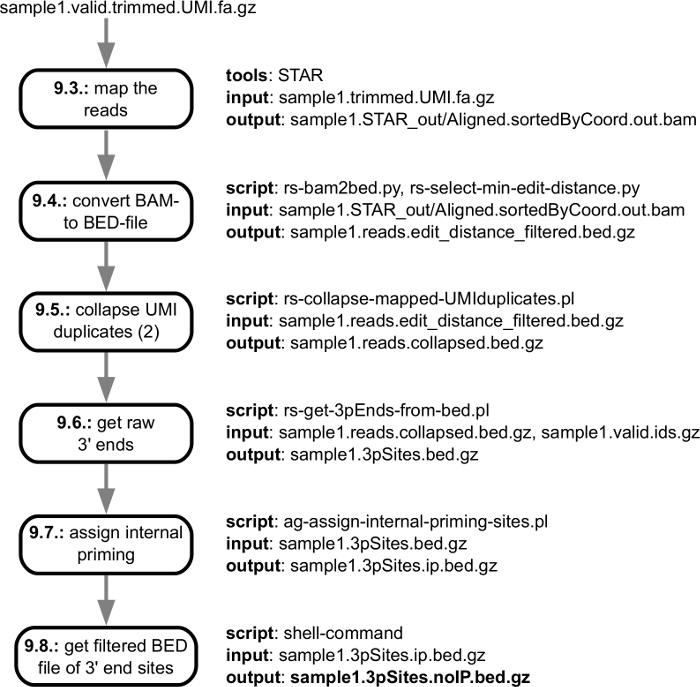
图 4: 序列读取处理的大纲, 从映射到基因组的步骤到单个3个端处理站点的生成.该图显示了管道中各个步骤的输入/输出规范, 并链接到了个体在 "数据处理" 一节中所述的协议步骤。传递给用户的主输出文件以粗体标记。请单击此处查看此图的较大版本.
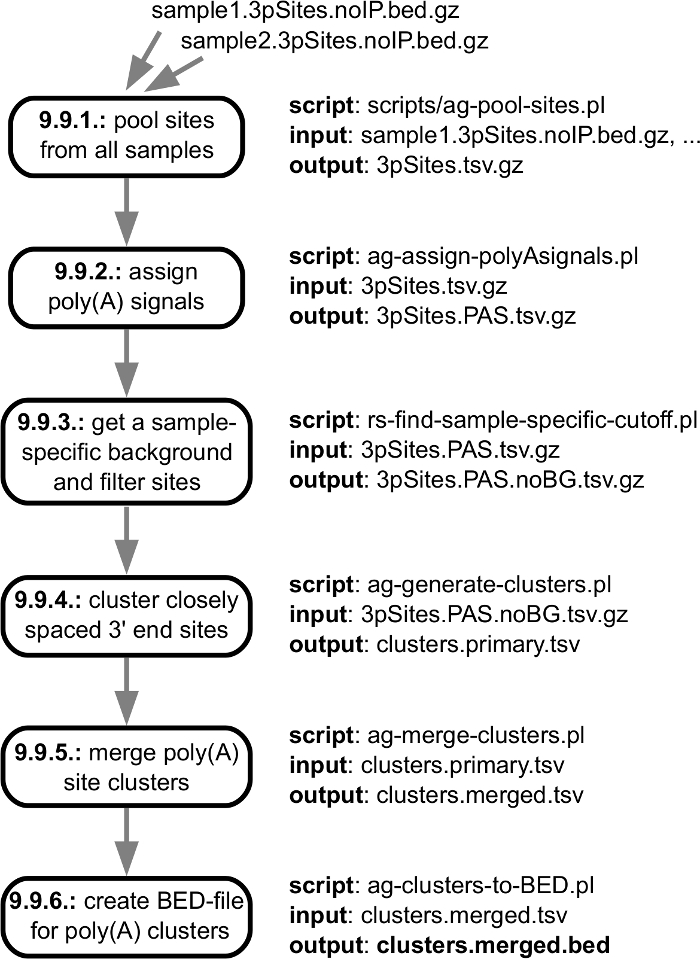
图 5: 为生成共同 3 "结束测序站点的群集而采取的步骤的概述.该图显示了管道中各个步骤的输入/输出规范, 并链接到了 "数据处理" 一节中描述的协议的各个步骤。主输出文件以粗体标记。请单击此处查看此图的较大版本.
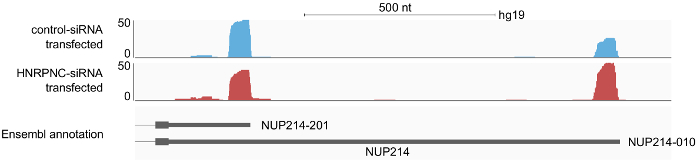
图 6: 3 ' 端处理的配置文件的示例结果沿 NUP214 基因的终端外显子读取, 如 IGV 16基因组浏览器中所示.A-seq2 读数是从两个样本的 HEK 293 细胞, 治疗要么与控制 siRNA 或与 HNRNPC siRNA。所记录的由分析管道批注的聚 (A) 站点的读取是以 BAM 格式保存的, 它用作 IGV 基因组浏览器的输入。读峰的3个端点映射到在 Ensembl 中标注的 mRNA 3 的端点。剖面图表明在 HNRNPC 击倒后, 长 3 UTR 型的使用增加。请单击此处查看此图的较大版本.
| si 控制复制1 | si 控制复制2 | |
| 编号: 29765 | 编号: 32682 | |
| 原始读取数 | 44210258 | 68570640 |
| 修剪和筛选后的有效读取数 | 14024538 | 21211793 |
| 唯一映射读取数 | 6953674 | 13946436 |
| 多个位点的读取映射数 | 2040646 | 2925839 |
| 单个3个处理站点的数量 | 1107493 | 1710353 |
表 1: 分析管线的输出示例.在各个步骤中获得的读取的摘要。
讨论
体3端处理所涉及的众多核心和辅助因素反映在相应的复杂多环境中。此外, 多也响应其他过程的变化, 如转录和剪接。3的 pre-mRNAs 的端裂部位通常是根据添加到 5 ' 裂解产物中的特征聚 (A) 尾来识别的。大多数方法使用可变长度的寡聚 (dT) 引物, 允许在反转转录反应中, 含有基因的基因的特定转换。这种方法的一个常见问题是内部启动的丰富的序列, 导致 artifactual 的裂解点。提出了两种在样品制备阶段规避工件的方法。在 3 p-seq 方法1中, 适配器是专门结扎到聚 (a) 尾的帮助下的夹板寡糖后, 部分核糖核酸 T1 消化和反向转录与在反应中作为唯一的核苷酸。由此产生的聚 (A)-聚 (dT) heteroduplexes, 然后消化与核糖核酸 H 和其余的 RNA 片段是孤立的, 结扎到适配器, 并测序。一个简单和优雅的方法, 2 p-seq, 使用自定义排序底漆跳过剩余的寡聚 (dT) 拉伸在排序反应中报告了相同的作者2。在一个相关的方法中, 3 ' 读取3, 一个不寻常的长底漆 5 Us 和 45 Ts, 也含有生物素退火到碎片 rna, 其次是严格的洗涤选择的 rna 分子与聚 (a) 的尾巴超过50核苷酸。尽管3的读数大大减少了内部启动的频率, 但它并没有完全消除它的3。还提出了直接 RNA 测序的协议, 但所产生的读数是短的, 并且有很高的错误率, 而且这种方法还没有被进一步开发18,19,20。波利亚序列和商品化的量子序列协议结合了基于寡集 (dT) 的启动与随机启动步骤的 cDNA 第二链合成20。使用模板开关反转转录反应与 Moloney 小鼠白血病病毒 (MMLV) 逆转录酶导致的基因与连接在一个单一的步骤, 从而没有适配器聚可以出现在 PAS-Seq 和 SAPAS 方法21,22。
这里提出的 A-seq2 方法在它的利用裂解核苷酸 (dU) 在化寡糖 (dT) 底漆之内站立了。此修改结合了丰富寡聚 (dt) 杂交、polyadenylated 目标的效用, 并将大部分的寡聚 (dt)25序列从孤立的片段中删除, 然后在库准备就绪并保存三 Ts 时,表示聚 (A) 尾的先验存在。相比之下, 利用核糖核酸 H 从 RNA 分子中去除聚 (A) 的方法会随机地留下数个 As。由于在 A-seq2, 测序是从义股的 3 ' 年底完成, 分裂点的位置预计将位于 NNNNTTT 主题后, 在原始序列开始读取。随机体不仅能使基础调用, 而且还能消除 PCR 扩增伪影。更长的 UMIs 也可以容纳。内部启动的可能性仍然在 A-seq2, 并处理计算, 首先通过丢弃 3 ' 两端与组编码, 丰富的下游序列, 然后通过丢弃 3 ' 结束集群, 可以解释的内部启动在丰富的聚 (a) 信号本身。最近对大量协议推断出的 (a) 站点的分析表明, A-seq2 独有的站点在基因中具有预期的核苷酸分布和位置, 类似于其他3端的测序协议。
在 A-seq2 的关键步骤是选择 polyadenylated rna 和去除核糖体 rna 和各种小 rna。这是最容易完成的一个 mRNA 隔离套件与寡糖 (dT)25磁珠。原则上, 与含酚的溶液分离的总 rna 也提供了高质量的 rna, 可以进一步选择由 mRNA 隔离试剂盒或寡核苷酸 (dT) 琼脂糖。在 A-seq2 中可以改变的一个步骤是碱性水解处理, 可以缩短或扩大, 以获得不同大小的 RNA 片段。关键的是, 增加 3 ' dATP 到 3 ' 的 RNA 片段由聚 (A) 聚合酶是有效的。在这里描述的协议中, 这种治疗适用于所有的 RNA 片段, 以避免 concatemerization 在结扎反应。最后, 我们注意到, 虽然 rna 连接1通常被用作 rna 连接, 它也 ligates 高效的单链 DNA, 正如我们在这里所做的, 结扎一个适配器到 5 ' 末端的 cDNA 分子。
因此, A-seq2 是一种高效且易于实施的协议, 用于识别体3的端处理站点。今后的发展可能包括进一步减少议定书的复杂性和所需材料的数量。相关的计算数据分析工具集进一步使 3 ' 端测序读取的齐次处理获得了广泛的协议。
披露声明
作者没有什么可透露的。
致谢
作者感谢 Mrs. Béatrice Dimitriades 对细胞培养的帮助。这项工作得到了瑞士国家科学基金会的资助 #31003A_170216 和 51NF40_141735 (NCCR RNA 和 #38; 疾病)。
材料
| Name | Company | Catalog Number | Comments |
| Materials | |||
| Agarose, ultra pure | Invitrogen | 16500-500 | |
| 2100 Bioanalyzer | Agilent | G2940CA | |
| Cordycepin triphosphate (3’ dATP) | SIGMA | C9137 | |
| DNA low bind vials, 1.5 ml | Eppendorf | 22431021 | |
| Dulbecco’s Phosphate Buffered Saline | SIGMA | D8637 | |
| Dynabeads mRNA-DIRECT Kit | Ambion | AM61012 | |
| GR-Green dye | Excellgen | EG-1071 | use 1:10,000 dillution |
| HiSeq 2500 or NextSeq 500 next generation sequencers | Illumina | inquire with supplier | |
| KAPA HiFi Hotstart DNA polymerase mix | KAPA/Roche | KK2602 | |
| Nuclease free water | Ambion | AM9937 | |
| Poly(A) polymerase, yeast | Thermo Fisher Scientific | 74225Z25KU | |
| Poly(A) polymerase, E.coli | New England Biolabs | M0276L | |
| Polynucleotide kinase | Thermo Fisher Scientific | EK0032 | |
| QIAEX II Gel Extraction Kit | Qiagen | 20021 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| RNA ligase 1, high concentration | New England Biolabs | M0437M | includes PEG-8000 |
| RNeasy MinElute RNA Cleanup kit | Qiagen | 74204 | |
| RNase H | New England Biolabs | M0279 | |
| RNasin Plus, ribonuclease inhibitor | Promega | N2618 | |
| Superscript IV reverse transcriptase | Thermo Fisher Scientiific | 18090050 | |
| Turbo DNase | Ambion | AM2238 | |
| USER enzyme mix | New England Biolabs | M5505 | |
| Dyna-Mag-2 magnetic rack | Thermo Fisher Scientific | 12321D | |
| Thermomixer C | Eppendorf | 5382000015 | Heated mixer with heated lid |
| MicroSpin columns | GE-Healthcare | 27-5325-01 | |
| Name | Company | Catalog Number | Comments |
| Buffers | |||
| Alkaline hydrolysis buffer, 1.5 x | Mix 1 part 0.1 M Na2CO3 and 9 parts 0.1 M NaHCO3. Add EDTA to 1 mM. Adjust pH to 9.2. Store aliquots at -20 °C. | ||
| 5x poly(A) polymerase buffer | Thermo Fisher Scientiific | 100 mM Tris-HCl, pH 7.0, 3 mM MnCl2, 0.1 mM EDTA, 1 mM DTT, 0.5 mg/ml acetylated BSA, 50% glycerol | |
| Biotin binding buffer | 20 mM TrisCl pH 7.5, 2 M NaCl, 0.1% NP40 | ||
| TEN buffer | 10 mM TrisCl, pH 7.5, 1 mM EDTA, 0.02% NP40 | ||
| Name | Company | Catalog Number | Sequence |
| Oligonucleotides according to Illumina TruSeq Small RNA Sample Prep Kits, for GA-IIx and Hiseq2000/2500 sequencers | Microsynth | ||
| revRA3 (RNA) | Microsynth | 5’ amino CCUUGGCACCCGAGAAUUCCA 3’ | |
| revDA5 | Microsynth | 5’ amino GTTCAGAGTTCTACAGTCCGAC GATCNNNN-3’ | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' (V = G, A or C) | |
| PCR primer forward, RP1 | Microsynth | 5' AATGATACGGCGACCACCGAGA TCTACACGTTCAGAGTTCTACAGTC CGA 3' | |
| PCR primer reverse, RPI1, barcode in bold | Microsynth | 5' CAAGCAGAAGACGGCATACGAGA TCGTGATGTGACTGGAGTTCCTTG GCACCCGAGAATTCCA 3' | |
| Name | Company | Catalog Number | Comments |
| Oligonucleotides according to Illumina TruSeq HT-Small RNA Sample Prep Kits, for HiSeq2000/2500 and NextSeq500 sequencers | |||
| HT-rev3A (DNA/RNA) | Microsynth | 5'-amino-GTGACTGGAGTTCAGACGTGTGCT CTTCCrGrAUrC-3' | |
| HT-rev5A | Microsynth | 5' amino-ACACTCTTTCCCTACACGACGCTCT TCCGATCTNNNN 3' | |
| Bio-dU-dT25, RT primer | Microsynth | 5' Biotin-TTTTTTTTTTTTTTTTTTTTTTTTTT-dU-TTTVN 3' | |
| PCR primers forward (D501-506) | Microsynth or Illumina | 5'-AATGATACGGCGACCACCGAGAT CTACAC[i5]ACACTCTTTCCCTACAC GACGCTCTTCCGATCT -3' | |
| PCR primers reverse (D701-D712) | Microsynth or Illumina | 5'-CAAGCAGAAGACGGCATACGAG A[i7]GTGACTGGAGTTCAGACGTG TGCTCTTCCGATC-3' | |
| Documentation for Illumina multiplexing: | Illumina | https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/experiment-design/illumina-adapter-sequences_1000000002694-01.pdf |
参考文献
- Jan, C. H., Friedman, R. C., Ruby, J. G., Bartel, D. P. Formation, regulation and evolution of Caenorhabditis elegans 3'UTRs. Nature. 469 (7328), 97-101 (2011).
- Spies, N., Burge, C. B., Bartel, D. P. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts. Genome Res. 23 (12), 2078-2090 (2013).
- Hoque, M., Ji, Z., et al. Analysis of alternative cleavage and polyadenylation by 3' region extraction and deep sequencing. Nat. methods. 10 (2), 133-139 (2013).
- Martin, G., Gruber, A. R., Keller, W., Zavolan, M. Genome-wide analysis of pre-mRNA 3’ end processing reveals a decisive role of human cleavage factor I in the regulation of 3' UTR length. Cell Rep. 1 (6), 753-763 (2012).
- Gruber, A. R., Martin, G., et al. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells. Nat. Commun. 5, 5465 (2014).
- Kivioja, T., Vähärautio, A., et al. Counting absolute numbers of molecules using unique molecular identifiers. Nat. methods. 9 (1), 72-74 (2011).
- Gruber, A. J., Schmidt, R., et al. A comprehensive analysis of 3' end sequencing data sets reveals novel polyadenylation signals and the repressive role of heterogeneous ribonucleoprotein C on cleavage and polyadenylation. Genome Res. 26 (8), 1145-1159 (2016).
- Lingner, J., Keller, W. 3'-end labeling of RNA with recombinant yeast poly(A) polymerase. Nucleic Acids Res. 21 (12), 2917-2920 (1993).
- Quail, M. A., Kozarewa, I., et al. A large genome center's improvements to the Illumina sequencing system. Nat. methods. 5 (12), 1005-1010 (2008).
- Rahmann, S. Snakemake--a scalable bioinformatics workflow engine. Bioinformatics. 28 (19), 2520-2522 (2012).
- . Anaconda Software Distribution Available from: https://continuum.io (2016)
- Dobin, A., Davis, C. A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Li, H., Handsaker, B., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Robinson, J. T., Thorvaldsdóttir, H., et al. Integrative genomics viewer. Nat. Biotechnol. 29 (1), 24-26 (2011).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ozsolak, F., Platt, A. R., et al. Direct RNA sequencing. Nature. 461 (7265), 814-818 (2009).
- Yao, C., Biesinger, J., et al. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation. Proc. Natl. Acad. Sci. U. S. A. 109 (46), 18773-18778 (2012).
- Lin, Y., Li, Z., et al. An in-depth map of polyadenylation sites in cancer. Nucleic Acids Res. 40 (17), 8460-8471 (2012).
- Shepard, P. J., Choi, E. -. A., Lu, J., Flanagan, L. A., Hertel, K. J., Shi, Y. Complex and dynamic landscape of RNA polyadenylation revealed by PAS-Seq. RNA. 17 (4), 761-772 (2011).
- Fu, Y., Sun, Y., et al. Differential genome-wide profiling of tandem 3' UTRs among human breast cancer and normal cells by high-throughput sequencing. Genome Res. 21 (5), 741-747 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。