Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Biology
Rekonstitution und Charakterisierung von Aktin-Mikrotubuli-Verbundwerkstoffen mit abstimmbarer motorgetriebener Dynamik und Mechanik
ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …Dieser Artikel stellt Protokolle für die Entwicklung und Charakterisierung abstimmbarer dreidimensionaler Verbundnetzwerke aus co-verschränkten Aktinfilamenten und Mikrotubuli vor. Verbundwerkstoffe unterliegen einer aktiven Umstrukturierung und ballistischen Bewegung, die von Myosin II- und Kinesinmotoren angetrieben wird, und werden durch die relativen Konzentrationen von Aktin, Mikrotubuli, Motorproteinen und passiven Vernetzern abgestimmt.
Das zusammengesetzte Zytoskelett, bestehend aus interagierenden Netzwerken aus semiflexiblen Aktinfilamenten und starren Mikrotubuli, restrukturiert und erzeugt Kräfte unter Verwendung von Motorproteinen wie Myosin II und Kinesin, um Schlüsselprozesse wie Migration, Zytokinese, Adhäsion und Mechanosensierung voranzutreiben. Während Aktin-Mikrotubuli-Interaktionen der Schlüssel zur Vielseitigkeit und Anpassungsfähigkeit des Zytoskeletts sind, ist ein Verständnis ihres Zusammenspiels mit Myosin- und Kinesinaktivität noch im Entstehen. Diese Arbeit beschreibt, wie abstimmbare dreidimensionale Verbundnetzwerke aus co-verschränkten Aktinfilamenten und Mikrotubuli entwickelt werden können, die einer aktiven Umstrukturierung und ballistischen Bewegung unterliegen, die von Myosin II- und Kinesinmotoren angetrieben werden und durch die relativen Konzentrationen von Aktin, Mikrotubuli, Motorproteinen und passiven Vernetzern abgestimmt sind. Protokolle für die Fluoreszenzmarkierung der Mikrotubuli und Aktinfilamente zur effektivsten Visualisierung der Kompositumstrukturierung und -bewegung mittels multispektraler konfokaler Bildgebung sind ebenfalls detailliert. Abschließend werden die Ergebnisse von Datenanalysemethoden vorgestellt, mit denen Nichtgleichgewichtsstruktur, Dynamik und Mechanik quantitativ charakterisiert werden können. Die Nachbildung und Untersuchung dieser abstimmbaren biomimetischen Plattform liefert wertvolle Erkenntnisse darüber, wie gekoppelte motorische Aktivität, Verbundmechanik und Filamentdynamik zu unzähligen zellulären Prozessen von der Mitose über die Polarisation bis hin zur Mechano-Empfindung führen können.
Das Zytoskelett ist ein dynamisches Verbundnetzwerk aus interagierenden Biopolymeren, das Zellen strukturell und mechanisch unterstützt. Assoziierte molekulare Motoren und Bindungsproteine restrukturieren und passen das Zytoskelett an, damit Zellen wachsen, ihre Form ändern, sich versteifen, bewegen und sogar selbst heilen können, was unzählige zelluläre Prozesse ermöglicht, die von Migration und Teilung bis hin zur Mechanosensorik reichen 1,2. Neben seiner Bedeutung in der zellulären Biophysik ist das Zytoskelett auch ein typisches Beispiel für aktive Materie mit potenziellen Materialanwendungen, die von der Wundheilung und Medikamentenabgabe bis hin zur Filtration und weichen Robotik reichen 1,3,4,5,6,7,8,9.
Die beiden Hauptmerkmale, die dem Zytoskelett seine einzigartige strukturelle und mechanische Vielfalt und Multifunktionalität verleihen, sind: 1) seine zusammengesetzte Natur, bestehend aus mehreren interagierenden Proteinfilamenten, wie halbflexiblen Aktinfilamenten und starren Mikrotubuli, sowie ihren zugehörigen Bindungs- und Vernetzungsproteinen 3,5,10; und 2) seine Fähigkeit, die filamentösen Proteine 1,7,11,12,13 kontinuierlich zu restrukturieren, zu bewegen, zu vergröbern und Arbeit auszuführen. Während diese elegante Komplexität es dem Zytoskelett ermöglicht, so unterschiedliche Prozesse wie Zellmotilität, Zytokinese und Wundheilung zu vermitteln 3,6,7,11, behindert sie die Fähigkeit der Forscher, die charakteristischen In-vivo-Eigenschaften des Zytoskeletts in rekonstituierten In-vitro-Systemen zu reproduzieren.
Aktuelle Grenzrekonstitutionsbemühungen konzentrieren sich auf Verbundwerkstoffe aus verschränkten und vernetzten Aktinfilamenten und Mikrotubuli 3,10,14,15,16,17, krafterzeugende Actomyosin-Netzwerke 2,8,18,19,20,21 und aktive Nematika, die von Kinesin-Mikrotubuli angetrieben werden Wechselwirkungen 22,23,24,25,26. Es wurde gezeigt, dass stationäre Aktin-Mikrotubuli-Verbundwerkstoffe emergente mechanische Eigenschaften15,16,27 aufweisen, wie eine verbesserte Filamentmobilität und erhöhte Steifigkeit im Vergleich zu Einkomponentensystemen 27. Studien zu In-vitro-Actomyosin-Systemen haben eine breite Palette von strukturellen und dynamischen Eigenschaften berichtet, die von den Konzentrationen von Aktin, Myosin und Vernetzern abhängen 28,29,30,31. Zum Beispiel unterliegen Actomyosin-Netzwerke bei ausreichender Vernetzung einer großflächigen Kontraktion und Vergröberung 2,28,30,32,33,34,35,36, während Netzwerke ohne Vernetzer einen schnellen, destabilisierenden Fluss und Bruch aufweisen 19,29 . Es wurde berichtet, dass rekonstituierte Mikrotubuli-basierte aktive Nematika, die Cluster von Kinesinmotoren verwenden, um Mikrotubulibündel zu vernetzen und an Mikrotubulibündeln zu ziehen, lang anhaltende turbulente Strömungen, Ausdehnung, Knicken, Brechen und Heilung aufweisen 12,22,23,24,25,37,38,39,40,41, 42,43,44,45,46,47.
In jüngerer Zeit wurde gezeigt, dass Aktin-Mikrotubuli-Komposite, die von Myosin-II-Minifilamenten angetrieben werden, zu einer geordneteren Kontraktion und Netzwerkintegrität führen als die Unordnung des Flusses und Netzwerkbruchs, die Actomyosin-Netzwerke ohne Vernetzer aufweisen 17,26,48. Darüber hinaus wird die Kombination aus Verbundrobustheit und Krafterzeugung optimiert, wenn Aktin und Mikrotubuli in vergleichbaren Konzentrationen vorliegen. Zu den wichtigsten Merkmalen in dieser Region des Formulierungsraums gehören eine verbesserte mechanische Festigkeit 26, eine koordinierte Bewegung von Aktin und Mikrotubuli26, eine stetige anhaltende Kontraktion und eine mesoskalige Umstrukturierung17.
Hier werden Protokolle beschrieben, um co-verschränkte und vernetzte Komposite aus Mikrotubuli und Aktinfilamenten zu entwickeln und abzustimmen, die durch Myosin-II-Minifilamente und Kinesin-Cluster, die auf Aktinfilamente bzw. Mikrotubuli wirken, aus dem Gleichgewicht gebracht werden (Abbildung 1). Die Dynamik, Struktur und Mechanik dieser Klasse von Verbundwerkstoffen können durch die relativen Konzentrationen der Filamente, Motoren und Vernetzer abgestimmt werden, um einen reichen Phasenraum aus advektiver und turbulenter Strömung, isotroper Kontraktion, Beschleunigung, Verzögerung, Entmischung, Versteifung, Entspannung und Bruch zu zeigen. Der Schwerpunkt dieser Arbeit liegt auf der Vorbereitung und Abstimmung dieser Klasse aktiver Zytoskelettkomposite. Um die Forscher beim Benchmarking und der Charakterisierung der beschriebenen aktiven Komposite zu unterstützen, werden jedoch auch effektive Bildgebungsverfahren mit multispektraler konfokaler Mikroskopie detailliert beschrieben. Schließlich werden die Ergebnisse der wichtigsten computergestützten Analysemethoden vorgestellt, mit denen die Dynamik, Struktur und Mechanik der Verbundwerkstoffe gemessen werden kann. Die Forscher werden ermutigt, diese Methoden zu übernehmen, zu denen differentielle dynamische Mikroskopie (DDM), räumliche Bildautokorrelation (SIA) und Partikelbildvelocimetrie (PIV) gehören, da sie optimiert wurden, um die komplexe Dynamik und strukturelle Vielfalt der Verbundwerkstoffe zu charakterisieren 17,26,49.
Die unten beschriebenen Schritte konzentrieren sich auf die Herstellung der Verbundwerkstoffe und deren Abbildung mittels konfokaler Mikroskopie. Protokolle, die die Datenanalyse nach der Erfassung und die Messungen der optischen Pinzette beschreiben, finden Sie in früheren Arbeiten 17,26,48,50 und werden auf Anfrage zur Verfügung gestellt. Alle Materialien sind in der bereitgestellten Materialtabelle aufgeführt.
1. Silanisierte Deckgläser und Objektträger vorbereiten, um die Adsorption von Proteinen an Kammeroberflächen zu verhindern
HINWEIS: Dies ist ein 2-tägiger Prozess. Silanisierte Objektträger können bis zu 1 Monat vor Gebrauch vorbereitet werden.
- Legen Sie Deckgläser Nr. 1 (24 mm x 24 mm) und Objektträger (1 Zoll x 3 Zoll) in ein dafür vorgesehenes Rack, das in den Plasmareiniger passt. Legen Sie das Gestell in den Plasmareiniger und lassen Sie es 20 Minuten laufen.
- Übertragen Sie Deckgläser und Objektträger in ein neues Rack, das nur für die Verwendung mit Silan bestimmt ist, und legen Sie das Gestell in einen Glasbehälter, um die Gläser wie unten beschrieben zu reinigen.
- Deckgläser und Pantoletten für 1 h in 100% Aceton eintauchen. Deckgläser und Pantoletten für 100 min in 100% Ethanol tauchen.
- Tauchen Sie Deckgläser und Pantoletten für 5 min in entionisiertes Wasser (DI). Wiederholen Sie die Reinigungsschritte noch zwei weitere Male.
- Deckgläser und Pantoletten für 15 min in frisch zubereitete 0,1 M KOH tauchen. Deckgläser und Dias für 5 min in frische DI tauchen. Wiederholen Sie diesen Schritt noch zwei weitere Male.
- Lufttrocknen von Deckgläsern und Pantoletten für 10 min. Behandeln Sie gereinigte Deckgläser und Objektträger mit Silan, um hydrophobe Oberflächen wie unten beschrieben zu erzeugen.
HINWEIS: Führen Sie die folgenden Schritte in einem Abzug aus.- Getrocknete Deckgläser und Objektträger in 2% Silan (gelöst in Toluol) für 5 min eintauchen. Verwenden Sie einen Trichter, um Silan zurück in die dafür vorgesehene Flasche zu gießen, um es bis zu fünf Mal wiederzuverwenden.
- Deckgläser und Pantoletten für 5 min in 100% Ethanol eintauchen. Ersetzen Sie Ethanol durch frisches Ethanol. Deckgläser und Dias für 5 min eintauchen.
- Deckgläser und Dias für 5 min in frische DI tauchen. Wiederholen Sie den Ethanol- und DI-Waschschritt zwei weitere Male mit jeweils frischem Ethanol und DI. Lufttrocknen von Deckgläsern und Pantoletten für 10 min.
2. Herstellung eines aktiven Aktin-Mikrotubuli-Komposits, das von Myosin-Minifilamenten angetrieben wird
- Entfernen Sie inaktives Myosin über Aktinfilamentbindung und führen Sie den Pulldown durch Ultrazentrifugation durch , wie unten beschrieben.
- Aktin zu Filamenten polymerisieren. Kombinieren Sie mit einer Präzisionsmikropipette und sterilen Pipettenspitzen in einem Mikrozentrifugenröhrchen: 1,87 μL DI, 1,3 μL 10x G-Puffer, 1,3 μL 10x F-Puffer, 1,63 μL 4 M KCl, 4,53 μL Aktin (47,6 μM) und 1,08 μL 100 μM Phalloidin.
HINWEIS: Um eine ausreichende Polymerisation zu gewährleisten, sollten die Aktinkonzentration und das Aktin:Phalloidin-Molverhältnis 18,4 μM bzw. 2:1 betragen. - Die Lösung vorsichtig auf und ab pipettieren, um zu mischen und dann im Dunkeln für ≥1 h auf Eis zu legen. Ultrazentrifuge auf 4 °C abkühlen. Myosin-Aliquot von -80 °C entfernen und auf Eis legen.
HINWEIS: Führen Sie Schritt 2.2 an dieser Stelle aus, während Aktin polymerisiert. - Nach ≥1 h Aktinpolymerisation werden dem polymerisierten Aktin 1,3 μL 10 mM ATP und 2 μL 19 μM Myosin zugegeben.
HINWEIS: Das Aktin:Myosin-Molverhältnis sollte >5 betragen, um eine ausreichende Entfernung von inaktiven Myosinmotoren (d.h. toten Köpfen) zu gewährleisten. - Pipeten Sie die Lösung vorsichtig auf und ab, um zu mischen. Überführung in ein Ultrazentrifugenröhrchen.
Zentrifugieren bei 4 °C und 121.968 x g für 30 min.
- Aktin zu Filamenten polymerisieren. Kombinieren Sie mit einer Präzisionsmikropipette und sterilen Pipettenspitzen in einem Mikrozentrifugenröhrchen: 1,87 μL DI, 1,3 μL 10x G-Puffer, 1,3 μL 10x F-Puffer, 1,63 μL 4 M KCl, 4,53 μL Aktin (47,6 μM) und 1,08 μL 100 μM Phalloidin.
- Bereiten Sie ein co-verschränktes Verbundnetzwerk aus Aktinfilamenten und Mikrotubuli wie unten beschrieben vor.
HINWEIS: Beginnen Sie 30 Minuten vor dem Myosin-Spindown (Schritt 2.1.4).- Stellen Sie einen Heizblock auf 37 °C ein. Verwenden Sie eine Präzisionsmikropipette und sterile Pipettenspitzen, um einem Mikrozentrifugenröhrchen Folgendes hinzuzufügen: 13,9 μL PEM, 3 μL 1% Tween20, 1,55 μL 47,6 μM Aktin, 0,36 μL 34,8 μM R-Aktin, 0,3 μL 250 mM ATP, 0,87 μL 100 μM Phalloidin, 1,91 μL 5-488-Tubulin, 0,3 μL 100 mM GTP, und 0,75 μL 200 μM Taxol auf ein Gesamtvolumen von 23 μL.
HINWEIS: Die aufgeführten Konzentrationen von Aktin und Tubulin gelten für ein Kompositum mit 2,9 μM Aktin und 2,9 μM Tubulin. Die Gesamtproteinkonzentration beträgt c = c A + c T = 5,8 μM und die molare Aktinfraktion ist c A/(c A + cT) = Φ A = 0,5. Siehe Schritt 2.5, um diese Werte anzupassen. - Die Lösung vorsichtig auf und ab pipen, um sie zu mischen und 1 h lang auf einen lichtgeschützten 37 °C-Heizblock zu legen.
- Stellen Sie einen Heizblock auf 37 °C ein. Verwenden Sie eine Präzisionsmikropipette und sterile Pipettenspitzen, um einem Mikrozentrifugenröhrchen Folgendes hinzuzufügen: 13,9 μL PEM, 3 μL 1% Tween20, 1,55 μL 47,6 μM Aktin, 0,36 μL 34,8 μM R-Aktin, 0,3 μL 250 mM ATP, 0,87 μL 100 μM Phalloidin, 1,91 μL 5-488-Tubulin, 0,3 μL 100 mM GTP, und 0,75 μL 200 μM Taxol auf ein Gesamtvolumen von 23 μL.
- Bereiten Sie Probenkammern für konfokale Bildgebungsexperimente vor, wie unten beschrieben.
HINWEIS: Führen Sie die Schritte 2.1.4 und 2.2.2 während der Wartezeiten aus.- Legen Sie zwei silanisierte Objektträger nebeneinander auf eine Heizplatte (ausgeschaltet), legen Sie zwei Streifen thermoplastischer Siegelfolie über die Objektträger im Abstand von ~3 mm und legen Sie zwei silanisierte Deckgläser über die thermoplastische Siegelfolie, um eine Probenkammer zu bilden.
- Drehen Sie die Heizplatte auf niedrige Stufe, bis Deckgläser fest mit Objektträgern mit geschmolzener thermoplastischer Siegelfolie verbunden sind (~1-2 min). Drücken Sie mit gleichmäßigem Druck nach unten, um die Verklebung unter Beibehaltung eines Abstands von ~ 100 μm zwischen den beiden Oberflächen sicherzustellen.
- Entfernen Sie die Kammern und schalten Sie die Heizplatte aus. Etikettenkammern mit (+) und (-). Die (+) Kammer ist für die aktive Probe (mit Myosin) und die (-) Kammer wird die Kontrolle sein (kein Myosin). Stellen Sie sicher, dass jede Kammer ≤10 μL Flüssigkeit aufnehmen kann.
- Bereiten Sie die Beispiele wie unten beschrieben vor.
HINWEIS: Es ist wichtig, diesen Schritt unmittelbar nach Abschluss der Schritte 2.1 und 2.2 abzuschließen.- Die Myosin-Aktin-Probe wird vorsichtig aus der Ultrazentrifuge entnommen (Schritt 2.1.4) und sofort die oberen 7,5 μl des Überstands aufgeblasen und in ein neues Mikrozentrifugenröhrchen überführt.
- Nehmen Sie die Aktin-Mikrotubuli-Probe aus dem Hitzeblock und mischen Sie vorsichtig 1,5 μL 10x D-Glucose, 1,5 μL 10x GOC und 1,5 μL 1 mM Blebbistatin hinein. Die Lösung wird in zwei 13,7-μL-Aliquots aufgeteilt und als (+) und (-) bezeichnet.
- 1,28 μL des Überstands aus Schritt 2.4.1 zu (+) aliquot mischen. Mischen Sie 1,28 μL DI zum (-) Aliquot. Jede Lösung wird langsam durch Kapillarwirkung in die entsprechende Kammer (Schritt 2.3) fließen lassen. Achten Sie darauf, keine Luftblasen in den Kanal einzuführen.
- Versiegeln Sie die beiden offenen Enden jedes Kanals mit schnell trocknendem Epoxid- oder UV-Kleber. Stellen Sie sicher, dass der Klebstoff vollständig trocken ist, bevor Sie ihn auf das Mikroskop legen. Bild sofort wie in Schritt 3 beschrieben.
HINWEIS: UV-Kleber ist vorteilhaft, da er bei UV-Einwirkung fast sofort aushärtet. Da Blebbistatin jedoch UV-empfindlich ist, ist es wichtig, den Klebstoff (an den Rändern der Probenkammer) nur lokal mit einem kleinen UV-Stab zu beleuchten, um eine Deaktivierung des Blebbistatins zu vermeiden.
- Optional: Variieren Sie die Proteinkonzentrationen, um die Dynamik und Struktur der Verbundwerkstoffe abzustimmen.
HINWEIS: Die folgenden Schritte werden als Änderungen an den oben genannten Schritten vorgeschlagen, um die Konzentrationen von Aktin, Mikrotubuli und Myosin zu variieren, falls gewünscht.- Führen Sie die oben beschriebenen Schritte aus, mit Ausnahme der folgenden Änderungen in den Schritten 2.2.1 und 2.4.3.
- Um die Konzentrationen von Aktin und Mikrotubuli zu variieren und dadurch c und Φ A einzustellen, wird das Volumen von Aktin, R-Aktin und 5-488-Tubulin, die in Schritt 2.2.1 verwendet werden, wie gewünscht erhöht oder verringert26. Wenn Sie die Aktinkonzentration variieren, passen Sie die molaren R-Aktin- und Phalloidinkonzentrationen proportional an, um die gleichen molaren Verhältnisse mit Aktin aufrechtzuerhalten. Stellen Sie das Volumen von PEM so ein, dass das Endvolumen der Mischung 23 μL bleibt. Alle anderen Komponentenvolumina und -konzentrationen bleiben gleich.
- Um die Myosinkonzentration zu variieren, wird die Menge des Myosins, das dem (+)-Aliquot in Schritt 2.4.3 zugesetzt wird, wie gewünscht angepasst. Passen Sie das DI-Volumen an, das dem (-) Aliquot hinzugefügt wird, entsprechend. Passen Sie das PEM-Volumen in Schritt 2.2.1 an, um die Zunahme oder Abnahme des Myosin- (+) und DI-Volumens (-) zu berücksichtigen, und stellen Sie sicher, dass das Endvolumen jeder Probe ((+) und (-)) 14,98 μl beträgt.
3. Bildgebung und Charakterisierung aktiver Komposite mittels konfokaler Mikroskopie
- Um in Schritt 2 hergestellte Actomyosin-Mikrotubuli-Verbundwerkstoffe abzubilden, verwenden Sie ein konfokales Laserscanning-Mikroskop (LSCM) oder ein ähnliches Mikroskop mit einem 60x 1,4 NA Öl-Tauchobjektiv. Um Aktinfilamente und Mikrotubuli gleichzeitig in separaten Fluoreszenzkanälen sichtbar zu machen, verwenden Sie einen 561 nm Laser mit 565/591 nm Anregungs-/Emissionsfiltern und einen 488 nm Laser mit 488/525 nm Anregungs-/Emissionsfiltern.
- Platzieren Sie die Probenkammer so auf dem Mikroskop, dass der Kontrollkanal direkt über dem Objektiv positioniert ist. Stellen Sie sicher, dass zwischen dem Objektiv und dem Deckglas eine Ölschnittstelle vorhanden ist.
- Verwenden Sie die Bühnensteuerungen, um das Kontrollkomposit in den Fokus zu bringen, und suchen Sie dann beide Oberflächen der Probenkammer. Verschieben Sie die z-Position in die Mitte der Probenkammer. Überprüfen Sie, ob klare filamentöse Netzwerke vorhanden sind, wie in Abbildung 2 dargestellt.
- Visualisieren Sie weiterhin die Kontrollkammer und passen Sie die Intensität jedes Lasers an, um die gleichzeitige Visualisierung von Aktinfilamenten und Mikrotubuli zu ermöglichen. Halten Sie die niedrigstmögliche Laserintensität aufrecht, um Photobleiche (häufiger im Aktinkanal) und Durchbluten (typischerweise von den Mikrotubuli in den Aktinkanal) zu verhindern.
- Um die inaktive Kontrollprobe zu charakterisieren, sammeln Sie drei Zeitreihen (Videos) mit 256 x 256 Quadratpixelbildern (213 μm x 213 μm) bei 2,65 fps für insgesamt ≥1000 Bilder. Sammeln Sie jede Zeitreihe in einem anderen Bereich der Probenkammer, der durch ≥500 μm getrennt ist. Stellen Sie sicher, dass es eine minimal erkennbare Bewegung und keine Strömung oder Umstrukturierung gibt.
- Schalten Sie den 488-nm-Laser aus und verwenden Sie die Bühnensteuerung, um zur (+)-Kammer zu gelangen.
- Visualisieren Sie mit dem 568-nm-Laser die Mikrotubuli im (+)-Kanal, um eine ordnungsgemäße Netzwerkbildung sicherzustellen (Abbildung 2), und identifizieren Sie das axiale Zentrum der Probenkammer (das sich von der mittleren z-Position der Kontrollkammer unterscheiden kann).
- Schalten Sie den 488-nm-Laser ein und wiederholen Sie Schritt 3.5 oben mit den folgenden Änderungen. Erfassen Sie Zeitreihen für bis zu 45 Minuten und stoppen Sie die Aufnahme, wenn sich die Probe entweder aus dem Sichtfeld bewegt, Brüche oder Fotobleiche auftreten. Zeichnen Sie 5-10 Zeitreihen auf und verfolgen Sie den Zeitpunkt, zu dem jede Zeitreihe relativ zum Beginn der ersten Zeitreihe beginnt.
- Analysieren Sie Daten mit DDM, SIA und PIV, wie in Abbildung 3, Abbildung 4, Abbildung 5 und zuvor 17,48,50,51 beschrieben.
HINWEIS: Der 488-nm-Laser aktiviert lokal die Myosin-ATPase-Aktivität durch Deaktivierung von Blebbistatin, daher sollte er nur zu Beginn der Datenerfassung so eingeschaltet werden, dass t = 0 am Anfang der Zeitreihe steht. Diese Erfassungsparameter sind wie bisher für die Analyse der differentiellen dynamischen Mikroskopie (DDM) optimiert26.
4. Herstellung aktiver Aktin-Mikrotubuli-Verbundwerkstoffe, die von Kinesinmotoren angetrieben werden
HINWEIS: Die folgenden Schritte erzeugen Aktin-Mikrotubuli-Verbundwerkstoffe, die durch Kinesinmotoren oder eine Kombination aus Kinesin und Myosin50 aus dem Gleichgewicht gebracht werden.
- Bereiten Sie Kinesin- und Myosinmotoren wie unten beschrieben vor.
- Wenn Sie Myosin einbeziehen, führen Sie Schritt 2.1 aus.
- Um Kinesin-Motorcluster zu bilden, die Kräfte zwischen Mikrotubulipaaren binden und ausüben, verwenden Sie eine Mikropipette und sterile Pipettenspitzen, um einem sterilen 1,5-ml-Mikrozentrifugenröhrchen Folgendes hinzuzufügen:1,16 μL PEM, 2,74 μL 8,87 μM Kinesin-Dimere, 7,29 μL 83,3 μM NeutrAvidin, 0,81 μL 2mM DTT . Mischen Sie vorsichtig, indem Sie die Lösung nach oben und unten pipettieren und 30 min bei 4 °C lichtgeschützt inkubieren (verwenden Sie ein schwarzes Mikrozentrifugenröhrchen oder wickeln Sie sie in Folie).
HINWEIS: Das molare Verhältnis von Kinesin-Dimeren zu NA beträgt 1:25.
- Führen Sie Schritt 2.3 aus, um Probenkammern vorzubereiten, und stellen Sie drei statt zwei Kammern her. Führen Sie diesen Schritt während der Kinesinkubation (Schritt 4.1.2) und der Myosin-Ultrazentrifugation (Schritt 4.1.1) durch.
- Bereiten Sie ein co-verschränktes Verbundnetzwerk aus Aktinfilamenten und Mikrotubuli vor.
- Stellen Sie den Heizblock auf 37 °C ein. Verwenden Sie eine Mikropipette und sterile Pipetenspitzen, um einem sterilen 1,5-ml-Mikrozentrifugenröhrchen Folgendes hinzuzufügen: 3,21 μL PEM, 4,5 μL 1% Tween20, 2,18 μL 47,6 μM Aktin, 3,46 μL 5-R-Tubulin, 4,5 μL 100 mM ATP, 4,5 μL 10 mM GTP, 1,13 μL 200 μM Taxol und 1,57 μL 20 μM 488-Phalloidin. Stellen Sie sicher, dass das Gesamtvolumen 25 μL beträgt.
- Die Lösung vorsichtig auf und ab pipen, um sie zu mischen und 1 h lang auf den lichtgeschützten 37 °C-Heizblock zu legen. Nehmen Sie das Röhrchen aus dem Kühlblock und mischen Sie mit einer Mikropipette 0,84 μL 100 μM Phalloidin vorsichtig ein. 5-10 min bei Raumtemperatur lichtgeschützt inkubieren.
HINWEIS: Die Zugabe von Phalloidin in diesem Schritt und nicht in Schritt 4.3.1 verbessert die Fluoreszenzmarkierung von Aktinfilamenten, da 488-Phalloidin nicht mit unmarkiertem Phalloidin um Aktinbindungsstellen konkurrieren muss.
- Bereiten Sie aktive Komposite für die konfokale Bildgebung vor.
- 1,13 μL 200 μM Blebbistatin, 1,35 μL 10x Glu und 1,35 μL 10x GOC in die Lösung aus Schritt 4.3.2 geben und vorsichtig durch Pipettieren auf und ab mischen. Die Lösung wird in drei 10-μL-Aliquots aufgeteilt und als (K), (K+M) und (-) bezeichnet.
- Mischen Sie 2,54 μL Myosin aus Schritt 2.1.4 zum (K+M)-Aliquot. 2,54 μL PEM zu (K) und (-) Aliquots mischen.
- Verwenden Sie eine Mikropipette und sterile Pipettenspitzen, um 2,5 μL Kinesincluster aus Schritt 4.1.2 zu (K) und (K+M) Aliquots hinzuzufügen. Zum Mischen auf und ab pipen. Mischen Sie 2,5 μL PEM zu (-) mit der gleichen Technik.
HINWEIS: Die aufgeführten Konzentrationen von Aktin und Tubulin gelten für ein Kompositum mit 2,32 μM Aktin und 3,48 μM Tubulin. Die Gesamtproteinkonzentration beträgt c = c A + c T = 5,8 μM und die molare Aktinfraktion ist c A/(c A + cT) = Φ A = 0,4. Kinesin- und Myosinkonzentrationen betragen 0,35 μM bzw. 0,47 μM. In Schritt 2.5 finden Sie allgemeine Richtlinien zum Anpassen von cA, cT, c und ΦA. - Mit einer Mikropipette wird jede Lösung langsam durch Kapillarwirkung in den entsprechenden Kanal der vorbereiteten Probenkammern (Schritt 4.2) fließen. Drücken Sie die Pipette sehr langsam und vorsichtig nach unten, um keine Luftblasen in den Kanal einzuführen.
- Versiegeln Sie die beiden offenen Enden jedes Kanals mit schnell trocknendem Epoxidharz oder UV-härtendem Kleber. Stellen Sie sicher, dass der Klebstoff vollständig trocken ist, bevor Sie ihn auf das Mikroskop legen.
HINWEIS: Es ist wichtig, dass dieser Schritt schnell durchgeführt wird, um die Zeit zu minimieren, in der das Kinesin wirkt, ohne überwacht zu werden. Aus diesem Grund wird Epoxidharz empfohlen, das in 1 min (statt 5 oder 10 min) aushärtet. UV-härtender Klebstoff ist in dieser Hinsicht vorteilhaft, da er bei UV-Einwirkung nahezu sofort aushärtet.
- Bildvorbereitende Muster sofort nach Schritt 3, mit Ausnahme der folgenden wichtigen Änderungen. Da Kinesin nicht durch Lichtaktivierung gesteuert wird, beginnt es sofort nach Schritt 4.4.3 zu arbeiten, also markieren Sie diese Zeit als t = 0. Um das Komposit so nah wie möglich am anfänglichen inaktiven Zustand (t = 0) abzubilden, bilden Sie zuerst die Kanäle (K) und (K+M) ab und notieren Sie die Zeit, die zwischen Schritt 4.4.3 und dem Beginn der Datenerfassung (Schritt 3.8) verstrichen ist. In der Praxis beträgt diese verstrichene Zeit ~5 min.
5. Einbau passiver Vernetzer in aktive Verbundwerkstoffe
HINWEIS: Diese Schritte beschreiben, wie biotinylierte Aktin- und Tubulin-Untereinheiten und NeutrAvidin (NA) verwendet werden, um Aktin passiv zu Aktin (A-A) oder Mikrotubuli zu Mikrotubuli (M-M) in den in Schritt 4 beschriebenen aktiven Kompositen zu vernetzen.
- A-A- oder M-M-Vernetzerkomplexe mit biotinylierten Proteinen (Biotin-Aktin oder Biotin-Tubulin), NA und Biotin im Verhältnis 2:2:1 Biotin-Aktin/Tubulin:Biotin:NA herstellen. Starten Sie diesen Prozess vor Schritt 4 .
- Verwenden Sie für A-A-Vernetzer eine Mikropipette und sterile Pipettenspitzen, um 2 μL 11,6 μM Biotin-Aktin, 1,39 μL 8,33 μM NA, 2,27 μL 1,02 μM Biotin und 4,34 μL PEM in ein Mikrozentrifugenröhrchen zu geben. Mischen Sie vorsichtig, indem Sie auf und ab pipettieren.
- Verwenden Sie für M-M-Vernetzer eine Mikropipette und sterile Pipettenspitzen, um 1,86 μL 4,55 μM Biotin-Tubulin, 1,11 μL 8,33 μM NA, 1,82 μL 1,02 μM Biotin und 5,21 μL PEM in ein Mikrozentrifugenröhrchen zu geben. Mischen Sie vorsichtig, indem Sie auf und ab pipettieren.
- Wickeln Sie das/die Röhrchen(e) aus Schritt 5.1.1 und/oder 5.1.2 in thermoplastische Dichtungsfolie, um eine wasserdichte Abdichtung zu erzeugen. In ein Flotationsfloß in einem temperaturgeregelten Sonikatorbad auf 4 °C geben.
- 90 min bei 4 °C beschallen. In der Praxis ist es am besten, den Ultraschall in einen kalten Raum zu stellen und dem Ultraschallbad Eisbeutel hinzuzufügen, um eine niedrige Temperatur aufrechtzuerhalten.
- Um Vernetzerkomplexe für die Bildgebung in Proben zu integrieren, folgen Sie Schritt 4.3 und modifizieren Sie Schritt 4.3.1 wie unten beschrieben für A-A-Vernetzung (Schritt 5.2.1) oder M-M-Vernetzung (Schritt 5.2.2).
- Für die A-A-Vernetzung kombinieren Sie Folgendes in einem Mikrozentrifugenröhrchen: 1,94 μL PEM, 4,50 μL 1% Tween20, 2,18 μL 47,6 μM Aktin, 3,46 μL 45,5 μM 5-R-Tubulin, 1,13 μL A-Vernetzer (Schritt 5.1.1), 4,50 μL 100 mM ATP, 4,50 μL 10 mM GTP, 1,13 μL 200 μM Taxol, und 1,57 μL 20 μM 488-Phalloidin. Stellen Sie sicher, dass das Gesamtvolumen 25 μL beträgt.
- Für die M-M-Vernetzung kombinieren Sie Folgendes in einem Mikrozentrifugenröhrchen: 1,97 μL PEM, 4,50 μL 1% Tween20, 2,18 μL 47,6 μM Aktin, 3,76 μL 45,5 μM 5-R-Tubulin, 1,13 μL 1:4 Verdünnung von M-M-Vernetzern (Schritt 5.1.2), 4,50 μL 100 mM ATP, 4,50 μL 10 mM GTP, 1,13 μL 200 μM Taxol, und 1,57 μL 20 μM 488-Phalloidin. Stellen Sie sicher, dass das Gesamtvolumen 25 μL beträgt.
- Führen Sie die Schritte 4.3.2-4.5 mit den spezifischen Konzentrationen für ein Vernetzer-Aktin-Mol-Verhältnis von RA = 0,02 und ein Vernetzer:Tubulin-Molverhältnis von RT = 0,005 aus. Diese RA- und RT-Werte ergeben ähnliche Längen zwischen Vernetzern entlang Aktinfilamenten und Mikrotubuli (d A 60 nm und d MT 67 nm), geschätzt unter Verwendung von d A = I Monomer/2R A
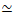 , wobeiI-Monomer die Länge eines Aktinmonomers ist, und dMT
, wobeiI-Monomer die Länge eines Aktinmonomers ist, und dMT 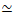 = I-Ring/26RT, wobeiI-Ring die Länge eines Rings von 13 Tubulinen15 ist. 17.
= I-Ring/26RT, wobeiI-Ring die Länge eines Rings von 13 Tubulinen15 ist. 17.
Um die erfolgreiche Herstellung aktiver Verbundwerkstoffe (Abbildung 1) zu bestimmen und deren Dynamik und Struktur zu charakterisieren, wird ein Laser-Scanning-Fluoreszenzmikroskop mit mindestens zwei Fluoreszenzkanälen verwendet, um die Aktinfilamente und Mikrotubuli gleichzeitig sichtbar zu machen (Abbildung 2 und Abbildung 6). Alle Aktinfilamente und Mikrotubuli in den Verbundwerkstoffen sind spärlich markiert, anstatt in Tracer-hellen Filamenten zu dotieren, wie es oft in In-vitro-Studien der Fall ist. Diese Methode stellt sicher, dass die gemessene Dynamik und Struktur repräsentativ für den Verbundwerkstoff selbst sind und nicht für die Tracer, die unter anderen Bedingungen als die Verbundwerkstoffe gebildet werden. Aus diesem Grund können einzelne Aktinfilamente und Mikrotubuli typischerweise nicht aufgelöst werden, sondern Bilder zeigen die mesoskalige Netzwerkstruktur (Abbildung 2 und Abbildung 6).
Dieser Markierungsansatz wurde für räumliche Bildautokorrelation (SIA) und differentielle dynamische Mikroskopie (DDM) optimiert, die die Dynamik und Struktur im reziproken Fourierraum untersuchen (Abbildung 4, Abbildung 5 und Abbildung 8)52,53,54,55. Die Partikelbild-Velocimetrie (PIV) kann auch zur Darstellung und Charakterisierung von Dynamik und Strömungsfeldern verwendet werden (Abbildung 3 und Abbildung 7), erfordert jedoch Pixel-Binning (geringere räumliche Auflösung) und größere Verzögerungszeitschritte (niedrigere zeitliche Auflösung) als SIA und DDM, um fehlerhafte Vektoren zu eliminieren, die durch Rauschen in den dichten, signalarmen Bildern entstehen. Dennoch wird PIV zur qualitativen Untersuchung von Strömungsfeldern und zur Bestätigung der DDM-Ergebnisse empfohlen (Abbildung 4 und Abbildung 8)26,50.
Die Probencharakterisierung der beschriebenen Netzwerke unter Verwendung dieser Analysen (d.h. DDM, SIA, PIV) wird bereitgestellt, um den Forschern zu helfen, ähnliche Analysen zum Benchmarking und zur Charakterisierung ihrer Proben anzuwenden. Detaillierte Beschreibungen dieser Techniken würden jedoch den Rahmen dieser Arbeit sprengen. Detaillierte Beschreibungen der Durchführung von DDM auf diesen und anderen ähnlichen Systemen, einschließlich benutzerfreundlichen Python-Codes, finden Sie in früheren Arbeiten 17,26,49,50 und den darin enthaltenen Referenzen. Für Einzelheiten zur Durchführung von SIA und PIV auf den hier beschriebenen Systemen wird der Leser auf frühere Arbeiten17,50 verwiesen.
Mehrere Kontrollen, die unten beschrieben werden, sollten durchgeführt werden, um sicherzustellen, dass die Verbundwerkstoffe wie erwartet funktionieren. Ein Komposit ohne Myosin oder Kinesin sollte im Wesentlichen statisch mit minimalen thermischen Fluktuationen oder Drift erscheinen. Aktinfilamente und Mikrotubuli sollten miteinander verschränkt und homogen verteilt erscheinen, mit minimaler Bündelung, Aggregation oder Phasentrennung von Aktin und Mikrotubuli über ein Sichtfeld von ~200 μm x 200 μm (Abbildung 2, ganz links)17. Ein ähnliches Ergebnis sollte man für Komposite erwarten, die Myosin enthalten, aber nicht 488 nm Licht ausgesetzt sind (um das Blebbistatin zu deaktivieren).
Nach Einbau von Myosin und Exposition gegenüber 488 nm Licht durchlaufen die Verbundwerkstoffe eine Kontraktion, die für Aktin und Mikrotubuli weitgehend isotrop und ähnlich ist, wie in Mikroskopbildern vor und nach der Myosinaktivität (Abbildung 2) sowie in entsprechenden PIV-Flussfeldern für unterschiedliche Zeiten während der Aktivität zu sehen ist (Abbildung 3). Um festzustellen, ob die Bewegung ballistisch, diffusiv, subdiffusiv usw. ist, wird die charakteristische Dekorrelationszeit τ(q), die aus DDM bestimmt wird, als Funktion des Wellenvektors (d.h. des reziproken Raums) ausgewertet. Siehe wie zuvor ausführlich beschrieben 17,26,49. Abbildung 4 zeigt auch, wie DDM zur Charakterisierung dieser Verbundwerkstoffe verwendet wird. Die Skalierung τ(q)~1/vq β mitβ = 1 zeigt eine ballistische Bewegung mit der Geschwindigkeit v an. Als Referenz stellt β = 2 die Diffusionsdynamik dar, wobei v der Diffusionskoeffizient ist. Alle aktiven Verbundwerkstoffe weisen eine ballistische Skalierung auf (Abbildung 4A) mit Geschwindigkeiten, die durch die Konzentrationen von Aktin und Myosin abgestimmt sind (Abbildung 4B) und die während der Aktivität zeitlich variieren können, entweder beschleunigend oder verlangsamend (Abbildung 4C, D).
Netzwerkumstrukturierung und Clusterbildung, sichtbar in Abbildung 2 und deutlicher für höhere Aktin- und Myosinkonzentrationen, können mit SIA charakterisiert werden, wie in Abbildung 5 dargestellt und zuvorbeschrieben 17,48,50. Kurz gesagt, eine Korrelationslänge ξ, die ein Maß für die charakteristische Größe von Features in einem Bild ist, kann bestimmt werden, indem jede räumliche Intensitäts-Autokorrelationskurve g(r) an eine Exponentialfunktion des Abstands r zwischen Pixeln angepasst wird. Größere g(r)-Peaks, die über längere Strecken bestehen bleiben, deuten auf größere strukturelle Merkmale hin (z.B. Bündelung, Clusterbildung der einzelnen Filamente). Wie in Abbildung 5 gezeigt, spiegelt sich bei höheren Aktinfraktionen und Myosinkonzentrationen eine signifikante Umstrukturierung und Aggregation im Anstieg von ξ im Zeitverlauf wider.
Die viskoelastischen Eigenschaften und die nichtlineare mechanische Reaktion der aktiven Verbundwerkstoffe können auch mittels optischer Pinzettenmikrorheologie (OTM) gemessen werden. Protokolle und repräsentative Ergebnisse für diese Experimente liegen jedoch außerhalb des Rahmens dieser Arbeit. Interessierte Leser werden auf frühere Arbeiten48,56 verwiesen, die ausführlich beschreiben, wie OTM-Messungen durchzuführen sind und welche Ergebnisse zu erwarten sind.
Mit dem oben beschriebenen Programm von Experimentier- und Analysewerkzeugen wird im folgenden Abschnitt beschrieben, wie sich Dynamik und Struktur ändern, wenn Kinesinmotoren und Biotin-NA-Vernetzer in die Verbundwerkstoffe eingebaut werden (Abbildung 6, Abbildung 7 und Abbildung 8). Abbildung 6 zeigt repräsentative konfokale Bilder von Kompositen, die entweder von Kinesin (K) oder Kinesin und Myosin (K+M) angetrieben werden, mit und ohne passive Vernetzung (XL) von Aktinfilamenten oder Mikrotubuli.
Der Einbau von Kinesin in Verbundwerkstoffe führt zunächst zu einer ähnlichen Dynamik und Umstrukturierung wie myosingetriebene Verbundwerkstoffe, wie in der oberen Reihe von Abbildung 7 (Klasse 1) zu sehen ist. Die Dynamik geht jedoch typischerweise in eine großräumige anisotrope Strömung (Abbildung 7 mittlere Reihe, Klasse 2), Beschleunigung und Verzögerung (Abbildung 7 untere Reihe, Klasse 3) über. Diese Merkmale koppeln nach 5-30 min mit mesoskaligem Clustering und Aggregation (Abbildung 6 und Abbildung 8B). PIV-generierte Strömungsfelder und zeitliche Farbkarten in Abbildung 7 zeigen Beispiele für isotrope Umstrukturierung (Klasse 1, oberes Bild), gerichtete Strömung (Klasse 2, mittlere Felder) und bidirektionale Beschleunigung (Klasse 3, untere Felder).
Die Geschwindigkeiten von Aktin und Mikrotubuli zu unterschiedlichen Zeitpunkten während der Aktivität, die über Anpassungen an τ(q)-Kurven bestimmt werden, veranschaulichen die Beschleunigung gefolgt von der Verzögerung (Abbildung 8), die von der Vernetzung abhängt. Wie auch in Abbildung 8 gezeigt, wenn beide Motorproteine eingebaut sind, ist die Dynamik tatsächlich langsamer als bei reinen Kinesin-Kompositen, und es kommt zu einem verzögerten Beginn des mesoskaligen Flusses. Myosin unterstützt auch eine homogenere gegenseitige Durchdringung von Aktin- und Mikrotubuli-Netzwerken während der gesamten Aktivitätsdauer sowie eine geringere Aggregation und Umstrukturierung. Diese Effekte sind in den Bildern in Abbildung 6 zu sehen und werden durch die über SIA berechneten zeitvariablen Korrelationslängen quantifiziert, die in Gegenwart von Myosin im Allgemeinen kleiner sind (Abbildung 8B).
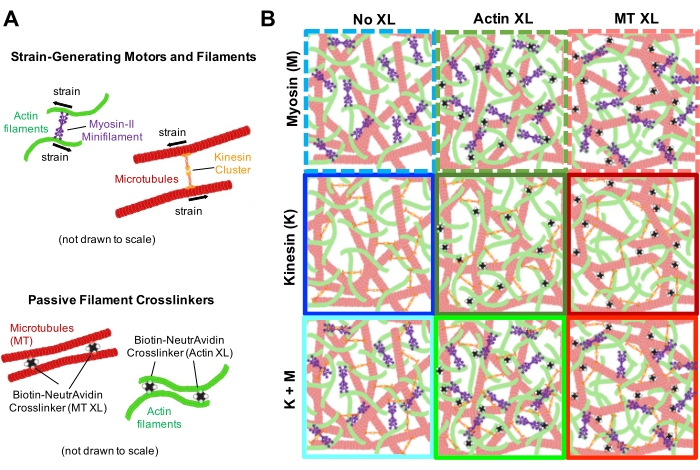
Abbildung 1. Design und Charakterisierung von aktiven Aktin-Mikrotubuli-Verbundwerkstoffen mit mehreren krafterzeugenden Motoren und passiven Vernetzern. (A) Aktinmonomere und Tubulindimere werden bei molaren Konzentrationenc A und c T von 0,73-11,6 μM und molaren Fraktionen von Aktin Φ A = c A/ (c A + c T) = 0, 0,25, 0,5, 0,75 und 1 co-verschränkte Netzwerke von Aktinfilamenten (grün) und Mikrotubuli (rot) copolymerisiert. Passive Vernetzung wird unter Verwendung von NA erreicht, um biotinylierte Aktinfilamente (Aktin XL) oder Mikrotubuli (MT XL) bei Vernetzer-Protein-Molverhältnissen von R A= 0,01-0,08 bzw. RMT= 0,001-0,01 für Aktin bzw. Mikrotubuli zu verknüpfen. Myosin-II-Minifilamente (violett) und Kinesin-Cluster (orange) drücken und ziehen bei Konzentrationen von c M = 0,12 - 0,48 μM und cK = 0,2 - 0,7 μM auf die Filamente, um die Verbundwerkstoffe aus dem stationären Zustand zu treiben. (B) Schematische Darstellung des Formulierungsraums. Myosin-II-Minifilamente (M), Kinesin-Cluster (K) oder beide Motoren (K + M) werden in Verbundwerkstoffe ohne passive Vernetzer (No XL), Aktin-Aktin-Vernetzungen (Aktin XL) und Mikrotubuli-Mikrotubuli-Vernetzungen (MT XL) eingebaut. Alle Cartoons sind nicht maßstabsgetreu gezeichnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
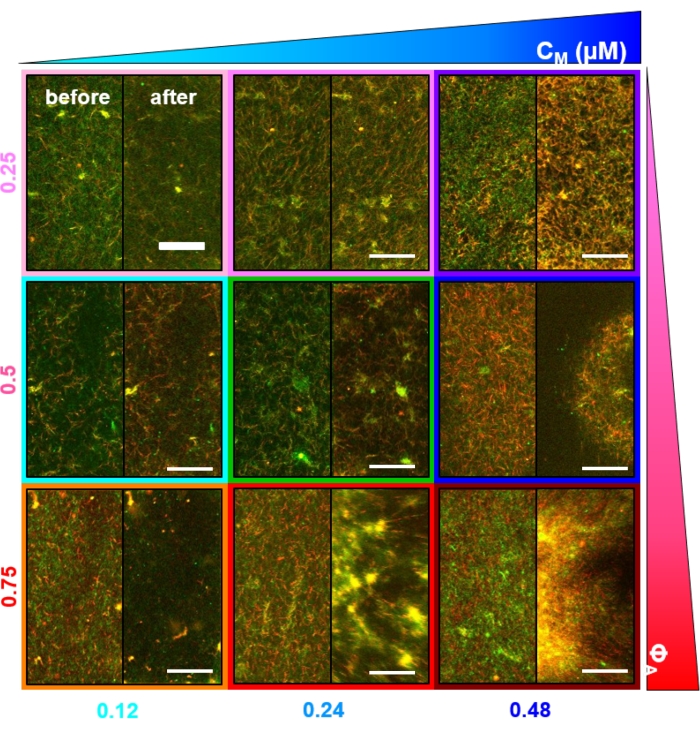
Abbildung 2. Zweifarbige konfokale Bildgebung von Myosin-getriebenen Zytoskelett-Kompositen mit unterschiedlichen Myosinkonzentrationen cM und molaren Aktinfraktionen ΦA. (A) 256 x 128 Quadratpixel (212 x 106 μm2) zweifarbige konfokale Mikroskopiebilder zeigen, wie Komposite aus Aktinfilamenten (grün) und Mikrotubuli (rot) über myosinmotorische Aktivität neu angeordnet werden. Es sind keine Kinesinmotoren oder passive Vernetzer vorhanden. In jedem Panel werden Bilder gezeigt, die zu Beginn (links, vorher) und am Ende (rechts, nachher) der 45-minütigen Myosinaktivierung (durch Beleuchtung mit 488 nm Licht zur Deaktivierung von Blebbistatin) aufgenommen wurden. Die Paneele sind geordnet, indem die molare Konzentration von Myosin (cM) von links nach rechts erhöht und der molare Anteil des Aktins (ΦA) von oben nach unten erhöht wird. Die Farben, die jedes Bedienfeld umreißen, entsprechen der in Abbildung 4 und Abbildung 5 verwendeten Farbcodierung. Maßstäbe sind 50 μM. Um Dynamik und Struktur für die Analyse bestmöglich zu erfassen, verwenden wir Bildraten von 1-5 fps, ROIs mit 50-250 μm Seiten und Zeitreihendauern von 5-45 min, abhängig von der Kontraktions- und Umlagerungsrate. Panels, in denen die Vorher-Nachher-Bilder ähnlich aussehen, weisen auf eine minimale Umstrukturierung hin, wie in den rosa-, magenta- und cyanfarbenen Panels zu sehen ist. Kleinräumige Clusterbildung, die durch erhöhte Heterogenität und das Vorhandensein heller punktförmiger Merkmale belegt wird, ist in den orangen, grünen und roten Feldern zu sehen. Eine großflächige Kontraktion, die als gleichmäßig schrumpfendes Netzwerk angesehen wird, ist in den blauen und violetten Feldern offensichtlich. Diese Zahl wurde gegenüber Referenznummer17 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
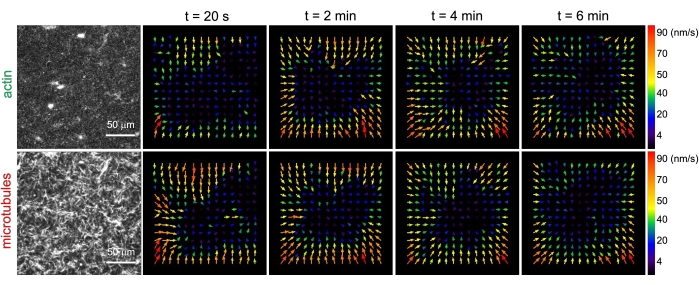
Abbildung 3. Die Particle Image Velocimetry (PIV) zeigt, dass die Actomyosinaktivität eine koordinierte kontraktile Dynamik von Aktin und Mikrotubuli in co-verschränkten Kompositen auslöst. PIV-Strömungsfelder für Aktin (obere Reihe) und Mikrotubuli (untere Reihe) in einem Myosin-getriebenen Komposit mit (ΦA, cM) = (0,5, 0,24) zu steigenden Zeiten während einer 6-minütigen Zeitreihe. Flow-Felder wurden mit dem Fiji/ImageJ PIV-Plugin mit einer Verzögerung von 20 s und 2 Pixel x 2 Pixel Binning generiert. Sowohl Aktin als auch Mikrotubuli zeigen während der gesamten Filmdauer eine gleichmäßige Bewegung, die auf den mittleren Bereich des Sichtfeldes gerichtet ist. Maßstäbe in allen Bildern sind 50 μm. Unterschiedliche Pfeilfarben entsprechen unterschiedlichen Geschwindigkeiten, wie in der Farbskala rechts neben den Vektorfeldern angegeben. Diese Zahl wurde gegenüber Referenznummer26 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
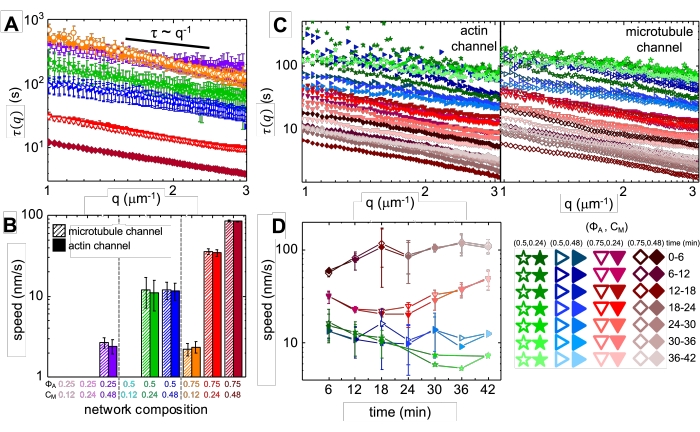
Abbildung 4. Die zeitaufgelöste differentielle dynamische Mikroskopie (DDM) misst die Bewegungsgeschwindigkeit und -art von Aktin und Mikrotubuli in aktiven Verbundwerkstoffen. (A) DDM wird auf Mikrotubuli (obere, offene Symbole) und Aktinkanälen (unten, gefüllte Symbole) von Zeitreihen durchgeführt, um charakteristische Zerfallszeiten τ vs Wellenzahl q sowohl für Aktin (gefüllte Symbole) als auch für Mikrotubuli (offene Symbole) zu bestimmen, wie zuvor beschrieben17,26. Alle Kurven folgen der τ ~ q-1-Skalierung, die eine ballistische Bewegung anzeigt, mit Geschwindigkeiten v, die über Anpassungen an τ(q) = (vq)-1 bestimmt werden. Höhere Geschwindigkeiten entsprechen kleineren τ(q)-Werten für ein beliebiges q. Symbolfarben und -formen entsprechen den in B dargestellten Kombinationen (ΦA, cM). (B) Die Kontraktionsgeschwindigkeiten v werden durch Anpassungen an τ(q)-Kurven bestimmt, die über alle Verzögerungszeiten für die Dauer jeder 45-minütigen Zeitreihe gemittelt werden. (C) Time-resolved DDM (trDDM) quantifiziert, wie sich die Dynamik im Laufe der Zeit ändert, indem τ(q) für Aktin (gefüllte Symbole, links) und Mikrotubuli (offene Symbole, rechts) für aufeinanderfolgende 6-Minuten-Intervalle (gekennzeichnet durch verschiedene Schattierungen derselben Farbe) während der 45-minütigen Aktivierungszeit ausgewertet wird. trDDM wird für jede Kombination (ΦA, cM) (gekennzeichnet durch verschiedene Symbole und Farben) durchgeführt, wie in der Legende unten rechts beschrieben. τ(q)-Kurven in C folgen ähnlichen Skalierungen und Trends wie die in A, zeigen aber auch Zeitabhängigkeit für bestimmte (Φ A, cM) Zusammensetzungen, insbesondere für Φ A = 0,75. (D) Die Kontraktionsgeschwindigkeiten für Aktinfilamente (geschlossene Symbole) und Mikrotubuli (offene Symbole) werden aus Anpassungen an entsprechende τ(q)-Kurven bestimmt. Fehlerindikatoren in allen Diagrammen stellen den Standardfehler von Werten über drei bis fünf Replikate dar. Diese Zahl wurde gegenüber Referenznummer17 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
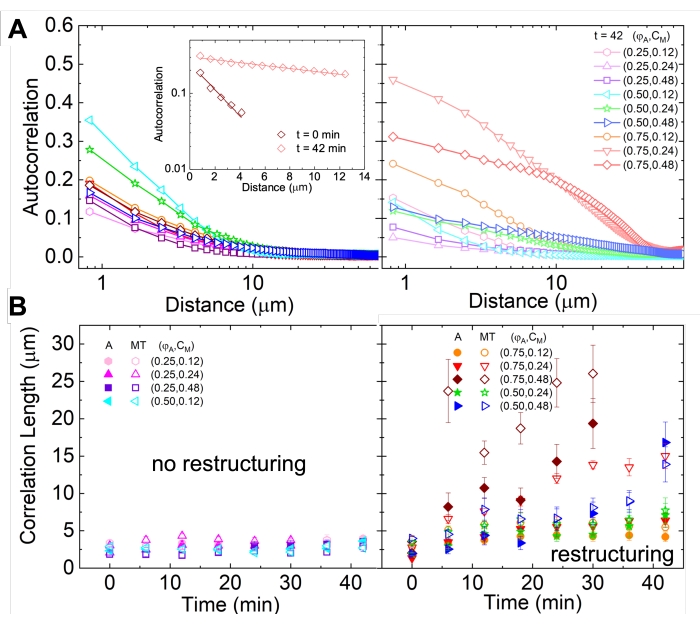
Abbildung 5. Die räumliche Bildautokorrelation (SIA) quantifiziert die motorgetriebene Umstrukturierung aktiver Zytoskelettkomposite. (A) Autokorrelation g(r) für die Mikrotubuli am Anfang (links, t = 0 min, dunkle Farbtöne) und am Ende (rechts, t = 42 min, helle Farbtöne) des Experiments für (ΦA, cM) Formulierungen, die in der Legende aufgeführt sind. Einschub: Beispielanpassungen von Daten an 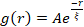 die Anfangs- und Endzeit für (ΦA, cM) = (0,75, 0,12). (B) Durchschnittliche Korrelationslängen ξ für Aktin (geschlossene Symbole) und Mikrotubuli (offene Symbole) für jedes (Φ A, cM), bestimmt durch exponentielle Anpassungen jeder g(r)-Kurve, wie im Einschub in A gezeigt. Fehlerindikatoren in A und B stellen den Standardfehler über drei bis fünf Replikate dar. Diese Zahl wurde gegenüber Referenznummer17 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
die Anfangs- und Endzeit für (ΦA, cM) = (0,75, 0,12). (B) Durchschnittliche Korrelationslängen ξ für Aktin (geschlossene Symbole) und Mikrotubuli (offene Symbole) für jedes (Φ A, cM), bestimmt durch exponentielle Anpassungen jeder g(r)-Kurve, wie im Einschub in A gezeigt. Fehlerindikatoren in A und B stellen den Standardfehler über drei bis fünf Replikate dar. Diese Zahl wurde gegenüber Referenznummer17 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
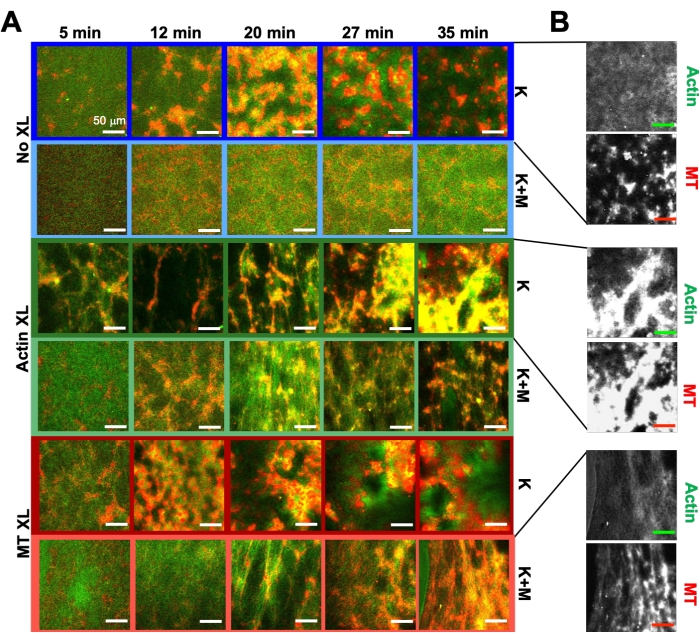
Abbildung 6. Integration von Kinesinmotoren und passiven Vernetzern in aktive Verbundwerkstoffe, um die Programmierbarkeit zu erhöhen und den Phasenraum von Dynamik und Struktur zu erweitern. (A) Zweifarbige konfokale Bilder von Aktin (grün) und Mikrotubuli (rot) in aktiven Verbundwerkstoffen zeigen eine komplexe formulierungsabhängige Umstrukturierung über die Zeit (in min angegeben). Die fünf Bilder in jeder Reihe entsprechen fünf Bildern einer Zeitreihe von 2000 Bildern, die für ein Kompositum aufgenommen wurden, das von Kinesin (K, Zeilen 1, 3, 5) oder Kinesin und Myosin (K+M, Zeilen 2, 4, 6) angetrieben wird, und entweder keine passiven Vernetzer (No XL, Zeilen 1, 2), Aktin-Aktin-Vernetzungen (Aktin XL, Zeilen 3, 4) oder Mikrotubuli-Mikrotubuli-Vernetzungen (MT XL, Zeilen 5, 6). Maßstabsbalken sind alle 50 μm. Die Umrissfarben entsprechen dem Farbschema in Abbildung 8. (B) Getrennte Aktin- und Mikrotubuli-Fluoreszenzkanäle für die reinen Kinesin-Komposite zeigen unterschiedliche Strukturen sowohl mit Aktin-MT-Kolokalisierung als auch mit Mikrophasentrennung. Die gezeigten Bilder sind für Verbundwerkstoffe mit c A= 2,32 μM, c T= 3,48 μM, c K = 0,35 μM, cM= 0,47 μM (Zeilen 2, 4, 6), R A= 0,02 (Zeilen 3, 4) und R MT = 0,005 (Zeilen 5, 6). Alle Komposite beginnen mit gleichmäßig verteilten, sich gegenseitig durchdringenden Netzwerken von Aktin und Mikrotubuli (Spalte 1). Kinesin-getriebene Verbundwerkstoffe ohne Vernetzer (Reihe 1) bilden lose verbundene amorphe Cluster, die MT-reich sind. Aktin kolokalisiert zunächst in den Zentren dieser Aggregate, wird dann aber aus den MT-reichen Regionen verdrängt, die sich weiterhin zusammenziehen und voneinander trennen. Die Aktin-Aktin-Vernetzung (Reihe 3) behindert diese mikroskalige Aktin-MT-Trennung, stattdessen werden MT-reiche Aggregate über lange Aktinstränge verbunden. Die Aktinvernetzung ermöglicht auch eine langsame Aufnahme von Aktin in die MT-reichen Regionen, so dass das Komposit zu einem verbundenen Netzwerk von kolokalisierten Aktin- und MT-Clustern wird. Mikrotubuli-Vernetzung (Reihe 5) führt zu einer amorphen Clusterbildung von MTs, die im Laufe der Zeit zusammenwachsen, was zu einer größeren Phasentrennung von Aktin und MTs führt. Die Zugabe von Myosin (Zeilen 2, 4, 6) reduziert die kinesingesteuerte Entmischung und Umstrukturierung. Ohne Vernetzer (Reihe 2) zeigen Verbundwerkstoffe im Laufe der Stunden wenig Umlagerung. Die Vernetzung erhöht die Umstrukturierung und Kolokalisierung von Aktin und Mikrotubuli (Zeilen 4, 6). Insbesondere wenn Mikrotubuli vernetzt sind (Reihe 6), kommt es zu einer signifikanten Durchdringung und Reorganisation in netzartige Fasernetzwerke. Diese Zahl wurde gegenüber Referenznummer50 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
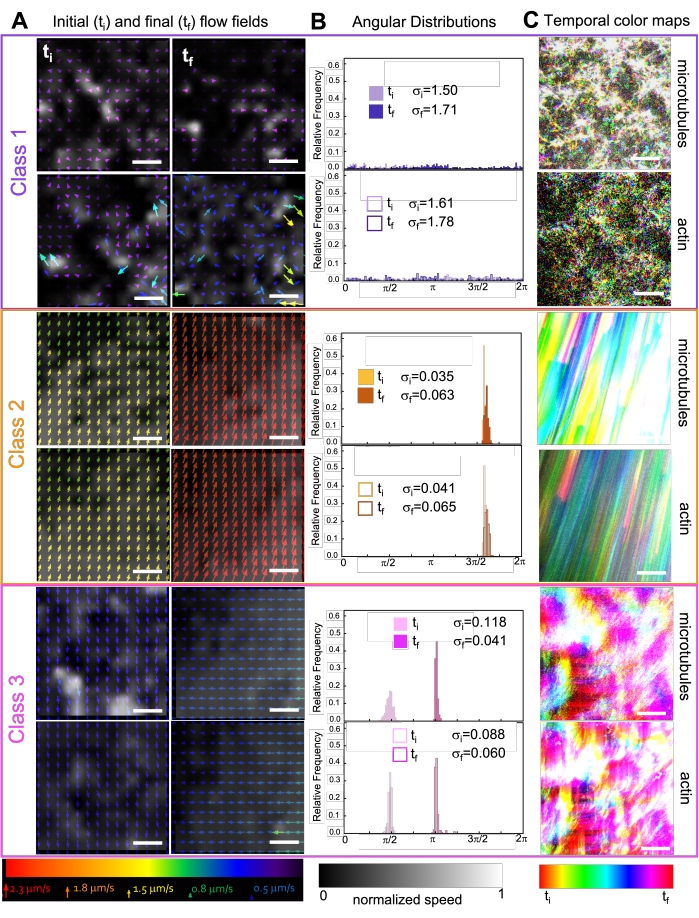
Abbildung 7. PIV zeigt, dass aktive Verbundwerkstoffe drei Klassen von räumlich unterschiedlichen Strömungsfeldern aufweisen. (A) PIV-Strömungsfelder für den ersten (t i) und letzten (tf) Rahmen von drei repräsentativen Zeitreihen, die die verschiedenen dynamischen Klassen zeigen, die die in Abbildung 6 gezeigten Verbundwerkstoffe aufweisen. PIV-Strömungsfelder für Mikrotubuli (oben) und Aktin (unten) für Beispielvideos der Klassen 1 (oben, lila), Klasse 2 (Mitte, orange) und Klasse 3 (unten, Magenta), mit Pfeilfarben, die der universellen Geschwindigkeitsskala unten entsprechen, und der Graustufen-Farbkarte, die die räumliche Geschwindigkeitsverteilung zeigt, die für jedes Strömungsfeld entsprechend der unten gezeigten Skala separat normalisiert wird. Maßstabsbalken sind alle 50 μM. (B) Winkelverteilungen von Geschwindigkeitsvektoren von A (in Einheiten des Bogenmaßes) mit aufgelisteten Anfangs- und Endstandardabweichungen σ i und σf. (C) Zeitliche Farbkarten für die in A und B analysierten Videos zeigen die Bild-zu-Bild-Position jedes Pixels relativ zu seinem Ausgangspunkt. Karten der Klasse 1 zeigen kleinräumige zufällige Bewegungen; Karten der Klasse 2 zeigen schnelle unidirektionale Bewegungen mit minimalen räumlichen oder zeitlichen Schwankungen; Karten der Klasse 3 weisen Merkmale der Klasse 1 und 2 auf. Diese Zahl wurde gegenüber Referenznummer50 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
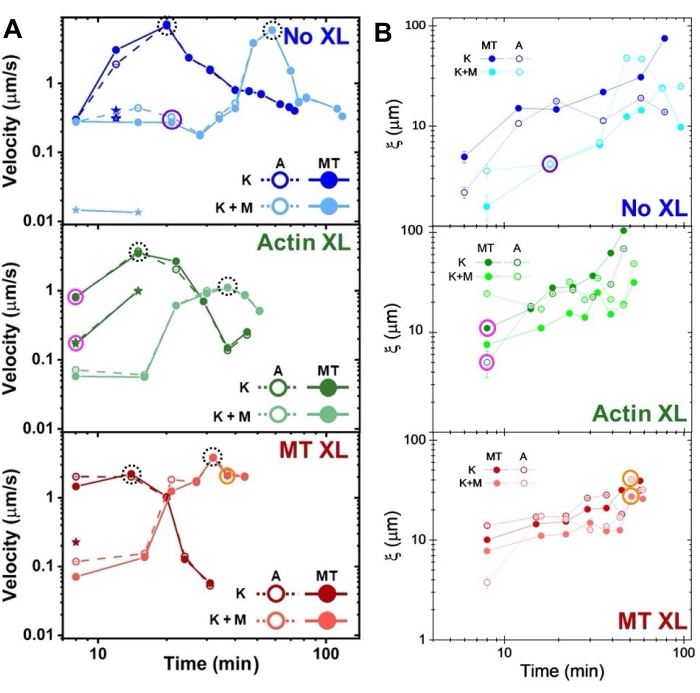
Abbildung 8. DDM und SIA messen die zeitvariable Dynamik und Struktur von zweimotorigen Aktin-Mikrotubuli-Verbundwerkstoffen. (A) Die in Abbildung 6 und Abbildung 7 beschriebenen Geschwindigkeiten für Verbundwerkstoffe, gemessen über DDM, zeigen die Beschleunigung und Verzögerung von Verbundwerkstoffen, programmiert durch Vernetzung und Myosinaktivität. Die Geschwindigkeiten von Mikrotubuli (MT, geschlossene Kreise) und Aktin (A, offene Kreise) werden als Funktion der Aktivitätszeit in Kompositen ohne Vernetzung (oben, blau), Aktinvernetzung (Mitte, grün), Mikrotubulivernetzung (unten, rot), ohne Myosin (K, dunklere Farbtöne) und mit Myosin (K+M, hellere Farbtöne) aufgetragen. Für Fälle der Klasse 3, die zwei Geschwindigkeiten haben, wird die langsamere Geschwindigkeit durch einen Stern angezeigt. Datenpunkte, die von gestrichelten schwarzen Kreisen umschlossen sind, entsprechen der maximalen Geschwindigkeit vmaxfür jede Formulierung. Fehlerbalken (die meisten zu klein, um sie zu sehen) sind der Standardfehler über die Potenzgesetzanpassungen des entsprechenden τ(q). (B) Strukturelle Korrelationslängen ξ, bestimmt über SIA, versus Aktivitätszeit, für den gleichen Satz von Zeitreihen, die in A ausgewertet wurden. Jeder Datenpunkt ist ein Durchschnitt der Korrelationslängen, die für den ersten und letzten Frame der entsprechenden Zeitreihe ermittelt wurden. Im Allgemeinen nimmt ξ sowohl für Aktin als auch für Mikrotubuli in allen Verbundsystemen zu, und Verbundwerkstoffe, die ausschließlich von Kinesin angetrieben werden, haben größere Korrelationslängen als diejenigen, in denen auch Myosin vorhanden ist. Datenpunkte in A und B, die den drei in Abbildung 7 analysierten Zeitreihen entsprechen, sind in der entsprechenden Klassenfarbe eingekreist (1 = lila, 2 = orange, 3 = magenta). Diese Zahl wurde gegenüber Referenznummer50 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ein wichtiger Fortschritt des oben beschriebenen rekonstituierten Systems ist seine Modularität und Abstimmbarkeit, so dass Benutzer ermutigt werden, die Konzentrationen von Proteinen, Motoren, Vernetzern usw. an ihre gewünschten Ergebnisse anzupassen, sei es, um einen bestimmten zellulären Prozess zu emulieren oder ein Material mit spezifischer Funktionalität oder mechanischen Eigenschaften zu entwickeln. Die Grenzen des Konzentrationsbereichs von Aktin und Tubulin werden an der unteren Grenze durch die kritische Konzentration festgelegt, die zur Polymerisation von Aktin (~0,2 μM)57,58,59 und Tubulin (~3 - 4 μM)60 erforderlich ist, und an der oberen Grenze durch den Übergang zur nematischen Ausrichtung von Aktinfilamenten (~90 μM)61,62 oder Mikrotubuli (~35 μM)63 . Aktinmonomere und Tubulindimere sollten zusammen zu Filamenten polymerisiert werden, anstatt nach der Polymerisation miteinander zu vermischen, um sicherzustellen, dass sie homogen durchdringende perkolierte Netzwerke bilden, die sich synergistisch gegenseitig unterstützen. Die neuartige Dynamik, die die Verbundwerkstoffe aufweisen, beruht auf dieser Wechselwirkung. Während es im Allgemeinen wichtig ist, alle im Protokoll beschriebenen Schritte zu befolgen, um die gezeigten Ergebnisse erfolgreich zu reproduzieren, sind einige Schritte anspruchsvoller, während andere Raum für Änderungen und Anpassungen an spezifische Bedürfnisse und verfügbare Ressourcen bieten.
Ein wichtiger Schritt, um reproduzierbare Ergebnisse zu gewährleisten, ist beispielsweise die ordnungsgemäße Vorbereitung und Lagerung der Reagenzien gemäß den Richtlinien der Materialtabelle. Zytoskelettproteine (Aktin, Tubulin, Myosin, Kinesin) sind labil und sollten aliquotiert, mit flüssigem Stickstoff schockgefroren und bei -80 °C in Einwegaliquoten gelagert werden. Nach der Entfernung von -80 °C sollten Aliquots auf Eis gehalten werden. Zytoskelettproteine behalten ihre Funktion nach zusätzlichen Gefrier-Tau-Zyklen nicht zuverlässig.
Mikrotubuli sind empfindlicher gegenüber Depolymerisation und Denaturierung als Aktin. Nach der Entfernung von -80 °C sollte Tubulin vor der Polymerisation auf Eis gehalten und innerhalb von 12 h verwendet werden. Nach der Polymerisation sollten Mikrotubuli bei Raumtemperatur gehalten werden. Es ist auch wichtig, Mikrotubuli mit Taxol zu stabilisieren, um eine Depolymerisation zu verhindern. Die Phalloidin-Stabilisierung von Aktinfilamenten ist ebenfalls wichtig, um das ATP-verbrauchende Aktin-Laufband zu unterdrücken, das mit der Myosin- und Kinesinaktivität konkurriert.
Die Ultrazentrifugation von Myosinmotoren ist ein weiterer kritischer Schritt, da sie inaktive Myosin-Totköpfe entfernt. Die Nichtentfernung der enzymatisch inaktiven Monomere führt zu einer passiven Vernetzung des Aktinnetzwerks und einem Aktivitätsverlust. Um die ATPase-Aktivität von Motoren zu verlängern, kann ein ATP-Regenerationssystem wie Kreatinphosphat und Kreatinphosphokinase64 eingebaut werden.
Schließlich erfordert die Aufrechterhaltung der Kompositaktivität die Hemmung der Adsorption von Filamenten und Motoren an den Wänden der Probenkammer, was durch Passivierung der Mikroskopdeckgläser und Objektträger erreicht werden kann. Motorproteine sind besonders anfällig für Adsorption, was dazu führt, dass der Verbundwerkstoff an die Oberfläche der Probenkammer gezogen wird, sich aus dem Sichtfeld bewegt, zu 2D kollabiert und nicht mehr aktiv wird. Das Silanisieren der Deckgläser und Objektträger ist eine effektive Möglichkeit, die Oberflächen zu passivieren und Adsorption zu verhindern (siehe Schritt 1). Eine alternative Passivierungsmethode, die effektiv in In-vitro-Zytoskelett-Experimenten eingesetzt wird, ist die Beschichtung der Oberfläche mit einer Lipiddoppelschicht, ähnlich der Zellmembran18. Diese Methode ist vorteilhaft, wenn man Proteine an die Oberfläche binden oder andere spezifische Protein-Oberflächen-Wechselwirkungen einführen möchte, da die Doppelschicht funktionalisiert werden kann. Für optische Pinzettenexperimente ist die Passivierung der Mikrosphären ebenfalls kritisch und kann durch Beschichtung carboxylierter Mikrokugeln mit BSA oder PEG mittels Carbodiimid-Vernetzerchemie48 erreicht werden.
Es gibt einige Aspekte der vorgestellten Protokolle, die Forscher an ihre Bedürfnisse anpassen können. Erstens können Forscher nicht-native Biotin-NA-Vernetzer durch biologische Vernetzer wie Alpha-Actinin oder MAP65 ersetzen, die Aktin und Mikrotubuli vernetzen, bzw. 28,65,66. Die Verwendung von nicht-nativen Vernetzern in den hier beschriebenen Verbundwerkstoffen ist durch ihre verbesserte Reproduzierbarkeit, Stabilität und Abstimmbarkeit im Vergleich zu nativen Vernetzern motiviert. Aufgrund der starken Biotin-NA-Bindung kann davon ausgegangen werden, dass Vernetzer dauerhaft sind, und nicht die meisten nativen Vernetzer, die vorübergehend an weitreichende Umsatzraten binden. Die Dynamik der transienten Vernetzung erschwert das Parsen der Beiträge von Vernetzern und Motoren zur Dynamik. Darüber hinaus können Biotin-NA-Linker vielseitig zur Vernetzung von Aktin und Mikrotubuli sowie zur Vernetzung von Aktin zu Mikrotubuli eingesetzt werden. Auf diese Weise kann ein eindeutiger Vergleich zwischen Vernetzungsmotiven vorgenommen werden, wobei alle anderen Variablen (z. B. Vernetzergröße, Bindungsaffinität, Stöchiometrie usw.) fixiert bleiben. Schließlich sind die Reagenzien, die benötigt werden, um Biotin-NA-Linker zu integrieren, weit verbreitet, gut charakterisiert und häufig in vielen biophysikalischen Labors verwendet. Eine der wichtigsten Stärken der hier beschriebenen In-vitro-Plattform ist jedoch ihre Modularität, so dass Forscher in der Lage sein sollten, Biotin-NA-Linker nahtlos durch native Linker zu ersetzen, wenn sie dies wünschen.
Zweitens werden im aktuellen Protokoll Aktinmonomere und Tubulindimere zusammen in einem Zentrifugenröhrchen zu Filamenten polymerisiert, bevor sie der Probenkammer hinzugefügt werden. Das Fließen der Lösung verschränkter filamentöser Proteine in die Probenkammer kann zu einer Strömungsausrichtung, insbesondere der Mikrotubuli, führen, wodurch die gewünschte Isotropie und Homogenität der Verbundwerkstoffe gebrochen wird. In der Tat war ein wichtiger Fortschritt in früheren Arbeiten an stationären Aktin-Mikrotubuli-Verbundwerkstoffen die Fähigkeit, Aktin und Mikrotubuli in situ (in der Probenkammer) zu copolymerisieren, um die Bildung isotroper interpenetrierender Netzwerke von Aktin und Mikrotubuli sicherzustellen15,16,27. Die Erweiterung dieses Ansatzes auf aktive Verbundwerkstoffe würde jedoch erfordern, dass die Motoren vor der Aktin- und Tubulinpolymerisation zur Probe hinzugefügt werden und die gesamte Probe vor den Experimenten bei 37 °C inkubiert wird. Tests dieser Variation des Protokolls haben zu einer reduzierten Aktinpolymerisation und keiner erkennbaren motorischen Aktivität geführt, wahrscheinlich aufgrund der konkurrierenden ATPase-Aktivität und der verlängerten Inkubation der Motoren um 37 °C. Glücklicherweise gibt es keine erkennbare Strömungsausrichtung von Verbundwerkstoffen, wenn die aktuellen Protokolle befolgt werden, wie in Abbildung 2, Abbildung 3 und Abbildung 6 zu sehen ist. Dennoch werden die Forscher ermutigt, Protokolle zu entwerfen, die die In-situ-Bildung aktiver Verbundwerkstoffe ermöglichen.
Ein weiterer Punkt ist das Fluoreszenzmarkierungsschema, bei dem alle Aktinfilamente und Mikrotubuli im Netzwerk spärlich markiert werden. Dieser Markierungsansatz wurde optimiert, um die Struktur des Netzwerks direkt zu visualisieren, anstatt Struktur und Dynamik über Tracerfilamente oder Mikrosphären abzuleiten. Der Nachteil besteht jedoch darin, dass einzelne Filamente nicht hell beschriftet und auflösbar sind. Ein Ansatz, den Forscher sowohl zur Auflösung einzelner Filamente als auch zur Visualisierung der Netzwerkstruktur verfolgen könnten, besteht darin, vorgeformte Filamente zu dotieren, die mit einem anderen Fluorophor markiert sind, so dass sowohl das umgebende Netzwerk als auch einzelne Filamente gleichzeitig abgebildet werden können. Bei Verwendung von mehr als zwei Fluorophoren und Anregungs-/Emissionskanälen ist das Durchbluten zwischen den Kanälen jedoch oft schwer zu eliminieren, so dass bei der Auswahl der Fluorophore, Filter und Laserintensitäten Vorsicht geboten ist.
Eine damit verbundene Einschränkung ist die Unfähigkeit, die Myosin- oder Kinesinmotoren in den Verbundwerkstoffen sichtbar zu machen. Die verwendeten fluoreszenzmarkierten Aktinmonomere und Tubulin-Dimere sind kommerziell erhältlich, während die Visualisierung von Myosin oder Kinesin in Verbundwerkstoffen eine interne Markierung erfordert. Die Forscher werden ermutigt, den nächsten Schritt zu unternehmen, um Motoren zu kennzeichnen, wie es zuvor18,67 getan wurde, um die motorische Aktivität und Bindung eindeutig mit der Dynamik und den Strukturen unserer Verbundwerkstoffe verknüpfen zu können.
Schließlich ist es wichtig zu beachten, dass im aktuellen Protokoll der Beginn und die Dauer der Kinesinaktivität nicht kontrolliert werden. Da die Myosinaktivität durch Photodeaktivierung von Blebbistatin, wie oben beschrieben, gesteuert wird, um eine ähnliche Lichtaktivierung von Kinesin aufzubauen, kann man lichtaktiviertes ATP einbauen.
Um die Komplexität der hier beschriebenen Designs aufzubauen, zelluläre Bedingungen besser nachzuahmen und den dynamischen Struktur-Funktions-Parameterraum zu erweitern, werden sich zukünftige Arbeiten auf die Integration von Zwischenfilamenten wie Vimentin68,69 sowie anderen Motoren wie Dynein13,70 konzentrieren. Gelsolin wird auch in verschiedenen Konzentrationen eingebaut, um die Aktinlänge14 zu kontrollieren, sowie Tau-Protein, um die Steifigkeit der Mikrotubuli zu kontrollieren.
Zusammenfassend beschreiben die vorgestellten Protokolle, wie die Dynamik, Struktur und Mechanik von Zytoskelett-inspirierten aktiven Materiesystemen entworfen, erstellt und charakterisiert werden kann, die zwei separate aktive Kraft erzeugende Komponenten enthalten, die auf verschiedene Substrate in einem einzigen System wirken. Diese abstimmbare und modulare Plattform bringt die Rekonstitutionsbemühungen der Nachahmung des zellulären Zytoskeletts einen wichtigen Schritt näher und bietet die einzigartige Möglichkeit, seine Eigenschaften über einen weiten Phasenraum zu programmieren, indem die verschiedenen Komponenten unabhängig voneinander integriert, entfernt und abgestimmt werden. Darüber hinaus sind alle Komponenten dieses vielseitigen Systems kommerziell erhältlich (siehe Materialtabelle), mit Ausnahme der Kinesin-Dimere, die im Ross-Labor gereinigt werden, wie zuvorbeschrieben 50, und auf Anfrage erhältlich sind. Schließlich ist der gesamte Analysecode über GitHub49 frei verfügbar und basiert auf freien Programmiersprachen und Software (Python und Fidschi). Die transparente Verbreitung von Protokollen zur Gestaltung dieser Systeme wird diese Plattform hoffentlich für eine vielfältige Gruppe von Nutzern mit unterschiedlichen Fachkenntnissen, Hintergründen, institutionellen Zugehörigkeiten und Forschungszielen zugänglicher machen.
Die Autoren haben nichts zu verraten
Wir danken Maya Hendija und Dr. Jonathan Michel für die Unterstützung bei der Datenanalyse und Dr. Janet Sheung, Dr. Moumita Das und Dr. Michael Rust für hilfreiche Diskussionen und Anleitungen. Diese Forschung wurde durch einen William M. Keck Foundation Research Grant und den NSF DMREF Award (DMR 2119663) unterstützt, der an RMRA und JLR und die National Institutes of Health R15 Grants (R15GM123420, 2R15GM123420-02) vergeben wurde, die an RMR-A und RJM vergeben wurden.
| Name | Company | Catalog Number | Comments |
| (-)-Blebbistatin Abbreviation used in paper: blebbistatin | Sigma Aldrich | B0560 | Stock Concentration: 200 μM in DMSO Storage: dessicated, in DMSO, -20ºC Stock and Experiment Recipes: dissolve 1 mg of powder to 200 μM in DMSO Storage, Handling, Troubleshooting Notes: limited shelf-life, typically stops functioning reliably after 3-4 months. purchase and prepare new solution every 3 months. |
| 1:20 488-tubulin:tubulin mixture Abbreviation used in paper: 5-488-tubulin | NA | NA | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: mix tubulin and 488-tubulin at a 20:1 ratio, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| 1:20 R-tubulin:tubulin mixture Abbreviation used in paper: 5-R-tubulin | NA | NA | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: mix tubulin and rhodamine tubulin at a 20:1 ratio, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| actin (biotin): skeletal muscle Abbreviation used in paper: biotin-actin | Cytoskeleton | AB07 | Stock Concentration: 1 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: (1) immediately prior to use dilute to 0.5 mg/ml in PEM, (2) once removed from -80ºC, store aliquot on ice at 4ºC for up to 1 week |
| actin (rhodamine): rabbit skeletal muscle Abbreviation used in paper: R-actin | Cytoskeleton | AR05 | Stock Concentration: 1.5 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1.5 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: once removed from -80ºC, store aliquot on ice at 4ºC, can be used for up to 1 week |
| adenosine triphosphate Abbreviation used in paper: ATP | Thermo Fisher Scientific | A1048 | Stock Concentration: 100 mM Storage: in solution (pH 7), -20ºC Stock and Experiment Recipes: reconsitute in DI H20, bring pH to 7 with NaOH Storage, Handling, Troubleshooting Notes: routinely check pH and adjust as needed, hydrolyzes over time, replace every ~6-12 months |
| AlexaFluor488 Phalloidin Abbreviation used in paper: 488-phalloidin | Thermo Fisher Scientific | A12379 | Stock Concentration: 100 μM DMSO Storage: protected from light, dessicated, -20ºC Stock and Experiment Recipes: reconstitute to 100 μM with DMSO Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 20 μM in PEM (1 μL in 4 μL PEM) |
| AlexaFluor488–labeled actin Abbreviation used in paper: 488-actin | Thermo Fisher Scientific | A12373 | Stock Concentration: 1.5 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1.5 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: this item has been discontinued |
| Basic Plasma Cleaner Abbreviation used in paper: plasma cleaner | Harrick Plasma | PDC-32G | |
| Bemis Parafilm M Laboratory Wrapping Film Abbreviation used in paper: transparent film | Thermo Fisher Scientific | 13-374-5 | |
| D-(+)-Glucose Abbreviation used in paper: | Thermo Fisher Scientific | A1682836 | Stock Concentration: 100x Storage: store at stock concentration (100x) or 10x concentration, dessicated, at -20ºC Stock and Experiment Recipes: reconstitute powder to 4.5 mg/ml in DI H20 Storage, Handling, Troubleshooting Notes: final concentration in solution should 45 μg/mL |
| D-Biotin Abbreviation used in paper: biotin | Fisher Scientific | BP232-1 | Stock Concentration: 1.02 mM in PEM Storage: dessicated, 4ºC |
| deionized nanopure water Abbreviation used in paper: DI | |||
| Dimethyldichlorosilane Abbreviation used in paper: silane | Thermo Fisher Scientific | D/3820/PB05 | Stock Concentration: 2% dissolved in Toulene |
| Dithiothreitol Abbreviation used in paper: DTT | Thermo Fisher Scientific | R0861 | Stock Concentration: 1 M in DMSO Storage: dessicated, -20ºC Stock and Experiment Recipes: dilute to 2 mM in PEM immediately before each experiment |
| DMSO Anhydrous Abbreviation used in paper: DMSO | Thermo Fisher Scientific | D12345 | |
| F-Buffer Abbreviation used in paper: F-buffer | NA | NA | Stock Concentration: 10x Storage: dessicated, -20ºC Stock and Experiment Recipes: 10 mM Imidazole (pH 7.0), 50 mM KCl, 1 mM MgCl2, 1 mM EGTA, 0.2 mM ATP |
| G-Buffer Abbreviation used in paper: G-buffer | NA | NA | Stock Concentration: 10x Storage: dessicated, -20ºC Stock and Experiment Recipes: 2.0 mM Tris (pH 8), 0.2 mM ATP, 0.5 mM DTT, 0.1 mM CaCl2. Store at -20°C. |
| glass microscope slide Abbreviation used in paper: slide | Thermo Fisher Scientific | 22-310397 | |
| Glucose oxidase + catalase + β-mercaptoethanol Abbreviation used in paper: GOC | Sigma Aldrich | G2133-250KU, C1345, 63689 | Stock Concentration: 100x Storage: store at stock concentration (100x) or 10x concentration, dessicated, at -20ºC Stock and Experiment Recipes: For 100x: 4.3 mg/ml glucose oxidase, 0.7 mg/ml catalase, 0.5% v/v β-mercaptoethanol in DI H20 Storage, Handling, Troubleshooting Notes: final concentration in solution should be: 0.005% β-mercaptoethanol, 43 μg/mL glucose oxidase, 7 μg/mL catalase |
| glu-GOC oxygen scavenging system Abbreviation used in paper: glu-GOC | NA | NA | Stock Concentration: 100x Storage: prepare fresh each time Stock and Experiment Recipes: mix equal parts Glu and GOC and add at 1/100 final sample volume immediately before imaging Storage, Handling, Troubleshooting Notes: prepare from Glu and GOC immediately before imaging |
| Guanosine triphosphate Abbreviation used in paper: GTP | Thermo Fisher Scientific | R0461 | Stock Concentration: 100 mM Storage: 100 μL aliquots at -20ºC |
| Instant Mix 1-minute epoxy Abbreviation used in paper: epoxy | Loctite | 1366072 | |
| Kinesin-1 401 BIO 6x HIS Abbreviation used in paper: kinesin | Prepared in JL Ross Lab at Syracuse University | NA | Stock Concentration: 8.87 μM in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Storage, Handling, Troubleshooting Notes: biotinylated dimers form kinesin clusters, each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| NeutrAvidin Abbreviation used in paper: NA | Thermo Fisher Scientific | 31000 | Stock Concentration: 5 mg/ml in PEM Storage: dessicated, -20ºC Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM |
| No 1. glass coverslips (24 mm x 24 mm) Abbreviation used in paper: coverslip | Thermo Fisher Scientific | 12-548-CP | |
| Paclitaxel Abbreviation used in paper: Taxol | Thermo Fisher Scientific | P3456 | Stock Concentration: 2 mM in DMSO Storage: protected from light, dessicated, -20ºC Stock and Experiment Recipes: reconstitute to 2 mM with DMSO Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 200 μM in DMSO (0.4 μL in 3.6 μL DMSO) |
| PEM-100 Abbreviation used in paper: PEM | NA | NA | Stock Concentration: 1x Storage: room temperature (RT) Stock and Experiment Recipes: 100 mM K-PIPES (pH 6.8), 2 mM EGTA, 2 mM MgCl2 Storage, Handling, Troubleshooting Notes: use KOH to adjust pH to 6.8, recheck pH often and adjust accordingly |
| phalloidin Abbreviation used in paper: phalloidin | Thermo Fisher Scientific | P3457 | Stock Concentration: 100 μM in DMSO Storage: protected from light, dessicated, -20ºC, adhere closely to storage/handling conditions Stock and Experiment Recipes: reconstitute to 100 μM with DMSO Storage, Handling, Troubleshooting Notes: susceptible to impurities in its preparation and denaturing, identifiable as large amorphous aggregates of actin in samples |
| porcine brain tubulin Abbreviation used in paper: tubulin | Cytoskeleton | T240 | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| Potassium Chloride Abbreviation used in paper: KCl | Thermo Fisher Scientific | AM9640G | Stock Concentration: 4 M Storage: RT |
| Rabbit skeletal actin Abbreviation used in paper: actin | Cytoskeleton | AKL99 | Stock Concentration: 2 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 2 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: once removed from -80ºC, store aliquot on ice at 4ºC, can be used for up to 1 week |
| Rabbit skeletal myosin II Abbreviation used in paper: myosin | Cytoskeleton | MY02 | Stock Concentration: 10 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 10 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: monomers form minifilaments at low KCl, each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| Tubulin (biotin): porcine brain Abbreviation used in paper: biotin-tubulin | Cytoskeleton | T333P | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 0.5 mg/ml in PEM |
| Tubulin (fluorescent HiLyte 488): porcine brain Abbreviation used in paper: 488-tubulin | Cytoskeleton | TL488M | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| tubulin (rhodamine): porcine brain Abbreviation used in paper: R-tubulin | Cytoskeleton | TL590M | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| Tween 20 Abbreviation used in paper: Tween20 | Thermo Fisher Scientific | J20605.AP | Stock Concentration: 1% v/v in DI H20 Storage: RT |
| ultracentrifuge grade microtubes Abbreviation used in paper: Beckman-Coulter Optima Max XP | Beckman Coultier | 343776 | Storage, Handling, Troubleshooting Notes: 8x34 mm PC |
| UV light curing glue Abbreviation used in paper: UV glue | Pharda | SKG-2869 |
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Koenderink, G. H., Paluch, E. K. Architecture shapes contractility in actomyosin networks. Current Opinion in Cell Biology. 50, 79-85 (2018).
- Dogterom, M., Koenderink, G. H. Actin-microtubule crosstalk in cell biology. Nature Reviews Molecular Cell Biology. 20 (1), 38-54 (2019).
- Burla, F., Mulla, Y., Vos, B. E., Aufderhorst-Roberts, A., Koenderink, G. H. From mechanical resilience to active material properties in biopolymer networks. Nature Reviews Physics. 1 (4), 249-263 (2019).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State and Materials Science. 15 (5), 177-182 (2011).
- Xiao, Q., Hu, X., Wei, Z., Tam, K. Y. Cytoskeleton molecular motors: structures and their functions in neuron. International Journal of Biological Sciences. 12 (9), 1083-1092 (2016).
- Ajeti, V. et al. Wound healing coordinates actin architectures to regulate mechanical work. Nature Physics. 15 (7), 696-705 (2019).
- Jung, W. et al. Dynamic motions of molecular motors in the actin cytoskeleton. Cytoskeleton. 76 (11-12), 517-531 (2019).
- Pollard, T. D., O'Shaughnessy, B. Molecular mechanism of cytokinesis. Annual Review of Biochemistry. 88 (1), 661-689 (2019).
- Huber, F., Boire, A., López, M. P., Koenderink, G. H. Cytoskeletal crosstalk: when three different personalities team up. Current Opinion in Cell Biology. 32, 39-47 (2015).
- Rivero, F. et al. The role of the cortical cytoskeleton: F-actin crosslinking proteins protect against osmotic stress, ensure cell size, cell shape and motility, and contribute to phagocytosis and development. Journal of Cell Science. 109 (11), 2679-2691 (1996).
- Duclos, G. et al. Topological structure and dynamics of three-dimensional active nematics. Science. 367 (6482), 1120-1124 (2020).
- Baclayon, M. et al. Optical tweezers-based measurements of forces and dynamics at microtubule ends. Optical Tweezers. 1486, 411-435 (2017).
- Gurmessa, B., Fitzpatrick, R., Falzone, T. T., Robertson-Anderson, R. M. Entanglement density tunes microscale nonlinear response of entangled actin. Macromolecules. 49 (10), 3948-3955 (2016).
- Francis, M. L. et al. Non-monotonic dependence of stiffness on actin crosslinking in cytoskeleton composites. Soft Matter. 15 (44), 9056-9065 (2019).
- Ricketts, S. N. et al. Varying crosslinking motifs drive the mesoscale mechanics of actin-microtubule composites. Scientific Reports. 9 (1), 12831 (2019).
- Lee, G. et al. Active cytoskeletal composites display emergent tunable contractility and restructuring. Soft Matter. 17 (47), 10765-10776 (2021).
- Murrell, M. P., Gardel, M. L. F-actin buckling coordinates contractility and severing in a biomimetic actomyosin cortex. Proceedings of the National Academy of Sciences. 109 (51), 20820-20825 (2012).
- Soares e Silva, M. et al. Active multistage coarsening of actin networks driven by myosin motors. Proceedings of the National Academy of Sciences. 108 (23), 9408-9413 (2011).
- Sonn-Segev, A., Bernheim-Groswasser, A., Roichman, Y. Dynamics in steady state in vitro acto-myosin networks. Journal of Physics: Condensed Matter. 29 (16), 163002 (2017).
- Ideses, Y., Sonn-Segev, A., Roichman, Y., Bernheim-Groswasser, A. Myosin II does it all: assembly, remodeling, and disassembly of actin networks are governed by myosin II activity. Soft Matter. 9 (29), 7127 (2013).
- Fürthauer, S. et al. Self-straining of actively crosslinked microtubule networks. Nature Physics. 15 (12), 1295-1300 (2019).
- Lemma, L. M. et al. Multiscale microtubule dynamics in active nematics. Physical Review Letters. 127 (14), 148001 (2021).
- Fan, Y., Wu, K.-T., Aghvami, S. A., Fraden, S., Breuer, K. S. Effects of confinement on the dynamics and correlation scales in kinesin-microtubule active fluids. Physical Review E. 104 (3), 034601 (2021).
- Triclin, S. et al. Self-repair protects microtubules from destruction by molecular motors. Nature Materials. 20 (6), 883-891 (2021).
- Lee, G. et al. Myosin-driven actin-microtubule networks exhibit self-organized contractile dynamics. Science Advances. 7 (6), eabe4334 (2021).
- Ricketts, S. N., Ross, J. L., Robertson-Anderson, R. M. Co-entangled actin-microtubule composites exhibit tunable stiffness and power-law stress relaxation. Biophysical Journal. 115 (6), 1055-1067 (2018).
- Bendix, P. M. et al. A quantitative analysis of contractility in active cytoskeletal protein networks. Biophysical Journal. 94 (8), 3126-3136 (2008).
- Linsmeier, I. et al. Disordered actomyosin networks are sufficient to produce cooperative and telescopic contractility. Nature Communications. 7 (1), 12615 (2016).
- Stam, S. et al. Filament rigidity and connectivity tune the deformation modes of active biopolymer networks. Proceedings of the National Academy of Sciences. 114 (47), E10037-E10045 (2017).
- Yadav, V. et al. Filament nucleation tunes mechanical memory in active polymer networks. Advanced Functional Materials. 29 (49), 1905243 (2019).
- Ennomani, H. et al. Architecture and connectivity govern actin network contractility. Current Biology. 26 (5), 616-626 (2016).
- Alvarado, J., Sheinman, M., Sharma, A., MacKintosh, F. C., Koenderink, G. H. Molecular motors robustly drive active gels to a critically connected state. Nature Physics. 9 (9), 591-597 (2013).
- Alvarado, J., Cipelletti, L., Koenderink, G. H. Uncovering the dynamic precursors to motor-driven contraction of active gels. Soft Matter. 15 (42), 8552-8565 (2019).
- Jung, W., Murrell, M. P., Kim, T. F-actin cross-linking enhances the stability of force generation in disordered actomyosin networks. Computational Particle Mechanics. 2 (4), 317-327 (2015).
- Lenz, M., Thoresen, T., Gardel, M. L., Dinner, A. R. Contractile units in disordered actomyosin bundles arise from f-actin buckling. Physical Review Letters. 108 (23), 238107 (2012).
- Memarian, F.L. et al. Active nematic order and dynamic lane formation of microtubules driven by membrane-bound diffusing motors. Proceedings of the National Academy of Sciences. 118 (52), e2117107118 (2021).
- Needleman, D., Dogic, Z. Active matter at the interface between materials science and cell biology. Nature Reviews Materials. 2 (9), 17048 (2017).
- Foster, P. J., Fürthauer, S., Shelley, M. J., Needleman, D. J. Active contraction of microtubule networks. eLife. 4, e10837 (2015).
- Thijssen, K. et al. Submersed micropatterned structures control active nematic flow, topology, and concentration. Proceedings of the National Academy of Sciences. 118 (38), e2106038118 (2021).
- Sanchez, T., Chen, D. T. N., DeCamp, S. J., Heymann, M., Dogic, Z. Spontaneous motion in hierarchically assembled active matter. Nature. 491 (7424), 431-434 (2012).
- Colen, J. et al. Machine learning active-nematic hydrodynamics. Proceedings of the National Academy of Sciences. 118 (10), e2016708118 (2021).
- Mitchell, K. A., Tan, A. J., Arteaga, J., Hirst, L. S. Fractal generation in a two-dimensional active-nematic fluid. Chaos: An Interdisciplinary Journal of Nonlinear Science. 31 (7), 073125 (2021).
- Pandolfi, R. J., Edwards, L., Johnston, D., Becich, P., Hirst, L. S. Designing highly tunable semiflexible filament networks. Physical Review E. 89 (6), 062602 (2014).
- Tan, A. J. et al. Topological chaos in active nematics. Nature Physics. 15 (10), 1033-1039 (2019).
- Roostalu, J., Rickman, J., Thomas, C., Nédélec, F., Surrey, T. Determinants of polar versus nematic organization in networks of dynamic microtubules and mitotic motors. Cell. 175 (3), 796-808.e14 (2018).
- Ndlec, F. J., Surrey, T., Maggs, A. C., Leibler, S. Self-organization of microtubules and motors. Nature. 389 (6648), 305-308 (1997).
- Sheung, J. Y. et al. Motor-driven restructuring of cytoskeleton composites leads to tunable time-varying elasticity. ACS Macro Letters. 10 (9), 1151-1158 (2021).
- McGorty, R. PyDDM v0.2.0. Zenodo. (2022).
- Achiriloaie, D. H. et al. Kinesin and myosin motors compete to drive rich multi-phase dynamics in programmable cytoskeletal composites. arXiv. (2021).
- Wulstein, D. M., Regan, K. E., Garamella, J., McGorty, R. J., Robertson-Anderson, R. M. Topology-dependent anomalous dynamics of ring and linear DNA are sensitive to cytoskeleton crosslinking. Science Advances. 5 (12), eaay5912 (2019).
- McGorty, R. Image-Correlation. at <https://github.com/rmcgorty/Image-Correlation>. (2020).
- Robertson, C. Theory and practical recommendations for autocorrelation-based image correlation spectroscopy. Journal of Biomedical Optics. 17 (8), 080801 (2012).
- McGorty, R. Differential Dynamic Microscopy - Python. at <https://github.com/rmcgorty/Differential-Dynamic-Microscopy---Python>. (2021).
- Cerbino, R., Trappe, V. Differential dynamic microscopy: probing wave vector dependent dynamics with a microscope. Physical Review Letters. 100 (18), 188102 (2008).
- Robertson-Anderson, R. M. Optical tweezers microrheology: from the basics to advanced techniques and applications. ACS Macro Letters. 7 (8), 968-975 (2018).
- Pollard, T. D. Polymerization of ADP-actin. Journal of Cell Biology. 99 (3), 769-777 (1984).
- Coué, M., Brenner, S. L., Spector, I., Korn, E. D. Inhibition of actin polymerization by latrunculin A. FEBS Letters. 213 (2), 316-318 (1987).
- Pollard, T. D. Actin and actin-binding proteins. Cold Spring Harbor Perspectives in Biology. 8 (8), a018226 (2016).
- Kumar, N. Taxol-induced polymerization of purified tubulin. Mechanism of action. Journal of Biological Chemistry. 256 (20), 10435-10441 (1981).
- Käs, J. et al. F-actin, a model polymer for semiflexible chains in dilute, semidilute, and liquid crystalline solutions. Biophysical Journal. 70 (2), 609-625 (1996).
- Viamontes, J., Narayanan, S., Sandy, A. R., Tang, J. X. Orientational order parameter of the nematic liquid crystalline phase of F -actin. Physical Review E. 73 (6), 061901 (2006).
- Hitt, A. L., Cross, A. R., Williams, R. C. Microtubule solutions display nematic liquid crystalline structure. Journal of Biological Chemistry. 265 (3), 1639-1647 (1990).
- Andexer, J. N., Richter, M. Emerging enzymes for ATP regeneration in biocatalytic processes. ChemBioChem. 16 (3), 380-386 (2015).
- Farhadi, L. et al. Actin and microtubule crosslinkers tune mobility and control co-localization in a composite cytoskeletal network. Soft Matter. 16 (31), 7191-7201 (2020).
- Falzone, T. T., Lenz, M., Kovar, D. R., Gardel, M. L. Assembly kinetics determine the architecture of α-actinin crosslinked F-actin networks. Nature Communications. 3 (1), 861 (2012).
- Thoresen, T., Lenz, M., Gardel, M. L. Reconstitution of contractile actomyosin bundles. Biophysical Journal. 100 (11), 2698-2705 (2011).
- Sanghvi-Shah, R., Weber, G. F. Intermediate filaments at the junction of mechanotransduction, migration, and development. Frontiers in Cell and Developmental Biology. 5, 81 (2017).
- Shen, Y. et al. Effects of vimentin intermediate filaments on the structure and dynamics of in vitro multicomponent interpenetrating cytoskeletal networks. Physical Review Letters. 127 (10), 108101 (2021).
- Laan, L., Roth, S., Dogterom, M. End-on microtubule-dynein interactions and pulling-based positioning of microtubule organizing centers. Cell Cycle. 11 (20), 3750-3757 (2012).
Erratum
Erratum: Reconstituting and Characterizing Actin-Microtubule Composites with Tunable Motor-Driven Dynamics and MechanicsAn erratum was issued for: Reconstituting and Characterizing Actin-Microtubule Composites with Tunable Motor-Driven Dynamics and Mechanics. The Authors section was updated.
Mehrzad Sasanpour1
Daisy H. Achiriloaie1,2
Gloria Lee1
Gregor Leech1
Christopher Currie1
K. Alice Lindsay3
Jennifer L. Ross3
Ryan J. McGorty1
Rae M. Robertson-Anderson1
1Department of Physics and Biophysics, University of San Diego
2W. M. Keck Science Department, Scripps College, Pitzer College, and Claremont McKenna College
3Department of Physics, Syracuse University
to:
Mehrzad Sasanpour1
Daisy H. Achiriloaie1,2
Gloria Lee1
Gregor Leech1
Maya Hendija1
K. Alice Lindsay3
Jennifer L. Ross3
Ryan J. McGorty1
Rae M. Robertson-Anderson1
1Department of Physics and Biophysics, University of San Diego
2W. M. Keck Science Department, Scripps College, Pitzer College, and Claremont McKenna College
3Department of Physics, Syracuse University
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved