Summary
Abstract
Introduction
Protocol
Representative Results
Discussion
Acknowledgements
Materials
References
Cancer Research
Многопараметрический скрининг опухолевых органоидных препаратов с использованием широкоугольной визуализации живых клеток для объемного и одноорганоидного анализа
Этот протокол описывает полуавтоматизированный метод скрининга органоидных препаратов со средней и высокой пропускной способностью и не зависящее от микроскопа автоматизированное программное обеспечение для анализа изображений для количественной оценки и визуализации многопараметрических, одноорганоидных реакций на лекарственные средства для захвата внутриопухолевой гетерогенности.
Органоиды опухолей, полученные от пациента (PDTO), имеют большие перспективы для доклинических и трансляционных исследований и прогнозирования ответа пациента на терапию от скрининга лекарств ex vivo . Однако современные анализы скрининга лекарств на основе аденозинтрифосфата (АТФ) не отражают сложность лекарственного ответа (цитостатического или цитотоксического) и внутриопухолевой гетерогенности, которая, как было показано, сохраняется в ФДО из-за объемного считывания. Визуализация живых клеток является мощным инструментом для преодоления этой проблемы и более глубокой визуализации реакций на лекарства. Однако программное обеспечение для анализа изображений часто не адаптировано к трехмерности PDTO, требует флуоресцентных красителей жизнеспособности или не совместимо с форматом микропластин с 384 лунками. В этой статье описывается полуавтоматизированная методология для посева, обработки и изображения PDTO в высокопроизводительном формате 384 скважины с использованием обычных, широкоугольных систем визуализации живых клеток. Кроме того, мы разработали программное обеспечение для анализа изображений без маркеров жизнеспособности для количественной оценки показателей лекарственного ответа на основе скорости роста, которые улучшают воспроизводимость и корректируют вариации скорости роста между различными линиями PDTO. Используя нормализованную метрику лекарственного ответа, которая оценивает реакцию на лекарство на основе скорости роста, нормализованной до положительного и отрицательного контрольного состояния, и флуоресцентного красителя гибели клеток, цитотоксические и цитостатические реакции на лекарства могут быть легко различимы, что значительно улучшает классификацию респондентов и неответчиков. Кроме того, гетерогенность лекарственного ответа может быть количественно определена на основе анализа реакции на одноорганоидное лекарственное средство для выявления потенциальных резистентных клонов. В конечном счете, этот метод направлен на улучшение прогнозирования ответа на клиническую терапию путем захвата многопараметрической сигнатуры лекарственного ответа, которая включает в себя кинетическую остановку роста и количественную оценку гибели клеток.
В последние годы открытие лекарств от рака in vitro , скрининг лекарств и фундаментальные исследования переходят от использования традиционных двумерных (2D) моделей рака с увековеченными клеточными линиями к более физиологически значимым трехмерным (3D) моделям рака. Это стимулировало принятие опухолевых сфероидов с установленными линиями раковых клеток, которые воссоздают более сложные межклеточные взаимодействия и структуры, присутствующие в солидных опухолях. В настоящее время полученные от пациента опухолевые органоиды (PDTO) являются наиболее передовой и физиологически значимой 3D-моделью рака, доступной для исследований рака in vitro , поскольку они обеспечивают дополнительные преимущества по сравнению с сфероидами опухоли, а именно гетерогенность, обнаруженную у больных раком1. ФДО устанавливаются из опухолевой ткани, происходящей от больных раком, и, следовательно, сохраняют как фенотип опухоли, так и генотип. Таким образом, PDTO становятся бесценными для фундаментальных и трансляционных исследований рака и имеют потенциал для значительного улучшения точности онкологии2.
Несмотря на их многообещающий потенциал, эти сложные 3D-модели рака in vitro часто недостаточно используются из-за отсутствия передовых методов анализа. Наиболее часто используемый анализ определяет количество жизнеспособных клеток в PDTO посредством количественной оценки внутриклеточной АТФ3. Эти анализы обычно представляют собой одноточечный, объемный анализ, таким образом, игнорируя критические зависящие от времени ответы и пренебрегая клональными реакциями. В частности, способность контролировать рост ФДО (темпы роста) и их реакцию на специфические методы лечения представляет большой интерес 4,5. Нормализованный ответ на лекарство (NDR), который оценивает ответ на лекарство на основе скорости роста, нормализованной до положительного (ctrl +) и отрицательного контрольного (ctrl-) состояния, также недавно был зарегистрирован как решающий показатель для оценки чувствительности к лекарствам от рака с помощью скрининга на основе клеток, хотя это было преимущественно сделано для 2D-клеточных линий6. Поэтому необходимы более сложные методы анализа, чтобы в полной мере использовать эти более клинически репрезентативные и сложные 3D-модели рака. Микроскопия считается мощным подходом к изучению сложности этих органоидных моделей7.
В этой статье описывается метод мониторинга кинетических реакций лекарств в 3D-моделях рака с использованием обычных широкоугольных микроскопов и систем визуализации живых клеток. Адаптация была сделана к протоколу, описанному Driehuis et al.4 , чтобы быть совместимым с автоматизацией с использованием робота-пипетки, цифрового дозатора лекарств и системы визуализации живых клеток для повышения воспроизводимости и сокращения количества «практических» часов труда. Этот метод позволяет проводить скрининг лекарств со средней и высокой пропускной способностью как сфероидов опухоли с установленными линиями раковых клеток (см. Дополнительную таблицу S1 для тестируемых клеточных линий), так и PDTO в 384-луночном микропластине и мультиорганоидном формате. Используя сверточный сетевой процесс машинного обучения, автоматическая идентификация и отслеживание отдельных сфероидов опухолей или PDTO могут быть выполнены исключительно с помощью визуализации brightfield и без использования флуоресцентных красителей для маркировки живых клеток8. Это очень выгодно, так как большая часть идентификации с помощью визуализации яркого поля требует ручной аннотации (что является трудоемким и трудоемким) или требует добавления флуоресцентных красителей, которые могут запутать реакции лекарств, связанные с окислительным стрессом, вызванным фотоксичностью9.
Полученное программное обеспечение для анализа изображений, разработанное собственными силами, расширяет функциональность обычных систем визуализации живых клеток, поскольку модули анализа 3D-изображений либо недоступны, либо ограничены платформой, либо несовместимы с 384-луночными микропластинами и цельно-луночной визуализацией. Кроме того, эти модули часто имеют высокую цену и предлагают ограниченные объемные считывания органоидов. Поэтому этот метод очень актуален для ученых, которые имеют доступ к широко доступным системам визуализации живых клеток и стремятся извлечь больше информации о лекарственном ответе по сравнению с золотым стандартом, но рудиментарным анализом на основе АТФ. С добавлением специфических показателей гибели клеток реакции цитостатического препарата можно отличить от цитотоксических реакций, что обеспечивает дальнейшее понимание механистических действий лекарств, которые в настоящее время недостижимы из анализа с одной временной точкой. Наконец, визуализация живых клеток позволяет отслеживать отдельные органоиды для получения показателей ответа на отдельные органоидные препараты для захвата гетерогенности ответа и выявления потенциальных резистентных субклонов.
Целью этого метода и связанного с ним программного обеспечения для анализа изображений является внедрение недорогой автоматизации скрининга органоидных препаратов для ограничения вмешательства пользователя и снижения вариативности в обработке, анализе изображений и анализе данных. Чтобы сделать это программное обеспечение доступным для исследователей, оно не зависит от микроскопа и платформы, а также доступно облачное приложение. Таким образом, поддерживая обычные системы визуализации живых клеток, мы также стремимся улучшить их функциональность для приложений 3D-культивирования и анализа.
Использовались органоиды аденокарциномы протоков поджелудочной железы человека (PDAC), полученные от пациентов. Фрагменты резекции тканей были получены от пациентов, перенесших лечебную операцию в университетской больнице Антверпена. Письменное информированное согласие было получено от всех пациентов, и исследование было одобрено Этическим комитетом UZA (ref. 14/47/480). Подробная информация, относящаяся ко всем материалам, реагентам, оборудованию и программному обеспечению, используемым в этом протоколе, приведена в Таблице материалов. Обзор рабочего процесса представлен на рисунке 1. Пример данных приведен в дополнительном материале для воспроизведения протокола.
1. День 0: Препарат 2- или 3-дневных органоидов
- Разогрейте микропластины при 37 °C в течение ночи и разморозьте внеклеточный матрикс (ECM) при 4 °C.
- Приготовьте полную органоидную культуральную среду PDAC: дополнить ADF+++ (Advanced DMEM/F12, 1% добавка глутамина, 1% HEPES и 1% пенициллина/стрептомицина) с 0,5 нМ WNT суррогатного белка Fc-Fusion, 4% кондиционированной средой Noggin-Fc Fusion Protein, 4% кондиционированной средой Rpso3-Fc Fusion Protein, 1x B27, 1 мМ N-ацетилцистеина (NAC), 5 мМ никотинамида, 500 нМ A83-01, 100 нг/мл FGF10 и 10 нМ гастрина).
- Установите PDTO в соответствии с методом выбора.
ПРИМЕЧАНИЕ: Подробный протокол предоставлен Driehuis et al., который описывает традиционный метод установления, культивирования и прохождения PDTO в куполах ECM4. - Ферментативно диссоциируют органоиды в куполах ECM.
- Аспирировать среду и промыть 1 раз фосфатно-буферным физиологическим раствором (PBS). Добавьте фермент диссоциации (например, 2 мл в 6-луночной микропластине) и пипетку вверх и вниз 10x с пипеткой 1 мл для механической диссоциации органоидов и куполов ECM.
- Инкубируйте в течение 10 мин при 37 °C, пипеткой вверх и вниз, и проверьте, не диссоциированы ли органоиды на отдельные клетки. При необходимости повторите этот шаг.
- Соберите клеточную суспензию в пробирку объемом 15 мл, добавьте АДФ+++ к объему 10 мл, центрифугу в течение 5 мин при 450 × г при комнатной температуре и аспирируйте супернатант пипеткой Пастера и всасывающим насосом.
- Повторно суспендируют гранулу в 100-200 мкл полной среды в зависимости от размера гранулы и подсчитывают количество клеток по выбранному способу. Например: смешайте 10 мкл клеточной суспензии + 10 мкл Trypan Blue и подсчитайте с помощью автоматизированного счетчика клеток.
- Пластинчатые одиночные ячейки в куполах ECM.
- Разбавить клеточную суспензию и добавить 2/3 ECM согласно таблице 1. Пипетка до десяти капель по 20 мкл на лунку в предварительно нагретой 6-луночной пластине. Переверните пластину и инкубируйте в течение 30 мин при 37 °C.
- Накрыть полной средой, дополненной 10 мкМ Y-27632 и инкубировать в течение 2-3 дней в инкубаторе.
ПРИМЕЧАНИЕ: Десяти куполов, содержащих по 75 000 клеток каждый, обычно достаточно для заполнения одной микропластины из 384 скважин при концентрации 200 органоидов/лунка, исключая скважины на краю.
2. Дни 2 - 3: Сбор урожая и посев 2- или 3-дневных органоидов
- Соберите неповрежденные органоиды из куполов ECM.
ПРИМЕЧАНИЕ: Органоиды, как правило, прилипают к пластиковым поверхностям (например, трубкам, наконечникам пипеток). Чтобы избежать этого, пластиковую посуду можно предварительно промыть 0,1% раствором бычьего сывороточного альбумина (BSA) / PBS.- Аспирировать среду и промыть 1 раз PBS. Добавьте 1-2 мл холодного (4 °C) раствора для извлечения органоидов в 6-луночную пластину в зависимости от количества куполов ECM и высиживайте на льду на встряхивающей платформе в течение 10 мин.
- Пипетка вверх и вниз с пипеткой 1 мл, чтобы диссоциировать купола ECM, инкубировать в течение дополнительных 10 минут на льду и визуально проверить под микроскопом, не диссоциирован ли ECM.
- Необязательно: если предпочтительно более равномерное распределение по размеру, фильтруйте суспензию через клеточный сетчатый фильтр 70 мкм перед центрифугированием.
- Соберите органоиды в 15 мл пробирку, предварительно покрытую 0,1% BSA/PBS, добавьте адФ+++ до 10 мл и центрифугу в течение 5 мин при 200 × г при 4 °C. Аспирировать супернатант и повторно суспендировать гранулу в до 1000 мкл полной органоидной среды PDAC в зависимости от размера гранулы для получения концентрации >6000 органоидов/мл.
- Подсчитайте органоиды, используя любой метод подсчета по выбору, предпочтительно на основе изображения.
- Засейте органоиды.
ПРИМЕЧАНИЕ: Смотрите Таблицу материалов для минимального объема/скважины для двух различных типов 384-луночных микропластин, используемых в этом протоколе.- Предварительно охладите всю пластиковую посуду при -20 °C или на льду в течение не менее 20 минут перед использованием, чтобы избежать затвердевания ECM.
- Готовят посевной раствор из 1 мл раствора органоидного сырья (стадия 2.1.6), используя полную среду для посева ~200 органоидов на лунку в 50 мкл, минимальный объем, используемый для заполнения лунки. Используйте дополнительный файл 1 для расчета количества органоидного посевного раствора. Добавьте остаточный объем 1 500 мкл при использовании резервуара объемом 25 мл и многоканальной пипетки или пипеточного робота.
- Засейте органоиды с помощью робота-пипетки.
ПРИМЕЧАНИЕ: Как посевной раствор, так и микропластину необходимо охлаждать при 4°C во время пипетки, чтобы избежать затвердевания ECM. Поэтому резервуар объемом 25 мл и держатель микропластины были напечатаны на 3D-принтере для использования в сочетании с роботом-пипеткой, который может удерживать охлаждающие элементы, перечисленные в Таблице материалов. Предоставляются STL-файлы для 3D-печати пользовательского лабораторного ПО (Дополнительный файл 2 и Дополнительный файл 3) и пользовательские лабораторные JSON-файлы для робота-пипетки (Дополнительный файл 4 и Дополнительный файл 5).- Разработайте протокол дозирования с помощью интерактивного конструктора протоколов. Приведен пример JSON-файла (Дополнительный файл 6), в который уже загружено пользовательское лабораторное ПО и используется восьмиканальная пипетка p300 (Gen2) с соответствующими наконечниками пипеток.
- Откройте приложение управления роботом-пипеткой, выберите протоколы, нажмите «Импорт» и перетащите дополнительный файл 6 в указанное поле.
- Выберите импортированный протокол и поместите все лабораторное оборудование, включая охлаждающие элементы и пластиковую посуду, в колоды в соответствии с макетом, показанным в поле Deck Setup . Используйте левый слот для резервуара 25 мл и охлаждающего элемента, как показано в дополнительном файле 7.
- Нажмите «Запустить протокол» и перейдите к установке. Откройте вкладку Labware Setup, нажмите «Выполнить проверку положения Labware» и следуйте инструкциям по калибровке робота-пипетки на новое оборудование.
ПРИМЕЧАНИЕ: Данные смещения Labware могут быть сохранены на более поздний срок, но рекомендуется запускать проверку положения labware перед каждым запуском. - Заполните резервуар объемом 25 мл (размещенный поверх охлаждающего элемента) охлажденным раствором для посева органоидов и нажмите « Начать запуск».
ПРИМЕЧАНИЕ: Верхний и нижний колодцы также заполняются раствором органоидной суспензии из-за использования восьмиканальной пипетки. - Центрифугируйте микропластинку в течение 1 мин при 100 × г при 4 °C.
- Инкубировать при 37 °C в течение не менее 30 мин.
- Наполните наружные пустые колодцы не менее чем 50 мклH2Oво избежание испарения.
- Инкубируйте при 37 °C в течение ночи, чтобы удалить любые пузырьки в скважине, которые могут помешать анализу изображения.
3. День 4: Лечение лекарств и отпуск реагентов с помощью цифрового диспенсера для лекарств
- Создайте протокол дозирования лекарств с помощью программного обеспечения для управления цифровым диспенсером лекарств.
- Наведите курсор на пластину 1 над макетом пластины, выберите атрибуты редактирования пластины и заполните тип пластины: 384 скважины, дополнительный объем (мкл): 50 и предел DMSO (%): 1.
- Добавьте жидкости, нажав на кнопку + рядом с Fluids. Дважды щелкните по вновь созданной жидкости и назовите ее; выберите класс (на основе ДМСО или водный +Tween 20) и концентрацию.
ПРИМЕЧАНИЕ: Все лекарственные средства и реагенты должны быть растворены в 100% ДМСО или 0,3% Твин-20. Можно использовать запасной раствор 1-10 мМ с учетом максимальной концентрации ДМСО <1%. В таблице 2 приведены примеры требуемых разведений для обычных флуоресцентных реагентов и методов лечения. - Компоновка плит
- Для титрования препарата выберите колодцы и нажмите на Титрование. Для жидкости выберите интересующий препарат, выберите самую высокую концентрацию (например, 2000 нМ) и самую низкую концентрацию (например, 10 нМ); для реплик выберите минимум 2 и выберите желаемый шаблон титрования.
ПРИМЕЧАНИЕ: Структура титрования будет зависеть от многих факторов, в том числе от того, сколько соединения должно быть помещено в одну пластину, должны ли скважины быть рандомизированы, а также от количества реплик и контрольных элементов. - Для положительного контроля выберите три скважины, нажмите «Установить значение» и заполните 2 мкМ ставроспорина из запаса 10 мМ в ДМСО, что вызовет максимальную гибель клеток.
- Для Cytotox Green выберите все используемые скважины, нажмите «Установить значение» и введите 60 нМ/лунка.
ПРИМЕЧАНИЕ: Флуоресцентное окрашивание Cytotox Green указывает на клетки, которые погибли, и, следовательно, не будет мешать мониторингу реакции на лекарство. Здесь не требуется флуоресцентный маркер для живых клеток. - Для отрицательного контроля и нормализации DMSO выберите все скважины с дополнительными четырьмя скважинами для управления транспортным средством, щелкните правой кнопкой мыши, выберите нормализацию, выберите класс нормализуемой жидкости: на основе DMSO и нормализуйте до объема самого высокого класса для получения равной концентрации DMSO в каждой скважине.
ПРИМЕЧАНИЕ: Концентрации ДМСО должны составлять <1%. Приведен пример файла титрования лекарственного средства TDD (Дополнительный файл 8). - Нажмите на стрелку в разделе «Выполнить» в левом верхнем углу, выберите «Всегда имитировать» и нажмите « Имитировать », чтобы выявить любые ошибки и получить объемы каждого препарата, который необходимо подготовить.
ПРИМЕЧАНИЕ: Чтобы преодолеть предупреждение, когда начальный объем дозирования слишком мал, «Для каждой жидкости на каждой пластине рекомендуется дозировать предупреждающий колодец 30 нл или более», выберите два отверстия на краю, заполненные водой, выберите «Установить значение» и введите 10 мкМ препарата, для которого происходит предупреждение. Это приводит к прайму картриджа с объемом выше 30 нЛ. Эти же скважины могут быть использованы для заправки картриджа DMSO путем нормализации до % от общего объемного значения (например, 0,5%).
- Для титрования препарата выберите колодцы и нажмите на Титрование. Для жидкости выберите интересующий препарат, выберите самую высокую концентрацию (например, 2000 нМ) и самую низкую концентрацию (например, 10 нМ); для реплик выберите минимум 2 и выберите желаемый шаблон титрования.
- Снимите флажок Всегда имитировать под кнопкой Выполнить ; нажмите « Выполнить », чтобы запустить протокол дозирования лекарств и следуйте инструкциям.
- Нанесите уплотнительную мембрану на микропластинку, чтобы предотвратить испарение.
- Инкубируют краситель Cytotox Green через 1-2 ч при 37 °C в инкубаторе и переходят к этапу 4.
4. Получение изображений с помощью теискателя live-cell
ПРИМЕЧАНИЕ: Для скорости роста и NDR сканирование в точке времени 0 (T0 = начало лечения) должно быть получено через 1-2 ч после добавления Cytotox Green.
- Откройте программное обеспечение Управления образом live-cell, выберите Новый редактор методов, перейдите к файлу > импорта и выберите пример XML-файла метода (Дополнительный файл 9). Кроме того, создайте новый файл и выберите Пластина: (CORE384fb_OpticalImaging) - Corning 384 Flat Black (Corning #4588), Без крышки и без кассеты влажности; Применение: только изображения; Цель: 4x; Выкройка: Центральная; Проверьте каналы Brightfield и Green (интенсивность светодиодов (%) = 40; Время экспозиции (мс) = 200).
ПРИМЕЧАНИЕ: Настройки зеленого канала хорошо работают для концентрации 60 нМ Cytotox Green. Опция Live Viewer может использоваться для настройки смещения фокуса и/или настроек светодиода в режиме реального времени. - Нажмите кнопку Пуск , чтобы начать сканирование в T0.
- Повторяйте сканирование каждые 24 часа в течение 5 дней, используя тот же метод. Кроме того, чтобы автоматически запустить измерение таймлапса, настройте метод в программном обеспечении управления образом live-cell для кинетического эксперимента, щелкнув и перетащив вкладку Kinetic Loop в поле метода . Аналогичным образом, вкладки «Температура» и «Газ » должны быть перетащены в поле метода , чтобы установить систему на 37 ° C и 5% CO2 , чтобы обеспечить правильные условия в тепловизоре живых клеток во время эксперимента.
5. Анализ изображений и данных
- Объединение и сжатие данных
- Программное обеспечение управления образчиком live-cell создает папку для каждого сканирования в каждой точке времени. Создайте новую папку, скопируйте отдельные папки эксперимента в эту новую родительскую папку и добавьте _0h, _24h, _48h, _72h, _96h и _120h к соответствующим именам папок эксперимента.
- Подготовьте карту пластин XLSX из программного обеспечения digital drug dispenser Control, щелкнув правой кнопкой мыши на макете карты пластин из протокола дозирования лекарств и скопируйте все колодцы; вставьте данные в XLSX-файл. Удалите данные Cytotox Green и staurosporine и добавьте матрицу для Cell Line и Replicate. Введите ctrl- и ctrl+. Пример карты пластин см. в дополнительном файле 10 .
- Откройте инструмент сжатия данных, нажмите « Обзор», выберите родительскую папку и нажмите « Выполнить », чтобы инициировать сжатие данных изображения. Все файлы изображений TIFF для разных точек времени сжимаются в один HDF5 для каждой скважины в новой папке наборов данных в родительской папке.
- Анализ изображений
- Перейдите на платформу веб-приложения для анализа изображений, войдите в систему и нажмите «Добавить новый проект» на вкладке « Главная ». Введите имя проекта, продолжите, выберите Добавить новый эксперимент и отправьте папку наборов данных, содержащую файлы HDF5.
- После загрузки перейдите в папку проекта и эксперимента и нажмите «Загрузить карту тарелок» для получения дополнительных функций. Нажмите «Выполнить анализ», выберите «Анализ нескольких органоидов», «Параметры по умолчанию» и нажмите «Анализ», чтобы начать анализ изображений.
- Нажмите «Загрузить результаты», чтобы загрузить таблицы необработанных данных, которые содержат измерения для каждой скважины (например, общая площадь яркого поля, общая зеленая зона флуоресценции и т. Д.), А также сегментированные изображения / видео для подтверждения точности анализа и дальнейшей обработки данных.
- Показатели лекарственного ответа на основе темпов роста и нормализованные ответные меры на лекарства
- Выберите файл Raw_NDR.xlsx из папки результатов (требуется сопоставление пластин) (дополнительный файл 11) и загрузите его в Official_NDR_7point R-script (дополнительный файл 12) для автоматического создания таблиц значений GR (нормализованных до ctrl-) и NDR (нормализованных до ctrl- и ctrl+) (дополнительный файл 13, дополнительный файл 14, дополнительный файл 15 и дополнительный файл 16 ). Значения GR и NDR вычисляются из параметра, как показано в уравнении (1), с помощью R-скрипта (Дополнительный файл 12).
Общая площадь выживания = Общая площадь Брайтфилда - Общая зеленая зона (1)
Где 0 < NDR <1 = цитостатический эффект (остановка роста), а NDR < 0 = цитотоксический ответ (гибель клеток).
ПРИМЕЧАНИЕ: R-скрипт был адаптирован из Gupta et al.6. - Из таблицы clonal_data.xlsx извлеките данные ответа с одним органоидом и постройте их в виде пузырькового графика.
- Используйте Z-фактор10 для оценки качества лекарственного скрининга при прогоне (см. уравнение (2)). Откажитесь от эксперимента с Z-фактором < 0,5.
 (2)
(2)
- Выберите файл Raw_NDR.xlsx из папки результатов (требуется сопоставление пластин) (дополнительный файл 11) и загрузите его в Official_NDR_7point R-script (дополнительный файл 12) для автоматического создания таблиц значений GR (нормализованных до ctrl-) и NDR (нормализованных до ctrl- и ctrl+) (дополнительный файл 13, дополнительный файл 14, дополнительный файл 15 и дополнительный файл 16 ). Значения GR и NDR вычисляются из параметра, как показано в уравнении (1), с помощью R-скрипта (Дополнительный файл 12).
Протокол автоматизированного дозирования обеспечивает равномерное распределение PDAC_060 PDTO во всех колоннах 384-луночной микроплиты (рисунок 2A). Как и ожидалось, между скважинами наблюдалось изменение числа и средней площади PDTO (рисунок 2A,B). Общая площадь выживания (total brightfield area - total green area) сочетает в себе сегментацию органоидов без меток с флуоресцентным сигналом гибели клеток и, по нашему опыту, является наиболее надежным параметром для изучения реакций лекарств с течением времени (рисунок 2C)8. Чтобы учесть различия в посеве клеток и размере органоидов, следует использовать метрики, основанные на скорости роста, для уменьшения вариаций между репликациями, как показано уменьшенными полосами ошибок на рисунке 2D по сравнению с рисунком 2C, и более высоким Z-фактором, указывающим на значительно улучшенное качество лекарственного экрана (рисунок 2E).
Кривая доза-реакция NDR (рисунок 2G), нормализованная до ctrl- и ctrl+, явно превосходит кривую доза-реакция GR (рисунок 2F), нормализованную до ctrl-, поскольку она увеличивает разделение кривых лекарственного ответа и более точно представляет цитотоксические реакции на лекарственные средства. Пример связанных изображений для PDTO, обработанных паклитакселом ctrl-, ctrl+ и 400 нМ/80 нМ, обработанных паклитакселом, показан на рисунке 3. Интересное наблюдение заключается в том, что цитотоксический эффект гемцитабина был доминирующим в комбинированной терапии, поскольку не наблюдалось добавленной ценности паклитаксела.
Затем были использованы две дополнительные линии PDTO, PDAC_052 и PDAC_087. Наблюдалась четкая разница в темпах роста между этими линиями (рисунок 4А), что подтверждает использование gr-метрик. Опять же, кривые доза-реакция NDR (рисунок 4C) привели к увеличению динамического диапазона и разделения между тремя различными пациентами по сравнению с кривыми GR (рисунок 4B). Кроме того, протокол позволяет определять NDR с течением времени и показывает, что PDAC_052 и PDAC_060 имели очень похожий цитостатический лекарственный ответ на низкую дозу gem-pac (рисунок 4D), в то время как четкий дифференциальный цитостатический и цитотоксический ответ можно было наблюдать для средних (Рисунок 4E) и высоких доз (Рисунок 4F) gem-pac. Эти реакции на препарат соответствовали клиническим реакциям, наблюдаемым у пациентов (рисунок 4G).
Наконец, основным преимуществом подхода и программного обеспечения является то, что реакции на одноорганоидные лекарственные средства могут быть количественно определены для изучения гетерогенности ответа и выявления потенциально устойчивых субклонов. Рисунок 5 дает четкий обзор клональной динамики различных пациентов и показывает, что PDAC-087 имел наиболее устойчивые субклоны после лечения, что согласуется с агрессивным и высокорезистентным заболеванием, наблюдаемым у пациента. Интересно, что этот пациент также был наименее чувствителен к ctrl+ ставроспорину.
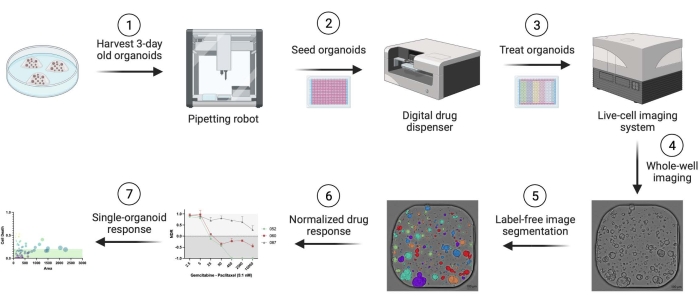
Рисунок 1: Обзор рабочего процесса. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
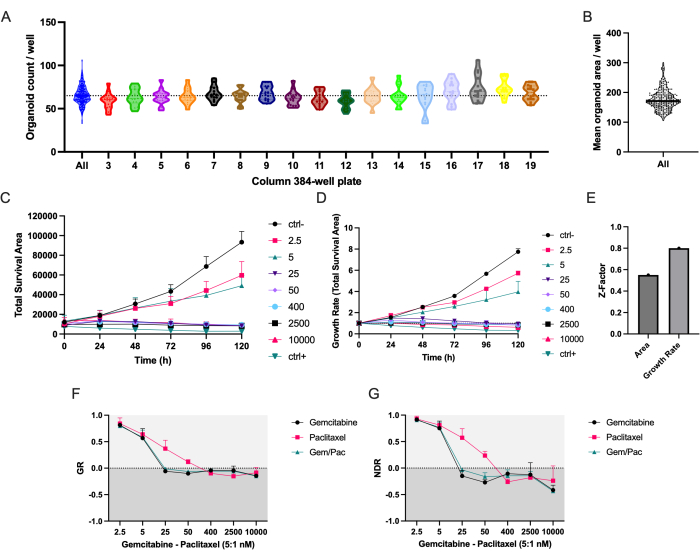
Рисунок 2: Точность посева и показатели реакции на лекарства. (A) Количество органоидов / скважина PDAC_060 PDTO, посеянных в микропластине из 384 скважин с использованием пипеточного робота. Каждая точка представляет собой количество в одной скважине, а участки разделены колоннами микропластин из 384 скважин. (B) Среднее значение PDTO площади/скважины. (C) Общая площадь выживания (общая площадь яркого поля - общая зеленая зона) и (D) скорость роста (общая площадь выживания нормализована до T0 = 1) PDAC_060 ФДО, обработанных соотношением гемцитабин/паклитаксел 5:1. (E) Z-фактор как показатель качества анализа. (F) Кривая скорости роста-дозы реакции нормализована до ctrl- и (G) нормализованной кривой лекарственного ответа, нормализованной до ctrl- и ctrl+. Полосы ошибок указывают на среднее ± SD двух скважин. Сокращения: PDAC = аденокарцинома протоков поджелудочной железы; PDTO = органоид опухоли, полученный из пациента; GR = темпы роста; NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
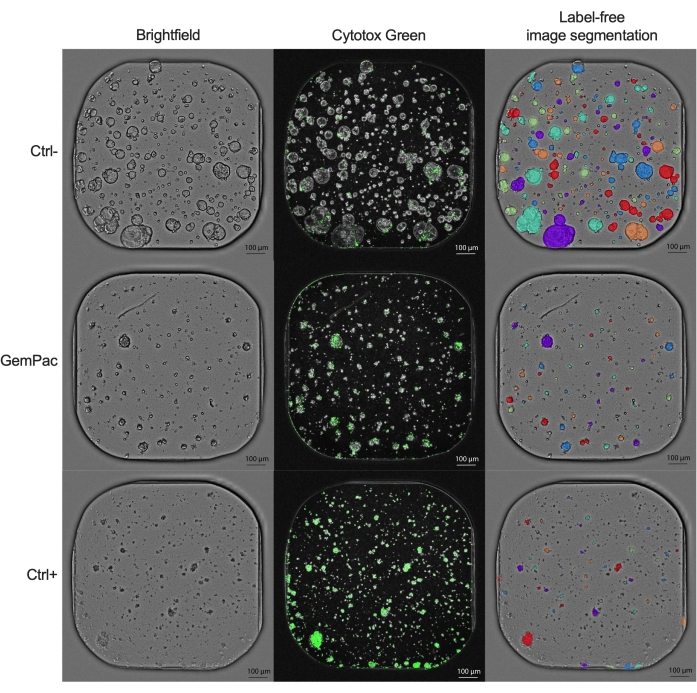
Рисунок 3: Примеры изображений. Репрезентативные изображения PDAC_060 PDTO, обработанных транспортным средством (ctrl-), 400 нМ гемцитабина / 80 нМ паклитаксела и 2 мкМ ставроспорина (ctrl +). Левый столбец показывает изображения яркого поля, средний столбец показывает флуоресцентный сигнал Cytotox Green, а правый столбец показывает аннотированные изображения brightfield без меток с использованием модуля органоидного анализа. Шкала стержней = 100 мкм. Сокращения: PDAC = аденокарцинома протоков поджелудочной железы; PDTO = органоид опухоли, полученный из пациента; GemPac = гемцитабин/паклитаксел. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
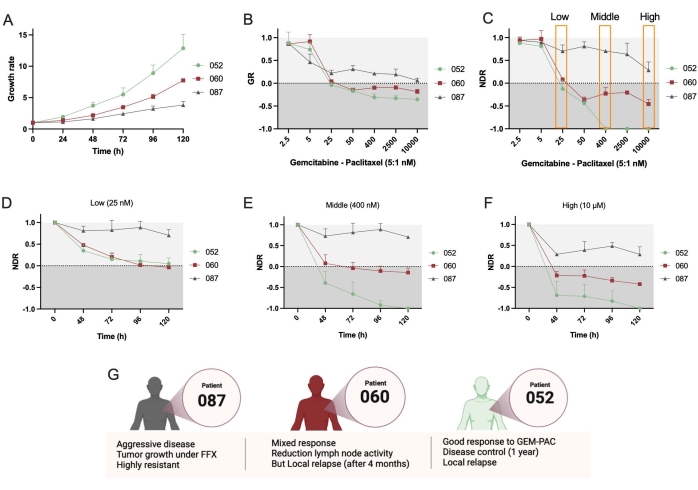
Рисунок 4: Сравнение межпаципанитного лекарственного ответа. (A) Сравнение темпов роста (на основе общей площади выживаемости) линий PDAC_052, PDAC_060 и PDAC_087 PDTO. (B) Кривая скорости роста-дозы реакции нормализована до ctrl- и (C) нормализованной кривой реакции лекарственного средства, нормализованной до ctrl- и ctrl+. Кинетический NDR (D) низкой, (E) средней и (F) высокой дозы гемцитабина/паклитаксела (соотношение 5:1). (G) Клинические характеристики пациентов с PDAC. Полосы ошибок указывают на среднее ± SD двух скважин. Сокращения: PDAC = аденокарцинома протоков поджелудочной железы; PDTO = органоид опухоли, полученный из пациента; GR = темпы роста; NDR = нормализованный ответ на наркотики; FFX = фолфиринокс. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
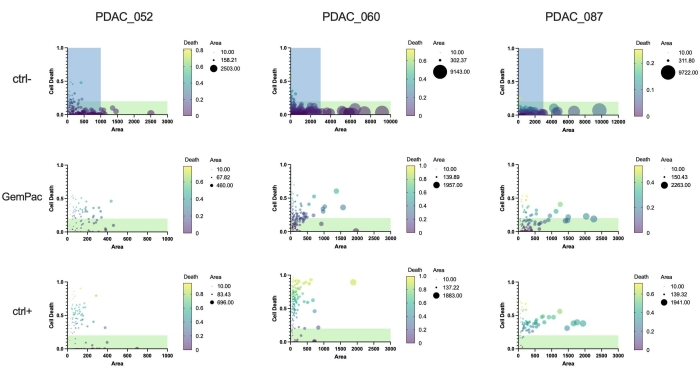
Рисунок 5: Одиночные органоидные метрики. Реакция на дозу одного органоида, основанная на гибели клеток (зеленая зона / область яркого поля) и области (яркое поле) PDAC_052, PDAC_060 и PDAC_087 PDTO, обработанных транспортным средством (ctrl-), 400 нМ гемцитабина / 80 нМ паклитаксела и 2 мкМ ставроспорина (ctrl +). Зеленые области указывают на жизнеспособные органоиды; синие области обозначают диапазон x-as графиков GemPac и ctrl+. Сокращения: PDAC = аденокарцинома протоков поджелудочной железы; PDTO = органоид опухоли, полученный из пациента; GemPac = гемцитабин/паклитаксел. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| Запас клеточной суспензии | Ячейки/Капля | # Капли (20 мкл) | Сток (1/3) | ECM (2/3) |
| 1,13 × 107 ячеек/мл | 75,000 | 10 | 75 ул | 150 мкл |
| 1,13 × 107 ячеек/мл | 75,000 | 5 | 40 ул | 80 мкл |
Таблица 1: Разбавление для нанесения покрытий в куполах ECM. Аббревиатура: ECM = внеклеточный матрикс.
| Соединение | Концентрация запасов | Разбавление | Рабочая концентрация | Растворитель | Концентрация скважин | Комментарии |
| Цитотокс Грин | 1 мМ (DMSO) | 1/10 | 10 мкМ | ДМСО | 60 нМ | Маркер гибели клеток |
| Цитотокс Красный | 1 мМ (DMSO) | 1/10 | 10 мкМ | ДМСО | 250 нМ | Маркер гибели клеток |
| Каспаза 3/7 Зеленый | 5 мМ (ДМСО) | 1/2 | 2,5 мМ | ДМСО | 2,5 мкМ | Апоптотический маркер |
| Хёхст | 20 мМ (H2O) | 1/200 | 100 мкМ | 0,33% Анимация движения/PBS | 50 нМ | Ядерный маркер |
| Ставроспорин | 10 мМ (DMSO) | / | 1 - 10 мМ | / | 2 – 5 мкМ | Позитивный контроль |
| Гемцитабин | 10 мМ (DMSO) | / | 1 - 10 мМ | / | Титрование | Химиотерапия |
| Паклитаксел | 10 мМ (DMSO) | / | 1 - 10 мМ | / | Титрование | Химиотерапия |
| Цисплатин | 5 мМ (0,9% NaCl) | 1/2 | 2,5 мМ | 0,6% Анимация движения/PBS | Титрование | Химиотерапия |
Таблица 2: Примеры разведений часто используемых лекарственных средств и флуоресцентных реагентов. Каждое соединение должно быть растворено либо в 100% DMSO, либо в 0,3% Tween/PBS.
Дополнительная таблица S1: Обзор совместимых линий раковых клеток. Статический: сфероиды не являются мигрирующими. Слияние: сфероиды мигрируют навстречу друг другу и сливаются вместе. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 1: Инструмент расчета раствора для посева органоидов. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 2: STL-файл для 3D-печати пользовательского лабораторного поттера «Держатель микропластины». Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 3: STL-файл для 3D-печати пользовательского лабораторного оборудования «Держатель резервуара 2 х 25 мл». Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 4: JSON-файл для пользовательского робота-пипетки лабораторного оборудования 'Microplate Holder'. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 5: JSON-файл для пользовательского робота для пипетки лабораторного оборудования '2 x 25 мл Резервуар Holder_WithCooler'. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 6: JSON-файл для пипетирования протокола робота 'Plating_ PDO_384well_Cooled_Row2-23'. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 7: Обзор настройки рабочего стола робота-пипетки. (A) Охлаждающие элементы и (B) резервуар и микропластина. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 8: Файл TDD для протокола цифрового диспенсера лекарств. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 9: XML-файл для протокола тепловизора живых клеток для визуализации ярких полей и флуоресцентных изображений. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 10: Пример карты пластин. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 11: Пример входного файла для сценария R отчета о недоставке. Аббревиатура: NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 12: Нормализованный сценарий NDR R реакции на наркотики. Аббревиатура: NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 13: Пример выходного файла значений GR скрипта NDR R. Сокращения: GR = темпы роста; NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 14: Пример выходного файла скрипта NDR R с транспонированными значениями GR. Сокращения: GR = темпы роста; NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Supplementary File 15: Пример выходного файла значений отчета О недоставке R сценария отчета о недоставке. Аббревиатура: NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 16: Пример выходного файла сценария R отчета о недоставке с перенесенными значениями отчета о недоставке. Аббревиатура: NDR = нормализованный лекарственный ответ. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Скрининг на лекарства PDTO со средней и высокой пропускной способностью часто опирается на показания, которые извлекают только часть информации, которую органоиды потенциально могут предоставить. Становится все более очевидным, что для того, чтобы быстро развивающаяся органоидная технология реализовала больший научный и клинический потенциал, критически необходимы более продвинутые 3D-анализы, показания и методы анализа. Здесь описан усовершенствованный конвейер скрининга, который не только повышает воспроизводимость, но и значительно повышает клиническую переводимость за счет включения считывания изображений живых клеток на основе ИИ. В дополнение к программному обеспечению для анализа, разработанному собственными силами, реализовано использование нормализованной метрики лекарственного ответа (NDR), которая ясно демонстрирует ее способность определять специфические для пациента различия в ответе на лечение6.
Включение этого показателя нормализации, несомненно, будет иметь огромное значение, напоминая, что многочисленные исследования направлены на то, чтобы очертить ответы на лечение на основе незначительных различий в площади под кривой (AUC) или полумаксимальной ингибирующей концентрации (IC50) (поскольку большинство кривых «доза-реакция» перекрываются / расположены близко друг к другу)11,12 . Показатели скорости роста уже внедрены в протоколы скрининга органоидных препаратов с использованием анализа на основе АТФ, но полагаются на нормализацию эталонных скважин, лизированных в точке времени 04. Напротив, этот метод позволяет нормализовать скорость роста внутри скважины, что учитывает не только межпациентные различия в скорости роста PDTO, но и межскважинные различия, возникающие в результате изменений плотности посева и эффектов, зависящих от местоположения пластин, для повышения воспроизводимости. Кроме того, мы адаптировали NDR для дальнейшего увеличения разделения межпациентного ответа PDTO путем включения положительного контроля для нормализации 6,8.
Кроме того, анализ, который совместим с высокопроизводительными и автоматизированными форматами, может точно обнаруживать отдельные органоидные реакции, что позволяет количественно оценить субклональную резистентность - основную движущую силу рецидива и прогрессирования опухоли13. Например, хотя PDAC052 и PDAC060 показали хороший ответ на лечение in vitro (на основе NDR), дополнительный одноорганоидный анализ смог обнаружить небольшую (большую популяцию с PDAC060) популяцию субклонов, которые не реагируют на лечение. Интересно, что это в значительной степени соответствовало клиническим наблюдениям, учитывая, что PDAC052 и PDAC060 имели длительный ответ (опухолевая активность не обнаружена), но в конечном итоге оба были диагностированы с местной опухолевой прогрессией (из-за присутствия резистентных клонов). По сравнению с обычными 3D-показаниями (анализ на основе АТФ и размер / числа), ожидается, что этот усовершенствованный конвейер скрининга повысит прогностическую производительность за счет извлечения более клинически значимой информации из этих «пациентов в лаборатории». Эта гипотеза в настоящее время проверяется путем скрининга клинических образцов PDTO в лаборатории авторов с помощью этого метода, чтобы соотнести ex vivo с ответом in vivo и клиническим исходом.
Чтобы получить больше информации о механизмах лекарственного ответа, обычные флуоресцентные реагенты визуализации живых клеток, в дополнение к красителям цитотоксичности, совместимы с этим методом для изучения механизмов гибели клеток. Ранее мы показали совместимость этого метода с зеленым реагентом Sartorius Caspase 3/7 для изучения каспазозависимой индукции апоптоза после лечения цисплатином8. Совместимость с другими красителями для изучения окислительного стресса (реагенты CellROX) или гипоксии (реагенты Image-iT Hypoxia) еще предстоит проверить. Однако эти реагенты уже успешно использовались в 3D моделях in vitro 14,15.
Программное обеспечение для анализа изображений также совместимо с другими форматами пластин или методами культивирования (например, пластинами микрополости, куполами ECM), если могут быть получены четкие, сфокусированные изображения органоидов. Это часто является сложной задачей для органоидов, культивируемых в куполах, поскольку они растут в разных z-плоскостях, что требует функциональности z-укладки микроскопа, которая не всегда доступна. Поэтому мы советуем использовать микропластины с плоским дном ULA 384-well для обеспечения изображения достаточного качества.
Кроме того, анализ совместим с другими системами визуализации живых клеток, как было показано ранее для фазоконтрастных изображений, снятых с помощью системы IncuCyte ZOOM8. Ограничением системы визуализации живых клеток Spark Cyto, которая использовалась в этой рукописи, является емкость одной пластины для кинетических измерений. Тем не менее, расширение Spark Motion увеличивает его емкость до 40 микропластин, которые могут быть экранированы оптом. Совместимость программного обеспечения, разработанного собственными силами, будет распространена на эти и другие системы, чтобы предложить платформенно-независимое решение с целью стандартизации и автоматизации конвейеров анализа изображений и данных. Веб-приложение также будет включать интерактивные графические инструменты и автоматизированные расчеты метрик лекарств, как показано в этом документе, для сокращения времени ручного анализа.
Алгоритм сегментации PDTO без меток был обучен и протестирован на различных собственных моделях сфероидов и PDTO с различными морфологическими различиями (твердое, полутвердое, цистическое) и, следовательно, может обнаруживать их с высокой точностью8. Ограничение модели заключается в том, что включение кистозных PDTO увеличило нежелательное обнаружение пузырьков, присутствующих в скважине после посева. Тем не менее, ночной инкубации было достаточно, чтобы удалить большинство из этих пузырьков, что позволило провести качественное сканирование точки времени 0. Точность сегментации органоидного изображения и метода должна быть проверена другими пользователями, и на основе их отзывов программное обеспечение может быть обучено дополнительно для получения надежного и автоматизированного алгоритма анализа изображений. Кроме того, мы стремимся получить больше клинических данных для корреляции лекарственного ответа ex vivo , количественного с помощью этого метода, с клиническим ответом у пациента, чтобы определить наилучшие параметры для прогнозирования ответа на терапию и дальнейшей разработки этого метода для функциональной прецизионной медицины рака16.
Авторы заявляют об отсутствии конфликта интересов.
Часть этого исследования финансировалась за счет пожертвований от различных доноров, включая Дедерта Шильда vzw и г-на Вилли Флорена. Эта работа частично финансируется Фламандским исследовательским фондом, 12S9221N (A.L.), G044420N (S.V., A.L., E.G), 1S27021N (M.L),и Фондом промышленных исследований Университета Антверпена, PS ID 45151 (S.V., A.L., C.D.). Рисунок 1 был создан с помощью BioRender.com.
| Name | Company | Catalog Number | Comments |
| 6-well plate | Greiner | 657160 | |
| 8-Channel p300 (GEN 2) pipette | Opentrons | ||
| 300 µL Tips | Opentrons | ||
| 384-well flat-bottom ULA microplate | Corning | 4588 | minimum volume 50 µL |
| 384-well flat-bottom ULA Phenoplate | Perkin Elmer | 6057802 | minimum volume 75 µL |
| A8301 | Tocris Bioscience | 2939 | |
| ADF+++ | Advanced DMEM/F12, 1% GlutaMAX, 1% HEPES, 1% penicillin/streptomycin | ||
| Advanced DMEM/F-12 | ThermoFisher Scientific | 12634 | |
| B27 | ThermoFisher Scientific | 17504044 | |
| Breathe easy sealing membrane | Sigma-Aldrich | Z380059 | |
| Caspase 3/7 Green | Sartorius | 4440 | |
| Cell Counting Slides for TC10/TC20 | Bio-Rad Laboratories | 1450017 | |
| CellTiter-Glo 3D | Promega | G9681 | ATP-assay |
| Cooler for 25 mL reservoir | VWR (Diversified Biotech) | 490006-908 | |
| Cooling element 12 x 8 x 3 cm | Bol.com | 9200000107744702 | For custom microplate holder OT-2 |
| Cultrex Organoid Harvesting Solution | R&D systems | 3700-100-01 | |
| Cultrex PathClear Reduced Growth Factor BME, Type 2 | R&D systems | 3533-010-02 | extracellular matrix (ECM) |
| Cytotox Green | Sartorius | 4633 | |
| Cytotox Red | Sartorius | 4632 | |
| D300e | Tecan | Digital drug dispenser | |
| D300e Control v3.3.5 | Tecan | Control software D300e | |
| FGF10 | Peprotech | 100-26 | |
| Full Medium | ADF+++ supplemented with 0.5 nM WNT surrogate-Fc-Fusion protein, 4% Noggin-Fc Fusion Protein conditioned medium, 4% Rpso3-Fc Fusion Protein conditioned medium, 1x B27, 1 mM N-acetyl cysteine (NAC), 5 mM nicotinamide, 500 nM A83-01, 100 ng/mL FGF10, and 10 nM Gastrin | ||
| Gastrin | Sigma-Aldrich | G9145 | |
| Gemcitabine | Selleck Chemicals | S1714 | |
| GlutaMAX | ThermoFisher Scientific | 35050 | |
| HEPES | ThermoFisher Scientific | 15630056 | |
| Hoechst 33342 Solution (20 mM) | ThermoFisher Scientific | 62259 | |
| Human pancreatic ductal adenocarcinoma (PDAC) patient-derived organoids | Biobank@uza (Antwerp, Belgium; ID: BE71030031000; Belgian Virtual Tumorbank funded by the National Cancer Plan) | ||
| N-acetyl-cysteine | Sigma-Aldrich | A9165-25G | |
| Nicotinamide | Sigma-Aldrich | N0636-100G | |
| Noggin-Fc Fusion Protein conditioned medium | Immunoprecise | N002 | |
| Opentrons App v6.0.1 | Opentrons | OT-2 control software | |
| Opentrons Protocol Designer Tool | Opentrons | https://designer.opentrons.com/ | |
| Orbits data compression tool | www.orbits-oncology.com or contact corresponding author | ||
| Orbits image analysis webapp | University of Antwerp | www.orbits-oncology.com or contact corresponding author | |
| OT-2 | Opentrons | Pipetting robot | |
| Paclitaxel | Selleck Chemicals | S1150 | |
| Pasteur Pipette 230 mm | Novolab | A33696 | |
| Peniciline-Streptomycin | ThermoFisher Scientific | 15140 | |
| Prism 9 | GraphPad | ||
| Rspo3-Fc Fusion Protein conditioned medium | Immunoprecise | N003 | |
| Spark Cyto 600 | Tecan | Live-cell imaging and multi-mode platereader | |
| SparkControl v3.1 | Tecan | Spark Cyto control software | |
| Staurosporine | Tocris Bioscience | 1285 | |
| Sterile 25 mL reservoir | VWR (Diversified Biotech) | 10141-922 | |
| T8 plus cassette | Tecan | ||
| TC20 | Bio-Rad Laboratories | automated cell counter | |
| TrypLE | ThermoFisher Scientific | 12604-021 | dissociation enzyme |
| Tween-20 | Acros Organics | 233360010 | |
| WNT Surrogate-Fc-Fusion protein | Immunoprecise | N001 | |
| Y-27632 | Selleck Chemicals | S1049 |
- Drost, J., Clevers, H. Organoids in cancer research. Nature Reviews Cancer. 18 (7), 407-418 (2018).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Le Compte, M., et al. Patient-derived organoids as individual patient models for chemoradiation response prediction in gastrointestinal malignancies. Critical Reviews in Oncology/Hematology. 157, 103190 (2021).
- Driehuis, E., Kretzschmar, K., Clevers, H. Establishment of patient-derived cancer organoids for drug-screening applications. Nature Protocols. 15 (10), 3380-3409 (2020).
- Hafner, M., Niepel, M., Chung, M., Sorger, P. K. Growth rate inhibition metrics correct for confounders in measuring sensitivity to cancer drugs. Nature Methods. 13 (6), 521-527 (2016).
- Gupta, A., Gautam, P., Wennerberg, K., Aittokallio, T. A normalized drug response metric improves accuracy and consistency of anticancer drug sensitivity quantification in cell-based screening. Communications Biology. 3 (1), 42 (2020).
- Rios, A. C., Clevers, H. Imaging organoids: a bright future ahead. Nature Methods. 15 (1), 24-26 (2018).
- Deben, C., Cardenas, E., et al. OrBITS: label-free and time-lapse monitoring of patient derived organoids for advanced drug screening. Cellular Oncology. , (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), (2017).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Engel, R. M., et al. Patient-derived colorectal cancer organoids upregulate revival stem cell marker genes following chemotherapeutic treatment. Journal of Clinical Medicine. 9 (1), 128 (2020).
- Hennig, A., et al. Detecting drug resistance in pancreatic cancer organoids guides optimized chemotherapy treatment. The Journal of Pathology. 257 (5), 607-619 (2022).
- D'Alterio, C., Scala, S., Sozzi, G., Roz, L., Bertolini, G. Paradoxical effects of chemotherapy on tumor relapse and metastasis promotion. Seminars in Cancer Biology. 60, 351-361 (2020).
- Njoroge, R. N., et al. Organoids model distinct Vitamin E effects at different stages of prostate cancer evolution. Scientific Reports. 7 (1), 16285 (2017).
- Godet, I., et al. Fate-mapping post-hypoxic tumor cells reveals a ROS-resistant phenotype that promotes metastasis. Nature Communications. 10 (1), 4862 (2019).
- Letai, A. Functional precision cancer medicine-moving beyond pure genomics. Nature Medicine. 23 (9), 1028-1035 (2017).
Tags
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved