Análise Imuno-Histoquímica Multiplex da Paisagem Espacial de Células Imunes do Microambiente Tumoral
* These authors contributed equally
In This Article
Summary
Este protocolo descreve em detalhes como é realizada a caracterização das células imunes do microambiente tumoral usando imuno-histoquímica multiplex.
Abstract
A paisagem de células imunes do microambiente tumoral potencialmente contém informações para a descoberta de biomarcadores prognósticos e preditivos. A imuno-histoquímica multiplex é uma ferramenta valiosa para visualizar e identificar diferentes tipos de células imunes em tecidos tumorais, mantendo suas informações espaciais. Aqui, fornecemos protocolos detalhados para analisar populações de células linfocitárias, mieloides e dendríticas em seções de tecido. Desde o corte de seções embebidas em parafina fixadas em formalina, procedimentos automáticos de coloração multiplex em uma plataforma automatizada, digitalização das lâminas em um microscópio de imagem multiespectral, até a análise de imagens usando um algoritmo de aprendizado de máquina desenvolvido internamente ImmuNet. Esses protocolos podem ser aplicados a uma variedade de amostras de tumor simplesmente trocando os marcadores tumorais para analisar células imunes em diferentes compartimentos da amostra (tumor versus margem invasiva) e aplicar a análise do vizinho mais próximo. Esta análise não se limita a amostras de tumores, mas também pode ser aplicada a outros tecidos (não) patogênicos. As melhorias no equipamento e no fluxo de trabalho nos últimos anos reduziram significativamente os tempos de produção, o que facilita a aplicação futura desse procedimento no ambiente de diagnóstico.
Introduction
As células imunes desempenham um papel crucial na proteção contra patógenos, como vírus e bactérias, mas também contra células cancerígenas1. Portanto, o sistema imunológico dentro do microambiente tumoral (TME) é muito promissor para a descoberta de biomarcadores prognósticos e preditivos2. Os infiltrados de células imunes têm sido correlacionados ao prognóstico em vários tipos de câncer, embora isso ainda não tenha sido implementado no atendimento clínico 3,4. Na maioria dos tipos de tumores, um alto número de células T citotóxicas e células T auxiliares 1 e/ou baixo número de células T reguladoras estão ligados a bons prognósticos. Esforços estão em andamento para incorporar o chamado "Immunoscore" no estadiamento TNM do câncer colorretal, transformando-o no estadiamento TNM-I 5,6. O Immunoscore é derivado do número total de células T (detectadas com CD3) e células T citotóxicas (detectadas com CD8) em duas regiões tumorais diferentes: o núcleo do tumor versus a margem invasiva (IM) dos tumores. O Immunoscore também foi proposto como tendo valor prognóstico em outros tipos de câncer, como melanoma, câncer de pulmão e câncer de mama 6,7,8,9. Além disso, os infiltrados de células imunes também podem se correlacionar com a resposta à imunoterapia de bloqueio de checkpoint10. No entanto, esses biomarcadores preditivos devem ser validados em estudos prospectivos antes de poderem ser implementados rotineiramente na prática clínica. Além disso, também foi proposto que um único biomarcador será insuficiente para uma previsão significativa11. Portanto, a criação de um mapa completo de uma amostra de paciente pela combinação de diferentes biomarcadores foi proposta como um biomarcador preditivo mais abrangente no chamado "imunograma de câncer"12.
Dentre os métodos de estudo de células imunes dentro do TME, a técnica mais antiga e conhecida é a imuno-histoquímica (IHQ), utilizada rotineiramente para testes diagnósticos em diversas doenças, especialmente câncer13. Essa técnica foi limitada ao uso de um ou apenas alguns marcadores14 por um longo tempo e, portanto, foi superada em ambientes de pesquisa por outras técnicas, como citometria de fluxo e perfil de expressão gênica (GEP). No entanto, os tecidos tumorais fixados em formalina e embebidos em parafina (FFPE) normalmente usados em diagnósticos e pesquisas de rotina não são (idealmente) adequados para citometria de fluxo e GEP. Além disso, embora o GEP e a citometria de fluxo forneçam muitas informações sobre o fenótipo e a função celular, a falta de informações espaciais é uma grande desvantagem. Portanto, a heterogeneidade dentro de uma amostra, como diferenças nas áreas infiltradas por células imunes versus áreas excluídas por células imunes de um tumor, pode passar despercebida15. Novas plataformas foram desenvolvidas para análise multiplex de tecidos FFPE, como IHC multiplex, citometria de massa por imagem e CO-Detection por indEXing (CODEX) que podem ser usadas para detectar vários marcadores simultaneamente dentro de uma seção de tecido16. As células imunes no TME estão sendo amplamente estudadas para encontrar os melhores biomarcadores para imunoterapia. No entanto, as técnicas multiplex e a análise automatizada de imagens representam obstáculos próprios.
Nosso laboratório tem uma vasta experiência em coloração IHC multiplex usando o método de amplificação de sinal Opal/Tyramide (TSA) e automatizou isso em uma plataforma IHC (consulte a Tabela de Materiais)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. Otimizamos os painéis de células imunes para a detecção de diferentes subconjuntos de linfócitos, células mieloides e células dendríticas (DCs). Os tecidos que contêm áreas densas de células imunes - para linfócitos ou morfologias celulares complexas (ou seja, células mieloides e DCs) - são particularmente difíceis de analisar, com risco de superestimar ou subestimar o número de células imunes presentes. Para superar esse problema, o software de análise ImmuNet foi desenvolvido pelo nosso grupo32, e esse pipeline de aprendizado de máquina melhorou imensamente a qualidade da detecção desses diferentes tipos de células imunológicas. Um protocolo detalhado desde a obtenção do material FFPE até a análise das densidades de células imunes em diferentes compartimentos teciduais e distâncias entre os tipos de células imunes é descrito aqui.
Este protocolo descreve como os painéis multiplex IHC são realizados no Radboud University Medical Center desde a implementação do gerador de imagens de patologia digital em 2022. Os painéis multiplex IHQ descritos podem ser usados para diferentes carcinomas (por exemplo, pulmão, próstata, colorretal, bexiga, mama) com o uso de um anticorpo pan-citoqueratina como marcador tumoral ou para melanoma com o uso de anticorpos associados a melanócitos como marcadores tumorais. Esses protocolos IHQ multiplex foram cuidadosamente otimizados em termos de concentração de anticorpos primários, combinações de fluoróforos e a sequência do procedimento de coloração. Nós e outros descrevemos a otimização do painel IHC multiplex anteriormente 17,33,34,35. Os painéis multiplex IHC podem ser adaptados, mas os pipelines de análise descritos precisam ser avaliados e potencialmente ajustados ou retreinados de acordo. Os protocolos IHC multiplex de sete cores descritos fazem uso dos fluoróforos Opal Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 e 4',6-diamidino-2-fenilindol (DAPI), de modo que a fácil separação e a varredura rápida no gerador de imagens são habilitadas com "Imunofluorescência Multiespectral de Um Toque" (MOTiF). A coloração e a varredura de nove cores não são descritas neste protocolo, pois isso requer ainda mais ajustes da configuração experimental e outro modo de varredura no gerador de imagens que usa o filtro sintonizável de cristal líquido.
Protocol
O material do paciente mostrado para este protocolo fazia parte de um estudo conduzido anteriormente e foi oficialmente considerado isento de aprovação ética médica pelo Comitê de Ética Médica local de Radboudumc concomitante com a legislação holandesa (número de arquivo 2017-3164)30.
1. Coleta de material FFPE, seleção de blocos e preparação de amostras
- Recupere identificadores de bloco FFPE de arquivos de pacientes por meio de médicos assistentes ou patologistas. Verifique com os regulamentos locais se a permissão ética é necessária.
- Solicite blocos FFPE do arquivo de patologia local ou hospital(es) externo(s).
NOTA: Também é possível que o material tumoral ou uma biópsia seja adquirido para um estudo específico. Este pode ser o caso de pequenos ensaios clínicos ou estudos em animais. Nesses casos, o processamento da amostra de tecido pode ser de responsabilidade do pesquisador. - Quando vários bloqueios FFPE estiverem disponíveis, selecione o bloqueio FFPE mais representativo contendo tecido tumoral viável, preferencialmente com tecido estromal circundante presente, avaliando as lâminas coradas com hematoxilina e eosina (HE) (Figura 1).
NOTA: É aconselhável obter uma opinião especializada para esta seleção (por exemplo, um patologista). É possível que os HEs não estejam disponíveis para avaliação do conteúdo de um bloco FFPE e novos precisem ser feitos para a seleção. Vá para a seção 2 para obter uma descrição. - Corte fitas FFPE de 4 μm de espessura em um micrótomo.
NOTA: A espessura pode estar entre 1 μm e 6 μm sem impacto perceptível de manchas; no entanto, 4 μm é o mais padrão. - Montar as amostras em lâminas de vidro numa posição favorável à fluídica do corador automático (figura 2A-C), utilizando um dos métodos a seguir descritos:
- Coloque as secções sobre a superfície da água destilada a 40 °C num banho-maria para esticar e retire-as com uma lâmina de vidro.
OU
Coloque as lâminas de vidro em uma placa de aquecimento a 40 °C, certificando-se de cobrir o local onde a seção deve ser montada na lâmina com uma gota de água destilada. Coloque a seção em cima desta gota com uma pinça e deixe-a esticar. Absorva a água destilada usando uma toalha de papel e remova o excesso de água batendo na lâmina.
NOTA: Colocar seções de tecido muito próximas ao rótulo da lâmina resultará em coloração abaixo do ideal (Figura 2D, E). Tendemos a montar de 6 a 10 lâminas de vidro por amostra para realizar os diferentes painéis IHC multiplex e ter um backup.
- Coloque as secções sobre a superfície da água destilada a 40 °C num banho-maria para esticar e retire-as com uma lâmina de vidro.
- Deixe as lâminas de vidro montadas secarem a 56 °C por 1 h ou durante a noite a 37 °C.
- Utilizar as lâminas de vidro montadas para a experiência ou guardá-las em caixas a 4 °C.
NOTA: Em nossa experiência até agora, essas lâminas montadas podem ser armazenadas por anos antes que a coloração IHC multiplex seja realizada.
2. Gerando lâminas coradas com hematoxilina e eosina
NOTA: Todas as etapas seguintes da seção 2 devem ser executadas em uma capela de exaustão.
- Desparafinizar lâminas em xileno (2 x 5 min).
- Reidratar em etanol (99,6% 1 x 5 min; 95% 1 x 5 min; 70% 1 x 2 min). Como alternativa, mergulhe as lâminas 3x em etanol a 99,6%.
- Lave as lâminas em água destilada (2 min).
- Corar os núcleos com hematoxilina (10 min).
- Lave as lâminas com H2O destilado (5 min).
- Manchar as lâminas com eosina (5 min).
- Desidrate as lâminas mergulhando 3x em etanol a 99,6%.
- Mergulhe as lâminas 2x em xileno.
- Adicione algumas gotas de meio de montagem e sele com uma lamínula.
- Deixe as lâminas endurecerem e retire-as da hotte quando todos os produtos químicos tiverem evaporado.
3. Realização de IHQ monoplex e multiplex no autostainer
- Calcule a quantidade de reagente necessária dependendo do número de amostras a serem coradas.
NOTA: Por execução, o corador automático tem capacidade para 30 lâminas e leva ~ 18 h para completar o protocolo IHC multiplex com seis anticorpos. Quando mais lâminas precisam ser coradas, vários lotes podem ser colocados todas as noites da semana (de trabalho); 4 noites de 30 slides = 120 slides por semana.- Prepare todos os reagentes necessários no início da semana. O sistema de coloração automática dispensa 150 μL de reagente por lâmina. Use os recipientes de titulação de 6 mL para reagentes de anticorpos e opala e os recipientes de 30 mL para o reagente de bloqueio e a peroxidase secundária de anticorpo-rábano.
NOTA: Os recipientes de 6 mL têm inserções convenientes que podem ser facilmente retiradas e substituídas quando necessário. Com cálculos de reagentes, deve-se considerar o volume morto de 1,6 mL ou 300 μL para o recipiente de 30 mL ou recipiente de titulação de 6 mL, respectivamente. - Diluir todos os fluoróforos de opala e digoxigenina (DIG) 1:100 no diluente fornecido; diluir Opal780 1:25 no diluente de anticorpos. Dilua todos os anticorpos primários em diluente de anticorpos, com diluições especificadas no Arquivo Suplementar 1.
- Prepare todos os reagentes necessários no início da semana. O sistema de coloração automática dispensa 150 μL de reagente por lâmina. Use os recipientes de titulação de 6 mL para reagentes de anticorpos e opala e os recipientes de 30 mL para o reagente de bloqueio e a peroxidase secundária de anticorpo-rábano.
- Para seguir este protocolo, execute IHC monoplex (Arquivo Suplementar 2) em lâminas contendo tecido de controle de amígdala e outros tipos de tecido (tumor) de interesse antes de iniciar o experimento IHC multiplex real para garantir que todos os reagentes estejam bem preparados.
NOTA: O Monoplex IHC leva ~ 3.5 h e pode ser verificado antes do final do dia quanto aos padrões e intensidade do sinal. Se certos sinais forem muito fracos (Figura 3), ajustes nos reagentes podem ser feitos. - Para correção de autofluorescência, prepare uma lâmina com tecido (tumoral) contendo estruturas autofluorescentes, como sangue e colágeno. Prepare esta lâmina simultaneamente com lâminas IHC monoplex, mas com reagente de bloqueio substituindo o anticorpo e os reagentes Opal (Arquivo Suplementar 3).
NOTA: Em princípio, essa lâmina pode ser reutilizada para imagens multiespectrais até que a correção de autofluorescência não seja mais ideal. No entanto, com tecidos altamente autofluorescentes, como o cérebro e o fígado, é aconselhável usar esse tecido para a correção da autofluorescência. - Com cada execução de IHC multiplex, carregue 29 amostras no sistema de coloração automática com uma lâmina de tecido de controle para verificar o desempenho de cada execução de IHC multiplex.
- Baixe os protocolos IHC multiplex do website do autostainer na guia Downloads e ajuste-os para caber em cada painel IHC multiplex personalizado36. Para IHC multiplex, consulte o Arquivo Suplementar 4 para o protocolo e para painéis IHC multiplex personalizados, consulte o Arquivo Suplementar 1.
- Após a conclusão do protocolo de coloração, retire as lâminas do corador automático e coloque-as em um recipiente com tampão de lavagem.
- Para evitar a contaminação do sistema de coloração automática com DAPI, pois as amostras já estão coradas em concentrações muito baixas, aplique o DAPI manualmente antes de cobrir as lâminas com lamínulas. Adicione duas gotas de DAPI por mL de tampão de lavagem e incube por 5 min em temperatura ambiente no escuro.
NOTA: Para construir bibliotecas espectrais, é importante não ter nenhum DAPI corado nas amostras. Uma gota de DAPI por mL de tampão de lavagem e 10 min de incubação em RT também é possível. - Lave as lâminas 3x com tampão de lavagem.
- Coloque as lâminas em toalhas de papel e retire o excesso de tampão de lavagem das lâminas.
- Pipet algumas gotas de meio de montagem no tecido.
- Coloque uma lamínula de vidro suavemente em cima do meio de montagem para cobrir o slide em um ângulo para evitar bolhas de ar.
- Remova o excesso de meio de montagem e bolhas de ar empurrando suavemente a lamínula de vidro com uma pinça ou uma ponta de pipeta limpa.
- Deixe as lâminas intactas por ~24 h antes que o meio de montagem solidifique, horizontalmente em uma placa de lâminas de microscopia ou carregue-as diretamente no microscópio para geração de imagens.
- Depois que o meio de montagem estiver solidificado ou após a imagem das lâminas, armazene as lâminas em caixas de microscopia a 4 °C.
4. Imagiologia utilizando o gerador de imagens de patologia digital e anotação de ficheiros de digitalização
- Ligue o gerador de imagens pressionando o botão liga/desliga à direita da máquina. Após pelo menos 20 s, inicie o software.
NOTA: Aguarde 20 s para permitir que o hardware seja inicializado corretamente. - Coloque os slides nos por quatro slides.
- Opcional: Insira os slides em um arquivo .csv para o qual um modelo pode ser baixado (Arquivo Suplementar 5). Para carregar o arquivo .csv no programa, salve-o em C:\Users\Public\Akoya\VectraPolaris\States.
NOTA: Um máximo de 20 ou 80 slides podem ser carregados simultaneamente.
- Opcional: Insira os slides em um arquivo .csv para o qual um modelo pode ser baixado (Arquivo Suplementar 5). Para carregar o arquivo .csv no programa, salve-o em C:\Users\Public\Akoya\VectraPolaris\States.
- Configurações de referência
- Abra o Check Dashboard no menu principal.
NOTA: Um com lâminas de referência é fornecido pelo fabricante e pode ser mantido permanentemente no slot 20. - Defina as referências de campo claro no slide fornecido uma vez por semana de acordo com as instruções do fabricante (leva alguns minutos).
- Defina as referências de fluorescência na lâmina fornecida uma vez por mês de acordo com as instruções do fabricante (leva mais de 1 h).
- Abra o Check Dashboard no menu principal.
- Fazendo ou ajustando o protocolo
- Volte ao menu principal e clique em Editar protocolo para criar um protocolo.
- Clique em Novo... e selecione Fluorescência como Modo de imagem, Varredura de lâmina multiespectral e Opal Polaris 5, 6 e 7 cores na opção Coloração .
- Dê um nome ao protocolo em Nome do protocolo e salve-o em um estudo selecionando um estudo em Estudos disponíveis ou crie um estudo em Criar novo estudo | Nome do estudo.
- Conclua selecionando Criar protocolo.
- Para este tipo de digitalização, use apenas a janela esquerda Configurações de digitalização de slides multiespectrais; ignore a janela à direita Configurações de campo multiespectral.
- Digitalize os slides em diferentes ampliações. Para seguir este protocolo, digitalize com ampliação de 20x, deixando a resolução de pixel em 0,50 μm (20x).
- Defina os tempos de exposição selecionando Scan Exposures.
- Carregue o no qual os slides são mantidos selecionando o slot correto na opção Carregar transportador .
- Para ajudar a navegar pelos slides, selecione Obter visão geral para adquirir uma imagem de visão geral da portadora que contém os slides depois que a portadora for carregada. Para ativar ou desativar isso automaticamente, clique no ícone de engrenagem no canto superior direito, vá para Preferências... e marque a opção ativar ou desativar em Imagem de visão geral de navegação para ativar a portadora de imagem automaticamente ao carregar para tarefas interativas.
- Defina os tempos de exposição por filtro nas lâminas coradas IHC monoplex correspondentes selecionando Definir exposições de varredura e encontrando pontos diferentes com um sinal positivo. Foque manualmente ou use o Foco automático e selecione Autoexpor depois de alternar para o filtro compatível para esse sinal. Selecione o tempo de exposição mais baixo para evitar a superexposição e tire fotos de cada slide para referência depois que todos os tempos de exposição forem definidos (Figura 3).
NOTA: Ignore a opção Definir exposições de campo para este tipo de digitalização. - Defina os tempos de exposição em uma lâmina manchada multiplex verificando todos os filtros em alguns locais com sinal positivo. Reduza o tempo de exposição automática mais baixo em 10% para evitar a superexposição e tire algumas fotos depois que todos os tempos de exposição forem definidos.
- Tire instantâneos da lâmina não corada para compensação de autofluorescência usando o filtro AF de amostra para navegar (Figura 3H).
NOTA: Locais com eritrócitos e estruturas de colágeno são de interesse. O tempo de exposição do filtro Opal480 pode precisar ser reduzido para regiões autofluorescentes fortes. Se o sinal Opal480 for forte o suficiente, ele ainda deve ser bem separado (consulte a seção 6) das estruturas autofluorescentes devido à implementação do filtro AF de amostra proprietário. - Avalie a qualidade da coloração e da imagem usando o software (consulte as seções 5 e 6; Figura 4, Arquivo Suplementar 6: Figura Suplementar S1 e Figura Suplementar S2).
- Selecione o botão Salvar... para certificar-se de que o protocolo e seus tempos de exposição ajustados sejam salvos no protocolo.
NOTA: Quando o protocolo já está salvo, nenhuma notificação extra de ajustes não salvos é fornecida pelo software até o momento.
- Digitalização automática de slides
- Volte ao menu principal e clique em Digitalizar slides para digitalizar os slides.
- Insira manualmente os nomes/IDs dos slides e as tarefas e protocolos correspondentes em Configurar tarefas ou automaticamente a partir do arquivo .csv criado anteriormente com Configuração de carga.
- Clique em Digitalizar para iniciar a digitalização.
- Aguarde até que uma janela apareça para salvar a configuração da verificação. Clique em Salvar para usar as configurações padrão e iniciar a digitalização.
NOTA: A digitalização usando este método leva ~10-20 minutos por slide. Dependendo do número de slides, a digitalização pode levar até um dia inteiro. - Verifique se a verificação dos slides foi bem-sucedida para todos os slides procurando por mensagens de erro. Para saber se a digitalização foi bem-sucedida, procure um arquivo de digitalização de lâminas inteiras Akoya salvo (.qptiff) da digitalização e o tecido completo na digitalização.
5. Anotação de dados usando o visualizador de slides
- Volte para o menu principal e clique em Iniciar fenográfico para abrir o visualizador de slides.
- Se os arquivos de digitalização não estiverem diretamente visíveis, indique sua localização clicando primeiro no ícone de engrenagem no canto superior direito, vá para Alterar localização do navegador... e selecione aleatoriamente um dos arquivos .qptiff do conjunto de dados de interesse.
NOTA: Os dados são armazenados por padrão em D:\Data\VectraPolaris. - Carregue um slide selecionando-o e clicando em Carregar no canto superior direito ou clicando duas vezes nele.
- Faça login clicando no botão Login no canto superior direito.
NOTA: O nome de usuário pode ser apenas as iniciais ou o nome e é usado para acompanhar quem fez quais anotações. - Para realizar a desmixagem, clique no botão Unmixing na parte superior e selecione a opção Opal + AF .
NOTA: Isso é útil para se livrar de alguns dos sinais autofluorescentes perto do canal Opal 480, mas não todos. - Para gerar um algoritmo para processamento em lote dos dados, selecione imagens representativas usando o Carimbo usando a opção para imagens 1 x 1 do inForm Projects (tamanho da imagem: 928 μm x 696 μm).
NOTA: Alguns selos representativos contendo tumor, estroma, fundo e diferentes tipos de células imunes são selecionados em todo o conjunto de dados para terminar com ~ 20-30 imagens. - Dependendo do que precisa ser analisado no tecido, selecione uma região de interesse usando a opção ROI e selecione para inForm Batch. Exclua manualmente as imagens que não precisam ser analisadas, como imagens muito distantes do tumor ou em segundo plano.
NOTA: Tendemos a desenhar um ROI ao redor de todo o tumor e selecionar uma imagem extra longe da região do tumor para poder analisar um IM de ~ 0,5 mm.
Se o ROI desenhado for relativamente pequeno, o ROI consistirá em 2 a 9 imagens 20x mescladas. Como isso não é preferido por nós, carimbe manualmente o tecido de interesse (selecionado para o inForm Batch) para contornar isso. - Quando terminar de anotar, deixe as anotações serem salvas automaticamente e carregue o próximo slide.
- Durante o processo de anotação, verifique se os slides estão digitalizados corretamente.
- Se um arquivo .qptiff estiver faltando ou uma lâmina não for digitalizada com sucesso, verifique se há algum tecido presente na lâmina, limpe a lâmina com etanol a 70% e digitalize novamente.
- Se o tecido não estiver totalmente escaneado, faltando uma região potencialmente importante (tumor), ou se o escaneamento da região importante estiver fora de foco, limpe a lâmina com etanol a 70% e escaneie novamente.
NOTA: Em ambos os casos, também pode ajudar circundar o tecido com um marcador na parte superior da lamínula para ajudar o sistema a localizar o tecido e tentar digitalizar novamente (Arquivo Suplementar 6: Figura Suplementar S3). Em nossas mãos, um marcador vermelho fino funcionou melhor do que um marcador preto grosso.
- Depois que a digitalização e a anotação de todas as amostras forem concluídas, faça backup dos dados armazenando-os em um computador ou disco externo diferente.
6. Mistura espectral
- Abra o software de análise de imagem automatizada inForm.
- Carregue as imagens no software por File | Abrir imagem; Selecione arquivos .qptiff. Deixe os carimbos, marcados como Projetos do inForm na etapa 5.6, serem carregados no projeto.
- Carregue os arquivos .qptiff que são criados para a compensação de autofluorescência.
- Para compensar a autofluorescência, use a ferramenta selecionar autofluorescência na imagem para desenhar uma linha na imagem da lâmina não corada através de diferentes tipos de estruturas autofluorescentes, como eritrócitos e colágeno.
- Na seção Editar marcadores e cores... , atribua nomes de marcadores que correspondam ao fluoróforo Opala e ajuste a cor para o preferido.
- Para desfazer a mistura dos fluoróforos, selecione Preparar tudo no canto inferior esquerdo.
- Percorra as imagens e verifique se todos os sinais estão visíveis nas imagens e se a mistura correu bem. Selecione o ícone do globo ocular para desligar e ligar todos os marcadores, um por um, para verificar a qualidade.
- Opcionalmente, treine os algoritmos para segmentação de tecidos, segmentação celular e fenotipagem.
- Vá para a guia Exportar e crie um novo diretório de exportação vazio clicando no botão Procurar... no Diretório de Exportação.
- Em Imagens a serem exportadas:, selecione Imagem composta e Imagens de componente (TIFF de várias imagens).
- Selecione Arquivo | Salvar | Projeto para salvar o algoritmo em um determinado local.
- Vá para a guia Análise em lote verticalmente à esquerda para o processamento em lote de slides.
- Selecione Criar diretórios separados para cada item em Opções de exportação.
- Para adicionar slides para análise, selecione arquivos .qptiff no botão Adicionar slides... e carregue-os na análise em lote.
- Selecione Executar para iniciar o processamento em lote de slides.
7. Desenho do ROI
- Crie uma pasta apenas com os arquivos de componentes da seção 6, mas mantenha a estrutura hierárquica de pastas intacta (os arquivos de componentes estão em pastas nomeadas por amostra/slide).
- Abra o software visualizador de diapositivos QuPath.
- Clique em Criar projeto à esquerda e selecione/crie uma nova pasta vazia com um nome adequado.
- Clique em Automatizar e selecione Mostrar editor de script.
- Copie e cole o script que está disponível no Arquivo Suplementar 7. Na linha 34, altere o local para onde estão as pastas de slides que contêm todos os arquivos de componentes (a pasta criada na etapa 7.1.
- Selecione Executar e retornar quando a costura em lote de slides for concluída (no dia seguinte ou mais tarde) para continuar.
- Arraste os arquivos de .ome.tif gerados para o projeto QuPath e salve-os como um projeto.
- Quando uma nova janela aparecer automaticamente, selecione Definir tipo de imagem | Fluorescência e clique em Importar.
- No menu à esquerda, observe a lista de amostras; clique duas vezes em um para abrir a amostra (Figura 5A).
- Para ajustar a intensidade dos canais para torná-los mais visíveis, clique no ícone de contraste.
- Selecione todos os canais e clique em Redefinir.
- Desative a autofluorescência.
- Para começar a desenhar um ROI para o tumor, clique no ícone de contraste e selecione Mostrar escala de cinza. Selecione o canal do marcador tumoral e ajuste a intensidade para torná-lo visível de maneira ideal (Figura 5B).
- Clique na ferramenta pincel para desenhar um ROI do tumor aproximadamente.
- Ao selecionar a ferramenta de varinha, clique fora do ROI enquanto pressiona a tecla alt para suavizar o ROI do lado de fora (Figura 5C).
- Mescle pedaços de tumor separados com o mesmo ROI.
- Dê ao ROI um nome adequado, como tumor , clicando com o botão direito do mouse na anotação na lista à esquerda; selecione Definir propriedades e insira o nome.
- Para fazer um ROI para o IM, expanda o ROI existente da região do tumor selecionando: Objetos | Anotações... | Expanda as anotações.
- Selecione o tamanho do raio de expansão e selecione Remover interior e Restringir ao pai (Figura 5D).
- Clique no ícone de contraste, selecione o canal de autofluorescência e ajuste a intensidade para torná-lo visível de maneira ideal.
- Clique na varinha e ajuste o ROI enquanto pressiona a tecla alt para suavizar o ROI do lado de fora e remover qualquer plano de fundo que não deva fazer parte desse ROI.
- Dê ao ROI um nome adequado, como margem invasiva ou IM , clicando com o botão direito do mouse na anotação na lista à esquerda, selecione Definir propriedades, insira o nome e, opcionalmente, altere sua cor para verde.
- Salve as anotações: Arquivo | Exportar objetos | Exporte todos os objetos e clique em OK com a seleção padrão em Exportar como FeatureCollection e salve-o em um local preferencial.
8. Detecção de células imunes
- Como o ImmuNet usa dados de componentes (arquivos TIFF multicanal) para treinamento e inferência, divida as anotações em conjuntos de treinamento e validação. Para treinar o modelo, siga as etapas descritas no arquivo Leiame do repositório, substituindo o conjunto de dados e as anotações de exemplo pelos dados desejados. Além de diferentes células imunes, forneça ao modelo exemplos negativos fazendo anotações de fundo em locais que não devem ser reconhecidos como uma célula de interesse: células tumorais, outras células ou "sem células" (estruturas que podem ser confundidas com células de interesse); consulte a publicação ImmuNet para obter detalhes32.
- Usando as anotações de validação, certifique-se de que o desempenho seja satisfatório. Observe a taxa de erro por tipo de anotação - uma parcela de anotações de validação que o modelo não detectou - a métrica de avaliação mais direta. Avalie o desempenho em relação a falsos positivos fazendo alguns ROIs totalmente anotados e calculando as pontuações F.
- Além da avaliação quantitativa, inspecione visualmente a previsão para obter uma noção qualitativa dos erros que o modelo tende a cometer (Figura 6, Arquivo Suplementar 6: Figura Suplementar S4 e Figura Suplementar S5). Se o desempenho do modelo for considerado insuficiente, visualize a previsão de alguns blocos, conforme descrito no repositório, e verifique quais sites são mais propensos a erros. Faça mais anotações nesses sites e execute novamente o treinamento e a avaliação do modelo.
- Quando o desempenho desejado for alcançado, execute a inferência para todo o conjunto de dados, conforme descrito na seção Inferência para todo o conjunto de dados do Leiame do repositório. Use os arquivos .csv obtidos com a previsão do modelo como entrada para análise de dados (escreva um script Python ou R para isso).
9. Fenotipagem de predição e análise de dados
NOTA: Nesta seção, damos um exemplo de análise de dados simples para uma única amostra de melanoma corada com o painel de linfócitos, que combina as localizações das células imunes identificadas pelo ImmuNet (seção 8) e ROIs delineadas com o QuPath (seção 7). A análise foi realizada no R 4.1.1 (um script é fornecido como Arquivo Suplementar 8). O script requer os pacotes: plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 e RColorBrewer 1.1-2, que podem ser instalados com o comando install.packages(). Como entrada, é necessário um ficheiro .csv com a previsão da ImmuNet de uma amostra e um ficheiro com ROIs exportados do QuPath. As etapas 9.1 a 9.6 descrevem a análise de uma única amostra realizada no script fornecido e as seções 9.7 a 9.9 descrevem as opções para a análise de várias amostras.
- Depois de carregar a previsão do ImmuNet em R, determine os limites para a expressão do marcador previsto, plotando os marcadores que definem os fenótipos uns contra os outros e selecionando os limites que separam melhor as populações.
NOTA: A estratégia de gating usada para a amostra fornecida é mostrada na Figura 7B. As estratégias de bloqueio para os painéis de células mieloides e dendríticas são mostradas no Arquivo Suplementar 6: Figura Suplementar S6 e Figura Suplementar S7. - Depois de determinar os limites, use-os para atribuir a cada previsão do ImmuNet um fenótipo definido em um painel. Em algumas previsões, observe que nenhum dos marcadores previstos está acima do limiar ou a combinação de marcadores considerados expressos após o limiar pode ser inconsistente (por exemplo, predições de CD3+ CD20+ nos painéis de linfócitos). Se um bom desempenho do modelo for alcançado na etapa 8.3, a fração de tais previsões será pequena; filtre-os antes da análise.
- Para analisar separadamente as ROIs do tumor e sua margem invasiva de até 100 μm desenhadas no QuPath, carregue os arquivos GeoJSON correspondentes em R e, para cada previsão, determine o ROI no qual a previsão se enquadra.
- Para uma verificação de sanidade e como parte da análise exploratória de dados, visualize as células imunes encontradas em uma amostra separadamente nas ROIs correspondentes, juntamente com os limites das ROIs (Figura 7A).
- Agora, calcule as densidades de diferentes células imunes separadamente para cada ROI. As densidades encontradas na amostra são mostradas na Tabela 1.
- Se várias amostras estiverem disponíveis, visualize a distribuição das densidades celulares. Transforme os valores de densidade para obter valores normalmente distribuídos.
NOTA: Quando as contagens de determinados fenótipos são 0, elas não podem ser transformadas em Log, levando a valores ausentes. Para superar esse problema, a suavização LaPlaciana pode ser aplicada adicionando 0,5 a todas as contagens de células antes de dividir pela área da superfície. - Analise os valores de densidade e plote-os usando o software de sua escolha (Figura 8).
- Localizações preservadas das células permitem a análise espacial. Por exemplo, para cada célula imune detectada, encontre um vizinho mais próximo e, em seguida, para cada fenótipo, calcule a porcentagem de casos em que os diferentes fenótipos ocorrem como o vizinho mais próximo.
NOTA: Como o número de células natural killer (NK) encontradas nesta amostra foi muito pequeno, nós as excluímos desta análise. Os resultados obtidos para ROIs de tumor e IM são apresentados na Figura 9.
Representative Results
Os blocos de FFPE contendo tecido tumoral foram selecionados com base em relatórios de patologia e lâminas coradas com HE. Quando múltiplas lesões tumorais são ressecadas do paciente e/ou as amostras tumorais são grandes, elas são divididas em vários blocos FFPE. Preferimos analisar as células imunes tanto no compartimento do tumor quanto no que é conhecido como margem invasiva (IM) do tumor. O IM é um tecido estromal não canceroso adjacente ao tumor. Portanto, quando há vários blocos FFPE disponíveis para uma amostra de tumor, os blocos FFPE que contêm os dois tipos de tecido são selecionados. Como visto nas lâminas coradas com HE, um bloco FFPE continha tecido tumoral e tecido estromal adjacente ao tumor (Figura 1A). Outro bloqueio FFPE do mesmo tumor continha muito menos tecido estromal circundante (Figura 1B). No entanto, para algumas amostras de tecido, não há escolha em blocos FFPE ou o IM não está presente em nenhum dos blocos FFPE. Este é frequentemente o caso das biópsias (com agulha), que devem ser mantidas em mente durante a interpretação dos dados.
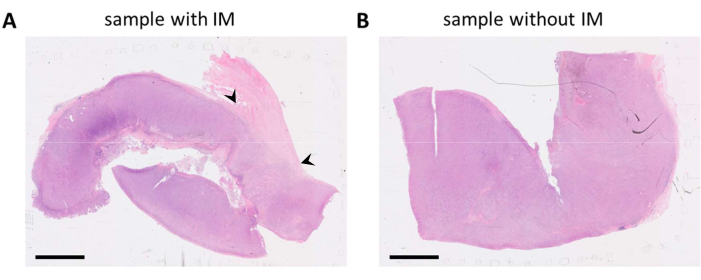
Figura 1: Lâminas coradas com HE de uma amostra de tumor de melanoma. (A) Um exemplo de uma amostra de tumor com tecido estromal adjacente ao tumor (IM) no canto superior direito da amostra (indicado com pontas de setas pretas). (B) Outra amostra da mesma lesão tumoral com pouco ou nenhum tecido estromal presente na amostra. Barras de escala = 5 mm. Abreviaturas: HE = hematoxilina e eosina; Como primário = margem invasiva. Clique aqui para ver uma versão maior desta figura.
A coloração IHQ multiplex com um painel proposto de sete cores (Arquivo Suplementar 4) pode ser realizada manualmente em um processo de coloração de 3 dias (considerando o horário normal de trabalho) ou durante a noite em um corador automático. Ao usar o corador automático, as seções devem ser montadas em um local específico na lâmina de vidro que permita a fluidez ideal do sistema (Figura 2A). Quando as seções são montadas corretamente em lâminas (Figura 2B), elas serão uniformemente coradas (Figura 2C). Se as seções não forem montadas de maneira ideal na lâmina de vidro (Figura 2D), isso geralmente resulta em um padrão de coloração abaixo do ideal (Figura 2E) porque os fluidos do corador automático não atingem o tecido (completo). Isso pode acontecer quando as amostras são muito grandes ou quando os slides montados são fornecidos por alguém que não está ciente desse problema. Nestes casos, apenas a parte bem manchada da lâmina deve ser selecionada para análise. Outra opção para esses tipos de amostras pode ser corá-las manualmente para espalhar os líquidos de maneira ideal.
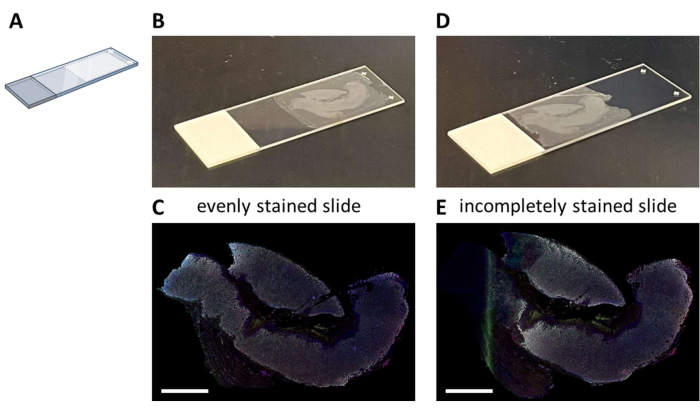
Figura 2: Montagem da seção FFPE na corrediça de vidro e impacto. (A) Esquema de onde montar na corrediça de vidro para uma coloração ideal no corador automático. (B) Exemplo de um slide montado corretamente. (C) Lâminas montadas corretamente resultam em uma seção de tecido uniformemente corada. (D) Exemplo de uma corrediça montada abaixo do ideal. (E) Lâminas montadas abaixo do ideal podem resultar em uma seção de tecido manchado incompleta, como visto no lado esquerdo desta imagem. Barras de escala = 5 mm. Abreviatura: FFPE = fixado em formalina e embebido em parafina. Clique aqui para ver uma versão maior desta figura.
Quando grandes experimentos de IHC multiplex são realizados em várias rodadas de coloração e grandes quantidades de soluções precisam ser preparadas, é melhor primeiro testar esses reagentes em uma corrida de IHC monoplex antes de prosseguir para a IHC multiplex. O IHC monoplex é verificado com o gerador de imagens de patologia digital para padrões de coloração esperados e os tempos de exposição são definidos com os filtros correspondentes nas lâminas de controle (Figura 3A-H). O tecido da amígdala é usado como controle positivo para a maioria dos marcadores de células imunológicas. Como o tempo de exposição ao DAPI no tecido controle das amígdalas é sempre maior do que em outros tecidos (Figura 3G), o tempo de exposição ao DAPI deve ser definido no tipo de tecido a ser estudado. Os tempos de exposição regulares com este tipo de varredura estão entre 1 ms e 30 ms, dependendo do fluoróforo e do filtro (Figura 3I). Quando uma IHQ monoplex excede esses números ou o padrão de coloração não é tão claro quanto o esperado, a solução de anticorpos deve ser ajustada ou substituída. No exemplo mostrado aqui, decidimos aumentar a concentração de FOXP3 (Figura 3C e Figura 3I) para ter a intensidade mais dentro da faixa com os outros marcadores. A autofluorescência também pode ser mais forte em outros tecidos do que no tecido de controle da amígdala. Em nossa configuração, o tempo de exposição para o filtro AF de amostra está entre 25 ms e 50 ms (Figura 3H,I).

Figura 3: Configuração dos tempos de exposição em IHQ monoplex e amostras de controle não coradas. (A) Sinal CD20 - Opal 480 no tecido controle da amígdala. (B) CD3 - Sinal Opal 520 no tecido controle das amígdalas. (C) FOXP3 - Sinal Opal 570 no tecido controle das amígdalas. (D) CD56 - sinal Opal 620 no tecido controle das amígdalas (E) CD8 - sinal Opal 690 no tecido controle das amígdalas. (F) Marcador tumoral - sinal Opal 780 no tecido controle da amígdala. (G) O sinal DAPI no tecido de controle da amígdala é frequentemente mais fraco do que o tipo de tecido de interesse. (H) Autofluorescência - amostra de sinal AF no tecido de controle do tumor. (I) Captura de tela dos tempos de exposição antes de ajustá-la com 10% e verificar as lâminas multiplex manchadas de IHC. Barras de escala = 100 μm. Abreviaturas: AF = autofluorescência; IHQ = imuno-histoquímica; DAPI = 4'6-diamidino-2-fenilindol. Clique aqui para ver uma versão maior desta figura.
Depois que o IHC multiplex é executado, os tempos de exposição são ajustados a partir das configurações de IHC monoplex, verificando algumas lâminas IHC multiplex e selecionando a exposição automática. Com este tipo de digitalização, não há opção de proteção contra saturação e, portanto, é extremamente importante evitar definir a exposição muito alta, evitando assim a superexposição. A superexposição dificulta a separação espectral dos fluoróforos. Muitas vezes não definimos tempos de exposição que excedem os tempos de exposição baseados no IHC monoplex e apenas diminuímos os tempos de exposição para marcadores que são mais fortes no IHC multiplex (Figura 3G e Figura 4A). Ao auto-expor em diferentes locais em alguns slides, pode-se observar que os tempos de exposição de alguns filtros ainda são muito altos. Estes devem ser ajustados para o número mais baixo observado ao usar a configuração de exposição automática e subtrair outros 10% do valor para evitar a superexposição em outros locais invisíveis (Figura 4A). Com este método, os tempos de exposição podem ser menores para certos filtros do que os que foram definidos no IHC monoplex. No entanto, com um experimento IHC multiplex bem-sucedido, todos os marcadores devem ser observáveis, pelo menos na lâmina de controle (Figura 4B-H, Arquivo Suplementar 6: Figura Suplementar S1 e Figura Suplementar S2). Considere que certos marcadores podem não estar presentes em todas as amostras. Ao incluir uma lâmina de controle contendo pelo menos uma seção de amígdala, a coloração bem-sucedida de todos os marcadores dos painéis padrão e a intensidade do sinal podem ser verificadas.
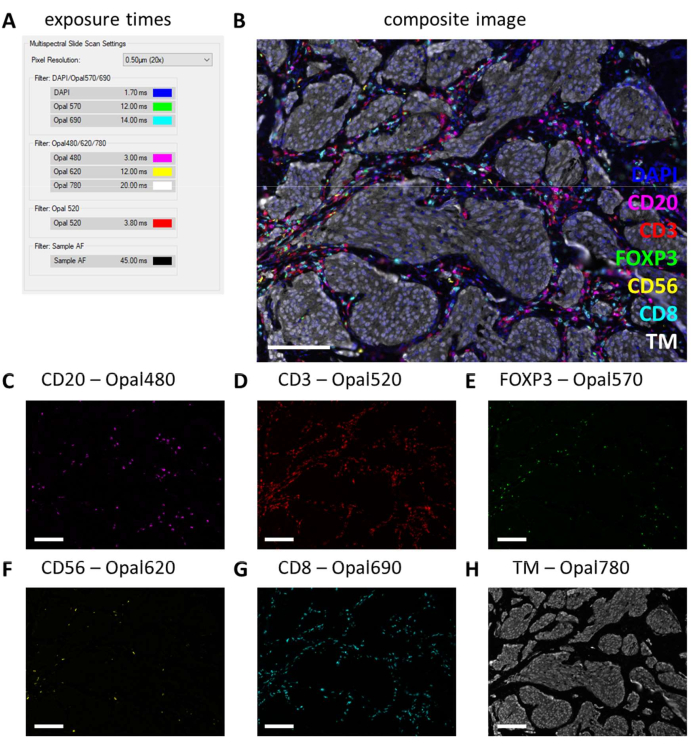
Figura 4: Exemplo de uma seção corada com sucesso com o painel de linfócitos em uma amostra de tumor de melanoma. (A) Tempos de exposição usados para registrar esta amostra multiplex de IHC. (B) Imagem composta do painel multiplex de linfócitos IHC dentro do tecido tumoral. (C) CD20 - Sinal Opal 480 em magenta. (D) CD3 - Sinal Opal 520 em vermelho. (E) FOXP3 - Sinal Opal 570 em verde. (F) CD56 - Sinal Opal 620 em amarelo. (G) CD8 - Sinal Opal 690 em ciano. (H) TM - Opal 780 em branco. Barras de escala = 100 μm. Abreviatura: TM = marcador tumoral; IHQ = imuno-histoquímica. Clique aqui para ver uma versão maior desta figura.
As lâminas IHC multiplex são totalmente digitalizadas pelo gerador de imagens digital. Os blocos para análise subsequente são selecionados no visualizador de slides. No entanto, quando regiões mais específicas precisam ser analisadas, como tumor versus IM, essas regiões de interesse (ROIs) podem ser desenhadas usando o QuPath. Após a conclusão do processamento em lote dos blocos selecionados no visualizador de slides, os arquivos de componente são mesclados novamente (Figura 5A e Arquivo Suplementar 7). Usando o canal marcador tumoral (Figura 5B) e a ferramenta varinha mágica no QuPath, o contorno do tumor pode ser rastreado para formar a "ROI do tumor" (Figura 5C). Em seguida, a ROI do tumor pode ser expandida com uma certa distância, neste caso, 500 μm, para criar uma "ROI de margem invasiva" (Figura 5D). Qualquer fundo indesejado (não tecido) é removido desse ROI com a ferramenta varinha mágica observando o sinal de autofluorescência (Figura 5E). Tanto o ROI do tumor quanto o ROI do IM são salvos como um arquivo GeoJSON para processamento posterior (Figura 5F).
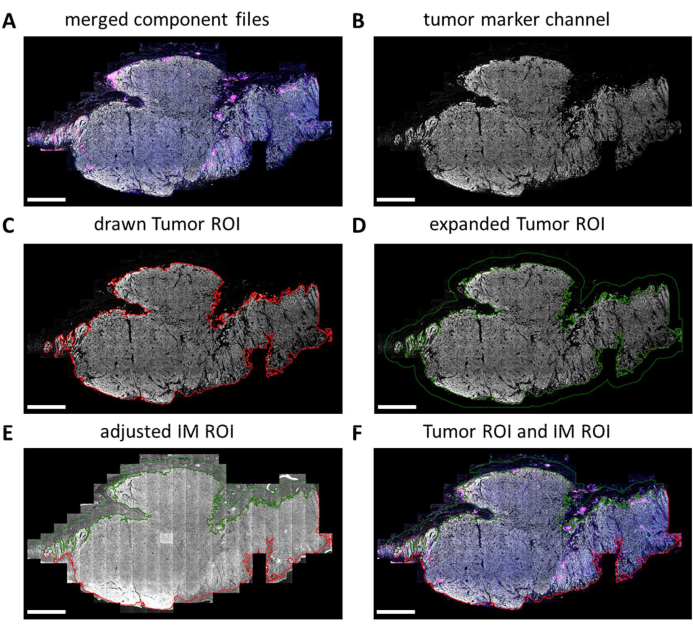
Figura 5: ROI do tumor e processo de desenho de ROI de margem invasiva no QuPath. (A) Arquivos de componentes mesclados. (B) Imagem em escala de cinza mostrando apenas o canal marcador tumoral. (C) A ROI do tumor é desenhada em torno do sinal do marcador tumoral. (D) Um novo ROI é feito expandindo o ROI do tumor em 100-500 μm para formar o ROI IM. (E) O ROI IM é ajustado para incluir apenas tecido estromal, excluindo o fundo (sinal negativo) e outras grandes estruturas de tecido, como gordura, vasos sanguíneos e folículos pilosos. (F) O ROI do tumor resultante e o ROI IM são salvos e exportados para arquivos GeoJSON para processamento posterior das regiões. O ROI do tumor é exibido com um contorno vermelho e o ROI do IM com um contorno verde. Barras de escala = 2 mm. Abreviaturas: ROI = região de interesse; IM = margem invasiva; GeoJSON = Notação geográfica de objeto JavaScript. Clique aqui para ver uma versão maior desta figura.
As redes ImmuNet podem ser usadas para detectar células imunológicas. Para o painel de linfócitos, a imagem composta experimental (Figura 6A) pode ser comparada visualmente com as células imunes detectadas pelo software (Figura 6B). Comparações visuais semelhantes podem ser feitas para o painel mieloide (Arquivo Suplementar 6: Figura Suplementar S4) e o painel de células dendríticas (Arquivo Suplementar 6: Figura Suplementar S5).
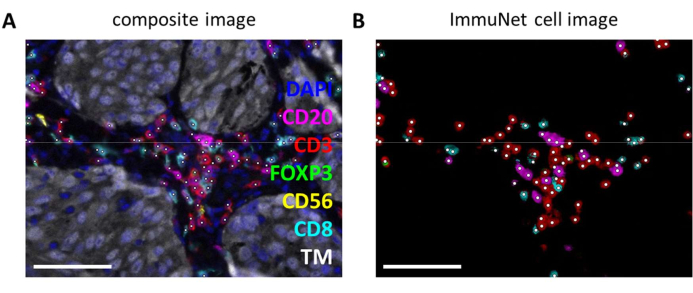
Figura 6: Linfócitos reconhecidos pelo ImmuNet. (A) Imagem composta da Figura 4B mostrando células reconhecidas pelo ImmuNet com pontos brancos. (B) Células reconhecidas pelo ImmuNet e subsequente expressão de marcadores detectados. Barras de escala = 50 μm. Abreviatura: TM = marcador tumoral. Clique aqui para ver uma versão maior desta figura.
As células imunes detectadas pelo ImmuNet e salvas em .csv formato podem ser importadas para qualquer linguagem de programação para análise posterior. Realizamos visualização espacial e gating em R (Arquivo Suplementar 8). As células detectadas podem então ser visualizadas espacialmente (Figura 7A, Arquivo Suplementar 6: Figura Suplementar S6 e Figura Suplementar S7). A expressão de pseudomarcadores pode ser realizada para fenotipar as células imunes individuais ( Figura 7B ).
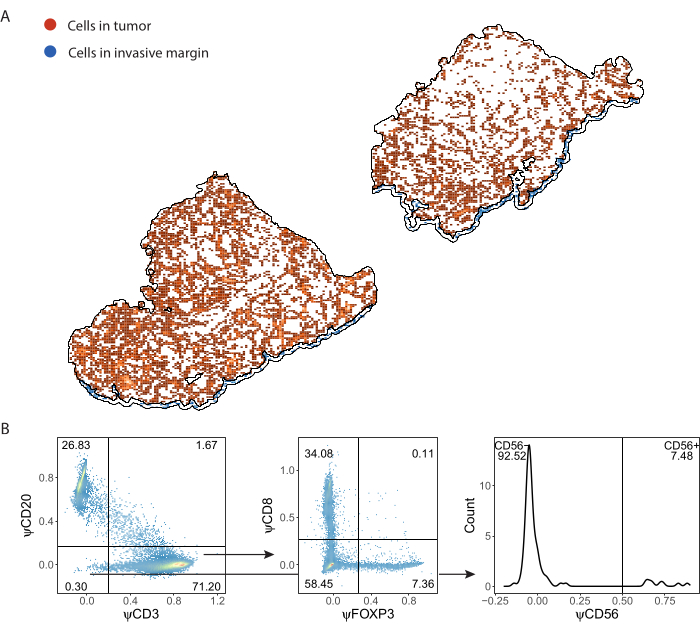
Figura 7: Estratégia de gating do painel de linfócitos. (A) Células imunes detectadas em regiões tumorais e de margem invasiva de interesse delineadas com QuPath. (B) Gating de todas as células detectadas pelo ImmuNet da parte A. Os linfócitos são primeiro bloqueados em células B CD20+ e células T CD3+. As células T CD3 + são ainda bloqueadas para a expressão de CD8 e FOXP3. A população CD20-CD3- é fechada para células assassinas naturais CD56+. Clique aqui para ver uma versão maior desta figura.
Quando os fenótipos das células previstas são determinados com gating, as densidades celulares de diferentes fenótipos podem ser calculadas dentro de diferentes ROIs. Isso é calculado dividindo o número total de células por fenótipo pela área de superfície do ROI (Tabela 1, Figura 8 e Arquivo Suplementar 8). Aqui, as células B são definidas como CD3-CD20+, as células T auxiliares como CD3+CD20-CD8-FoxP3-, as células T reguladoras como CD3+CD20-CD8-FoxP3+, as células T citotóxicas como CD3+CD20-CD8+FoxP3- e as células NK como CD3-CD20-CD56+.
| Fenótipo | Densidade no Tumor (células/mm2) | Densidade em IM (células/mm2) |
| Célula B | 185.74 | 145.62 |
| Célula T auxiliar | 301.46 | 157.51 |
| Célula T reguladora | 38.53 | 19.53 |
| Células T citotóxicas | 185.35 | 83.21 |
| Célula NK | 0.18 | 0 |
Tabela 1: Densidades de fenótipos em ROIs. Densidades de células de diferentes fenótipos encontradas em uma única amostra de melanoma corada com o painel de linfócitos. As densidades são calculadas separadamente em ROIs de tumor e IM. Abreviaturas: IM = margem invasiva; ROI = região de interesse.
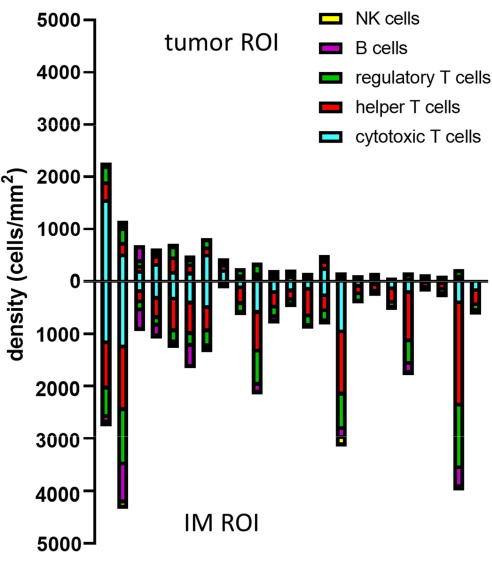
Figura 8: Exemplo de análise de dados para várias amostras. Análise de densidade de diferentes fenótipos de linfócitos no tumor e IM de 23 tumores primários de melanoma. Abreviaturas: IM = margem invasiva; ROI = região de interesse. Clique aqui para ver uma versão maior desta figura.
Para mergulhar mais nas informações espaciais dessas células imunes, também é possível determinar distâncias entre fenótipos identificados ou porcentagens de fenótipos de vizinhos mais próximos em uma amostra (Figura 9).
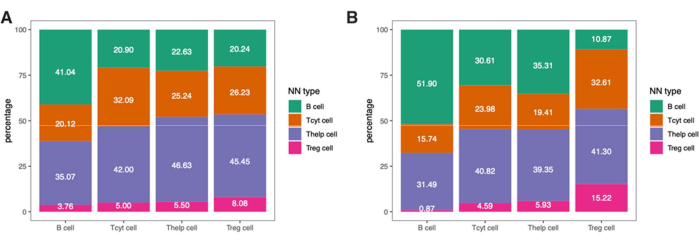
Figura 9: Exemplo de análise do vizinho mais próximo para uma única amostra. Porcentagem de fenótipos vizinhos mais próximos para diferentes tipos de células em (A) Tumor e (B) IM ROIs encontrados em uma única amostra de melanoma corada com o painel de linfócitos. Abreviaturas: IM = margem invasiva; ROI = região de interesse. Clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar 1: IHC multiplex resumindo as especificações do protocolo. Clique aqui para baixar este arquivo.
Arquivo suplementar 2: Protocolo de coloração automática para monoplex. Clique aqui para baixar este arquivo.
Arquivo Suplementar 3: Protocolo de autocoloração para compensação de autofluorescência. Clique aqui para baixar este arquivo.
Arquivo Suplementar 4: Protocolo de autocoloração para imuno-histoquímica multiplex. Clique aqui para baixar este arquivo.
Arquivo Suplementar 5: Modelo .csv arquivo. Clique aqui para baixar este arquivo.
Arquivo Suplementar 6: Painéis de células mieloides e dendríticas em uma amostra de tecido de melanoma; marcação de lâminas em caso de falha de varredura; células mieloides e dendríticas reconhecidas pelo ImmuNet; estratégias de gating de painéis de células mieloides e dendríticas. Clique aqui para baixar este arquivo.
Arquivo Suplementar 7: script de ponto do QuPath. Clique aqui para baixar este arquivo.
Arquivo Suplementar 8: Script de análise de dados. Clique aqui para baixar este arquivo.
Discussion
A análise espacial do TME é uma técnica procurada para aprender mais sobre o compartimento das células imunes e descobrir novos biomarcadores prognósticos e preditivos, particularmente no campo da imuno-oncologia16. Muitas técnicas diferentes estão sendo desenvolvidas para esse fim, envolvendo a detecção de proteínas, transcritos de mRNA ou uma combinação dos dois, com estimativas de até 100-1.000 alvos. No entanto, uma multiplexação mais alta tem o custo de experimentos de menor rendimento, custos experimentais mais altos e desafios técnicos e, muitas vezes, apenas uma pequena parte do TME pode ser analisada. A IHQ multiplex usando o método baseado em TSA que descrevemos aqui, detecta seis marcadores diferentes + DAPI simultaneamente, é relativamente mais barata de realizar e seções de tecido inteiro são visualizadas em menos de 20 minutos, prontas para serem analisadas completamente. Essa técnica tornou-se menos complexa com a automação do procedimento de coloração. Melhorias no microscópio multiespectral, que incluem a adição de dois filtros extras, melhoraram tremendamente os tempos de separação e varredura espectral. É possível detectar até oito marcadores diferentes + DAPI simultaneamente. No entanto, ao expandir a multiplexação com mais marcadores, os benefícios mencionados acima desaparecem à medida que a separação espectral se torna mais desafiadora e os tempos de varredura para lâminas inteiras aumentam substancialmente. Esforços estão sendo realizados para padronizar a IHQ multiplex entre diferentes instituições para facilitar a implementação no ambiente de diagnóstico com mais facilidade. Para esta padronização do IHC multiplex, aconselhamos os usuários a aderir ao protocolo mais acessível com seis marcadores diferentes + DAPI. No entanto, ainda é necessário algum conhecimento técnico e a análise a jusante pode ser um desafio, para o qual desenvolvemos metodologias descritas neste protocolo.
A padronização começa com o desenvolvimento do painel IHC multiplex. A importância da escolha de anticorpos primários que detectam alvos proteicos específicos foi enfatizada antes17. Nossos painéis multiplex de IHC são desenvolvidos principalmente com clones de anticorpos primários que também são usados e validados para IHC em nosso departamento de diagnóstico. No entanto, no caso do painel IHQ multiplex de células dendríticas, a maioria dos anticorpos não foi usada no cenário diagnóstico (van der Hoorn et al., manuscrito em submissão). Para garantir a especificidade e minimizar as diferenças de lote, optamos por usar anticorpos monoclonais em vez de anticorpos policlonais e também validamos a maioria dos anticorpos usando linhagens celulares transfectadas e células primárias. Ao longo dos anos, diferentes versões de painéis IHC multiplex foram utilizadas em vários estudos usando o sistema Vectra 3 18,21,23,24,25,26,27,28,29,30,31,32. Para implementar esses painéis IHC multiplex de maneira ideal no sistema PhenoImager HT, alguns ajustes tiveram que ser feitos nas combinações de anticorpos primários e fluoróforos. Para se beneficiar de uma melhor separação espectral e tempos de varredura mais rápidos de seções de tecido inteiro, é necessária a implementação dos mais recentes fluoróforos Opal480 e Opal780 e evitar o uso de fluoróforos Opal540 e Opal650 em painéis IHC multiplex de sete cores. Os tempos de varredura são ~ 3-10 vezes mais rápidos, dependendo do tamanho da seção de tecido. Os ajustes do painel IHC multiplex foram bastante fáceis de alcançar, mas algumas considerações precisam ser mantidas em mente. O espectro fluorescente do Opal480 se sobrepõe muito ao espectro de autofluorescência e, portanto, interfere na mistura espectral de eritrócitos e outras estruturas autofluorescentes. O uso de uma concentração aumentada do anticorpo primário emparelhado com o Opal480 resolveu esse problema na maioria dos casos. A implementação do filtro AF de amostra proprietário no PhenoImager HT facilita a separação de Opal480 e autofluorescência. No entanto, é melhor usar um anticorpo primário que produza um sinal claro quando usado com Opal480 para que seu sinal seja maior que a autofluorescência.
Embora esses painéis IHC multiplex estejam estabelecidos, a variação de lote para lote é algo que precisa ser considerado. Ao realizar controles de IHC monoplex antes de iniciar o experimento de IHC multiplex completo, às vezes observamos que os anticorpos primários têm um desempenho mais forte ou mais fraco de experimento para experimento. As razões para isso podem ser erros de pipetagem, condições de armazenamento de reagentes abaixo do ideal e prazo de validade. Resolvemos isso ajustando a solução de anticorpos primários com base em nossa experiência. Mesmo quando nenhum dos ajustes mencionados acima teve que ser feito, com cada experimento de lote IHC multiplex, é importante definir os tempos de exposição com base nas lâminas de controle coradas com IHC monoplex.
Como nossa pesquisa foi inicialmente focada em diferentes tipos de carcinomas e melanoma, os painéis multiplex IHC deveriam ser intercambiáveis entre os tipos de tumor com ajustes mínimos. Portanto, sempre incluímos vários tipos de tecido (tumoral) no processo de otimização e observamos que as diluições para anticorpos primários para marcadores de células imunes podem ser mantidas semelhantes entre diferentes tipos de tumor. No entanto, a detecção de tecido tumoral entre carcinomas e melanoma precisa de diferentes marcadores tumorais. Assim, o marcador tumoral sempre foi otimizado para funcionar no final de cada painel IHC multiplex e atualmente é sempre usado em conjunto com o Opal780, que coincidentemente também deve estar no último fluoróforo em um procedimento de coloração IHC multiplex. Ao usar o marcador tumoral consequentemente no final da IHQ multiplex, esses painéis IHQ multiplex podem ser facilmente trocados por outros tipos de tumor, como glioblastoma (ou seja, GFAP) e linfoma de Hodgkin (ou seja, CD30). Para o angiossarcoma, usamos este painel IHQ multiplex de linfócitos com gene relacionado à transformação de eritroblastos (ERG) como marcador tumoral com apenas dois experimentos de otimização25. A otimização incluiu a titulação do anticorpo primário ERG e o teste do painel IHC multiplex com ERG no final.
Outros ajustes nesses painéis multiplex IHC também podem ser feitos trocando um determinado marcador de célula imune por outro marcador imunológico ou funcional. Cada mudança requer otimização. O protocolo de otimização pode ser seguido conforme descrito anteriormente17. Certas alterações nos painéis IHC multiplex propostos irão interferir com os algoritmos ImmuNet que criámos. Dados suficientes devem ser gerados e tempo deve ser gasto para implementar essas mudanças no algoritmo (pelo menos 750 anotações para cada novo marcador e/ou fenótipo celular e 150 anotações para validação de marcadores previamente treinados). Os painéis aqui apresentados não contêm marcadores funcionais, embora a implementação de marcadores de checkpoint imunológico como PD-1 e PD-L1 em painéis multiplex IHQ seja realizada em nosso laboratório. No entanto, a análise de marcadores menos binários em sinais negativos e positivos tem se mostrado mais difícil e é uma área de pesquisa ativa em nosso grupo.
O número de marcadores que podem ser avaliados simultaneamente com IHQ multiplex é limitado em comparação com outras técnicas novas. Embora isso possa ser contornado analisando diferentes painéis em fatias consecutivas de um bloco FFPE, será difícil comparar essas fatias espacialmente. A orientação e os artefatos dobrados provavelmente não são os mesmos após a preparação do slide. No entanto, a IHQ multiplex é bastante acessível, o que a torna uma ferramenta atraente para mais instituições e pesquisadores e, portanto, mais adequada para implementação futura em um cenário diagnóstico. Com a padronização de painéis de células imunes IHC multiplex para vários tipos de tumores e pipelines de análise a jusante, mais conhecimento pode ser obtido sobre as diferenças no TME entre pacientes e tipos de tumor. Isso pode, por exemplo, levar a mais informações sobre o papel do TME na resposta antitumoral a tratamentos específicos. Isso pode até dar origem a novos biomarcadores para prever fatores como resposta ao tratamento e sobrevida esperada. No geral, isso pode permitir que a IHQ multiplex se torne uma ferramenta clínica para auxiliar na tomada de decisões clínicas, em uma abordagem de medicina personalizada. É certo que mais etapas do procedimento de análise provavelmente devem ser automatizadas e padronizadas para que seja viável para uso em um ambiente de diagnóstico diário, portanto, por enquanto, é principalmente uma perspectiva futurista.
A análise de vários marcadores em uma única lâmina de amostra pode ser uma ferramenta muito poderosa, apesar de seus desafios técnicos. Com protocolos experimentais padronizados e um método de análise robusto, como descrevemos aqui usando o ImmuNet, a quantificação de múltiplos marcadores o torna mais informativo do que o IHC clássico, enquanto o IHC multiplex permanece com um rendimento relativamente alto em comparação com os novos métodos experimentais de plex superior.
Disclosures
Os autores não têm conflitos de interesse a divulgar.
Acknowledgements
O PhenoImager HT foi adquirido por meio de financiamento fornecido pelo Centro Médico da Universidade Radboud e pelo Centro de Tecnologia Radboud para Microscopia. A FC é apoiada financeiramente por uma bolsa da Dutch Cancer Society (10673) e pela ERC Adv ARTimmune (834618). JT é apoiado financeiramente por uma bolsa NWO Vidi (VI.Vidi.192.084). Os autores gostariam de agradecer a Eric van Dinther e Ankur Ankan por sua ajuda na criação de fluxos de trabalho para armazenar dados IHC multiplex e Bengt Phung é agradecido por instruções sobre como implementar dados IHC multiplex no QuPath para desenho de ROI.
Materials
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
References
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205 (2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212 (2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032 (2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562 (2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762 (2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749 (2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938 (2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248 (2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498 (2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111 (2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133 (2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329 (2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511 (2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380 (2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238 (2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved