Визуализация одноцепочечных очагов ДНК в фазе G1 клеточного цикла
In This Article
Summary
В следующем протоколе представлено обнаружение очагов одноцепочечной ДНК в фазе G1 клеточного цикла с использованием синхронизации клеточного цикла с последующим иммунофлуоресцентным окрашиванием RPA2.
Abstract
ДНК имеет специальные пути клеточной репарации, способные справляться с повреждениями, которые могут возникать как из эндогенных, так и из экзогенных источников. Репарация ДНК требует сотрудничества между многочисленными белками, отвечающими за широкий спектр задач, от распознавания и сигнализации о наличии повреждения ДНК до его физического восстановления. Во время этого процесса часто образуются треки одноцепочечной ДНК (ssDNA), которые в конечном итоге заполняются ДНК-полимеразами. Природа этих треков мцДНК (как с точки зрения длины, так и с точки зрения количества), а также полимераза, рекрутируемая для заполнения этих пробелов, специфичны для пути репарации. Визуализация этих треков мцДНК может помочь нам понять сложную динамику механизмов репарации ДНК.
В этом протоколе представлен подробный метод подготовки G1-синхронизированных клеток для измерения образования очагов мцДНК при генотоксическом стрессе. Используя простой в использовании иммунофлуоресцентный подход, мы визуализируем ssDNA путем окрашивания на RPA2, компонент гетеротримерного комплекса репликации белка А (RPA). RPA2 связывается и стабилизирует промежуточные продукты мцДНК, которые возникают при генотоксическом стрессе или репликации, чтобы контролировать репарацию ДНК и активацию контрольных точек повреждения ДНК. Окрашивание 5-этинил-2'-дезоксиуридином (EdU) используется для визуализации репликации ДНК с целью исключения любых клеток S-фазы. Этот протокол обеспечивает альтернативный подход к традиционным неденатурирующим анализам на основе 5-бром-2'-дезоксиуридина (BrdU) и лучше подходит для обнаружения очагов мцДНК вне S-фазы.
Introduction
Чтобы поддерживать жизнь, клетки постоянно исследуют и восстанавливают ДНК, чтобы поддерживать свою геномную целостность. Клетки могут накапливать различные типы повреждений ДНК как из-за эндогенных (например, окисление, алкилирование, дезаминирование, ошибки репликации), так и из-за экзогенных (например, ультрафиолет, ионизирующее облучение) источников стрессоров ДНК. Неспособность восстановить эти повреждения приводит либо к апоптозу, либо к остановке клеточного цикла, либо к старению и может привести к заболеваниям1. Повреждения ДНК могут быть устранены любым из следующих основных путей репарации ДНК: DR (прямая реверсивная репарация), которая в основном восстанавливает алкилированные основания2; BER (коррекция эксцизии оснований), которая нацелена на негромоздкие ошибки оснований ДНК и одноцепочечные разрывы ДНК (SSB)3; NER (нуклеотидная эксцизионная репарация) для коррекции объемных, искажающих спираль повреждений ДНК4; MMR (репарация несоответствия), в основном нацеленная на несоответствие ДНК, петли вставки/делеции (IDL) и определенные повреждения основания5; NHEJ (негомологичное соединение концов) и HRR (гомологичная рекомбинационная репарация), которые активны на двухцепочечных разрывах ДНК (DSB)6; и TLS (translesion synthesis), который представляет собой механизм обхода повреждения ДНК7. Несмотря на то, что эти пути имеют различные особенности подложки, между ними есть определенные пересечения, чтобы обеспечить резервирование для эффективного ремонта. Понимание действия различных путей репарации ДНК в различных фазах клеточного цикла имеет решающее значение, поскольку эти факторы репарации ДНК могут служить важными мишенями для терапевтических подходов к лечению рака, старения и неврологических расстройств 8,9.
Одноцепочечная ДНК (ssDNA) генерируется на протяжении всего клеточного цикла в результате репарации повреждений ДНК, генерируемых как эндогенными, так и экзогенными повреждающими ДНК агентами. При генотоксическом стрессе мцДНК образуется в изобилии в фазах S и G2, где HRR и MMR имеют наибольшую активность, и когда механизм репликации останавливается или разрушается при столкновении с повреждениями ДНК 6,10,11. Другие пути репарации ДНК (например, NHEJ/микрогомологическое соединение концов (MMEJ)/одноцепочечный отжиг [SSA]) также генерируют ssDNA во время репарации DSB12. Эти треки мцДНК обычно возникают в результате резекции ДНК, осуществляемой экзонуклеазами, такими как EXO1, ДНК2 и CtIP во время HR и MMR, эндонуклеазами, такими как XPF и XPG во время NER, или в результате комбинированного действия POLB и FEN1 во время BER 4,13,14,15,16,17,18,19 . Благодаря работе механизма репликации, треки мцДНК также генерируются, когда геликазы ДНК раскручивают ДНК перед PCNA-связанными репликативными полимеразами20. Напротив, в фазе G1 недостаток HRR и репликации ДНК, а также ограниченная активность MMR уменьшают количество генерируемых треков мцДНК и, следовательно, их сложнее обнаружить 10,11,21.
Клеточные треки мцДНК являются высокочувствительными структурами, которые необходимо защищать, чтобы избежать образования DSB. Это достигается за счет покрытия дорожек ssDNA RPA. RPA представляет собой обильный гетеротримерный белковый комплекс, состоящий из нескольких субъединиц (RPA1, RPA2 и RPA3, также называемых RPA70, RPA32 и RPA14 соответственно), которые повсеместно экспрессируются на протяжении всего клеточного цикла22. Каждая субъединица RPA содержит ДНК-связывающий домен (DBD), способный взаимодействовать с 4-6 нуклеотидами, а объединенные субъединицы образуют стабильное тримеризационное ядро. В общей сложности RPA связывается примерно с 20-30 нуклеотидами с субнаномолярным сродством23,24.
Традиционные методы используют иммунофлуоресцентную (ИФ) микроскопию для визуализации очагов мцДНК путем мечения 5-бром-2'-дезоксиуридина (BrdU), включенного в геномную ДНК с помощью антител BrdU25. Этот подход основан на том факте, что антитела к BrdU могут обнаруживать BrdU только в экспонированной ssDNA25. Несмотря на то, что этот подход прост, он также имеет определенные ограничения. Например, клетки предварительно обрабатываются для включения BrdU перед началом эксперимента, что отнимает много времени и может мешать последующим эффекторам. Таким образом, обнаружение мцДНК на основе BrdU ограничено репликацией клеток и не может быть использовано для покоящихся клеток. Это исключает применение этого метода для изучения репарации ДНК в нереплицирующихся клетках, несмотря на его важность при некоторых заболеваниях, таких как рак и нейродегенерация 5,26. Кроме того, поскольку структуры BrdU и EdU очень похожи, большинство антител BrdU проявляют перекрестную реактивность по отношению к EdU, что необходимо учитывать при проведении экспериментов с двойным мечением27. Ранее окрашивание RPA использовалось для выявления очагов мцДНК в основном в клетках S-фазы; тем не менее, некоторые работы также успешно использовали его за пределами S-фазы 28,29,30,31,32,33,34,35. Следующий протокол эффективно использует свойства RPA, позволяя визуализировать очаги ssDNA после повреждения ДНК в фазе G1 клеточного цикла (хотя его можно использовать во всех фазах клеточного цикла).
Protocol
1. Поддержание hTERT-иммортализированных клеток пигментного эпителия сетчатки (RPE1)
- Поддерживайте клеточные линии RPE1 в модифицированной орлиной среде (DMEM) Dulbecco, дополненной 10% термически инактивированной фетальной бычьей сывороткой (Hi-FBS) и 100 мкг/мл пенициллина-стрептомицина (далее именуемого питательной средой) во увлажненном инкубаторе с 5%CO2 при 37 °C. Для рутинного культивирования выращивают клетки RPE1 в 15-сантиметровой чашке, обработанной культурой тканей, и разделяют при достижении 80-90% слияния (~16-18 ×10,6 клеток на 15-сантиметровую чашку).
- При расщеплении удалите среду и промойте клетки 10 мл 1x фосфатно-солевого буфера (PBS).
- Добавьте 3 мл 0,05% трипсина-ЭДТА, чтобы покрыть всю поверхность чашки. Держите клетки при температуре 37 °C с помощью трипсина до тех пор, пока они не отделятся.
- После трипсинизации клетки ресуспендируют с питательной средой и отжимают их при 150 × г в течение 5 мин при комнатной температуре (RT, 22-25 °C). Удалите надосадочную жидкость и аккуратно ресуспендируйте клетки в 10 мл питательной среды.
- Высевают 1,6-1,8 × 106 клеток в новую 15-сантиметровую чашку (~1 мл клеточной суспензии).
ПРИМЕЧАНИЕ: Все работы по культивированию тканей должны выполняться в соответствии с уровнями безопасности BSL-2. Инкубационный период трипсинизации зависит от слияния клеток. Обычно процесс занимает 2-3 минуты для 90% сливающейся пластины. Клетки следует регулярно проверять на контаминацию микоплазмами с помощью имеющихся в продаже наборов (см. примеры в таблице материалов).
2. Нокдаун миРНК интересующего гена (GOI)
- За день до трансфекции высевают 1,0 × 106 клеток RPE1 в 10-сантиметровую планшет, обработанную культурой тканей, с 10 мл питательной среды.
- В день трансфекции комплекс миРНК. Для 10-сантиметровой пластины используют конечную концентрацию 20 нМ siRPA2 и 12 мкл реагента для трансфекции на основе липидов в 500 мкл среды для трансфекции с низким содержанием сыворотки. Аккуратно перемешайте все компоненты, покрутив пробирку, и инкубируйте при RT (22-25 °C) в течение 5 минут.
- Добавьте комплексную смесь миРНК в клетки по каплям и инкубируйте клетки с миРНК в течение 48 часов.
3. Синхронизация ячеек RPE1 в фазу G0
- Трипсинизируйте клетки RPE1 из шага 2.3, как описано в разделе 1 (~2 × 106 ячеек).
- Переложите клеточную суспензию в центрифужные пробирки объемом 15 мл и центрифугируйте ее при 150 × г, RT (22-25 °C) в течение 5 мин.
- Удалите надосадочную жидкость и ресуспендируйте клетки в 12 мл PBS. Центрифугируют клетки при 150 × г при RT (22-25 °C) в течение 5 мин. Повторите удаление надосадочной жидкости и центрифугирование дважды.
- Ресуспендант клеток в 10 мл безсывороточного DMEM с добавлением 100 мкг/мл пенициллина-стрептомицина, 1 мМ пирувата натрия, 15 мМ HEPES и помещают их в 10-сантиметровую чашку для тканевой культуры.
ПРИМЕЧАНИЕ: Если клетки имеют тенденцию к слипанию, ресуспендируйте их всего в 1 мл DMEM без сыворотки и пипетируйте их вверх и вниз 5 раз с помощью наконечника P1000, чтобы удалить комки, прежде чем разбавить суспензию до окончательного объема 10 мл. - После 24-часового голодания сыворотки введите второй раунд сайленсинга, используя ту же процедуру, что описана в разделе 2, путем добавления комплексной миРНК к клеткам, испытывающим голодание в сыворотке.
- Выдержите клетки RPE1 в безсывороточном DMEM в течение 72 ч, прежде чем приступать к высвобождению G1.
4. Покрытие покровным стеклом и высвобождение ячеек в фазу G1
- Простерилизуйте пинцет 70% этиловым спиртом и поместите один стеклянный покровный листок (диаметром 12 мм и толщиной #1,5 [0,17 мм]) в лунку 24-луночной пластины.
- Разбавьте матрицу витроктинового покрытия PBS до получения конечной концентрации 10 мкг/мл. Добавляют 500 мкл раствора витронектина в каждую лунку, содержащую покровные стекла, и инкубируют в течение 1 ч при RT.
- Удалите раствор покрытия и промойте покровные стекла 1 мл PBS.
- Отделите клетки RPE1, испытывающие нехватку сыворотки, от 10-сантиметровой обработанной тканевой культуры, используя 1 мл 0,05% трипсина после промывки PBS в течение 1 мин при 37 °C.
ПРИМЕЧАНИЕ: Клетки отделяются гораздо быстрее после сывороточного голодания. Соблюдайте осторожность при промывке клеток PBS и используйте короткое время трипсинизации. - Чтобы инактивировать трипсин, ресуспендируйте клетки RPE1 в общей сложности в 6 мл питательной среды. Удаляют инактивированный трипсин, откручивая клетки с помощью 150 × г при RT (22-25 °C) в течение 5 мин.
- Ресуспендируйте клетки в 1 мл питательной среды и измерьте количество клеток.
- Высевают 4 × 104 клеток RPE1 на покрытое покрытием покровное стекло в общей сложности 500 мкл питательной среды.
ПРИМЕЧАНИЕ: Убедитесь, что жизнеспособность клетки выше 90%, прежде чем переходить к последующим этапам. Жизнеспособность клеток может быть быстро оценена с помощью окрашивания трипановым синим на этапе подсчета клеток. - Через 6 ч после введения клеток в питательную среду клетки, высвобождаемые G0 клетки будут находиться в ранней фазе G1. Проводите эксперименты в G1 в течение 6-12 часов до того, как клетки начнут входить в S-фазу.
- Перед введением повреждения ДНК в клетки проводят импульс с 10 мкМ 5-этинил-2'-дезоксиуридином (EdU) в течение 30 мин при 37 °С, разведенным в питательной среде.
- Удалите среду, содержащую EdU, и обработайте клетки 10 мкМ тимидином в течение 10 мин при 37 °C, чтобы предотвратить остаточное включение EdU во время индукции повреждения ДНК.
- Удаляют среду с тимидином и обрабатывают клетки 250 мкМH2O2в течение 1 ч, разведенным в питательной среде.
5. Иммунофлуоресцентное окрашивание мцДНК
- Промойте клетки один раз 1 мл RT (22-25 °C) PBS для удаления среды и компонентов сыворотки.
ПРИМЕЧАНИЕ: Будьте осторожны при мытье клеток, чтобы избежать отслоения и высыхания. Не обрабатывайте много лунок одновременно. - Предварительная экстракция: Промытые клетки инкубируют в 1 мл экстракционного буфера CSK (Таблица 1) в течение 5 мин при RT (22-25 °C).
ПРИМЕЧАНИЕ: Предварительная экстракция CSK удаляет все белки, не связанные с хроматином, включая растворимый RPA2.
ВНИМАНИЕ: Triton X-100 вреден при проглатывании и может вызвать раздражение кожи и повреждение глаз. - Удалите буфер CSK из ячеек и зафиксируйте их напрямую, добавив 0,5 мл 3,6% раствора параформальдегида (в PBS), содержащего 0,05% Triton X-100, в течение 10 мин при RT (22-25 °C).
ВНИМАНИЕ: Важно приготовить 3,6% PFA из 32% запаса PFA свежим. Параформальдегид может вызвать серьезное повреждение глаз, раздражение кожи и дыхательных путей. - Промойте клетки один раз 1 мл PBS, содержащего 0,05% Triton X-100, чтобы удалить PFA.
- Далее пермеабилизируют клетки с помощью 1 мл PBS, содержащего 0,5% Triton X-100, в течение 15 мин при RT (22-25 °C).
- Реакция EdU click-IT для визуализации реплицирующихся клеток (S-фаза)
- Раствор пермеабилизации удалить и промыть клетки 2 раза, используя 1 мл блокирующего буфера (табл. 1).
ВНИМАНИЕ: Бычий сывороточный альбумин (БСА) может вызвать раздражение дыхательных путей. - Добавьте 1 мл блокирующего буфера (табл. 1) и осторожно встряхните пластину, содержащую покровное стекло, в течение 10 мин при RT (22-25 °C).
- Удалите блокирующий буфер и добавьте 500 мкл коктейля клик-реакции, содержащего пиколилазид 647 (табл. 1). Инкубируйте покровные стекла в течение 30 мин при RT (22-25 °C) с легким покачиванием и проводите инкубацию ниже по течению в темноте.
ПРИМЕЧАНИЕ: При использовании антител к BrdU используйте удвоенное количество (1 мл) и время (60 мин) для клик-реакции в соответствии с рекомендациями производителя, чтобы убедиться, что реакция насыщена и включенный EdU помечен. Это ограничивает перекрестную реактивность антител BrdU27.
- Раствор пермеабилизации удалить и промыть клетки 2 раза, используя 1 мл блокирующего буфера (табл. 1).
- Удалить смесь клик-реакции и промыть клетки 2 раза PBS с 0,05% Triton X-100 в течение 10 мин при RT (22-25 °C) (рисунок 1 и рисунок 2).
- Добавьте 1 мл блокирующего буфера и инкубируйте при RT (22-25 °C) в течение 30 мин. В качестве альтернативы храните клетки в блокирующем буфере при температуре 4 °C в течение ночи.
- Применяют первичные антитела (крысы против RPA2, разведение 1:1,000) в течение 2 ч при RT (22-25 °C) в 250-500 мкл блокирующего буфера с легким покачиванием.
- Промойте клетки 2 раза PBS, содержащим 0,05% Triton X-100, чтобы быстро удалить большую часть раствора антител.
- Продолжайте промывать ячейки в течение 3 x 10 мин с блокирующим буфером при RT (22-25 °C).
- Применяют вторичные антитела (антикрысиное Alexa-488, разведение 1:1,000) в 250-500 мкл блокирующего буфера при RT (22-25 °C) в течение 2 ч при легком встряхивании.
- Промойте клетки блокирующим буфером 2 раза, чтобы быстро удалить большую часть вторичных антител. Продолжайте промывать ячейки в течение 3 x 10 мин PBS, содержащим 0,05% Triton X-100 при RT (22-25 °C).
- Для противоокрашивания ядер клетки однократно промывают PBS, содержащим 0,05% Triton X-100 и 1 мкг/мл 4',6-диамидино-2-фенилиндола (DAPI) в течение 10 мин при RT (22-25 °C). Промыть ячейки один раз PBS в течение 5 мин при RT (22-25 °C).
- Установите защитное стекло на предметные стекла микроскопа, используя 10 мкл монтажной среды/покровного стекла. Перед монтажом окуните покровные стекла в дистиллированную воду, чтобы избавиться от кристаллов соли. На следующий день сфотографируйте слайды и храните их при температуре 4 °C в течение нескольких недель (рисунок 3).
6. Получение изображений и количественная оценка
- Для получения изображений используйте любой доступный эпифлуоресцентный микроскоп, оснащенный стандартными наборами фильтров, для получения изображений каналов DAPI, FITC и Cy5 с увеличением не менее 60-63x, высокой числовой апертурой и масляными объективами для визуализации ядерных очагов.
ПРИМЕЧАНИЕ: Оптимальное возбуждение DAPI ~359 нм; Возбуждение Alexa 488 ~488 нм; в то время как возбуждение Alexa 647 составляет ~647 нм. - Для анализа изображений откройте файлы изображений в Fiji/ImageJ.
- Изготовьте ядерные маски с помощью окрашивания DAPI (рис. 4A-F и дополнительное видео S1).
- Откройте образ DAPI.
- Выберите Процесс | Улучшите контрастность и установите для параметра Насыщенный пиксель значение 0.35.
- Нажмите «Обработать» | Двоичный | Преобразовать в маску. Выберите двоичный файл | Заполните пробелы и нажмите Analyze | Анализ частиц. Установите размер на 10-Бесконечность.
- В менеджере ROI нажмите Показать все.
- Нахождение очагов RPA2 в ядре (рис. 4G, H и дополнительное видео S1)
- Откройте образ RPA2.
- Выберите Процесс | Найдите Maxima. Установите значение выпуклости , которое выделяет фокусы RPA2 (от 500 до 750), отделяя их от фона.
- Наконец, нажмите кнопку «Измерить » в Диспетчере рентабельности инвестиций.
- Вычислите общее количество ядерных фокусов ssDNA, разделив значение в столбце RawinDen на 255 (максимальное значение интенсивности пикселей в каждом фокусе).
- Выполняйте статистический анализ с помощью предпочтительного статистического программного инструмента.
ПРИМЕЧАНИЕ: Исключите из анализа все EdU-положительные клетки и неправильно сегментированные маски DAPI.
- Изготовьте ядерные маски с помощью окрашивания DAPI (рис. 4A-F и дополнительное видео S1).
Representative Results
Для преодоления ограничений детекции мцДНК в G1 мы использовали RPA2, который повышает как специфичность, так и интенсивность обнаружения очагов мцДНК35. Чтобы добиться точной синхронизации клеток, мы использовали клетки RPE1, которые могут быть эффективно обработаны сывороткой и синхронизированы в фазу G0. Затем их можно индуцировать к возвращению в клеточный цикл путем добавления сыворотки после депривации сыворотки. Для подтверждения эффективности синхронизации мы пометили клетки EdU, а содержание их ДНК — пропидий-йодидом. Кроме того, мы собрали качественные и количественные результаты с помощью проточной цитометрии (дополнительный рисунок S1A). Точечные диаграммы показывают, что после 72 ч сывороточного голодания ~98% клеток находятся в фазе G0. После добавления сывороточных сред в течение 6 ч клетки снова вступают в клеточный цикл (как видно по увеличению уровня р27 на рисунке 1A), имея ~97% клеток в G1, в то время как в фазе S только <1%, в фазе G2 — <2% клеток (дополнительный рисунок S1A). Через 20-28 ч после добавления сыворотки к клеткам они постепенно проходят через S-фазу, как показано на графиках проточной цитометрии (дополнительный рисунок S1A). Этот протокол клеточной синхронизации дает ~97% чистой популяции G1 (через 6 ч после добавления сыворотки после 72 ч сывороточного голодания). Для дальнейшей проверки эффективности синхронизации мы сравнили экспрессию маркеров клеточного цикла после высвобождения сыворотки крови с помощью вестерн-блоттинга (рис. 1A и дополнительный рисунок S1B) и параллельно провели анализ инкорпорации EdU для визуализации репликации ДНК. Окрашивание EdU также подчеркивает эффективность синхронизации и отсутствие репликации ДНК в фазе G1 (рис. 1B, C).
Традиционные методы обнаружения мцДНК в клетках млекопитающих основаны на обнаружении BrdU в мцДНК. На рисунке 2А показано, что при обработкеН2О2 и неокарциностатином (НКС) очаги BrdU обнаруживались только в клетках S-фазы, в то время как фокусы мцДНК не обнаруживались в клетках, не принадлежащих к S-фазе. Окрашивание антител к BrdU также показало заметное окрашивание ядрышкового фона, которое можно было обнаружить во всех ядрах, независимо от стадии клеточного цикла или применяемого лечения. Используя описанный здесь протокол щелчка EdU, мы не смогли обнаружить колокализацию очагов EdU и BrdU, что очевидно в необработанных образцах на рисунке 2A. Чтобы полностью исключить любой сигнал BrdU, возникший в результате перекрестной реакционной способности, мы избегали маркировки EdU и использовали циклин A2 в качестве маркера S-G2. Однако окрашивание циклином А2 не позволяло проводить предварительную экстракцию ЦСК, и при этом условии мы не наблюдали никаких очагов BrdU даже после генотоксического стресса (дополнительный рисунок S2A). Это подчеркивает тот факт, что предварительная экстракция CSK необходима для окрашивания мцДНК на основе анти-BrdU. В качестве контроля мы протестировали окрашивание антителами BrdU в условиях денатурации. Это открывает ДНК для обнажения инкорпорированного BrdU, что показывает, что BrdU был равномерно включен (дополнительный рисунок S2B).
Напротив, окрашивание RPA2 показывает образование NCS- и H2O2-зависимых очагов не только в S-фазе, но и в других фазах клеточного цикла (рис. 2B). В качестве контроля мы также обрабатывали клетки HU, который вызывает только накопление мцДНК в клетках, подвергающихся репликации. Как и ожидалось, мы обнаружили повышение сигнала только при обработке HU антителом RPA2 в EdU-положительных клетках, что подчеркивает специфичность этого подхода. Антитело RPA2 также может обнаруживать естественное образование мцДНК во время репликации при отсутствии экзогенного генотоксического стресса (рис. 2B). Высокая чувствительность антител RPA2 побудила нас попытаться использовать их в фазе G1, где обычное окрашивание BrdU не смогло обнаружить никаких сигналов генотоксического стресса (дополнительный рисунок S2C). На рисунке 3А показано, что образование очагов мцДНК при леченииH2O2было обнаружено при использовании антител к RPA2, даже в G1. Отмечено достоверное увеличение количества RPA2-очагов в этих ядрах при обработкеН2О2(рис. 3Б). Эти очаги были специфичны для RPA2, так как подавление RPA2 упраздняло сигнал ПЧ (рис. 3A,B). На рисунке 3C и дополнительном рисунке S1C показана эффективность подавления RPA2 в этих ячейках. По сравнению с традиционными методами, обнаружение мцДНК на основе RPA2 является высокочувствительным, поэтому его применение может быть расширено на клетки фазы G1.
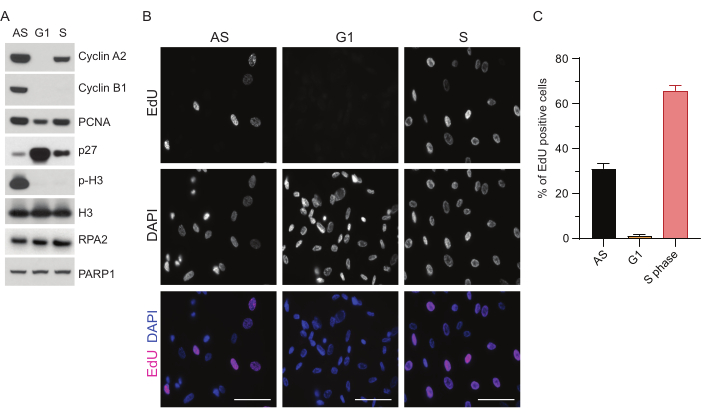
Рисунок 1: Эффективность синхронизации клеток RPE1 после сывороточного голодания. (A) Иммуноблоттинги показывают указанные уровни белка в асинхронных, синхронизированных с G1 и S-фазами клетках RPE1. (B) На репрезентативных изображениях показаны асинхронные, синхронизированные по фазе G1 и S ячейки RPE1, которые подвергались воздействию 10 мкМ EdU в течение 30 минут перед фиксацией и визуализировались с помощью реакции Click-IT. DAPI использовался для противодействия окрашиванию ядерной ДНК. Масштабные линейки = 50 мкм. (C) График показывает процентное соотношение EdU-положительных клеток к общей популяции клеток, оцененных DAPI. Столбик погрешности представляет собой стандартную ошибку среднего значения, а анализируемые числа ядер были следующими: AS n = 219, G1 n = 630, S n = 437. Сокращения: RPE1 = hTERT-иммортализированные пигментные эпителиальные клетки сетчатки; AS = асинхронный; EdU = 5-этинил-2'-дезоксиуридин; DAPI = 4',6-диамидино-2-фенилиндол. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
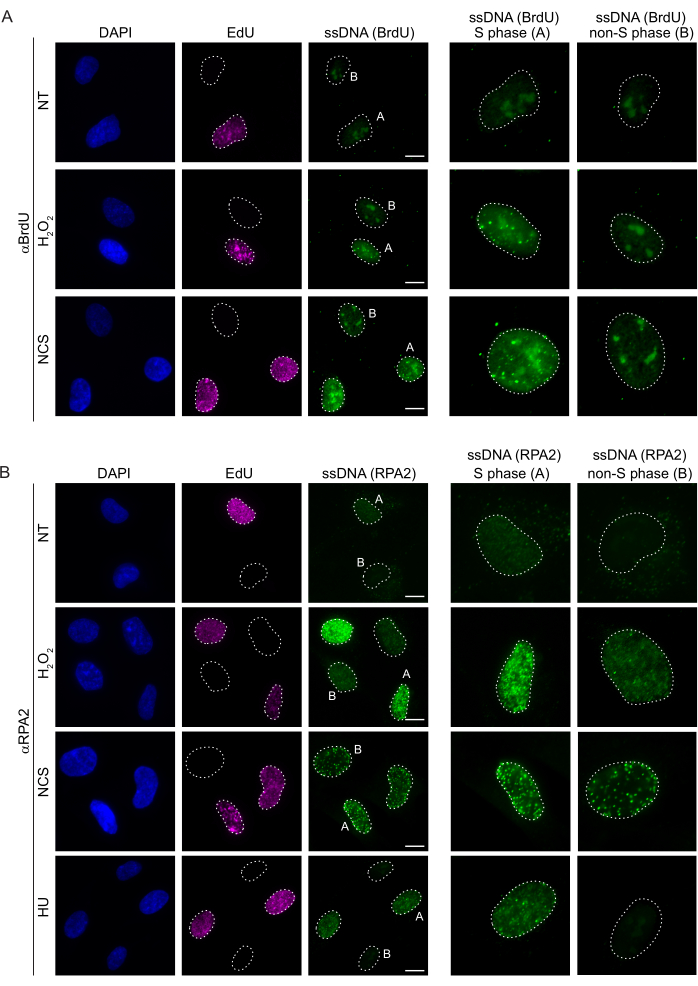
Рисунок 2: Обнаружение мцДНК с антителами BrdU или RPA2 при повреждении ДНК. (A) Репрезентативные изображения иллюстрируют фокусы мцДНК с использованием αBrdU (зеленый), клетки S-фазы выделены EdU (фиолетовый), а DAPI использовался для противодействия окрашиванию ядерной ДНК (синий). Клетки RPE1 хранили в 10 мкМ BrdU в течение 48 ч до любой дополнительной обработки. Через 48 ч клетки импульсировали 10 мкМ EdU в течение 30 мин с последующей обработкой H2O2 (250 мкМ) в течение 1 ч или неокарциностатином (0,5 мкг/мл) в течение 4 ч. Клетки фиксировались после предварительной экстракции ЦСК. Белой пунктирной линией обозначена граница каждого ядра. Масштабная линейка = 5 мкм. На панелях справа представлены увеличенные изображения обозначенных ядер S-фазы или не-S-фазы. (B) Репрезентативные изображения иллюстрируют фокусы мцДНК с использованием антител αRPA2 (зеленый цвет). Клетки S-фазы выделены EdU (фиолетовый), а DAPI использовался для противодействия окрашиванию ядерной ДНК (синий). Клетки RPE1 импульсировали 10 мкМ EdU в течение 30 мин, а затем 1 чH2O2 (250 мкМ), 4 ч гидроксимочевины (2 мМ) или 4 ч NCS (0,5 мкг/мл). Клетки фиксировались после предварительной экстракции ЦСК. Белой пунктирной линией обозначена граница каждого ядра. Масштабная линейка = 10 мкм. На панелях справа представлены увеличенные изображения обозначенных ядер S-фазы или не-S-фазы. Сокращения: ssDNA = одноцепочечная ДНК; BrdU = 5-бром-2'-дезоксиуридин; DAPI = 4',6-диамидино-2-фенилиндол; RPE1 = hTERT-иммортализированные клетки пигментного эпителия сетчатки; EdU = 5-этинил-2'-дезоксиуридин; NCS = неокарциностатин; HU = гидроксимочевина. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
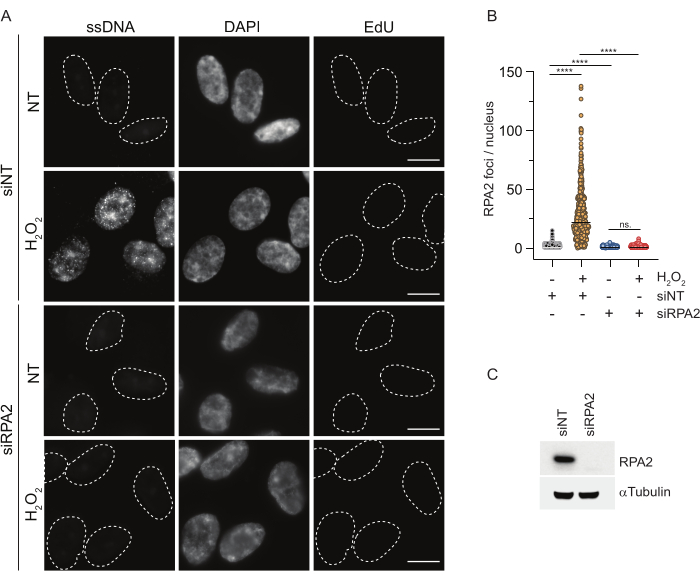
Рисунок 3: Обнаружение очагов мцДНК в фазе G1 с использованием антител RPA2. (A) Клетки RPE1 трансфицировали либо миРНК, нацеленными на RPA2, либо контрольными миРНК, а затем синхронизировали в G1 и импульсно мечили 10 мкМ EdU в течение 30 мин перед обработкой ихH2O2 (250 мкМ) в течение 1 ч при наличии показаний. DAPI использовался для противодействия окрашиванию ядерной ДНК. Клетки фиксировались после предварительной экстракции ЦСК. Белой пунктирной линией обозначена граница каждого ядра. Масштабная линейка = 5 мкм. (B) Измерения количества фокусов/ядер RPA2 были проведены на основе двух независимых экспериментов. При анализе учитывались только EdU-отрицательные клетки. Линии представляют среднее значение на графиках. Для статистического анализа был проведен непараметрический критерий ANOVA (Краскела-Уоллиса). Звезды обозначают P < 0,0001. Анализируемое количество ядер было следующим: siNT noH2O2 n = 513, siNTH2O2 n = 603, siRPA2 noH2O2 n = 266, siRPA2 H2O2 n = 536. (C) Эффективность нокдауна миРНК показана при иммуноблоттинге. Сокращения: siNT = нетаргетный контроль миРНК; BrdU = 5-бром-2'-дезоксиуридин; DAPI = 4',6-диамидино-2-фенилиндол; RPE1 = hTERT-иммортализированные клетки пигментного эпителия сетчатки; EdU = 5-этинил-2'-дезоксиуридин. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.

Рисунок 4: Количественная оценка очагов мцДНК на примере Фиджи. Подробные шаги на Фиджи, показывающие, как оценить количество очагов RPA2 в ядре. (А-Е) Создание ядерной маски с использованием канала DAPI. (Ф-Х) Пороговое значение для идентификации отдельных очагов ядерной мДНК по фоновому сигналу. Сокращения: ssDNA = одноцепочечная ДНК; DAPI = 4',6-диамидино-2-фенилиндол. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
| Цитоскелетный (CSK) буфер | |
| ТРУБЫ рН 7,0 | 10 мМ |
| NaCl | 100 мМ |
| ЭДТА рН 8 | 1 мМ |
| MgCl2 | 3 мМ |
| D-сахароза | 300 мМ |
| Тритон Х-100 | 0.20% |
| Коктейль ингибиторов фосфатазы | 1 таблетка на 10 мл |
| Коктейль ингибиторов протеазы | 1 таблетка на 10 мл |
| разбавленный в ддН2О | н/д |
| Промывочный буфер | |
| Тритон Х-100 | 0.05% |
| разбавленный в ПБС | н/д |
| Буфер пермеабилизации | |
| Тритон Х-100 | 0.50% |
| разбавленный в ПБС | н/д |
| Фиксирующий раствор | |
| Параформальдегид | 3.60% |
| Тритон Х-100 | 0.05% |
| разбавленный в ПБС | н/д |
| Блокирующий буфер | |
| Бычий сывороточный альбумин (BSA) | 5% |
| Тритон Х-100 | 0.10% |
| разбавленный в ПБС | н/д |
| Коктейль реакции Click-iT Plus | |
| 1x буфер реакций Click-iT | 435 мл |
| Решение Alexa Fluor PCA | 5 мл |
| CuSO4-медный защитный премикс | 10 мл |
| 1x буферная добавка Click-iT | 50 мл |
| Общий объем | 500 мл |
Таблица 1: Состав буферов, используемых в данном протоколе.
Дополнительный рисунок S1. (A) Клетки RPE1 синхронизировали с фазой G0 с помощью сывороточного голодания в течение 72 ч, а затем высвобождали в различные фазы клеточного цикла путем повторного введения сыворотки. Точечные диаграммы показывают клетки в фазах G0/G1, S или G2/M, где часы показывают время после повторного добавления сыворотки после сывороточного голодания. На графике справа показано процентное соотношение ячеек G0/G1, S и G2/M в каждом условии. Анализ FACS проводили с помощью коммерчески доступного набора для пролиферации клеток с использованием EdU и йодида пропидия в соответствии с рекомендациями производителя. (B) Необрезанные вестерн-блоттинги для рисунка 1. Цифры показывают маркеры молекулярной массы в кДа. PARP1 использовался в качестве регулятора загрузки и загружался в гель, который также был разработан против CCNA2, p27 (дополнительно очищенного от PCNA) и pH3 (S10) (дополнительно очищенного от H3) путем разрезания мембраны. CCNB1 и RPA2 загружали в отдельный гель с использованием одинакового количества лизата белка для обеспечения сопоставимости. (C) Необрезанные сканы вестерн-блоттинга для рисунка 3. Цифры показывают маркеры молекулярной массы в кДа. Аббревиатура: EdU = 5-этинил-2'-дезоксиуридин. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительный рисунок S2: (A) Репрезентативные изображения иллюстрируют фокусы мцДНК с использованием антител BrdU (зеленый); Ячейки S-фазы выделены циклином А2 (красный); и DAPI был использован для противодействия ядерному окрашиванию ДНК (синий). Клетки RPE1 хранили в 10 мкМ BrdU в течение 48 ч перед дальнейшим лечением. Через 48 ч клетки обрабатывалиН2О2(250 мкМ) в течение 1 ч или неокарциностатином (0,5 мкг/мл) в течение 4 ч до фиксации. Белой пунктирной линией обозначена граница каждого ядра. Масштабная линейка = 5 мкм. (B) Окрашивание BrdU клеток RPE1 в условиях денатурации и без нее. Асинхронные ячейки RPE1 предварительно обрабатывали 10 мкМ BrdU в течение 48 ч. Масштабная линейка = 10 мкм. (C) Измерения количества фокусов/ядер BrdU были проведены на основе двух независимых экспериментов в синхронизированных клетках RPE1 G1. При анализе учитывались только EdU-отрицательные клетки. Линии представляют среднее значение на графиках. Для статистического анализа был проведен непараметрический критерий ANOVA (Краскела-Уоллиса). Буква 'ns' указывает на несущественную разницу. Анализируемое число ядер было следующим: NT n = 52, NCS n = 105,H2O2 n = 82. Сокращения: siNT = нетаргетный контроль миРНК; BrdU = 5-бром-2'-дезоксиуридин; DAPI = 4',6-диамидино-2-фенилиндол; RPE1 = hTERT-иммортализированные клетки пигментного эпителия сетчатки; NCS = неокарциностатин. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительное видео S1: Запись экрана анализа фокусов RPA2 на Фиджи. Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Discussion
Поддержание здоровой, свободной от микоплазмы клеточной культуры имеет решающее значение для всех экспериментов, описанных выше. Клетки RPE1 имеют прочное прикрепление к пластиковой посуде, обработанной тканевой культурой, в обычных питательных средах; Однако их связывающие свойства значительно снижаются при хранении в условиях без сыворотки. Кроме того, для получения изображений фокусов мцДНК с высоким разрешением под микроскопом клетки должны быть нанесены на покровное стекло толщиной 0,17 мм, которое недостаточно гидрофильно для правильного прикрепления клеток RPE1. Без правильно уплощенных и равномерно распределенных клеток очень сложно визуализировать отдельные очаги мцДНК. Поэтому очень важно выбрать подходящий материал покрытия (например, витронектин) и оставить достаточное время (6-12 ч) для распространения и прикрепления клеток после их высвобождения в фазу G1.
Сложной частью протокола является получение однородных ячеек RPE1, синхронизированных с G1. Для этого необходимо выполнить два важных шага. Во-первых, для эффективного сывороточного голодания клетки должны быть трипсинизированы, тщательно промыты PBS и непосредственно высеяны в новые чашки для культивирования тканей с использованием безсывороточных сред. Промывание клеток непосредственно в чашках для культивирования тканей для удаления сыворотки не даст эффективной синхронизации G0. Во-вторых, при высвобождении клеток в фазу G1 клетки должны быть снова трипсинизированы и высеяны на планшеты для культуры свежих тканей. Аналогичным образом, простая смена среды и добавление в клетки питательной среды, содержащей сыворотку, не приведет к синхронной записи G1. Кроме того, для правильного входа в G1 плотность посева ячеек на покрытых покровных стеклах должна быть на определенном уровне слияния. В то время как идеальная синхронизация клеток, как правило, недостижима, этот протокол синхронизации, описанный здесь, дает ~97% чистой популяции G1. Рекомендуемая плотность засева для RPE1 на покровном стебле диаметром 12 мм составляет ~4 × 104 для получения однородного поля зрения для визуализации с примерно 70% слиянием. Более высокая плотность посева приводит к тому, что клетки отделяются и «отслаиваются» после экстракции CSK, что приводит к более высокому фоновому сигналу во время получения изображения.
Для снижения любого фонового сигнала и достижения благоприятного соотношения сигнал/шум необходима тщательная промывка после первичной и вторичной инкубации антител. Поскольку необходимо выполнить несколько этапов промывки, важно также не допустить пересыхания колодца на каждом этапе промывки. Мы минимизируем этот артефакт, применяя минимум 0,05% Triton X-100 на всех этапах промывки и инкубации. После того, как лунки высохли, в ячейках изменилось соотношение сигнал/шум; Это приводит к мозаичному рисунку под микроскопом и может помешать оценке. Получение изображений Z-стека в сочетании с деконволюцией может помочь в захвате фокусов в различных фокальных плоскостях для улучшения анализа.
Традиционные методы основаны на обнаружении инкорпорированного BrdU в неденатурирующих условиях. Эти методы, однако, зависят от предварительной обработки клеток высокими дозами BrdU в течение, по крайней мере, 1-2 дней (или времени, эквивалентного полному клеточному циклу в используемой клеточной линии) для обеспечения равномерной инкорпорации генома. Нежелательно, что обширное включение BrdU может вызвать интерференцию клеточного цикла36. Чтобы устранить эти ограничения, этот метод использует эндогенный RPA2 для обнаружения очагов мцДНК. Этот подход не требует репликационного включения BrdU, он также может быть использован в постмитотических клетках. Поскольку обширное внедрение BrdU не требуется, это экономит время и снижает сложность эксперимента. Используя окрашивание RPA2 для визуализации ssDNA, мы можем использовать 2'-дезокси-5-этинилуридин (EdU) и клик-химию для маркировки репликации ДНК, избегая при этом возможной перекрестной реактивности антител BrdU к EdU 27,37,38. Особое внимание следует уделять правильной маскировке инкорпорированного EdU во время клик-реакции, чтобы антитела BrdU не вступали в перекрестную реакцию с EdU 27,39.
Наконец, важным преимуществом использования RPA2 вместо BrdU является превосходное соотношение сигнал/шум по сравнению с окрашиванием BrdU вне S-фазы. Мы обнаружили, что неденатурирующее окрашивание BrdU и его способность визуализировать ssDNA ограничены S-фазой даже в реплицирующихся клетках (рис. 2). Антитела BrdU связываются только с достаточно подверженным воздействию BrdU на участках ssDNA. Связывание белков репарации, включая RPA2, с участками мцДНК может подавлять или препятствовать достаточному воздействию BrdU в мцДНК. Мы также обнаружили, что предварительная экстракция CSK необходима для визуализации ssDNA с использованием антител BrdU. Это возможно потому, что треки мцДНК недоступны для антитела без удаления из них слабосвязанных белковых компонентов.
Тем не менее, существуют некоторые ограничения, связанные с этим протоколом. Ограничением использования RPA2 для детектирования ssDNA является необходимость оптимизации этапа предварительной экстракции CSK. Несвязанный, избыток RPA2 должен быть вымыт из ДНК перед фиксацией клеток. С одной стороны, недоэкстракция приводит к высокому фону из-за белковой фракции RPA2, которая не связана с мцДНК. С другой стороны, чрезмерная экстракция приведет к потере сигнала. Для обнаружения BrdU это не является переменной, поскольку BrdU стабильно встраивается в ДНК и не может быть смыт предварительной экстракцией. Поэтому необходимо тщательно учитывать время предварительной экстракции CSK, количество Triton X-100 в буфере, объем и температуру, при которой выполняется предварительная экстракция. Предварительная экстракция CSK также ограничивает использование размера ядра для различения клеток G0/G1 от клеток S/G2.
Кроме того, мы не можем исключить возможность того, что часть сигнала, поступающего от RPA2, происходит из-за того, что он связан с другими взаимодействующими с хроматин-связывающими белками. Также необходимо учитывать видовую специфичность антител RPA2. Антитела, используемые в этом протоколе, могут распознавать RPA2 человека, мыши, крысы, хомяка и обезьяны. Еще одним ограничением этого подхода является то, что не все клеточные линии могут испытывать нехватку сыворотки для синхронизации G0. Большинство раковых клеточных линий могут обходить контрольные точки клеточного цикла и размножаться даже в среде, лишенной сыворотки. Несмотря на то, что сывороточное голодание полезно, поскольку оно не вызывает повреждения ДНК, необходимо тщательно следить за эффективностью синхронизации клеток, чтобы убедиться, что достигнуто надлежащее обогащение фаз клеточного цикла. Для клеток, которые не реагируют на депривацию сыворотки, необходимо рассмотреть другие методы клеточной синхронизации (например, митотическое встряхивание, ингибирование CDK1 для ареста G2 или неинвазивные методы, такие как центробежная элютриация). Другим возможным методом является использование визуализации с высоким содержанием для измерения содержания EdU и ядерной ДНК для профилирования клеточного цикла асинхронных клеток31. Необходимо учитывать последствия использования альтернативных методов синхронизации, чтобы предотвратить вмешательство в последующий анализ. Например, использование двойного тимидинового блока или афидиколина, часто используемого в литературе, приведет к репликационному стрессуи повреждению ДНК.
Исследование механизмов репарации ДНК продолжает оставаться предметом дискуссий в области рака и клеточной биологии. Представленный здесь протокол предлагает ценный подход к подготовке клеток, позволяющий визуализировать и количественно анализировать ssDNA при воздействии повреждающих ДНК агентов. Примечательно, что этот протокол подчеркивает использование белка, связывающего мцДНК, RPA2, демонстрируя его высокую специфичность для визуализации небольших количеств очагов мцДНК, избегая при этом нежелательной перекрестной реактивности во всех фазах клеточного цикла. Использование RPA2 дает множество преимуществ, особенно для исследователей, стремящихся анализировать клетки в фазе G1 клеточного цикла. Этот протокол учитывает несколько ограничений и решает проблемы, связанные с помехами сигнала, нежелательным фоновым шумом и перекрестной реактивностью при использовании окрашивания RPA2 или BrdU для обнаружения мцДНК.
Disclosures
У авторов нет конкурирующих интересов, которые они могли бы декларировать.
Acknowledgements
Авторы благодарят Микеле Пагано за его поддержку и полезные идеи, Эшли Чуй и Шэрон Кайсари за критическое прочтение рукописи, а также Джеффри Эстрада и Вильму Диас за их постоянную поддержку. Эта работа была поддержана приложением к гранту Национальных институтов здравоохранения GM136250.
Materials
| Name | Company | Catalog Number | Comments |
| Alpha-tubulin antibody | Sigma-Aldrich | T6074 | primary antibody (1:5,000) |
| Axio Observer Inverted Microscope | Zeiss | na | microscope |
| Bis-Tris Plus Mini Protein Gels, 4-12% | Invitrogen | NW04127BOX | Western Blot |
| Bovine Serum Albumin | Jackson ImmunoResearch | 001-000-162 | blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Sigma-Aldrich | B5002-100MG | nucleotide analogue |
| BrdU antibody BU1/75 | Abcam | ab6326 | primary antibody (1:500) |
| CellAdhere Dilution Buffer | Stemcell Technologies | 07183 | coating reagent |
| Click-iT Plus EdU Flow Cytometry Assay Kits | Invitrogen | C10632 | flow cytomery |
| Click-iT Plus EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Fisher Scientific | C10640 | click-reaction kit |
| cOmplete ULTRA Protease inhibitor tablets | Sigma-Aldrich | 5892791001 | reagent |
| Countess 3 Automated cell counter | Thermo Scientific | AMQAX2000 | cell counter |
| Coverslip | neuVitro | GG12PRE | tissue culture |
| Cyclin A2 antibody | Santa Cruz Biotechnology | sc-271682 | primary antibody (1:1,000) for IF and WB |
| Cyclin B1 antibody | Santa Cruz Biotechnology | sc-245 | primary antibody (1:5,000) |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650-100ML | vehicle control |
| DMEM, high glucose, with HEPES | Gibco | 12430051 | cell culture medium for RPE cells |
| DPBS, no calcium, no magnesium | Gibco | 14190144 | the PBS used throughout the protocol |
| D-Sucrose | Thermo Fisher Scientific | bp220-1 | reagent |
| Eclipse Ti2 Series Epifluorescent Microscope | Nikon | na | microscope |
| EdU (5-Ethynyl-2'-deoxyuridine) | Thermo Fisher Scientific | C10637 | nucleotide analog |
| Falcon 24-well plate | Corning | 351147 | tissue culture |
| Falcon Cell Culture Dishes 100 mm | Corning | 353003 | tissue culture |
| Fetal Bovine Serum, heat inactivated | Gibco | 16140071 | media supplement |
| Fiji (ImageJ) | NIH | version 1.54f | software and algorithms |
| FxCycle PI/RNase Staining Solution | Invitrogen | F10797 | PI staining |
| Goat anti-mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 555 | Thermo Fisher Scientific | A21422 | secondary antibody (1:1,000) |
| Goat anti-rat IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A48262 | secondary antibody (1:1,000) |
| Histone H3 antibody | Abcam | ab1791 | primary antibody (1:10,000) |
| hTERT RPE1 | ATCC | CRL-3216 | cell line |
| Hydrochloric acid | Sigma-Aldrich | H1758-100ML | reagent |
| Hydrogen peroxide 30% soultion | Sigma-Aldrich | H1009-100ML | reagent |
| Hydroxyurea,98% powder | Sigma-Aldrich | H8627-5G | reagent |
| Invitrogen Ultra Pure 0.5 M EDTA pH 8.0 | Thermo Fisher Scientific | 15-575-020 | reagent |
| Lipfectamine RNAiMAX Transfection Reagent | Invitrogen | 13778150 | transfection reagent |
| Magnesium chloride solution 1 M | Sigma-Aldrich | M1028-100ML | reagent |
| MycoFluor | Thermo Fisher | M7006 | Mycoplasma Detection Kit |
| Neocarzinostatin from Streptomyces carzinostaticus | Sigma-Aldrich | N9162-100UG | reagent |
| NuPage MES SDS Running Buffer (20x) | Invitrogen | NP0002 | Western Blot |
| onTARGETplus Human RPA2 siRNA | Dharmacon | L-017058-01-0005 | siRNA |
| p27 antibody | BD Biosciences | 610241 | primary antibody (1:1,000) |
| Paraformaldehyde aqueous solution (32%) | Electron Microscopy Sciences | 50-980-494 | fixative |
| PARP1 antibody | Cell Signaling Technology | 9542S | primary antibody (1:1,000) |
| PCNA antibody | Cell Signaling Technology | 13110S | primary antibody (1:2,000) |
| Penicillin-Streptomycin | Gibco | 15140163 | media supplement |
| pH3 antibody | Cell Signaling Technology | 3377S | primary antibody (1:2,000) |
| PhosSTOP phosphatase inhibitor tablets | Sigma-Aldrich | 4906837001 | reagent |
| PIPES Buffer 0.5 M solution, pH 7.0 | Bioworld | 41620034-1 | reagent |
| Precision Plus Protein Kaleidoscope Prestained Protein Standards | Bio-Rad | 1610395 | Western Blot |
| Prism | GraphPad | version 10 | statistical analysis and graph |
| ProLong Diamond Antifade Mountant | Thermo Scientific | P36961 | mounting media |
| Reduced serum media (Opti-MEM) | Gibco | 31985070 | used for transfection |
| Rpa32/rpa2 antibody (mouse) | EMD Millipore | NA19L | primary antibody (1:1,000) for WB |
| Rpa32/rpa2 antibody (rat) | Cell Signaling Technology | 2208S | primary antibody (1:1,000) for IF |
| Sodium Chloride solution (5 M) | Sigma-Aldrich | S5150 | reagent |
| Sodium Pyruvate (100 mM) | Gibco | 11360070 | media supplement |
| Sodium tetraborate decahydrate | Sigma-Aldrich | B3535-500G | reagent |
| Thermo Scientific Pierce DAPI Nuclear Counterstain | Thermo Scientific | 62248 | nucleic acid stain |
| Thymidine,powder | Sigma-Aldrich | T1985-1G | reagent |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | detergent |
| Trypsin-EDTA (0.5%), no phenol red | Gibco | 1540054 | cell dissociation agent |
| Vitronectin XF | Stemcell Technologies | 07180 | coating reagent |
| ZE5 Cell Analyzer | Bio-Rad | na | flow cytomery |
References
- Hakem, R. DNA-damage repair; the good, the bad, and ugly. EMBO J. 27 (4), 589-605 (2008).
- Gutierrez, R., O'Connor, T. R. DNA direct reversal repair and alkylating agent drug resistance. Cancer Drug Resist. 4 (2), 414-423 (2021).
- Krokan, H. E., Bjoras, M. Base excision repair. Cold Spring Harb Perspect Biol. 5 (4), a012583 (2013).
- Marteijn, J. A., Lans, H., Vermeulen, W., Hoeijmakers, J. H. Understanding nucleotide excision repair and its roles in cancer and ageing. Nat Rev Mol Cell Biol. 15 (7), 465-481 (2014).
- Li, G. M. Mechanisms and functions of DNA mismatch repair. Cell Res. 18 (1), 85-98 (2008).
- Hustedt, N., Durocher, D. The control of DNA repair by the cell cycle. Nat Cell Biol. 19 (1), 1-9 (2016).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Bhat, D. S., et al. Therapeutic disruption of RAD52-ssDNA complexation via novel drug-like inhibitors. NAR Cancer. 5 (2), zcad018 (2023).
- Gupta, P., Saha, B., Chattopadhyay, S., Patro, B. S. Pharmacological targeting of differential DNA repair, radio-sensitizes WRN-deficient cancer cells in vitro and in vivo. Biochem Pharmacol. 186, 114450 (2021).
- Pena-Diaz, J., et al. Noncanonical mismatch repair as a source of genomic instability in human cells. Mol Cell. 47 (5), 669-680 (2012).
- Schroering, A. G., Edelbrock, M. A., Richards, T. J., Williams, K. J. The cell cycle and DNA mismatch repair. Exp Cell Res. 313 (2), 292-304 (2007).
- Scully, R., Panday, A., Elango, R., Willis, N. A. DNA double-strand break repair-pathway choice in somatic mammalian cells. Nat Rev Mol Cell Biol. 20 (11), 698-714 (2019).
- Escribano-Diaz, C., et al. A cell cycle-dependent regulatory circuit composed of 53BP1-RIF1 and BRCA1-CtIP controls DNA repair pathway choice. Mol Cell. 49 (5), 872-883 (2013).
- Genschel, J., Modrich, P. Mechanism of 5'-directed excision in human mismatch repair. Mol Cell. 12 (5), 1077-1086 (2003).
- Hu, J., et al. Nucleotide excision repair in human cells: fate of the excised oligonucleotide carrying DNA damage in vivo. J Biol Chem. 288 (29), 20918-20926 (2013).
- Huertas, P., Jackson, S. P. Human CtIP mediates cell cycle control of DNA end resection and double strand break repair. J Biol Chem. 284 (14), 9558-9565 (2009).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in DNA replication with putative roles in cancer. Int J Mol Sci. 20 (1), 74 (2018).
- Symington, L. S. End resection at double-strand breaks: mechanism and regulation. Cold Spring Harb Perspect Biol. 6 (8), a016436 (2014).
- Liu, Y., et al. DNA polymerase beta and flap endonuclease 1 enzymatic specificities sustain DNA synthesis for long patch base excision repair. J Biol Chem. 280 (5), 3665-3674 (2005).
- Wold, M. S., Kelly, T. Purification and characterization of replication protein A, a cellular protein required for in vitro replication of simian virus 40 DNA. Proc Natl Acad Sci U S A. 85 (8), 2523-2527 (1988).
- Wienholz, F., Vermeulen, W., Marteijn, J. A. Amplification of unscheduled DNA synthesis signal enables fluorescence-based single cell quantification of transcription-coupled nucleotide excision repair. Nucleic Acids Res. 45 (9), e68 (2017).
- Wold, M. S. Replication protein A: a heterotrimeric, single-stranded DNA-binding protein required for eukaryotic DNA metabolism. Annu Rev Biochem. 66, 61-92 (1997).
- Chen, R., Wold, M. S. Replication protein A: single-stranded DNA's first responder: dynamic DNA-interactions allow replication protein A to direct single-strand DNA intermediates into different pathways for synthesis or repair. Bioessays. 36 (12), 1156-1161 (2014).
- Kang, Y., et al. Alteration of replication protein A binding mode on single-stranded DNA by NSMF potentiates RPA phosphorylation by ATR kinase. Nucleic Acids Res. 51 (15), 7936-7950 (2023).
- Kilgas, S., Kiltie, A. E., Ramadan, K. Immunofluorescence microscopy-based detection of ssDNA foci by BrdU in mammalian cells. STAR Protoc. 2 (4), 100978 (2021).
- Madabhushi, R., Pan, L., Tsai, L. H. DNA damage and its links to neurodegeneration. Neuron. 83 (2), 266-282 (2014).
- Liboska, R., Ligasova, A., Strunin, D., Rosenberg, I., Koberna, K. Most anti-BrdU antibodies react with 2'-deoxy-5-ethynyluridine -- the method for the effective suppression of this cross-reactivity. PLoS One. 7 (12), e51679 (2012).
- Biehs, R., et al. DNA double-strand break resection occurs during non-homologous end joining in G1 but is distinct from resection during homologous recombination. Mol Cell. 65 (4), 671-684.e5 (2017).
- Cruz-Garcia, A., Lopez-Saavedra, A., Huertas, P. BRCA1 accelerates CtIP-mediated DNA-end resection. Cell Rep. 9 (2), 451-459 (2014).
- Ercilla, A., et al. Physiological tolerance to ssDNA enables strand uncoupling during DNA replication. Cell Rep. 30 (7), 2416-2429.e7 (2020).
- Lezaja, A., et al. RPA shields inherited DNA lesions for post-mitotic DNA synthesis. Nat Commun. 12 (1), 3827 (2021).
- Mukherjee, B., Tomimatsu, N., Burma, S. Immunofluorescence-based methods to monitor DNA end resection. Methods Mol Biol. 1292, 67-75 (2015).
- Ochs, F., et al. 53BP1 fosters fidelity of homology-directed DNA repair. Nat Struct Mol Biol. 23 (8), 714-721 (2016).
- Raderschall, E., Golub, E. I., Haaf, T. Nuclear foci of mammalian recombination proteins are located at single-stranded DNA regions formed after DNA damage. Proc Natl Acad Sci U S A. 96 (5), 1921-1926 (1999).
- Forment, J. V., Walker, R. V., Jackson, S. P. A high-throughput, flow cytometry-based method to quantify DNA-end resection in mammalian cells. Cytometry A. 81 (10), 922-928 (2012).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Sci Rep. 6, 19567 (2016).
- Aten, J. A., Bakker, P. J., Stap, J., Boschman, G. A., Veenhof, C. H. DNA double labelling with IdUrd and CldUrd for spatial and temporal analysis of cell proliferation and DNA replication. Histochem J. 24 (5), 251-259 (1992).
- Podgorny, O., Peunova, N., Park, J. H., Enikolopov, G. Triple S-phase labeling of dividing stem cells. Stem Cell Reports. 10 (2), 615-626 (2018).
- Cappella, P., Gasparri, F., Pulici, M., Moll, J. Cell proliferation method: click chemistry based on BrdU coupling for multiplex antibody staining. Curr Protoc Cytom. Chapter 7, (2008).
- Ligasova, A., Koberna, K. Strengths and weaknesses of cell synchronization protocols based on inhibition of DNA synthesis. Int J Mol Sci. 22 (19), 10759 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved