Abordagem microfluídica para resolver a secreção simultânea e sequencial de citocinas de células polifuncionais individuais
* These authors contributed equally
In This Article
Summary
O protocolo descreve uma plataforma microfluídica avançada para medir quantitativamente a dinâmica da secreção de citocinas de células mononucleares individuais do sangue periférico humano. A plataforma mede até três citocinas em paralelo (IL-6, TNFα e IL-1β) para cada célula individual estimulada com lipopolissacarídeo como exemplo.
Abstract
Infecções, doenças autoimunes, respostas imunológicas desejadas e adversas ao tratamento podem levar a uma resposta complexa e dinâmica de citocinas in vivo. Essa resposta envolve inúmeras células imunes secretando várias citocinas para orquestrar a reação imunológica. No entanto, a dinâmica da secreção, quantidades e co-ocorrência das diferentes citocinas por vários subtipos celulares permanecem pouco compreendidas devido à falta de ferramentas apropriadas para estudá-las. Aqui, descrevemos um protocolo usando uma plataforma de gotículas microfluídicas que permite a medição quantitativa resolvida no tempo da dinâmica da secreção de várias citocinas em paralelo no nível de uma única célula. Isso é possível pelo encapsulamento de células individuais em gotículas microfluídicas, juntamente com um imunoensaio multiplexado para quantificação paralela das concentrações de citocinas, sua imobilização para imagens fluorescentes dinâmicas e a análise das respectivas imagens para derivar quantidades e dinâmicas secretadas. O protocolo descreve a preparação de nanopartículas magnéticas funcionalizadas, experimentos de calibração, preparação celular e encapsulamento das células e nanopartículas em gotículas para imagens fluorescentes e subsequente análise de imagem e dados usando o exemplo de células mononucleares do sangue periférico humano estimuladas por lipopolissacarídeos. A plataforma apresentada identificou um comportamento distinto de secreção de citocinas para células únicas e co-secretoras, caracterizando a heterogeneidade fenotípica esperada na amostra de células medida. Além disso, a natureza modular do ensaio permite sua adaptação e aplicação para estudar uma variedade de proteínas, citocinas e amostras de células, potencialmente levando a uma compreensão mais profunda da interação entre diferentes tipos de células imunes e o papel das diferentes citocinas secretadas dinamicamente para moldar a resposta imune rigidamente regulada. Esses novos insights podem ser particularmente interessantes nos estudos de desregulações imunológicas ou na identificação de populações-alvo em terapia e desenvolvimento de medicamentos.
Introduction
As infecções geralmente causam reações complexas do hospedeiro envolvendo os sistemas imunológicos inato e adaptativo 1,2. Após a infecção ou reconhecimento de agentes infecciosos, as células hospedeiras podem produzir uma gama diversificada de quimio- e citocinas, que são pequenas proteínas conhecidas como comunicadores críticos e moduladores do sistema imunológico3. As citocinas pró-inflamatórias são liberadas logo após a infecção para iniciar a resposta imune, seguidas posteriormente por citocinas anti-inflamatórias, que são essenciais para prevenir danos aos tecidos e doenças crônicas ou autoinflamatórias subsequentes. Esse equilíbrio entre a eliminação da ameaça e a proteção tecidual se manifesta como um amplo repertório de citocinas exercendo diferentes funções durante a infecção, permitindo um ajuste fino da resposta 4,5. Dentro dessa mistura, assinaturas únicas podem ser observadas dependendo do patógeno e dos sinais que induzem, da localização do tecido e das células imunes das quais se originam. No entanto, a liberação de citocinas também parece constituir um processo biológico multifuncional exclusivo para cada população celular, diverso na dinâmica de secreção e resposta individual. Essa heterogeneidade tem sido descrita na literatura há muitos anos, por exemplo, entre subpopulações de células T 6,7, onde investigações sobre doenças autoinflamatórias e infecções graves por COVID-19 exibiram uma grande diversidade funcional de marcadores inflamatórios dentro e entre os pacientes 8,9. Ultimamente, o advento do sequenciamento de célula única destacou a alta plasticidade e a diafonia entre subpopulações dentro de microambientes imunes que não eram aparentes anteriormente, indicando que métodos de célula única são necessários para capturar essa heterogeneidade10,11. Embora novos métodos estejam sendo desenvolvidos para analisar o transcriptoma, a análise fenotípica continua sendo um desafio, pois requer medições simultâneas, quantitativas e resolvidas no tempo da secreção de proteínas em um único nível de célula. Tais medições nos permitem investigar identidades de células secretoras, dinâmica e padrões de secreção (lento / rápido, precoce / tardio, simultâneo / sequencial) para um repertório ou um painel de citocinas. Ao permitir o estudo da dinâmica da liberação de citocinas durante uma resposta imune quantitativamente e com resolução temporal, os insights resultantes podem permitir uma compreensão do conjunto celular e da resposta induzida.
Em protocolos padrão, as citocinas são geralmente detectadas no sobrenadante de suspensões celulares e soro usando ensaio imunoenzimático (ELISA), produzindo quantidades secretadas em massa. As medições em massa não permitem quantificar as quantidades de citocinas produzidas por cada célula, um problema especialmente destacado em amostras de células heterogêneas. Métodos alternativos, como coloração de citocinas intracelulares, ensaio de imunospot ligado a enzima (ELISpot) ou ensaios microgravados (por exemplo, Isoplexis) detectam citocinas expressas por células individuais, mas fornecem apenas medições de desfecho12,13. Isso significa que a dinâmica da secreção e as mudanças que podem ocorrer no padrão de secreção celular ao longo do tempo de incubação são ignoradas. Além disso, as medições de endpoint não podem diferenciar entre secreção de citocinas simultânea e sequencial, portanto, a verdadeira extensão da polifuncionalidade simultânea das células imunes na secreção de citocinas permanece obscura usando esses métodos.
A resolução de célula única pode ser alcançada usando microfluídica de gotículas para gerar e processar compartimentos físicos do tamanho de picolitros, a fim de estudar células imunes em seus fenótipos únicos de secreção de citocinas no nível de célula única14,15. Esses compartimentos consistem em emulsões de água em óleo e podem ser gerados usando chips microfluídicos16,17. De fato, os ensaios microfluídicos baseados em gotículas demonstraram extrema versatilidade ao permitir a análise de diferentes amostras e repertórios biológicos no nível de célula única e sua integração com processos a montante (processamento de células e reagentes) e a jusante (classificação de célula única, proteômica ou sequenciamento) 18 , 19 , 20 , 21 , 22. Em particular, as configurações que permitem a imobilização de gotículas permitem a medição da funcionalidade de uma única célula ao longo do tempo, o que é valioso para a análise da secreção de proteínas18. Além disso, a integração de ensaios quantitativos multiplexados facilita investigações adicionais em dimensões anteriormente inacessíveis, em processos como a co-secreção e a identificação de células imunes polifuncionais23,24.
Neste protocolo, descrevemos um fluxo de trabalho de célula única baseado em gotículas imobilizadas para detectar, quantificar e medir temporariamente a secreção de até três citocinas em paralelo a partir de células individuais17,23. A tecnologia oferece a capacidade de monitorar as respostas de citocinas de mais de 20.000 células em paralelo.
O fluxo de trabalho apresentado consiste no encapsulamento microfluídico de células imunes únicas e nanopartículas funcionalizadas em gotículas de água em óleo de 60 pL. A imobilização de >100.000 gotículas em uma câmara de observação e a microscopia de fluorescência resolvida no tempo permitem a medição da dinâmica da secreção de citocinas dentro de cada gotícula e cada citocina (Figura 1A). Para cada célula individual dentro de uma gotícula, a secreção de citocinas é medida por um imunoensaio sanduíche, onde nanopartículas magnéticas funcionalizadas com um anticorpo de captura específico se ligam à citocina secretada, levando à subsequente realocação e ligação de anticorpos de detecção marcados com fluorescência ( Figura 1B , C ). Um beadline é formado pelo alinhamento das nanopartículas magnéticas, para as quais a realocação de fluorescência pode ser quantificada na presença de citocina. Aqui, a realocação de fluorescência é definida como a intensidade média de fluorescência encontrada na linha do grânulo dividida pela intensidade média de fluorescência da gota restante. Este ensaio pode ser multiplexado para várias citocinas, misturando lotes de nanopartículas funcionalizados de forma diferente e respectivos anticorpos de detecção marcados em diferentes canais de fluorescência23, resultando em realocações de fluorescência específicas nos diferentes canais. Com a ajuda de um script de análise personalizado, os valores de realocação de fluorescência podem ser extraídos e as imagens podem ser convertidas em perfis dinâmicos de secreção para cada célula e citocina individual. Portanto, os conjuntos de dados resultantes produzem inúmeras leituras, como a medição quantitativa da secreção ao longo do tempo, a identificação de subpopulações co-secretoras e as distribuições das células de acordo com quantidades, taxas e combinações secretadas de citocinas.
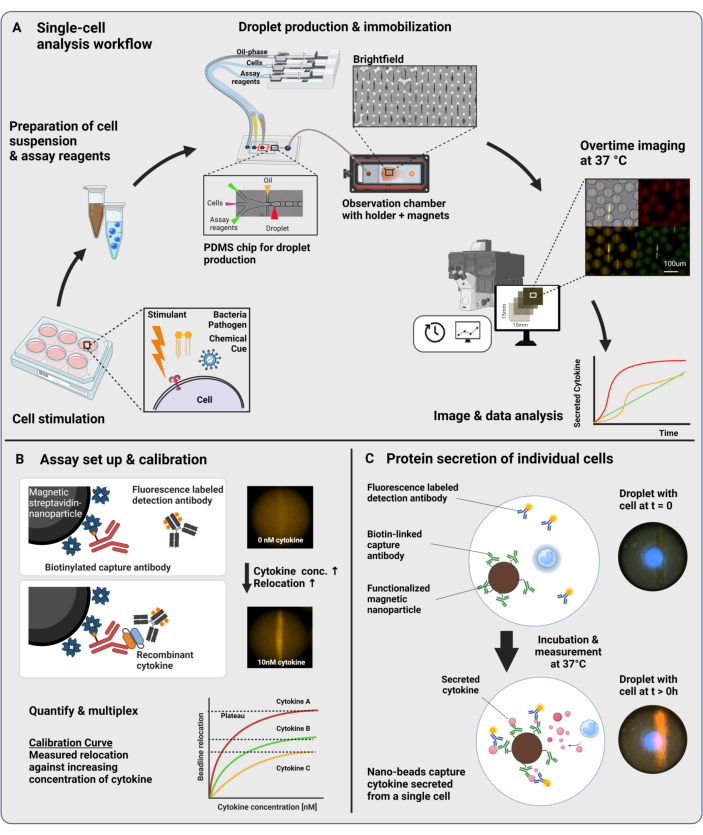
Figura 1: Fluxo de trabalho e princípio do ensaio. (A) Visão geral do fluxo de trabalho para analisar células secretoras de citocinas após a estimulação. Suspensões unicelulares e nanopartículas magnéticas são preparadas e encapsuladas em emulsões de óleo/água de 60 pL em volume (gotículas). As gotículas são imobilizadas e as nanopartículas alinhadas dentro de um campo magnético antes da medição por até 4 h a cada 30 min. Finalmente, as imagens são analisadas e os parâmetros para cada gota, ponto de tempo e canal fluorescente são extraídos. Este número foi modificado de17. (B) Princípio do bioensaio em sanduíche de gotículas. As nanopartículas funcionalizadas se ligam às citocinas secretadas, o que leva à subsequente realocação de anticorpos de detecção marcados com fluorescência para as nanopartículas. Essa realocação da fluorescência é quantificada e validada com experimentos de calibração realizados com citocinas recombinantes. A mistura de diferentes nanopartículas funcionalizadas permite a medição multiplexada de até três citocinas simultaneamente. (C) Em experimentos baseados em células, as gotículas são seguidas durante o tempo de medição e as células secretoras são identificadas por um aumento ao longo do tempo da realocação de fluorescência nas nanopartículas. Os esquemas não estão à altura. Figura criada com BioRender.com. Clique aqui para ver uma versão maior desta figura.
Protocol
Todos os experimentos foram realizados sob o acordo de ética EK202-N-56 e aprovados pela comissão de ética da ETH Zurich. O manuseio das células humanas foi realizado em um gabinete de fluxo laminar contido em um laboratório de nível de biossegurança 2.
NOTA: As seções a seguir detalham o protocolo para medir a secreção de citocinas resolvida no tempo em um nível de célula única. O procedimento descrito aqui é aplicado à estimulação de células mononucleares do sangue periférico (PBMC) com lipopolissacarídeo (LPS) e à medição paralela das citocinas IL-6, TNFα e IL-1β. No entanto, se necessário, o protocolo pode ser adaptado a outros tipos de células, estimulantes e citocinas.
1. Fabricação da câmara de observação
NOTA: Para evitar o movimento das gotículas durante a imagem, uma câmara de observação é preparada com uma altura cerca de 10% menor que o diâmetro da gota.
- Preparação da fita dupla face de corte e da lâmina de vidro superior
- Desenhe ou carregue o desenho desejado do recorte da câmara na guia Design do software de corte. Para as dimensões específicas usadas aqui, consulte a Figura 2E.
- Fixe a fita adesiva dupla face de 32 μm de espessura na fita adesiva com fita adesiva e coloque a fita adesiva na máquina de corte automática.
- Corte o desenho da câmara da fita, prestando atenção ao corte das bordas longas da câmara na mesma direção para facilitar o desprendimento na etapa 1.3.
- Armazene os recortes de fita em temperatura ambiente para armazenamento a longo prazo. Para armazenamento de curto prazo, armazene-os a -20 °C e remova-os apenas pouco antes da etapa 1.3. para facilitar o manuseio.
- Faça dois furos de cerca de 1 mm de diâmetro no meio de uma lâmina de microscópio padrão (76 mm x 26 mm x 1 mm), com a distância entre os dois furos de cerca de 3,5 cm.
- Limpeza e ativação por plasma de lâminas de vidro
- Limpe uma lâmina de vidro com furos e outra sem furos usando sabão. Enxágue bem com água destilada e seque com lenços de precisão sem fiapos.
- Coloque as lâminas de vidro em um limpador de plasma e trate as superfícies superiores a 55 W por 10 min. Remova as lâminas de vidro e prossiga para a etapa 1.3.
- Montagem da câmara
- Coloque a lâmina de vidro com orifícios em uma superfície limpa com o lado ativado por plasma voltado para cima, sem tocar na superfície ativada.
- Remova a camada protetora de um lado da fita adesiva dupla face, na mesma direção do corte. Sem tocar, alinhe o recorte da fita com as bordas da lâmina de vidro e os orifícios perfurados e coloque lentamente a fita em contato com a lâmina de vidro a partir da borda curta.
NOTA: Preste atenção para não gerar alongamentos ou dobras na fita, pois isso resultará em alturas incorretas da câmara. Como esta etapa é propensa a erros e requer alguma experiência prática, é aconselhável preparar várias lâminas de vidro em paralelo. - Remova a segunda camada protetora da fita, novamente na direção do corte, e coloque a segunda lâmina de vidro sem furos com a superfície ativada voltada para baixo. Pressione toda a superfície das duas lâminas de vidro juntas, colocando uma tábua plana em cima e pressionando para baixo com força na parte superior do corpo por cerca de 10 s.
- Depois de montar as duas lâminas de vidro, vire a câmara para que os dois orifícios fiquem voltados para você. Cole as nanoportas nos dois orifícios colocando uma pequena quantidade de cola curável por UV no anel abaixo da porta e colocando a porta em cima do orifício na lâmina de vidro. Adicione um anel de cola curável por UV ao redor da porta e cure a cola com uma lâmpada UV. A câmara agora deve ter a aparência representada na Figura 2E. Prossiga imediatamente para a etapa 1.4.
NOTA: A luz UV pode danificar os olhos e a pele. Use equipamento de proteção adequado.
- Revestimento fluorofílico da superfície da câmara
NOTA: Esta etapa deve ocorrer dentro de 1 h após o tratamento com plasma das lâminas de vidro (etapa 1.2.2) para garantir uma boa eficiência do revestimento.- Prepare 1 mL de solução de fluorosilano a 1% (1H, 1H, 2H, 2H-perfluorodeciltriclorossilano) em óleo fluorado (HFE-7500) e encha-o com uma seringa. Empurre a solução de revestimento através de um filtro de seringa de PTFE e uma agulha de 27G x 0.75 polegadas conectada a um microtubo de PTFE de 0.3 mm x 0.76 mm para a câmara de observação.
- Após 1 min de incubação, lave a solução de revestimento para fora da câmara usando pressão de nitrogênio sob uma capela de exaustão. Enxágue a câmara com óleo fluorado (somente HFE-7500) usando outro conjunto de seringa.
- Armazene a câmara cheia de óleo fluorado com entradas fechadas à temperatura ambiente (RT). Após cada experimento, lave as células e gotículas diretamente para garantir uma boa preservação do revestimento.
NOTA: O protocolo pode ser pausado aqui e as câmaras podem ser armazenadas e reutilizadas por vários meses.
- Suporte de câmara com ímãs
- Para o alinhamento das nanopartículas magnéticas, aplique um campo magnético estático à câmara de observação durante o encapsulamento de gotículas e a imagem. Para isso, coloque a câmara em um suporte de microscopia impresso em 3D personalizado (consulte a Figura 2D e o arquivo encontrado em Bounab et al.17 Dados Suplementares 4) que contém dois ímãs de neodímio ao longo dos lados longos da câmara.
2. Funcionalização de nanopartículas
NOTA: O processo de funcionalização das nanopartículas é semelhante para cada citocina, sendo a única diferença a adição de anticorpos de captura específicos para citocinas. A funcionalização de cada citocina é realizada em diferentes tubos de reação individuais em paralelo. Antes deste protocolo, o anticorpo de captura de TNFα e o anticorpo de detecção de IL-1β eram marcados internamente com biotina e Alexa Fluor 647, respectivamente. A conjugação foi realizada de acordo com o protocolo do fabricante encontrado no site do fornecedor (ver links na Tabela de Materiais) e os anticorpos foram aliquotados e armazenados a -20 °C.
- Adicione 50 μL de nanopartículas funcionalizadas com estreptavidina (diâmetro (Ø) 300 nm) no tubo destinado à detecção de TNFα, 50 μL para IL-1β e 100 μL para IL-6. Diluir a solução de nanopartículas 1:1 (v/v) em solução salina tamponada com fosfato (PBS).
- Adicionar a cada tubo 1/20 (v/v) do respectivo volume dos anticorpos de captura biotinilados (concentrações de reserva a 0,5 mg/ml) e incubar durante 30 min a RT.
NOTA: Ao adicionar pequenos volumes à solução de nanopartículas, deposite o volume na parte superior do tubo e lave-o várias vezes com a maior parte da solução. Isso garante uma mistura adequada e evita a formação de agregados. - Adicione 1/100 (v / v) de solução de D-biotina 1 mM ao tubo e incube por 5 min em RT. Isso resulta em uma concentração final de biotina de 10 μM.
NOTA: O excesso de biotina bloqueia os lados de ligação livres nas nanopartículas e reduz a formação de agregados indesejados. - Colete as partículas segurando um ímã de neodímio próximo ao tubo. Espere até que o sobrenadante esteja claro e descarte o sobrenadante.
NOTA: Os ímãs usados em todo o ensaio exibem forças atrativas muito fortes, que podem causar danos físicos se dois ímãs se encaixarem acidentalmente. - Para reduzir a adsorção não específica na superfície da nanopartícula, ressuspenda imediatamente as nanopartículas em 0,5x o volume final da etapa 2.1 do Pluronic F-127 (10%) e 0,5x o volume PBS. Incubar a solução durante 30 min em RT.
- Colete as partículas usando o ímã, descarte o sobrenadante e ressuspenda em 1x o volume do tampão de armazenamento (RPMI 1640, reposição de soro knockout a 5%, 1% de caneta / estreptococos, albumina sérica humana recombinante a 1% (HSA), HEPES 25 mM, 0,1% Pluronic F-127). Incubar a solução durante 30 min em RT.
NOTA: O protocolo pode ser pausado aqui e as partículas agora podem ser armazenadas por até 1 semana a 4 °C. - Imediatamente antes do encapsulamento, ressuspenda as partículas pipetando e misture as nanopartículas conjugadas na proporção de 2:1:1 (v/v) para IL-6:TNFα:IL-1β, respectivamente.
NOTA: As diferentes proporções de nanopartículas funcionalizadas dependem do par de anticorpos usado para cada citocina e foram determinadas experimentalmente por meio de amostras de calibração para obter uma faixa dinâmica ideal. - Lavar com meios completos (RPMI 1640, 10% FBS, 1% Pen/Strep, 25 mM HEPES) recolhendo as partículas com o íman, descartando o sobrenadante e ressuspendendo. Repita esta etapa, mas ressuspenda apenas em 0,5x o volume da mídia completa da etapa 2.7.
- Adicione os anticorpos de detecção de IL-6, TNFα e IL-1β marcados de forma diferente à solução para atingir uma concentração final de 10 nM cada. A solução agora está pronta para ser usada nos experimentos com gotículas.
3. Preparação celular
NOTA: PBMC foram isolados de um casaco amarelado recebido do banco de sangue de Zurique. As células foram congeladas e armazenadas em criogeniais (1 x 107 células/frasco) em nitrogênio líquido por vários meses.
- Descongelamento celular
- 1 h antes de iniciar o experimento, deixe o meio completo e o tampão MACS (EDTA 2 mM, BSA 0,5% em DPBS, filtrado estéril) para aquecer em RT. Prepare o tubo contendo as células adicionando 9 mL de meio completo em um tubo de 15 mL e mantendo-o em banho-maria a 37 ° C.
- Recupere um PBMC criovial (contendo ~ 1 x 107 células) de seu armazenamento em nitrogênio líquido. Agite o criotubo no banho-maria a 37 °C até que reste apenas uma pequena quantidade de gelo.
- Limpe o tubo com 70% de EtOH e transfira-o para o gabinete de fluxo laminar. Adicione 1 mL de meio completo pré-aquecido ao criogenial, misture delicadamente e transfira todas as células para o tubo contendo o meio completo quente. O criogenio pode ser lavado com 1 mL de meio completo quente para recuperar o número máximo de células.
- Girar as células a 500 x g durante 5 min à RT, rejeitar o sobrenadante e ressuspender suavemente com uma pipeta o sedimento celular com 1 ml de meio completo. Adicione 9 mL de meio completo.
- Gire as células a 500 x g por 5 min em RT. Descarte o sobrenadante e ressuspenda como anteriormente em 1 mL de meio completo.
- Conte as células usando o contador de células disponível. Nesse caso, foi utilizado um contador de células automatizado. As células foram contadas misturando 10 μL de suspensão celular com 10 μL de azul de tripano e transferindo 10 μL da mistura para a lâmina de contagem de células.
- Coloração e bloqueio de FcR
- Calcule o número total de células e o volume necessário para ressuspender as células em 2 x 106 células vivas / mL. Prepare a solução de coloração celular (CellTrace Violet) diluindo o estoque (5mM) 1000x em PBS (concentração de trabalho de 5 μM).
- Girar as células a 500 x g durante 5 minutos à RT. Rejeitar o sobrenadante e ressuspender as células no volume calculado da solução de coloração celular preparado no passo 3.2.1. Incubar as células a 37 °C durante 5 min.
- No final da incubação, tempere o corante restante na solução adicionando meio completo (pelo menos 2x o volume da solução de corante). Gire as células a 500 x g por 5 min em RT.
- Descarte o sobrenadante, ressuspenda o pellet celular em 60 μL de tampão MACS e adicione 20 μL de bloco FcR humano por 1 x 107 células. Incube as células por 10 min em RT.
- Encha o tubo até 10 mL com tampão MACS e gire as células a 500 x g por 5 min em RT.
- Rejeitar o sobrenadante e ressuspender as células em 1 ml de meio completo Contar as células conforme descrito no passo 3.1.6.
- Estimulação celular com LPS
- Usando a contagem de células, dilua as células a 1 x 106 células/mL e transfira 2 mL de células para cada poço em uma placa de 6 poços de ligação ultrabaixa.
- Diluir o LPS em meio completo e adicioná-lo ao poço que contém as células para uma concentração final de LPS de 1 μg/ml. Incubar as células durante 6 h a 37 °C.
- Preparação para encapsulamento
- No final do tempo de estimulação, transfira a suspensão celular para um novo tubo de 15 ml.
- Adicione 1 mL de meio completo no poço vazio. Com um raspador de células, desconecte as células restantes. Transfira as células para um novo tubo de 15 ml. Lave o poço com 1 mL de meio completo e transfira para outro tubo de 15 mL.
- Girar os dois tubos a 500 x g durante 5 min em RT e transferir 1 ml da solução sobrenadante não diluída (do primeiro tubo que contém as células não lavadas) para um novo tubo para análise posterior, se necessário (por exemplo, ELISA).
- Descarte o resto dos sobrenadantes.
- Ressuspenda os pellets em 0,5 mL de meio completo, combine as células do mesmo poço e transfira-as para um tubo de centrífuga. Conte as células conforme descrito na etapa 3.1.6.
- Gire as células a 500 x g por 5 min em RT e descarte a maior parte do sobrenadante (deixando aproximadamente 100 μL). Sem ressuspender o pellet, adicione com muito cuidado 200 μL de meio completo.
- Descarte o sobrenadante. Ressuspenda as células em meio completo a uma concentração de 6,6 a 13,3 x 106 células/mL para atingir um número médio de células por gotícula de λ = 0,2-0,4 para encapsulamento, conforme definido na etapa 8.6.
NOTA: As etapas 3.4.6 e 3.4.7 devem ser executadas imediatamente antes do encapsulamento para evitar a secreção de citocinas no sobrenadante. O número de células por gota segue uma distribuição de Poisson: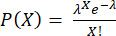 , onde P mostra a fração de gotículas contendo células X e λ é o número médio de células por gota.
, onde P mostra a fração de gotículas contendo células X e λ é o número médio de células por gota.
4. Encapsulamento e produção de gotículas
NOTA: O encapsulamento de células em gotículas é possibilitado por um chip gerador de gotículas microfluídicas, para o qual a fabricação é descrita em detalhes em outro lugar17. Alternativas estão disponíveis comercialmente (veja o exemplo na Tabela de Materiais). Um projeto de chip gerador de gotículas adequado tem duas entradas para fases aquosas, uma entrada para a fase oleosa e uma saída para as gotículas geradas. Além disso, um chip gerador de gotículas comercial adequado deve permitir a produção de água em gotículas de óleo fluorado de 40-60 pL de volume. O protocolo aqui descrito resulta em emulsões água/óleo (gotículas) com um diâmetro de 50 μm. O uso de várias opções para alterar o protocolo pode resultar em gotículas maiores ou menores.
- Preparação da bomba de seringa (Figura 2A)
- Encha uma seringa de 1 mL com 500 μL de fase contínua consistindo de 2% de fluorosurfactante 008 em óleo fluorado HFE-7500. Conecte uma agulha de 27G x 0.75 polegadas a um microtubo de PTFE de 0.30 mm x 0.76 mm e monte o conjunto na seringa e, posteriormente, na bomba da seringa.
NOTA: Certifique-se de que não permanece ar na seringa ou cânula, pois isso evita taxas de fluxo consistentes. - Prepare dois conectores de ponta de pipeta feitos sob medida para as fases aquosas (Figura 2B): Faça um furo com um punção de biópsia de Ø0,75 mm no meio de um recorte PDMS de ~ 5 mm de altura com Ø6 mm. Puxe ~3 cm de tubo de PTFE (0.56 mm de diâmetro interno, 1.07 mm de diâmetro externo) através do orifício no recorte PDMS e empurre o conjunto para dentro de uma ponta de pipeta de 200 μL. Conecte o outro lado do tubo a uma agulha de 23Gx 1.25 polegadas. Sele o conector espalhando cola curável por UV na parte superior da pipeta e cure-o com luz UV.
NOTA: Como a luz UV é prejudicial aos olhos, use óculos de proteção com proteção UV. - Encha duas seringas de 1 mL com 500 μL de óleo mineral leve, conecte duas agulhas 23G com os acessórios feitos sob medida e monte ambas na bomba da seringa.
- Aspire 30 μL de nanopartículas e 30 μL de solução celular nas pontas da pipeta das fases aquosas usando o software de controle da bomba de seringa.
- Prepare uma câmara de observação limpando a superfície com água para remover sujeira e poeira e seque-a com lenços de precisão. Prenda a câmara no suporte da câmara impresso equipado com dois ímãs de neodímio.
NOTA: Certifique-se de que os ímãs apontem na direção certa (atraindo uns aos outros) para formar um agregado alongado. - Incline ligeiramente a câmara (30°). Abra ambas as portas e conecte uma toalha de papel na porta superior para absorver o excesso de fase externa durante o enchimento.
- Encha uma seringa de 1 mL com 500 μL de fase contínua consistindo de 2% de fluorosurfactante 008 em óleo fluorado HFE-7500. Conecte uma agulha de 27G x 0.75 polegadas a um microtubo de PTFE de 0.30 mm x 0.76 mm e monte o conjunto na seringa e, posteriormente, na bomba da seringa.
- Produção de gotículas e enchimento da câmara
- Conecte a fase contínua via tubulação à entrada superior do chip microfluídico (Figura 2A, C, F). Lave o cavaco por cerca de 30 s com fase contínua usando uma vazão de 1800 μL/h.
- Conecte as pontas da pipeta das soluções aquosas às duas entradas intermediárias (Figura 2A, C, F).
- Inicie o fluxo da solução aquosa com 200 μL / h cada e deixe os canais e a saída serem preenchidos com líquido. Ao usar nanopartículas magnéticas, uma solução homogênea de cor marrom-avermelhada deve fluir para fora da saída do chip.
- Assim que o líquido aparecer na saída, inicie o fluxo da fase oleosa fluorada a 800 μL / h e aguarde até que uma produção de gotículas estável seja estabelecida, confirmada pela saída de uma solução homogênea, cinza e brilhante na saída.
- Uma vez estabelecida uma produção estável de gotículas, colete as gotículas produzidas conectando microtubos de PTFE (0,3 mm de diâmetro interno x 0,76 mm de diâmetro externo) à porta de saída e direcione-as para uma câmara de observação passando o microtubo através do módulo de ponteira de um encaixe de peça única apertado com os dedos (Figura 2A).
- Se ocorrer a produção adequada de gotículas, um líquido homogêneo e brilhante deve encher a câmara com uma frente reta de baixo para cima.
- Assim que a câmara estiver cheia, pare o fluxo e feche as portas com plugues de porta usando pressão apertada.
NOTA: Preste atenção para não fechar a câmara com muita força. O aprisionamento ou influxo de ar pode levar a movimentos das gotículas e, assim, comprometer o rastreamento durante a medição. - Após a produção de gotículas, lave o chip com óleo fluorado e sopre todos os lembretes de fluido com nitrogênio para preservar sua função. Os chips podem ser reutilizados várias vezes e armazenados por meses, desde que não estejam entupidos.
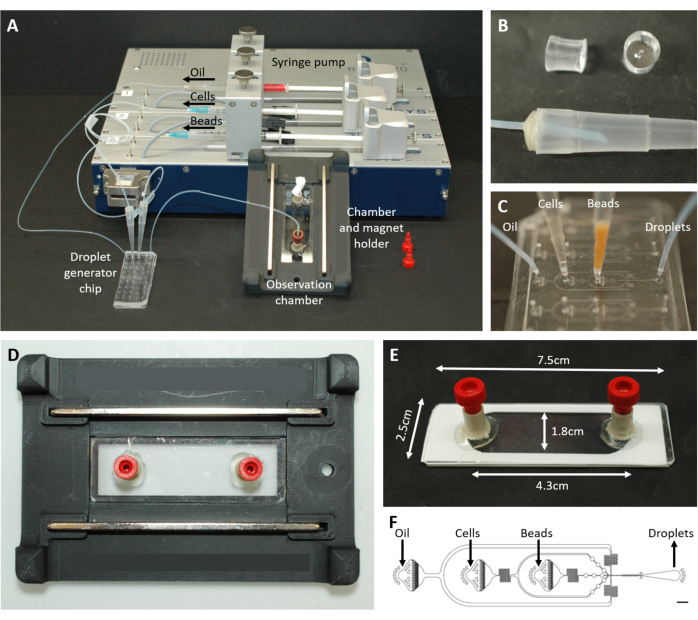
Figura 2: Visão geral da configuração microfluídica. (A) Configuração para encapsulamento de gotículas com a bomba de seringa, o chip de geração de gotículas e a câmara de observação e o suporte do microscópio. (B) Imagem do plugue PDMS perfurado (superior) para formar um conector para uma ponta de pipeta de 200 μL (inferior), conforme descrito na etapa 4.1.2 do protocolo. (C) Imagens da conexão das pontas da pipeta e da tubulação ao chip de geração de gotículas. (D) Imagem da câmara colocada dentro do suporte de microscópio impresso em 3D personalizado com dois ímãs na parte superior e inferior. (E) Foto da câmara de observação (com fita branca para ilustração). (F) Layout do chip microfluídico para criação de gotículas (barra de escala: 750 μm). Este número foi modificado de17. Clique aqui para ver uma versão maior desta figura.
5. Aquisição e medição de imagens
NOTA: A aquisição da imagem é realizada em um microscópio de epifluorescência invertido padrão fechado em uma incubadora, permitindo medições a 37 °C. As configurações aqui descritas são específicas para um microscópio Nikon Eclipse Ti2 rodando com o software NIS Elements (V. 5.30.04) equipado com uma câmera Orca Fusion, mas geralmente são adaptáveis a qualquer outro microscópio e câmera de fluorescência.
- Configurando parâmetros de medição
- Para definir o tamanho da imagem, selecione um tamanho de matriz de 10 x 10 imagens. Esta matriz conterá cerca de 50.000-70.000 gotículas. Use 1% de sobreposição e ative a mesclagem para costura de imagem.
- Para definir o número de canais medidos, selecione o canal DAPI para detecção de células, FITC, TRITC, canais Cy5 para detecção de citocinas (linhas de contas) e o canal BF para detecção de gotículas. Use o compartimento de pixels 2 x 2 e 16 bits para profundidade de bits. Adapte as configurações da câmera para obter valores de pixel de intensidade na gota que não atingem o máximo da câmera para cada canal de fluorescência.
NOTA: Os tempos de exposição exatos e as intensidades da lâmpada para cada canal dependem do modelo e dos reagentes usados e são estabelecidos antes de gerar curvas de calibração (etapa 7). Usar as mesmas configurações de aquisição na calibração e medições de células é importante para uma quantificação precisa. - Para configurar a medição resolvida no tempo, selecione uma medição a cada 30 minutos para 9 medições no total.
NOTA: Os parâmetros de medição podem diferir para células usadas, estimulantes, reagentes, citocinas medidas, temperatura de incubação e modelos de microscópio.
- Iniciando a medição
- Monte o suporte da câmara no microscópio com uma platina de formato de placa de poço (Figura 2D) e mude para o canal de campo claro (BF) usando a objetiva de 10x.
- Concentre-se nas gotículas imobilizadas no BF e certifique-se de que o conjunto esteja montado em um plano perfeito, girando e ajustando, se necessário. Vá para o meio da câmara para as etapas subsequentes.
- Ative o sistema de foco automatizado (PFS) e defina-o para o plano de medição ideal no canal BF para que as bordas das gotículas apareçam como círculos pretos e nítidos que podem ser facilmente distinguidos da fase oleosa e do fundo.
NOTA: As medições também são possíveis sem um sistema de foco automatizado, mas se o microscópio estiver equipado com um, é altamente recomendável usá-lo. Isso melhora a qualidade da medição para imagens grandes e muito costuradas. - Percorra todos os canais de fluorescência e defina o plano de medição ideal para cada um. Para medições de realocação nos canais FITC, TRITC e Cy5, certifique-se de que o agregado de nanopartículas esteja em foco perfeito, para o canal DAPI, certifique-se de que as células estejam em foco.
NOTA: Os planos focais ideais e os valores z podem diferir para todos os canais medidos. Certifique-se de salvar deslocamentos PFS individuais para cada canal. - Antes de iniciar a medição, percorra todos os canais para verificar novamente os focos individuais e aguarde 5 minutos para equilibrar, pois o movimento pode ocorrer inicialmente enquanto as soluções aquecem.
- Inicie a medição. Depois de gerar a primeira imagem, verifique se há irregularidades (foco, gotículas em movimento, canais errados, etc.). Reinicie a aquisição se necessário ou, no caso de ar, reabasteça a câmara (comece na etapa 4.1.4). Deixe o conjunto para obter imagens das gotículas por mais de 4 h.
6. Análise de imagem
- Instale o software de análise de imagem (DropMap Analyzer App v 4.023) no MatLab (https://github.com/ESPCI-LCMD/MiMB) e transfira o arquivo .nd2 gerado do experimento para um computador de análise.
- Abra o aplicativo. Selecione as configurações especificadas, caso contrário, deixe o valor padrão: CH1: DAPI, WD (gota inteira) selecionado; CH2: FITC, BL (beadline) selecionado; CH3: TRITC, BL selecionado; CH4: Cy5, BL selecionado; Diâmetro máximo de queda (μm): 70; Detecção de queda: Completo; Rastreamento: Sim. Pressione o botão Iniciar (ícone de fruta) para selecionar o local do arquivo .nd2 e iniciar a análise.
- Após alguns minutos, o programa mostrará uma seção de exemplo da imagem (Figura 3A). Pressione Espaço até encontrar um adequado para detecção de gotículas e pressione Enter. Na mesma seção da imagem, desenhe um retângulo em uma área representativa para encontrar parâmetros de limite para detectar gotículas.
- Após alguns minutos, outra janela será aberta mostrando a distribuição de intensidade do canal DAPI. Arraste e solte o controle deslizante para detectar apenas o sinal das células coradas e clique em Concluído no canto superior direito.
- Depois de segmentar a imagem em gotículas únicas com diâmetros menores que o diâmetro máximo da gota (μm), o programa agora executará as seguintes etapas para cada gota, ponto de tempo e canal de fluorescência sem mais entrada do usuário (consulte a Figura 3).
- O software calcula os pixels movidos da gota entre os pontos de tempo (gotículas que se movem mais de 40 pixels são excluídas automaticamente).
- O software mede o valor médio de fluorescência de toda a gota, detectando e medindo a intensidade média da linha do grânulo encontrando o pixel mais brilhante em uma linha horizontal e calculando a média de todas as intensidades de pixel em uma linha vertical de cima para baixo da gota. Isso é feito automaticamente e é usado para calcular os valores médios de realocação da linha de contas (Figura 3B) de acordo com a equação:
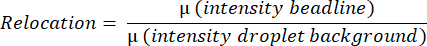
- O software calcula a porcentagem do total de pixels na área de gotículas acima do limite definido no canal DAPI.
- O arquivo .xslx resultante conterá as seguintes colunas de interesse para análise posterior: DropIdX (ID da gota rastreada ao longo do tempo), TrueCentroid_ t*2-1 e t+2 (coordenadas x e y, respectivamente, do centro da gota para o ponto de tempo t), DiameterMicrons (diâmetro da gota em μm), TrackingMove (número de pixels movidos durante todo o tempo de medição) FluoChannel_BL_Ratio_t (valor de realocação para FluoChannel no ponto de tempo t), DAPI_WD_PosPxlCount_t (número de pixels acima do limite em toda a gota no canal DAPI no ponto de tempo t).
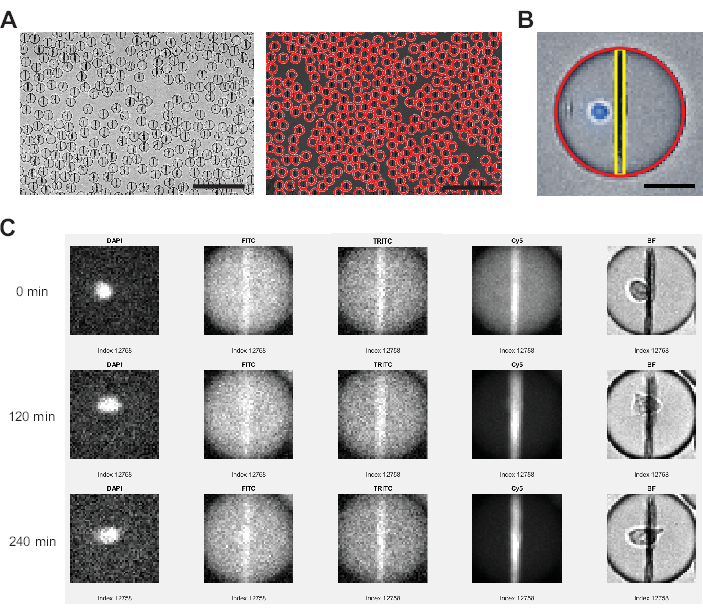
Figura 3: Análise de imagem realizada pelo software de análise de imagem. (A) As gotículas são detectadas no canal de campo claro (BF) usando uma transformação de Hough, marcando cada gota com um círculo vermelho. Barras de escala: 200 μm. (B) Dentro de cada gota, a linha de contas da nanopartícula é identificada através dos pixels mais brilhantes no plano horizontal e as intensidades de fluorescência médias para todos os pixels que abrangem de cima para baixo da gota. Além disso, a célula é identificada por meio de uma porcentagem de pixels >0 acima do limite para toda a área da gota. Barra de escala: 20 μm. (C) O software do analisador compara a intensidade de fluorescência nas nanopartículas com o fundo da gota para os canais FITC, TRITC e Cy5 em todos os pontos de tempo medidos para cada gota individual. São mostrados os pontos de tempo 0, 4 (120 min) e 9 (240 min). Para verificar manualmente a detecção correta de gotículas e células, os canais DAPI e BF também são exibidos. Clique aqui para ver uma versão maior desta figura.
7. Calibração
NOTA: Para uma leitura quantitativa, a calibração das concentrações de citocinas para valores de realocação de fluorescência precisa ser realizada uma vez, pois podem ocorrer diferenças entre diferentes configurações experimentais. Todas as etapas necessárias são detalhadas nas seções de protocolo anteriores, conforme referenciado.
- Prepare as nanopartículas conforme descrito na etapa 2.
- Reconstituir as proteínas recombinantes humanas de IL-6, TNFα e IL-1β de acordo com as instruções do fabricante.
NOTA: Certifique-se de que as alíquotas congeladas sejam descongeladas apenas uma vez e usadas prontamente. - Prepare uma série de diluição de 2 vezes para todas as três proteínas juntas usando meio completo (10% FBS, 1% Pen / Strep, 25 mM HEPES) com uma concentração inicial de 80 nM até 0,625 nM.
- Realize o encapsulamento conforme descrito na etapa 4 com nanopartículas funcionalizadas na primeira e RPMI apenas na segunda fase aquosa. Essa medida serve como o espaço em branco e o desvio padrão medido é usado para análise de dados posteriormente.
- Aguarde 5 minutos e crie uma imagem das gotículas conforme descrito na etapa 5. Tire 3 imagens com um tamanho de matriz de 2 x 2 nos respectivos canais de fluorescência.
- Repita as etapas 7.4 e 7.5 com todas as soluções de calibração preparadas, começando com a concentração mais baixa e terminando com a concentração mais alta.
- Analise as imagens conforme descrito na etapa 6. Não use a opção WD para o canal DAPI e defina Rastreamento como Não.
- A análise gera valores de realocação de fluorescência para cada gota medida em uma imagem. Extraia a mediana e o desvio padrão para cada canal de fluorescência. Calcule a média da mediana e do desvio padrão para cada imagem medida por concentração.
- Gere uma curva de calibração traçando a realocação mediana média em relação às concentrações medidas de cada proteína recombinante.
- Ajuste as curvas usando uma associação de uma fase:
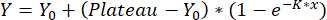 ,
,
com Y = relocalização em x, Y0 = relocalização da medição em branco e x a concentração utilizada. A curva de calibração obtida é usada para quantificar os valores de realocação conforme descrito na etapa 8.
NOTA: Ajuste apenas os valores até a realocação medida mais alta e exclua os valores de concentrações mais altas com realocação medida mais baixa. Espera-se uma diminuição nos valores de realocação medidos em concentrações mais altas e ocorre devido ao efeito Hook e à capacidade limitada de ligação das nanopartículas.
8. Análise dos dados
- Exclua gotículas com um valor TrackingMove superior a 10, ou seja, que se moveram mais de 10 pixels ao longo do curso de tempo da medição.
- Identifique gotículas contendo células coradas (canal DAPI) no primeiro ponto de tempo, classificando gotículas com valores superiores a 0 na coluna DAPI_WD_PosPxlPercent_1.
- Identifique gotículas contendo células secretoras aplicando os 3 critérios a seguir para a realocação de fluorescência de cada canal de fluorescência (FluoChannel_BL_Ratio_t colunas).
- Identificação de gotículas com valores crescentes de realocação, classificando uma inclinação positiva ao longo do tempo de medição.
- Identificação de gotículas com valores de realocação atingindo o limite de detecção (LOD). Uma gota é selecionada quando a realocação máxima de fluorescência ao longo do tempo de medição é superior ao LOD, calculado conforme descrito em outro lugar25:
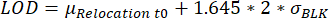 , onde μRealocação t0 é a mediana de todos os valores de realocação no ponto de tempo 0 e σBLK o desvio padrão do branco medido durante a calibração, cada um é específico de citocinas.
, onde μRealocação t0 é a mediana de todos os valores de realocação no ponto de tempo 0 e σBLK o desvio padrão do branco medido durante a calibração, cada um é específico de citocinas. - Verificar se o aumento no valor de realocação é significativo, verificando se a mudança entre a realocação de fluorescência máxima e mínima medida durante o tempo de medição é superior a:
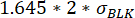 .
.
- Identifique as células co-secretoras atendendo aos critérios descritos na etapa 8.3. para mais de um canal de fluorescência simultaneamente.
- Repita a etapa 8.3. para todas as gotículas que não contêm célula (DAPI_WD_PosPxlPercent_1 = 0). Use essas gotículas para calcular a porcentagem de falsos positivos.
- Determine o valor λ preciso da medição selecionando aleatoriamente 200 - 500 gotículas e inspecionando-as com a função Verificar e classificar do software de análise de imagem. Conte o número de células nessas gotículas e calcule:
λ = número de células contadas / número de gotículas analisadas - Calcule o número total de células encapsuladas para a medição:
Contagem total de células = λ × número de gotículas analisadas - Calcule a porcentagem de células secretoras usando a contagem de células determinada. Além disso, calcule a porcentagem de falso-positivos para cada citocina (geralmente menos de 3% a 5% em relação ao número de positivos reais por canal) e use-os como um controle interno para consistência experimental e reprodutibilidade.
- Para calcular as concentrações de citocinas secretadas, converta os valores de realocação em concentração usando as equações de calibração estabelecidas na etapa 7.10.
- Calcule a taxa de secreção (SR) entre os pontos de tempo usando a seguinte equação.
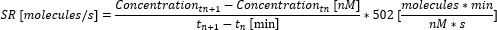
- Calcule a taxa média de secreção ao longo da medição, calculando a média das taxas de secreção individuais entre os pontos de tempo. Se a relocalização máxima mensurável tiver sido atingida antes do final da medição, ajustar a concentração para a concentração máxima mensurável (este valor é específico da citocina e corresponde à concentração máxima medida e utilizada na curva de calibração no passo 6.10) e não calcular a concentração adicional. Calcule a taxa de secreção e a média apenas até este ponto de tempo.
NOTA: Se menos de 50 células secretoras forem detectadas em um canal de fluorescência, as gotículas devem ser examinadas visualmente por meio da função de verificação e classificação e gotículas com agregados de fluorescência ou nanopartículas podem ser excluídas da análise. - Para extrair mais parâmetros da curva de secreção de cada célula, execute um ajuste de mínimo quadrado à curva de concentração de tempo para cada célula e citocina usando um script Python personalizado (disponível mediante solicitação). A função ajustada é uma curva sigmoidal seguindo a fórmula descrita abaixo (Ajustes com R2<0,95 são excluídos das seguintes etapas):
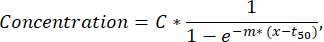
onde C corresponde ao platô de concentração [nM], t50 ao deslocamento do meio-máximo [s] e m à encosta da colina [min-1]. A partir desses parâmetros, os seguintes descritores de curva são extraídos conforme descrito abaixo.- Cmax [nM]: Concentração máxima medida.
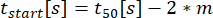 : Tempo de início da secreção, onde o ajuste atinge 10% de C.
: Tempo de início da secreção, onde o ajuste atinge 10% de C.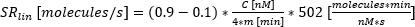 : taxa de secreção como a inclinação linear aproximada da curva entre 10% e 90% da curva tempo-concentração.
: taxa de secreção como a inclinação linear aproximada da curva entre 10% e 90% da curva tempo-concentração.
Representative Results
A plataforma funcional unicelular apresentada permitiu a medição de vários parâmetros. Primeiro, e semelhante às técnicas padrão, a frequência de células secretoras é representada no final da medição (Figura 4A). Após a estimulação com 1 μg/mL de lipopolissacarídeo (LPS) por 6 h de células mononucleares do sangue periférico (PBMC), 5,81% das células secretaram IL-6 (n= 1270), 4,55% TNFα (n= 995) e 6,06% IL-1β (n= 1326).
Para quantificar a secreção de citocinas, foram geradas curvas de calibração com concentrações conhecidas de citocinas recombinantes (Figura 4B). Essas curvas de calibração permitem a quantificação das concentrações de citocinas em gotículas ao longo do tempo. Exemplarmente, a concentração média de IL-6 em gotículas atingiu um platô após 90 min para PBMC estimulado por LPS, enquanto a IL-1β média em gotículas aumentou mais rapidamente a partir de 90 min, exibindo a resolução dinâmica da plataforma e a possibilidade de extrair subpopulações celulares secretando citocinas específicas (Figura 4C). À medida que a concentração muda entre os pontos de medição, é possível calcular as taxas de secreção dinâmica por citocina. Considerando a taxa média de secreção para cada citocina (Figura 4D), as células secretoras de IL-6 exibiram uma diminuição constante na taxa média de secreção, enquanto as células secretoras de TNFα e IL-1β mostraram um aumento na taxa de secreção após 90 min de tempo de medição e uma segunda diminuição após 150 min.
Além disso, é possível agrupar células em subpopulações dependendo das citocinas secretadas e co-secretadas ( Figura 4E ). Aqui, IL-6 e TNFα são secretados por 30,2% e 26,4% das células secretoras de IL-6 ou TNFα, respectivamente, enquanto as células de IL-1β secretoras de IL-1β constituem 68,8% de todas as células secretoras de IL-1β. Além disso, os efeitos da co-secreção nas concentrações secretadas e nas taxas de secreção podem ser resolvidos ( Figura 4F ). Ao observar as células secretoras de IL-6, diferentes quantidades de IL-6 foram secretadas se as células produzissem adicionalmente TNFα ou IL-1β. Da mesma forma, a distribuição das taxas médias de secreção ao longo da medição diferiu estatisticamente entre as células que secretam apenas IL-6 ou IL-6 ao lado de TNFα (taxas de secreção mais altas) e IL-1β (taxas de secreção de IL-6 mais baixas).
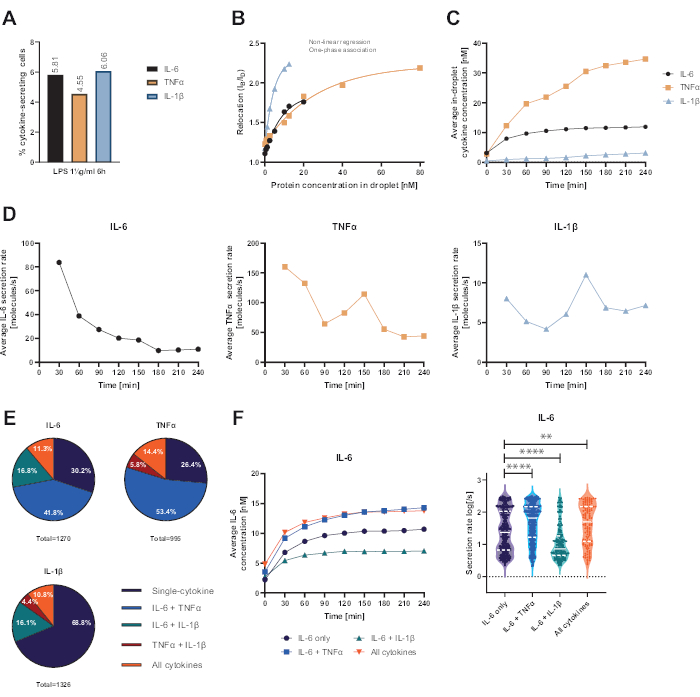
Figura 4: Resultados representativos de PBMC secretores de IL-6, TNFα e IL-1β após estimulação de 6 h com 1 μg/mL de LPS. (A) Porcentagem de PBMC secretores de IL-6, TNFα e IL-1β ao final da medição de 4 h. (B) Curvas de calibração de citocinas multiplexadas são geradas com concentrações conhecidas de citocinas recombinantes. Isso permite a quantificação de experimentos celulares calculando a partir do valor de realocação a concentração de citocinas dentro da gota. Os pontos foram ajustados usando um ajuste de curva de associação monofásica não linear, r2 = 0,9926 (IL-6), 0,9901 (TNFα), 0,9990 (IL-1β). (C) Concentrações médias secretadas de IL-6, TNFα e IL-1β liberadas pela secreção de PBMC durante o tempo de medição de 4 h. (D) Taxas médias de secreção de IL-6, TNFα e IL-1β durante o tempo de medição de 4 h. (E) Porcentagem relativa de células co-secretoras secretoras de IL-6, TNFα ou IL-1β e suas combinações. Normalizado para todas as células secretoras detectadas para cada citocina. (F) Concentrações médias de IL-6 ao longo do tempo de medição e distribuições médias da taxa de secreção (log) para células secretoras de IL-6 com resolução de co-secreção (n = 383 apenas para IL-6, n = 531 para IL-6 + TNFα, n = 213 para IL-6 + IL-1β e n = 143 para IL-6 + TNFα + IL-1β). As diferenças estatísticas nas distribuições das taxas de secreção foram avaliadas usando testes de Kolmogorov-Smirnov bilaterais, não pareados e não paramétricos com 95% de confiança, os valores de p são representados. ** (p <0,002) e **** (p <0,0001). A linha completa representa a mediana e a linha pontilhada os quartis. ncélulas totais = 21 866. Clique aqui para ver uma versão maior desta figura.
Para extrair informações adicionais sobre o nível de célula única, uma função sigmóide pode ser ajustada aos pontos de concentração-tempo de cada célula e citocina (Figura 5). Um conjunto de dados exemplar de concentração ao longo do tempo para uma célula e o ajuste sigmoidal correspondente é representado na Figura 5A. Aqui, o procedimento de ajuste de mínimos quadrados produz os seguintes parâmetros: C, correspondendo ao valor do platô superior da curva, t50 quantificando o deslocamento temporal da curva de zero, e a inclinação de Hill m, descrevendo a inclinação da parte ascendente da curva sigmóide com valores de concentração de 10% e 90% alcançados ao longo da medição. A partir desses parâmetros de ajuste, alguns descritores de curva podem ser extraídos, conforme explicado na etapa 7.12. produzindo o Cmax, o maior valor de concentração dos dados, tstart, o tempo de início da secreção, definido como atingindo 10% do valor de concentração do platô superior, e SRlin, a taxa de secreção durante a parte ascendente da curva.
Para classificar as subpopulações celulares, os descritores de curva obtidos de todos os ajustes de célula única foram classificados em três categorias cada: Os valores de Cmax foram agrupados em baixo, médio e alto para tstart em inicial, médio e tardio e SRlin em secretores lentos, médios e rápidos. Para ilustrar essa classificação, quatro curvas exemplares de secreção de célula única e seus descritores de curva correspondentes são mostrados (Figura 5A-D), onde a curva A exibe as características de um secretor baixo precoce de taxa média, a curva B é um secretor precoce, lento e alto, a curva C um secretor alto rápido precoce e a curva D mostra secreção baixa tardia. É importante observar que os pontos de corte para esses critérios são específicos de parâmetros de células, citocinas e ensaios e precisam ser adaptados para cada questão de pesquisa. Além disso, apenas a secreção de IL-6 de PBMC após estimulação de LPS de 1 μg/mL por 6 h foi considerada aqui, o que significa que a maioria das células eram secretoras precoces e altas com 80% e 79%, respectivamente (Figura 5E-F). Em relação à taxa de secreção, foi observada uma resposta bipolar com 55% das células secretoras de IL-6 sendo secretoras lentas e 39% como secretoras rápidas (Figura 5G).
Para caracterizar ainda mais o comportamento da secreção, os descritores de curva para cada célula foram plotados uns contra os outros e diferentes clusters foram extraídos (Figura 5H-J). Nenhuma correlação clara é dada entre tstart e Cmax (Figura 5H): as duas maiores populações eram secretores baixos precoces e secretores altos, independentemente do início da secreção. Considerando a relação entre tstart e SRlin (Figura 5I), a maioria das células eram secretoras lentas precoces com uma população clara de secretores altos iniciais e poucos secretores lentos / médios a tardios. Em relação às correlações SRlin e Cmax (Figura 5J), quase nenhum secretor rápido baixo a médio estava presente, com apenas uma população maior de secretores rápidos baixos. Além disso, havia uma grande população de secretores rápidos que não dependiam da concentração máxima medida, e duas populações de secretores altos secretavam lenta ou rápida. Em resumo, pode-se concluir que investigar a relação entre os descritores de curva para células individuais produz uma análise muito mais detalhada e pode potencialmente extrair novos achados biológicos de medições de secreção de célula única.
Com a análise apresentada acima, extraímos a dinâmica de secreção de células co-secretoras (Figura 6). Duas curvas de exemplo mostram diferentes dinâmicas de co-secreção para IL-6 e TNFα de duas células únicas com um início simultâneo de ambas as citocinas ( Figura 6A ), ou um início de secreção sequencial, com IL-6 sendo secretada primeiro ( Figura 6B ). Para classificar todas as células co-secretoras, foi definido um atraso de secreção de 60 min, onde todas as células que iniciam a secreção dentro dessa faixa são consideradas secretoras simultâneas e todas as células com atrasos mais longos são consideradas secretoras sequenciais. Essa análise também permitiu observar qual citocina foi secretada primeiro. Para IL-6 e TNFα, a cosecreção simultânea foi observada principalmente em 76% das células (Figura 6C), enquanto para IL-6 e IL-1β, a cosecreção sequencial foi observada em 86% das células, sendo a IL-6 a primeira citocina a ser secretada na maioria dos casos (Figura 6D).
Olhando para o tempo de início da secreção para as diferentes citocinas para todas as células co-secretoras individuais, nenhuma correlação clara entre os tempos de início da secreção foi observada nos experimentos realizados. Para a co-secreção de IL-6 e TNFα ( Figura 6E ), um cluster vertical maior em torno de 0 min estava presente, correspondendo às células co-secretoras mais predominantemente começando com IL-6. Para a co-secreção de IL-6 e IL-1β (Figura 6F), a maioria das células começou a secretar IL-6 no início da medição, enquanto a IL-1β foi secretada principalmente mais tarde. Em resumo, a análise aqui apresentada permitiu a identificação de diferentes subpopulações secretoras e dinâmicas complexas de co-secreção de citocinas.
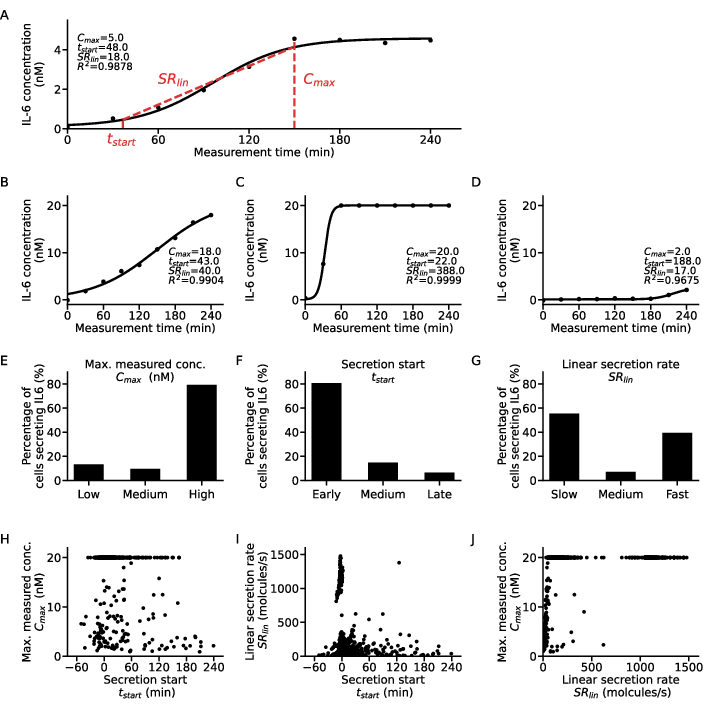
Figura 5: Análise detalhada de diferentes padrões dinâmicos de secreção para curvas de células secretoras de IL-6 únicas. (A) Dados representativos de concentração de citocinas de célula única ao longo do tempo de medição com a curva sigmóide ajustada e os parâmetros extraídos. (BD) Três curvas exemplares de concentração de citocinas de célula única para os diferentes tipos de secretores de citocinas encontrados para secreção de IL-6 após estimulação de LPS. (E-G) Porcentagens de células secretoras de IL-6 que são classificadas nos diferentes tipos de secretores com os seguintes critérios (n = 633): E. Cmax: baixo <5 nM, alto >19,5 nM, F. tstart: início <30 min, final >120min, G. SRlin: lento <250 moléculas/s, rápido >750 moléculas/s. (H-J) Relação entre os três descritores da curva de secreção Cmax, tstart e SRlin para cada célula individual (n=633). A grande população em Cmax = 20nM resulta de atingir o limite superior de detecção do ensaio. Clique aqui para ver uma versão maior desta figura.
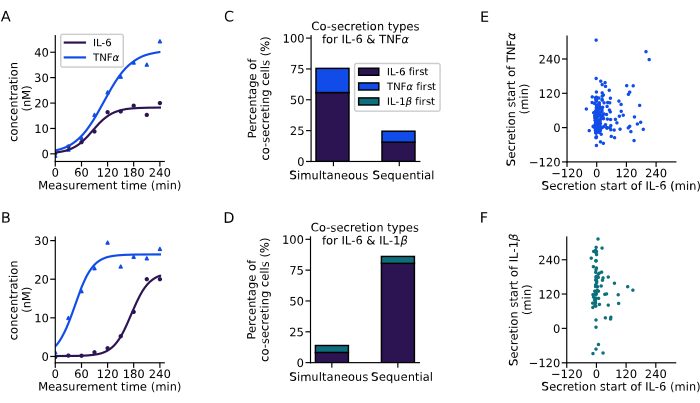
Figura 6: Extração de padrões de co-secreção de curvas de concentração de célula única. (AB) Curvas de concentração representativas para células únicas que secretam IL-6 e TNFα (A) simultaneamente e (B) sequencialmente, respectivamente. (CD) Porcentagem de células exibindo co-secreção simultânea e sequencial de IL-6 e TNFα (n=249), ou IL-6 e IL-1β (n=72), respectivamente. A secreção sequencial é definida através do atraso entre o início da secreção de citocinas de mais de 60 min. As cores indicam qual das citocinas iniciou a secreção primeiro. (E-F) Relação entre os horários de início da secreção para as diferentes citocinas para cada célula secretora (nIL6-TNFα=249, nIL6-IL1β=72). Clique aqui para ver uma versão maior desta figura.
Discussion
A liberação e secreção de citocinas são frequentemente investigadas em imunologia e medicina clínica3. A secreção desequilibrada de citocinas pode levar a efeitos prejudiciais para pacientes que sofrem de infecções, mas também em doenças neurológicas, inflamação ou câncer 26,27,28. Embora sua importância na saúde e na doença esteja bem estabelecida, o estudo de citocinas e suas células secretoras continua sendo um desafio, pois as metodologias atuais não são capazes de detectar e quantificar com precisão as citocinas originárias de uma única célula de maneira resolvida no tempo. Para o fluxo de trabalho aqui apresentado, foi utilizado um protocolo de estimulação estabelecido com PBMC e sua secreção de IL-6, TNF-α e IL-1β foi medida. A escolha de usar PBMCs em vez de subpopulações individuais e purificadas resultou da aplicação anterior para investigar síndromes de liberação de citocinas (CRS)23, uma condição caracterizada por concentrações plasmáticas altamente elevadas de citocinas pró-inflamatórias, incluindo IL-6, TNF-α e IL-1β29. Como a RSC geralmente não está ligada apenas a uma população, usamos PBMCs como estariam presentes in vivo. No entanto, as subpopulações celulares podem ser purificadas e avaliadas individualmente, se a questão científica exigir essa etapa. O tempo de incubação, as condições de estimulação e as faixas de ensaio dinâmico foram otimizados para medir a secreção das três citocinas de interesse. O fluxo de trabalho e os dados apresentados aqui demonstram como configurar, calibrar, quantificar, medir e analisar a secreção de célula única resolvida no tempo de várias citocinas. Este protocolo fornece um plano sobre como a análise multifuncional da secreção de citocinas pode permitir a grande diversidade funcional e dinâmica da citocina secretada nos pacientes.
Vários aspectos cruciais do protocolo de ensaio descrito permitem uma leitura biológica única. Primeiro, o encapsulamento de célula única em gotículas microfluídicas permitiu a extração de dados para cada célula individual. Eventos de encapsulamentos de múltiplas células podem ser detectados e classificados dentro ou fora por análise de imagem, dependendo da questão de pesquisa. Em segundo lugar, a inclusão de vários imunoensaios fluorescentes independentes em gotículas e o alinhamento das nanopartículas funcionalizadas permitiram a medição quantitativa de até três concentrações de citocinas em paralelo. Essa multiplexação permitiu a análise dos padrões de co-secreção de citocinas em um único nível de célula. Em terceiro lugar, a imobilização das gotículas permitiu a medição e correlação da secreção de citocinas para cada célula secretora e permitiu distinguir a secreção co-ocorrente da secreção sequencial. A resolução de tempo forneceu dados exclusivos sobre padrões de secreção e subpopulações de diferentes tipos de secretores. Por fim, a análise de imagem paralelizada permitiu a extração e o rastreamento eficientes de grandes quantidades de dados de medições com mais de 20.000 células individuais. A extração de curvas de secreção única permitiu ainda a descoberta de subpopulações e funcionalidades fenotípicas.
Além de sua leitura exclusiva, o ensaio também tem vantagens técnicas sobre a análise padrão de citocinas. Graças ao pequeno tamanho dos compartimentos de encapsulamento de cerca de 60 pL, quantidades absolutas de citocinas secretadas podem ser detectadas diretamente da fonte biológica com limites de detecção adequados à secreção celular. A miniaturização do ensaio também usa quantidades menores de biorreagentes caros. Além disso, a configuração requer muito pouco equipamento especializado, que muitas vezes já está disponível em laboratórios de biologia e bioengenharia. Os microscópios de fluorescência estão amplamente disponíveis e as bombas de seringa são frequentemente usadas em laboratórios de bioengenharia ou podem ser adquiridas a um custo relativamente baixo. Se houver uma cultura de células, o custo de todo o equipamento necessário para executar os experimentos é de cerca de 148.000 euros, com a maior parte contribuída pelo microscópio epifluorescente automatizado (130.000 euros). No entanto, esse instrumento pode ser frequentemente encontrado em laboratórios biológicos, e o restante do custo é distribuído para a bomba de seringa (13.000 euros, mas alternativas mais baratas estão disponíveis) e equipamentos menores. A fabricação do chip de gotículas e da câmara de observação é muito bem descrita17 e pode ser realizada fora de um ambiente de sala limpa com infraestrutura necessária, como fornos e limpadores de plasma presentes na maioria dos laboratórios de bioengenharia. Alternativamente, diferentes fornecedores estão disponíveis para fornecer aos laboratórios interessados chips geradores de gotículas. Devido aos pequenos volumes necessários, o ensaio é econômico e simples de configurar.
Para garantir o maior grau de reprodutibilidade, identificamos algumas etapas críticas para o sucesso do protocolo. Um problema comum para usuários iniciantes é o movimento das gotículas durante a medição. Embora o software de análise possa rastrear gotículas individuais até certo ponto, o movimento excessivo leva à perda de resolução de uma única célula e resultados imprecisos. O movimento pode ser evitado usando câmaras de medição herméticas adequadas, tamanho correto de gotículas e tamanhos de câmara, um curto período de equilíbrio antes de iniciar a medição e concentração adequada de surfactante. Outra etapa crítica é o foco preciso antes de iniciar a medição. A focagem inadequada leva a valores de realocação de fluorescência significativamente reduzidos e à subestimação da quantidade de citocina secretada. Finalmente, dependendo da pergunta e do protocolo em questão, o tempo correto entre as diferentes etapas é de extrema importância para a reprodutibilidade. Especialmente o tempo de espera entre o enchimento da câmara e o início da medição deve ser consistente, caso contrário, a janela de medição das citocinas secretadas pode ser perdida.
As limitações da tecnologia apresentada incluem a capacidade restrita de manipular ainda mais as células após o encapsulamento. Portanto, atualmente não é possível adicionar ou remover estimulantes, anticorpos ou reagentes adicionais. Além disso, como as células são encapsuladas em seu biorreator isolado, nenhuma interação entre as células (sinalização baseada em contato ou parácrina) pode ocorrer durante a medição. Essa limitação pode ser parcialmente superada com incubações em massa de antemão. Além disso, efeitos autócrinos aprimorados de citocinas secretadas também são possíveis e esses efeitos não podem ser quantificados ou excluídos com certeza, pois apenas citocinas secretadas detectadas por anticorpos são medidas. Portanto, a visão isolada sobre a secreção de citocinas deve sempre ser descrita no contexto da pergunta e aplicação correspondentes. No entanto, essa limitação também pode ser usada para o estudo detalhado de múltiplos, dupletos e trigêmeos encapsulados, se for de interesse. Isso forneceria uma configuração interessante útil para investigar o contato célula-célula ou questões baseadas em parácrinos. Por fim, também a faixa dinâmica do ensaio é limitada e precisa de adaptação à aplicação específica. Aqui, adaptamos a faixa dinâmica do ensaio à quantidade secretada esperada das citocinas medidas.
Para avançar ainda mais as capacidades e aplicabilidade do ensaio, vários desenvolvimentos podem ser abordados no futuro, em aspectos biológicos, técnicos e de análise de dados. No lado biológico, a medição de citocinas adicionais, outras proteínas secretadas, marcadores metabólicos ou de superfície celular pode ser integrada adaptando o ensaio. Além disso, este ensaio pode ser integrado a um fluxo de trabalho junto com outros ensaios baseados em células para ampliar as leituras (por exemplo, coloração ou sequenciamento por citometria de fluxo). Além disso, a usabilidade do ensaio pode ser simplificada, por exemplo, criando um chip microfluídico integrado para criação e observação de gotículas, permitindo assim uma aplicação mais ampla fora dos laboratórios de bioengenharia em um ambiente clínico. Em relação à análise de dados, a extração e o rastreamento de informações de imagens podem ser estendidos aprimorando a automação e usando abordagens de aprendizado de máquina, por exemplo, para detectar a presença e a posição da(s) célula(s) e da linha de contas em cada gotícula sem marcação fluorescente. Isso abriria canais fluorescentes adicionais que poderiam ser usados para imunoensaios, resultando na medição de ainda mais citocinas em paralelo.
O ensaio apresentado e os protocolos e análises associados podem ser aplicados para diversos casos de uso potenciais relacionados à dinâmica da secreção de citocinas. Mais especificamente, o ensaio poderia abordar questões imunológicas fundamentais, como identificar perfis de secreção de citocinas específicos do tipo de célula e da ativação, polifuncionalidade das células secretoras de citocinas ou os mecanismos de temporalidade e manutenção dos balanços de citocinas. Além disso, em aplicações clínicas, a plataforma pode permitir desvendar o papel das citocinas durante as respostas inflamatórias ativas ou crônicas, como observado no COVID-1930, ou fornecer uma ferramenta para estratificar pacientes e personalizar tratamentos com base em assinaturas únicas, como na autoinflamação31. Em conclusão, a avaliação quantitativa resolvida no tempo da secreção de citocinas de células individuais é um método muito necessário, pois elucida como um determinado medicamento, infecção, alteração genética ou estimulação ex vivo induz uma resposta específica.
Disclosures
Aspectos específicos, como as medições de beadline das células, foram patenteados.
Acknowledgements
Este projeto foi apoiado pela concessão # 2021-349 da Área de Foco Estratégico Saúde Personalizada e Tecnologias Relacionadas (PHRT) do Domínio ETH (Institutos Federais Suíços de Tecnologia), a bolsa inicial do Conselho Europeu de Pesquisa (concessão # 803,336) e a Fundação Nacional de Ciência da Suíça (concessão # 310030_197619). Além disso, agradecemos a Guilhem Chenon e Jean Baudry por seu trabalho e desenvolvimento do analisador DropMap inicial.
Materials
| Name | Company | Catalog Number | Comments |
| 008-FluoroSurfactant | RAN Biotechnologies | 008-FluoroSurfactant-10G | |
| 2-Stream flow-focusing droplet maker, 30 µm nozzle, PFOS hydrophobic surface treatment | Wunderli chips | ||
| Alexa Fluor 647 NHS Ester | ThermoFisher | A20006 | https://www.thermofisher.com/ch/en/home/references/protocols/cell-and-tissue-analysis/labeling-chemistry-protocols/fluorescent-amine-reactive-alexa-fluor-dye-labeling-of-igm-antibodies.html |
| Anti-Human IL-1β (Monoclonal Mouse), AF647 labelled in-house | PeproTech | 500-M01B | |
| ARcare92524 double-sided adhesive tape | Adhesvies Reasearch | ARcare92524 | |
| Bio-Adembeads Streptavidin plus 300nm | Ademtech | Cat#03233 | |
| Biotinylated Goat Anti-Human IL-1β | PeproTech | 500-P21BGBT | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A3059 | |
| Cell Scraper | TPP | 99002 | |
| CellTrace Violet Cell Proliferation Kit | Invitrogen | C34557 | Cell staining solution |
| Chromafil Xtra PTFE-45/25 syringe filters | Macherey-Nagel | 729205 | |
| Costar 6-well Clear Flat Bottom Ultra-Low Attachment | Corning | 3471 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| D-Biotin | Fluorochem | M02926 | |
| DPBS, no calcium, no magnesium | Gibco | 14196-094 | |
| epT.I.P.S. Standard 2-200 µl | Eppendorf | 30000889 | |
| Ethylenediaminetetraacetic acid disodium salt solution | Sigma-Aldrich | 3690 | |
| EZ-LINK-NHS-PEG4-Biotin | ThermoFisher | A39259 | https://www.thermofisher.com/order/catalog/product/20217 |
| FcR Blocking Reagent, human | Miltenyi Biotec | 130-059-901 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| Handy dish soap | Migros | 5.01002E+11 | |
| HEPES (1 M) | Gibco | 15630-080 | |
| HFE-7500 Oil 3M TM Novec | Fluorochem | B40045191 | |
| Idex F-120 Fingertight One-Piece Fitting, Standard Knurl, Natural PEEK, 1/16" OD Tubing, 10-32 Coned | Cole-Parmer | GZ-02014-15 | |
| IL-6 Monoclonal Antibody (MQ2-13A5 - Rat), FITC | ThermoFisher | 11-7069-81 | |
| IL-6 Monoclonal Antibody (MQ2-39C3), Biotin | ThermoFisher | 13-7068-85 | |
| KnockOut Serum Replacement | ThermoFisher | 10828-010 | |
| Loctite AA 3491 curable UV glue | Henkel AG & Co | 3491 | |
| Microscope slides (76x26x1mm, clear white) | Menzel Gläser | ||
| Mineral oil light | Sigma-Aldrich | 330779 | |
| NanoPort Assembly Headless, 10-32 Coned, for 1/16" OD | Idex | N-333 | |
| Neodymium block magnet | K&J Magnetics | BZX082 | |
| Omnifix-F Spritze, 1 ml, LS | Braun | 9161406V | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140-122 | |
| Phosphate buffered saline | Sigma-Aldrich | P4417 | |
| Pluronic F-127, 0.2 µm filtered (10% Solution in Water) | ThermoFisher | P6866 | |
| Precision wipes | Kimtech Science | 5511 | |
| PTFE microtubing 0.30 × 0.76 mm | FisherScientific | 1191-9445 | |
| PTFE microtubing 0.56 × 1.07 mm | FisherScientific | 1192-9445 | |
| Recombinant Human IL-1β | Peprotech | Cat#200-01B | |
| Recombinant Human IL-6 | Peprotech | Cat#200-06 | |
| Recombinant human serum albumine (HSA) | Sigma-Aldrich | A9731 | |
| Recombinant Human TNF-α | Peprotech | Cat#300-01A | |
| Reusable biopsy punch diameter 0.75 mm and 6 mm | Stiefel | 504529 and 504532 | |
| RPMI 1640 Medium, no phenol red | Gibco | 11835-030 | |
| Standard LPS, E. coli K12 | InvivoGen | tlrl-eklps | |
| Sterican needles 23 G for 0.56 mm diameter microtubing | FisherScientific | 15351547 | |
| Sterican needles 27 G for 0.30mm diameter microtubing | FisherScientific | 15341557 | |
| TNF alpha Monoclonal Antibody (MAb11), PE | ThermoFisher | 12-7349-81 | |
| TNF-alpha Monoclonal Antibody (MAb1), biotinylated in-house | ThermoFisher | 14-7348-85 | |
| Trypan Blue Stain (0.4%) for use with the Countess Automated Cell Counter | Invitrogen | T10282 | |
| Vacuum Filtration "rapid"-Filtermax | TPP | 99500 | |
| Devices | |||
| Cameo 4 automatic cutting machine | Silhouette | ||
| Cetoni Base 120 + 3x NEMESYS Low Pressure Syringe Pumps | Cetoni | NEM-B101-03 A | |
| Countess II Automated Cell Counter | ThermoFisher | ||
| Inverted Epi-fluorescence microscope Ti2 | Nikon | ECLIPSE Ti2-E, Ti2-E/B*1 | |
| OKO Lab Cage Incubator, dark panels | OKO Lab | ||
| ORCA-Fusion Digital CMOS camera | Hammatsu | C14440 | |
| SOLA Light Engine | Lumencor | sola 80-10247 |
References
- Chen, L., et al. Inflammatory responses and inflammation-associated diseases in organs. Oncotarget. 9 (6), 7204-7218 (2017).
- Cicchese, J. M., et al. Dynamic balance of pro- and anti-inflammatory signals controls disease and limits pathology. Immunol Rev. 285 (1), 147-167 (2018).
- Liu, C., et al. Cytokines: From clinical significance to quantification. Adv Sci. 8 (15), e2004433 (2021).
- Rojas, J. M., Avia, M., Martín, V., Sevilla, N. IL-10: A multifunctional cytokine in viral infections. J Immunol Res. 2017, 6104054 (2017).
- Kohanawa, Y. M. A regulatory effect of the balance between TNF-α and IL-6 in the granulomatous and inflammatory response to Rhodococcus aurantiacus infection in mice. J Immunol. 177 (1), 642-650 (2006).
- Geginat, J., et al. Plasticity of human CD4 T cell subsets. Front Immunol. 5, 630 (2014).
- Sallusto, F. Heterogeneity of human CD4+ T cells against microbes. Ann Rev Immunol. 34 (1), 317-334 (2016).
- Chetaille Nézondet, A. L., Poubelle, P. E., Pelletier, M. The evaluation of cytokines to help establish diagnosis and guide treatment of autoinflammatory and autoimmune diseases. J Leukocyte Biol. 108 (2), 647-657 (2020).
- Sims, J. T., et al. Characterization of the cytokine storm reflects hyperinflammatory endothelial dysfunction in COVID-19. J Allergy Clin Immunol. 147 (1), 107-111 (2021).
- Yasen, A., et al. Single-cell RNA sequencing reveals the heterogeneity of infiltrating immune cell profiles in the hepatic cystic echinococcosis microenvironment. Infection and Immunity. 89 (12), (2021).
- Jiang, Y., et al. Single-cell RNA sequencing highlights intratumor heterogeneity and intercellular network featured in adamantinomatous craniopharyngioma. Sci Adv. 9 (15), (2023).
- Tanguay, S., Killion, J. J. Direct comparison of ELISPOT and ELISA-based assays for detection of individual cytokine-secreting cells. Lymphokine Cytokine Res. 13 (4), 259-263 (1994).
- Bucheli, O. T. M., Sigvaldadóttir, I., Eyer, K. Measuring single-cell protein secretion in immunology: Technologies, advances, and applications. Eur J Immunol. 51 (6), 1334-1347 (2021).
- Brower, K. K., et al. Double emulsion flow cytometry with high-throughput single droplet isolation and nucleic acid recovery. Lab Chip. 20 (12), 2062-2074 (2020).
- Brower, K. K., et al. Double emulsion picoreactors for high-throughput single-cell encapsulation and phenotyping via FACS. Anal Chem. 92 (19), 13262-13270 (2020).
- Luo, X., Chen, J. Y., Ataei, M., Lee, A. Microfluidic compartmentalization platforms for single cell analysis. Biosensors. 12 (2), 58 (2022).
- Bounab, Y., et al. Dynamic single-cell phenotyping of immune cells using the microfluidic platform DropMap. Nat Protoc. 15 (9), 2920-2955 (2020).
- Eyer, K., et al. Single-cell deep phenotyping of IgG-secreting cells for high-resolution immune monitoring. Nat Biotechnol. 35 (10), 977-982 (2017).
- Gaa, R., et al. Versatile and rapid microfluidics-assisted antibody discovery. mAbs. 13 (1), 198130 (2021).
- Gerard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nat Biotechnol. 38 (6), 715-721 (2020).
- De Jonghe, J., et al. spinDrop: a droplet microfluidic platform to maximise single-cell sequencing information content. Nat Comm. 14 (1), 4788 (2023).
- Wheeler, M. A., et al. Droplet-based forward genetic screening of astrocyte-microglia cross-talk. Science. 379 (6636), 1023-1030 (2023).
- Portmann, K., Linder, A., Oelgarth, N., Eyer, K. Single-cell deep phenotyping of cytokine release unmasks stimulation-specific biological signatures and distinct secretion dynamics. Cell Rep Meth. 3 (7), 100502 (2023).
- Portmann, K., Linder, A., Eyer, K. Stimulation-induced cytokine polyfunctionality as a dynamic concept. eLife. 12, 89781 (2023).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. Clin Biochem Rev. 29, S49-S52 (2008).
- Yang, J., et al. New insight into neurological degeneration: Inflammatory cytokines and blood-brain barrier. Front Mol Neurosci. 15, 1013933 (2022).
- Kim, P. S., Ahmed, R. Features of responding T cells in cancer and chronic infection. Curr Opin Immunol. 22 (2), 223-230 (2010).
- Becher, B., Spath, S., Goverman, J. Cytokine networks in neuroinflammation. Nat Rev Immunol. 17 (1), 49-59 (2017).
- Cosenza, M., Sacchi, S., Pozzi, S. Cytokine release syndrome associated with T-cell-based therapies for hematological malignancies: Pathophysiology, clinical presentation, and treatment. Int J Mol Sci. 22 (14), 7652 (2021).
- Hu, B., Huang, S., Yin, L. The cytokine storm and COVID-19. J Med Virol. 93 (1), 250-256 (2021).
- Marcuzzi, A., et al. Autoinflammatory diseases and cytokine storms-Imbalances of innate and adaptative immunity. Int J Mol Sci. 22 (20), 11241 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved