Padrões dinâmicos de proteínas induzidas por luz em membranas modelo
In This Article
Summary
Aqui, é descrito um protocolo para gerar padrões proteicos regulados por luz e reversíveis com alta precisão espaço-temporal em membranas lipídicas artificiais. O método consiste na fotoativação localizada da proteína iLID (dímero induzível por luz melhorado) imobilizada em membranas modelo que, sob luz azul, se liga à sua proteína parceira Nano (SspB do tipo selvagem).
Abstract
A localização precisa e a ativação de proteínas na membrana celular em um determinado momento dão origem a muitos processos celulares, incluindo polarização, migração e divisão celular. Assim, métodos de recrutamento de proteínas para modelar membranas com resolução subcelular e alto controle temporal são essenciais na reprodução e controle de tais processos em células sintéticas. Aqui, é descrito um método para a fabricação de padrões de proteínas reversíveis reguladas pela luz em membranas lipídicas com alta precisão espaço-temporal. Para isso, imobilizamos a proteína fotocomutável iLID (dímero induzível por luz melhorado) em bicamadas lipídicas suportadas (SLBs) e na membrana externa de vesículas unilamelares gigantes (GUVs). Após a iluminação da luz azul local, o iLID se liga ao seu parceiro Nano (SspB do tipo selvagem) e permite o recrutamento de qualquer proteína de interesse (POI) fundida ao Nano da solução para a área iluminada na membrana. Essa ligação é reversível no escuro, o que fornece ligação dinâmica e liberação do POI. No geral, este é um método flexível e versátil para regular a localização de proteínas com alta precisão no espaço e no tempo usando luz azul.
Introduction
A formação de padrões proteicos nas membranas celulares dentro das regiões subcelulares dá origem a vários processos biológicos, incluindo migração, divisão e comunicação localizada célula a célula 1,2. Esses padrões de proteína são regulados no espaço e no tempo e são altamente dinâmicos. Replicar esses padrões de proteínas em células sintéticas é essencial para imitar os processos celulares que surgem delas e para obter uma melhor compreensão de como essa regulação funciona em nível molecular. Análogo ao que é observado para membranas em células vivas, os métodos para gerar padrões de proteínas em membranas artificiais devem capturar sua dinâmica e fornecer controle espaço-temporal preciso.
Dentre vários estímulos, a luz se destaca por proporcionar o maior controle espaço-temporal e várias vantagens adicionais3. Através da regulação com luz, é fácil iluminar uma área desejada a qualquer momento desejado com precisão incomparável. Além disso, a luz fornece alta sintonia como a intensidade da luz e as durações do pulso podem ser ajustadas. Além disso, a luz visível é inofensiva para as biomoléculas, incluindo proteínas, e é possível até abordar múltiplas funcionalidades com diferentes comprimentos de onda. Assim, abordagens responsivas à luz baseadas na luz visível surgem como caminhos promissores para a regulação controlada e biortogonal dos padrões de proteínas no espaço e no tempo 4,5,6. A utilização de pares de proteínas fotocomutáveis da optogenética, atuando como dimerizadores induzíveis por luz, fornece um método direto para recrutar proteínas específicas para as membranas. Em particular, os padrões de proteínas foram formados com sucesso em membranas artificiais usando a interação desencadeada por luz azul entre iLID (dímero induzível por luz melhorado, com base no domínio LOV2 fotocomutável de Avena sativa) e Nano (SspB de tipo selvagem) 7 , 8 , o sistema SpyTag induzível por luz azul (BLISS) 9 , o tetrâmero de proteína responsiva à luz verde CarH10 e a interação induzível por luz vermelha entre PhyB e PIF 611.
Foi demonstrado que a interação fotocomutável entre iLID e Nano5 pode ser usada para foto-padronizar proteínas em membranas modelo usando luz azul7. A interação iLID/Nano é reversível no escuro, altamente específica e opera em condições fisiológicas. A ancoragem do iLID em modelos de membrana lipídica, como vesículas unilamelares gigantes (GUVs) ou bicamadas lipídicas suportadas (SLBs), permite o recrutamento regulado pela luz de Nano para essas membranas, o que é reversível no escuro. Notavelmente, observamos que a introdução de um domínio desordenado no terminal N do iLID (resultando em uma proteína chamada disiLID) como uma ligação a uma membrana lipídica modelo aumenta a eficiência do recrutamento de Nano e a dinâmica de reversão8.
Ao empregar a interação disiLID/Nano, desenvolvemos um método para gerar padrões de alto contraste de proteínas de interesse (POI) nanofundidas em SLBs e nas membranas externas de GUVs. Este método permite a criação de padrões de proteínas com notável resolução espacial e temporal e alta reversibilidade em minutos. O protocolo detalhado descreve o processo de recrutamento local de proteínas em membranas artificiais. Especificamente, isso é conseguido imobilizando uma versão biotinilada de disiLID em SLBs e GUVs por meio da interação biotina-estreptavidina (SAv). Posteriormente, o Nano marcado com fluorescência (mOrange-Nano) é recrutado para essas membranas funcionalizadas disiLID sob iluminação de luz azul. Nosso protocolo experimental oferece uma abordagem direta e adaptável para obter o recrutamento de proteínas localizadas para as membranas. É importante ressaltar que essa metodologia não se limita às interfaces SLB e GUV relatadas ou mOrange-Nano; pode ser estendido a outros materiais e proteínas funcionalizados com disiLID fundidos ao Nano.
Protocol
1. Preparação experimental
- Expresse e purifique biotinilated-disiLID (b-disiLID) e mOrange-Nano (ver Tabela de Materiais) seguindo os procedimentos relatados anteriormente 7,8.
- Prepare misturas lipídicas em frascos de vidro com a composição e concentrações lipídicas selecionadas. Primeiro, dissolva os lipídios no clorofórmio para obter uma solução lipídica final com concentração de 1 mg / mL.
- Misturar lípidos de modo a obter uma composição de 94,9 mol% de 2-dioleoil-sn-glicero-3-fosfocolina (DOPC), 5 mol% de sal de sódio de 1,2-dioleoil-sn-glicero-3-fosfoetanolaminaN-(cap biotinil) (DOPE-biotina) e 0,1 mol% de 1,1'-dioctadecil-3,3,3',3'-tetrametilindodicarbocianina (DiD) (ver Tabela de Materiais).
NOTA: A mistura lipídica pode ser ajustada com proporções variadas e concentração de DOPE-biotina e/ou diferentes corantes de membrana. A concentração recomendada de DOPE-biotina (5 mol%) permite a formação de uma camada de estreptavidina de alta densidade (SAv) nas etapas seguintes.
- Misturar lípidos de modo a obter uma composição de 94,9 mol% de 2-dioleoil-sn-glicero-3-fosfocolina (DOPC), 5 mol% de sal de sódio de 1,2-dioleoil-sn-glicero-3-fosfoetanolaminaN-(cap biotinil) (DOPE-biotina) e 0,1 mol% de 1,1'-dioctadecil-3,3,3',3'-tetrametilindodicarbocianina (DiD) (ver Tabela de Materiais).
- Prepare pequenas vesículas unilamelares (SUVs) seguindo os métodos relatados anteriormente 7,8,12. Para esta etapa, recomenda-se preparar SUVs com diâmetro ≤100 nm.
- Para este estudo, o método de sonicação é empregado. Primeiro, evapore a solução de clorofórmio no frasco de vidro com um fluxo de nitrogênio enquanto gira os frascos para formar um filme lipídico fino. Em seguida, remova o clorofórmio residual por pelo menos 1 h sob vácuo.
- Reidrate o filme seco em água ultrapura com uma concentração final de 1 mg/mL de lipídios por vórtice. Por fim, sonicar a solução obtida durante 10 min até que a solução opaca fique límpida.
NOTA: Armazene a mistura lipídica em um tubo de microcentrífuga na geladeira por no máximo 2 semanas. Diferentes métodos de preparação de SUVs (por exemplo, método de extrusão) também podem ser usados, desde que o tamanho dos SUVs finais seja ≤ 100 nm.
- Reidrate o filme seco em água ultrapura com uma concentração final de 1 mg/mL de lipídios por vórtice. Por fim, sonicar a solução obtida durante 10 min até que a solução opaca fique límpida.
2. Recrutamento de mOrange-Nano para SLBs funcionalizados disiLID
- Adicione 150 μL de NaOH 2 M em cada poço da câmara de fundo de vidro de 18 poços de lâmina μ (consulte a Tabela de Materiais) e incube por 1 h à temperatura ambiente. Em seguida, remova o NaOH e lave os poços 3-5 vezes, primeiro com 150 μL de água ultrapura e, em seguida, 3 vezes com 150 μL de tampão (10 mM Tris pH 7,4, 100 mM de NaCl) contendo 10 mM de CaCl2.
- Adicione 15 μL de SUVs recém-preparados (concentração de estoque de 1 mg / mL em água) nos poços contendo 150 μL de tampão com 10 mM de CaCl2 para obter aproximadamente uma diluição de fator 10 de SUVs no tampão. Deixe os SUVs incubarem por 30 minutos em temperatura ambiente. Após o tempo de incubação, serão formados SLBs biotinilados.
- Lave os SLBs pelo menos 7 vezes com tampão (10 mM Tris pH 7,4, 100 mM NaCl) sem CaCl2 , removendo primeiro a solução e, posteriormente, adicionando tampão novo em cada etapa. Recomenda-se o uso de 80 μL de tampão para cada etapa de lavagem.
NOTA: Uma lavagem ideal dos SLBs é obtida pipetando a solução fresca para cima e para baixo várias vezes sem tocar na superfície. A pipetagem da solução nos poços que contêm os SLBs recém-formados deve ser suave para reduzir a formação de pequenas bolhas de ar que danificariam os SLBs formados. A partir deste momento, os poços devem conter um volume suficiente de tampão para evitar que os SLBs sequem. - Para a funcionalização adicional dos SLBs biotinilados com SAv, adicione uma solução de SAv a uma concentração final de 250 nM e incube por 30 min à temperatura ambiente. Em seguida, remova o excesso de SAv lavando-o com um tampão (10 mM Tris pH 7,4, 100 mM NaCl) pelo menos 5 vezes.
- A partir deste momento, mantenha as amostras sob uma luz vermelha protetora para evitar a fotoativação indesejada de proteínas fotocomutáveis. Adicione b-disiILD (consulte a Tabela de Materiais) a uma concentração final de 1 μM no poço. Após 30 min de incubação à temperatura ambiente, remova o excesso de proteína lavando com tampão pelo menos 5 vezes.
- Adicione mOrange-Nano (consulte a Tabela de Materiais) a uma concentração final de 200 nM e mantenha a amostra no escuro, cobrindo-a com papel alumínio.
- Coloque a lâmina μ sob o microscópio de fluorescência e ajuste as configurações de imagem. Defina o laser de 552 nm para excitação de mOrange-Nano. Ajuste a faixa de emissão para otimizar o sinal mOrange. A fotoativação do disiLID é obtida com um laser de 488 nm, utilizando pulsos de luz com intervalos de 2,58 s.
3. Preparação de GUVs
- Preparar uma solução a 5% (p/v) de álcool polivinílico (PVA, ver quadro de materiais) (MW: 145 000 g/mol) com sacarose 100 mM em água ultrapura, misturando durante a noite a 80 °C a 400 rpm.
- Prepare uma solução lipídica em clorofórmio com a composição desejada (concentração final de 10 mg/mL). Para este método, recomenda-se uma composição constituída por 10 mg/ml de POPC, 10 mol% de 1-palmitoil-2-oleoil-sn-glicero-3-fosfo-(1'-rac-glicerol) (POPG), 2% mol de DOPE-biotina e 1% mol% de DiD (ver Tabela de Materiais).
- Prepare GUVs com a técnica de hidratação 7,8. Primeiro, espalhe 40 μL da solução de PVA preparada como uma camada fina homogênea sobre uma lâmina de vidro de 60 mm x 24 mm, de preferência com uma ponta de pipeta. Em seguida, seque a camada fina a 50 °C por 30 min.
- Espalhe 5 μL da solução lipídica com uma agulha na camada de PVA e deixe secar a 30 ° C por 1 h.
- Monte uma câmara na lâmina de vidro funcionalizada usando um espaçador (~ 40 mm × 24 mm × 2 mm, consulte a Tabela de Materiais) e uma segunda lâmina de vidro.
- Adicione 1 mL de tampão de reidratação (10 mM Tris pH 7,4, 100 mM NaCl) na câmara por 1 h em temperatura ambiente para formar GUVs. Após 1 h, inverta a câmara e bata suavemente nas superfícies de vidro com uma ponta de pipeta.
- Remova cuidadosamente a lâmina de vidro de um lado para abrir a câmara construída e colher os GUVs com uma pipeta.
- Coloque a solução em um tubo de plástico e deixe os GUVs repousar por 2 h.
4. Recrutamento mOrange-Nano para GUVs funcionalizados disiLID
- Adicione uma solução de SAv aos GUVs recém-colhidos e deixe descansar por 30 min em temperatura ambiente.
NOTA: Os passos seguintes devem ser realizados com luz vermelha protetora para evitar a fotoativação do disiLID. - Adicione 1 μM de b-disiLID à solução de GUVs e coloque a amostra por 30 min no escuro, cobrindo-a com papel alumínio.
- Pré-trate uma câmara de fundo de vidro de 18 poços de lâmina μ com solução de BSA de 150 μL (3% p/v em água) por 10 min. Em seguida, remova a solução BSA e lave os poços com 150 μL de água ultrapura 3 vezes.
- Adicione 145 μL de 200 nM mOrange-Nano em tampão (10 mM Tris pH 7,4, 100 mM NaCl) ao poço.
- Em seguida, adicione 5 μL de GUVs decorados com b-disiLID à solução e aguarde ~15 min para que os GUVs assentem.
- Coloque a lâmina μ sob o microscópio confocal. Excite a amostra a 552 nm para visualizar a fluorescência mOrange (λex = 557 nm; λem = 576 nm) e a 638 nm para visualizar DiD (λex = 644 nm; λem = 665 nm) nas membranas GUV. O recrutamento mOrange-Nano é acionado com pulsos de luz azul (488 nm, intensidade 1%) a cada 5,3 s, a fim de minimizar os efeitos indesejados do fotobranqueamento.
NOTA: Os comprimentos de onda de excitação podem ser adaptados com base no tipo de microscópio em uso. Outros comprimentos de onda de excitação comuns disponíveis para microscópios também são lasers de 532 nm ou 561 nm e 633 nm, 647 nm, 639 nm ou 640 nm.
Representative Results
Os procedimentos descritos permitem a formação de SLBs para recrutar mOrange-Nano nas membranas sintéticas. A formação de mOrange-Nano definido padronizado nos SLBs funcionalizados com b-disiLID é mostrada na Figura 1A. Como uma região de interesse (ROI) quadrada (24 μm × 24 μm) no SLB é iluminada com luz azul de 488 nm, um rápido aumento do sinal de fluorescência é observado no canal mOrange (mostrado em vermelho) no ROI dentro de 200 s. O padrão mostra bordas muito definidas e nítidas (Figura 1B), indicando alto controle espacial sobre a área fotoativada. A interação é rápida e totalmente reversível, pois a iluminação com luz azul é interrompida. Este método também permite a formação de padrões ao longo de vários ciclos de iluminação (Figura 2). Ciclos alternados de ~ 200 s de luz azul e 200 s de escuridão levam ao recrutamento reversível de mOrange-Nano na área selecionada por várias vezes com valores comparáveis de intensidade Δ de fluorescência nos padrões.
A Figura 3 mostra a representação esquemática da preparação dos GUVs. O recrutamento de mOrange-Nano também é observado em GUVs. É mostrado que os GUVs colocados no escuro não exibem fluorescência mOrange ( Figura 4A ). Como os GUVs são iluminados globalmente com luz azul, a fluorescência mOrange é observada, colocalizando com o corante de membrana GUV (DiD). A interação é altamente reversível, pois a iluminação é encerrada. A quantificação da intensidade de mOrange na membrana GUV ao longo do tempo mostra o recrutamento rápido e eficaz de proteínas, bem como a reversibilidade total ( Figura 4B ).
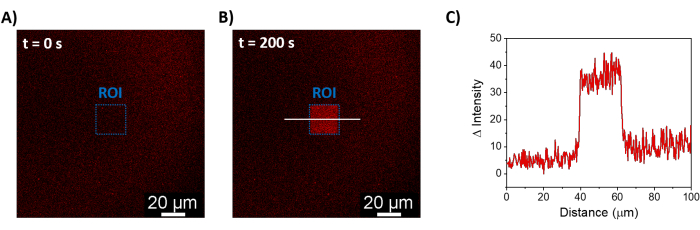
Figura 1: Imagens de microscopia de fluorescência de SLBs funcionalizadas com b-disiLID. As imagens de fluorescência na presença de mOrange-Nano antes (A) e durante (B) a iluminação local de luz azul (488 nm) na ROI. Barra de escala = 20 μm. (C) Intensidade de fluorescência de mOrange medida na ROI para SLBs funcionalizados com b-disiLID (a = 200 s). A figura é adaptada de Di Iorio et al.8. Clique aqui para ver uma versão maior desta figura.
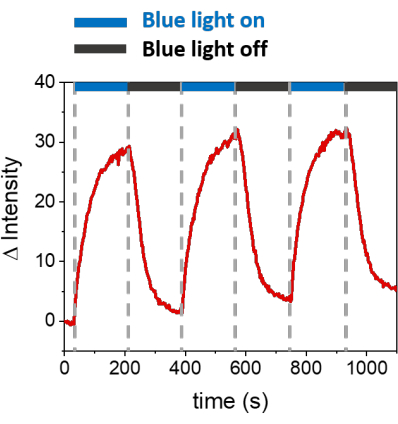
Figura 2: Intensidade de fluorescência de mOrange recrutado no ROI em SLBs decorados com b-disiLID durante três ciclos de recrutamento. Após cada etapa de fotoativação, a fluorescência mOrange-Nano aumentou dentro da ROI. O padrão atinge a saturação em 120 s e a fluorescência diminui em 120 s, quase para níveis de fundo. Nenhuma perda de qualidade do padrão é observada em diferentes ciclos de luz azul/escuridão. A figura é adaptada de Di Iorio et al.8. Clique aqui para ver uma versão maior desta figura.
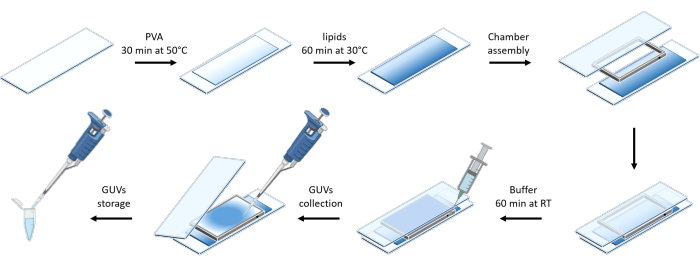
Figura 3: Representação esquemática da preparação dos GUVs usando o método de hidratação suave. O esquema oferece uma representação visual dos vários degraus e da câmara construída usando duas lâminas de vidro e um espaçador. Clique aqui para ver uma versão maior desta figura.
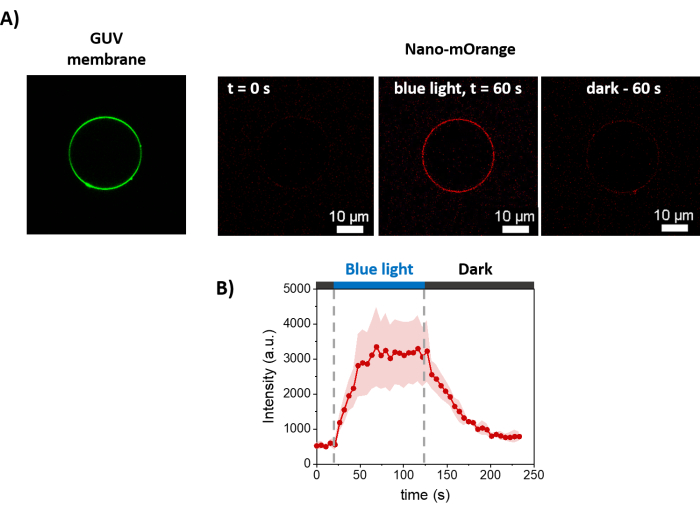
Figura 4: Medições de microscopia de fluorescência do recrutamento dependente de luz de mOrange-Nano em membranas GUVs. (A) Imagens de fluorescência de um GUV funcionalizado com disiLID na presença de mOrange-Nano. Em verde está o corante de membrana dos GUVs, e em vermelho está a fluorescência mOrange antes, durante e depois da iluminação da luz azul. Barras de escala = 10 μm. (B) Intensidade de fluorescência de mOrange localizada no GUV ao longo do tempo. Após a iluminação, a fluorescência mOrange (mostrada em vermelho) na membrana lipídica atinge a intensidade máxima em 60 s, com um aumento de 5,9 vezes na intensidade da fluorescência. À medida que a iluminação é interrompida, a fluorescência mOrange diminui para valores quase pré-iluminação em 60 s (com uma recuperação de 90%). A figura é adaptada de Di Iorio et al.8. Clique aqui para ver uma versão maior desta figura.
Discussion
Descrevemos um método para o recrutamento localizado de proteínas mOrange-Nano em membranas modelo, como bicamada lipídica suportada e vesículas unilamelares gigantes usando a proteína fotocomutável disiLID8. Aspectos que contribuem para a qualidade do padrão incluem a qualidade das proteínas, bem como a boa qualidade dos SLBs e GUVs.
Para garantir uma boa qualidade proteica após a expressão e purificação, é importante primeiro avaliar as propriedades fotocomutáveis do disiLID. Para isso, a absorção do cofator FMN deve ser medida no escuro e após a iluminação da luz azul. Espera-se que os espectros UV-Vis do disiLID mostrem o pico triplo característico do cofator FMN no escuro, que diminui significativamente com a iluminação da luz azul e se recupera no escuro13. Esse comportamento fotocomutável é crucial para obter recrutamento reprodutível e reversível nas etapas a seguir. Trabalhar com luz vermelha protetora e expor o disiLID à iluminação externa mínima durante a preparação das amostras aumenta o desempenho dos experimentos.
Outra etapa crítica, e talvez a mais crucial, é a formação de SLBs adequados. Defeitos nas membranas e / ou a formação de SLBs não homogêneos (ou seja, a presença de SLBs multicamadas ou remendados) afetarão a qualidade do padrão de proteína. Portanto, para usuários inexperientes, recomenda-se reproduzir o protocolo rotulando os SUVs com alguns corantes de membrana, como DiD e DiO, a fim de formar SLBs marcados com fluorescência. Desta forma, as propriedades e a qualidade dos SLBs podem ser bem caracterizadas com microscopia de fluorescência. As medições FRAP representam uma abordagem típica para avaliar a qualidade de um SLB, avaliando a fluidez das membranas. Alternativamente, no caso de SLBs biotinilados, como os descritos neste protocolo, o SAv marcado com fluorescência (por exemplo, Atto 488-SAv) pode ser usado para visualizar e avaliar a qualidade dos SLBs.
A primeira parte do protocolo descreve a formação de padrões em SLBs. Para garantir um resultado ideal, é importante adicionar mOrange-Nano aos SLBs e deixar a amostra incubar no escuro por 15 min. Durante a fotoativação, a seleção do ROI não se restringe a um tamanho específico. No entanto, a intensidade do laser e o tempo de exposição precisam ser regulados para reduzir o fotobranqueamento indesejado das proteínas fluorescentes.
Este método não se restringe a proteínas biotiniladas, e outras abordagens podem ser usadas para ancorar disiLID em SLBs. Por exemplo, o disiLID marcado com His pode ser expresso e ancorado em SLBs contendo Ni-NTA. No entanto, é crucial expressar Nano e disiLID com tags diferentes para evitar a substituição das proteínas nos SLBs. Este método também permite a possibilidade de inverter a ordem das proteínas, funcionalizando assim SLBs com Nano e recrutando disiLID (ou proteínas fundidas com disiLID) após iluminação de luz azul.
Para o controle dinâmico da localização da proteína, a localização reversível da proteína na região selecionada deve ser possível repetidamente. Para conseguir isso, a concentração de Nano (200 nM) na solução é um parâmetro crítico para obter alta reversibilidade.
Outra preocupação é o recrutamento de Nano para a superfície GUV funcionalizada com disiLID. Como no caso da padronização de proteínas em SLBs, esse método pode ser estendido a diferentes estratégias de funcionalização de membranas. Neste protocolo, todo o GUV foi iluminado com luz azul para recrutar mOrange-Nano em toda a superfície do GUV. No entanto, a seleção de pequenas ROIs localizadas na membrana GUV deve levar à localização precisa de proteínas em uma área mais restrita.
Este método apresenta apenas uma limitação relacionada à escolha do fluoróforo empregado para a imagem do recrutamento de Nano na membrana dos SUVs ou GUVs. Em particular, fluoróforos com um espectro de excitação na faixa de luz azul devem ser evitados, pois seu uso interferirá na fotoativação do (dis)iLID. Portanto, a escolha de fluoróforos na faixa de luz verde ou vermelha (por exemplo, mOrange ou Cy5) é recomendada para este tipo de experimento.
O design disiLID oferece uma maneira simples e adaptável de melhorar o recrutamento local de proteínas para as membranas e amplia a faixa dinâmica do iLID e Nano da optogenética4. Esses métodos se concentram no recrutamento de Nano em membranas mímicas, como bicamadas lipídicas e GUVs. No entanto, esta abordagem é extensível às inúmeras ferramentas optogenéticas em células onde (dis)iLID ou Nano estão ligados a uma membrana.
Disclosures
Os autores não têm nada a divulgar.
Acknowledgements
Este trabalho foi financiado pelo European Research Council ERC Starting Grant ARTIST (# 757593 S.V.W.). A DDI agradece à Fundação Alexander von Humboldt por uma bolsa de pós-doutorado.
Materials
| Name | Company | Catalog Number | Comments |
| µ-Slide 18 Well | Ibidi | 81817 | For SLB preparation |
| 25 µL Microliter Syringe | Hamilton | Model 702 N | For the preparation of lipid mixture and spreading the lipid solution on the PVA layer |
| Biotinyl Cap PE (1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl)) (sodium salt) | Avanti Polar Lipids | 870273C | For SUVs preparation |
| CaCl2 (Calcium chloride) | Sigma-Aldrich | C5670 | For SLB formation |
| Cover Slips 24 mm x 60 mm | Engelbrecht | K12460 | For GUVs formation |
| DiD (1,1'-Dioctadecyl-3,3,3',3'- Tetramethylindodicarbocyanine) | Thermo Fisher Scientific | D7757 | Membrane dye |
| disiLID | Sequence: MGGSGLNDIFEAQKIEWHEGGSH HHHHHGSMAATELRGVVGPGPAA IAALGGGGAGPPVGGGGGRGDA GPGSGAASGTVVAAAAGGPGPG AGGVAAAGPPAPPTGGSGGSGA GGSGSAGEFLATTLERIEKNFVIT DPRLPDNPIIFASDSFLQLTEYSR EEILGRNCRFLQGPETDRATVRK IRDAIDNQTEVTVQLINYTKSGKK FWNVFHLQPMRDYKGDVQYFIG VQLDGTERLHGAAEREAVMLIKK TAFQIAEAANDENYF | ||
| DOPC (1,2-di-(9Z-octadecenoyl)-sn-glycero-3-phosphocholine) | Avanti Polar Lipids | 850375P | For SUVs lipid composition |
| Eppendorf Protein LoBind microcentrifuge tubes | Merk | EP0030108116-100EA | For collecting freshly made GUVs |
| mOrange-Nano | Sequence: MRGSHHHHHHGSKIEEGKLVI WINGDKGYNGLAEVGKKFEKDT GIKVTVEHPDKLEEKFPQVAATG DGPDIIFWAHDRFGGYAQSGLLA EITPDKAFQDKLYPFTWDAVRYN GKLIAYPIAVEALSLIYNKDLLPNP PKTWEEIPALDKELKAKGKSALM FNLQEPYFTWPLIAADGGYAFKY ENGKYDIKDVGVDNAGAKAGLTF LVDLIKNKHMNADTDYSIAEAAFN KGETAMTINGPWAWSNIDTSKVN YGVTVLPTFKGQPSKPFVGVLSA GINAASPNKELAKEFLENYLLTDE GLEAVNKDKPLGAVALKSYEEELA KDPRIAATMENAQKGEIMPNIPQM SAFWYAVRTAVINAASGRQTVDEA LKDAQTNSSSNNNNNNNNNNLGI EGTTENLYFQGSVSKGEENNMAI IKEFMRFKVRMEGSVNGHEFEIE GEGEGRPYEGFQTAKLKVTKGG PLPFAWDILSPQFTYGSKAYVKH PADIPDYFKLSFPEGFKWERVMN FEDGGVVTVTQDSSLQDGEFIYK VKLRGTNFPSDGPVMQKKTMG WEASSERMYPEDGALKGEIKMR LKLKDGGHYTSEVKTTYKAKKPV QLPGAYIVGIKLDITSHNEDYTIVE QYERAEGRHSTGGMDELYKGG SGTSSPKRPKLLREYYDWLVDN SFTPYLVVDATYLGVNVPVEYVK DGQIVLNLSASATGNLQLTNDFIQ FNARFKGVSRELYIPMGAALAIYA RENGDGVMFEPEEIYDELNIG | ||
| NaCl (Sodium chloride) | Sigma-Aldrich | S9888 | For buffer |
| NaOH (Sodium hydroxide) | Sigma-Aldrich | 1064980500 | For surface activation in SLB formation |
| POPC (1-Palmitoyl-2- oleoylphosphatidylcholine) | Avanti Polar Lipids | 850457C | For GUVs lipid composition |
| POPG (1-palmitoyl-2-oleoyl-sn-glycero-3-phospho-(1'-racglycerol)) | Avanti Polar Lipids | 840457C | For GUVs lipid composition |
| PVA (Polyvinyl alcohol) fully hydrolyzed | Sigma-Aldrich | 8148940101 | For GUVs formation |
| SP8 confocal laser scanning microscope | Leica | ||
| Streptavidin | TermoFisher | 434301 | |
| Sucrose | Sigma-Aldrich | 84097 | For GUVs formation |
| Tris hydrochloride | Sigma-Aldrich | 10812846001 | For buffer |
References
- Kretschmer, S., Schwille, P. Pattern formation on membranes and its role in bacterial cell division. Curr Opin Cell Biol. 38, 52-59 (2016).
- Yang, H. W., Collins, S. R., Meyer, T. Locally excitable Cdc42 signals steer cells during chemotaxis. Nat Cell Biol. 18 (2), 191-201 (2016).
- Caldwell, R. M., et al. Optochemical control of protein localization and activity within cell-like compartments. Biochem. 57 (18), 2590-2596 (2018).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Guntas, G., et al. Engineering an improved light-induced dimer (iLID) for controlling the localization and activity of signaling proteins. Proc Natl Acad Sci. 112 (1), 112-117 (2015).
- Levskaya, A., Weiner, O. D., Lim, W. A., Voigt, C. A. Spatiotemporal control of cell signalling using a light-switchable protein interaction. Nature. 461 (7266), 997-1001 (2009).
- Bartelt, S. M., et al. Dynamic blue light-switchable protein patterns on giant unilamellar vesicles. Chem Commun. 54 (8), 948-951 (2018).
- Di Iorio, D., Bergmann, J., Higashi, S. L., Hoffmann, A., Wegner, S. V. A disordered tether to iLID improves photoswitchable protein patterning on model membranes. Chem. Commun. 59 (29), 4380-4383 (2023).
- Hartzell, E. J., Terr, J., Chen, W. Engineering a blue light inducible Spytag system (BLISS). J Am Chem Soc. 143 (23), 8572-8577 (2021).
- Xu, D., Bartelt, S. M., Rasoulinejad, S., Chen, F., Wegner, S. V. Green light lithography: a general strategy to create active protein and cell micropatterns. Mater Horiz. 6 (6), 1222-1229 (2019).
- Jia, H., et al. Light-induced printing of protein structures on membranes in vitro. Nano Lett. 18 (11), 7133-7140 (2018).
- Di Iorio, D., Verheijden, M. L., vander Vries, E., Jonkheijm, P., Huskens, J. Weak Multivalent Binding of influenza hemagglutinin nanoparticles at a sialoglycan-functionalized supported lipid bilayer. ACS Nano. 13 (3), 3413-3423 (2019).
- Kasahara, M., Torii, M., Fujita, A., Tainaka, K. FMN binding and photochemical properties of plant putative photoreceptors containing two LOV domains, LOV/LOV proteins. J Biol Chem. 285 (45), 34765-34772 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved