Avaliação rápida glicoqualitativa de proteínas recombinantes usando um sistema totalmente automatizado
In This Article
Summary
Um sistema para a análise automatizada e rápida de glicanos em proteínas foi desenvolvido anteriormente. Este artigo apresenta o protocolo para "análise glicoqualitativa", otimizado para um amplo espectro de usuários, como aqueles envolvidos na análise de estruturas de glicanos em biofármacos e outros materiais glicoconjugados.
Abstract
A glicosilação de proteínas, uma modificação pós-traducional crítica, influencia a estabilidade, eficácia e imunogenicidade de proteínas recombinantes, incluindo biofármacos. As estruturas de glicanos exibem heterogeneidade significativa, variando com os tipos de células de produção, condições de cultura e métodos de purificação. Consequentemente, o monitoramento e a avaliação das estruturas glicanos das proteínas recombinantes são vitais, principalmente na produção biofarmacêutica. O microarray de lectina, uma técnica complementar à espectrometria de massa, possui alta sensibilidade e facilidade de uso. No entanto, normalmente requer mais de um dia para produzir resultados. Para adaptá-lo à pesquisa não glicocientífica ou ao desenvolvimento de processos de medicamentos, é necessária uma alternativa automatizada e de alto rendimento. Portanto, foi desenvolvido o primeiro sistema de perfil de glicano baseado em lectina totalmente automatizado do mundo, utilizando o conceito de tecnologia "matriz de esferas em uma única ponta (BIST)". Este sistema permite a preparação e armazenamento de grânulos imobilizados por lectina em unidades de 1.000, com pedidos de inserção paralelos personalizáveis para diversos fins. Este artigo apresenta um protocolo prático para pesquisa envolvendo proteínas recombinantes "glico-qualificadas". Depois de testar sua reatividade contra 12 conjugados de poliacrilamida-glicano, 15 lectinas foram selecionadas para aumentar a versatilidade do sistema. Além disso, o processo de rotulagem da amostra foi otimizado com a mudança de Cy3 para biotina, reduzindo o tempo total de processamento em 30 minutos. Para qualificação imediata dos dados, os sinais de ligação à lectina são exibidos como um código de ponto no monitor superior. A confiabilidade do sistema foi confirmada por meio de testes diários de reprodutibilidade, testes de repetibilidade e testes de armazenamento de longo prazo, com um coeficiente de variação de <10%. Este glicoanalisador rápido e fácil de usar tem aplicações potenciais no monitoramento da qualidade de glicoproteínas endógenas para avaliação e validação de biomarcadores. Este método facilita a análise para aqueles que são novos na glicociência, ampliando assim sua utilidade prática.
Introduction
A glicosilação de proteínas é uma modificação pós-traducional crucial que precisa ser avaliada em biofármacos. Os perfis glicanos das proteínas podem variar com base nas condições de cultura, processos de purificação e células hospedeiras1. Instrumentos simples são necessários para qualificar a glicosilação dentro do pipeline de bioprocessos. Estima-se que mais de 50% das proteínas secretadas e de membrana in vivo sejam modificadas com glicanos múltiplos, que mudam de acordo com a linhagem celular, estágio de desenvolvimento e estado da doença, como o início da malignidade2. O monitoramento de perfis de glicanos tem um potencial significativo para identificar marcadores diagnósticos exclusivos e alvos de medicamentos. Instrumentos automatizados capazes de medir rapidamente tamanhos de amostras em larga escala estão em alta demanda para verificar e validar essa glicosilação aberrante de centenas de amostras de pacientes no pipeline de descoberta.
A tecnologia de microarray foi incorporada à glicômica para avaliar o perfil glicano das glicoproteínas3. Neste método, várias lectinas, que são proteínas de ligação ao glicano, são imobilizadas em uma superfície como uma lâmina de vidro. Essa tecnologia de análise de glicose baseada em interação não exige a liberação prévia de glicanos das proteínas centrais, simplificando o processo para pesquisadores novos na glicotecnologia. Apesar de seu uso generalizado, para aplicações industriais como a bioprodução, era necessário um sistema automatizado capaz de monitorar glicanos de forma rápida e fácil para um número maior de alvos de análise. Para resolver isso, um sistema automatizado de perfil de glicano baseado em um conceito único denominado "matriz de esferas em uma única ponta" (BIST), inicialmente desenvolvido para genotipagem, foi relatado anteriormente. Este sistema simplifica o processo com um auto-instrumento de alto rendimento do tipo caixaúnica 4. Usando pontas nas quais vários grânulos fixados por lectina são dispostos em paralelo 4,5, um método para analisar estruturas de glicanos modificadas em glicoproteínas foi estabelecido e denominado GlycoBIST (doravante denominado "sistema automático de perfil de glicanos") (Figura 1A). As lectinas podem ser fixadas em 1.000 grânulos e secas para manter a atividade por um ano, antes e depois de embalar uma ponta. Uma vez que as pontas e cartuchos contendo reagentes como o anticorpo antiestreptavidina marcado com HRP (SA-HRP) são colocados no instrumento protótipo de medição (um dispositivo de medição de reação automatizado, consulte a Tabela de Materiais), a ponta funciona como uma autopipeta. Um scanner de detecção de quimioluminescência na parte traseira interna do instrumento quantifica os sinais de oito pontas simultaneamente. Os dados quantitativos dessas oito pontas são exibidos de forma compacta e simultânea como códigos de pontos na tela sensível ao toque do instrumento para confirmação rápida dos resultados da medição. Além disso, o valor representado como o máximo do pico medido é transportado do instrumento como dados brutos e permite a representação gráfica por pesquisadores individuais (Figura 1A, painel inferior).
Neste artigo, os autores descrevem um método aprimorado de marcação de proteínas com biotina, reduzindo o tempo de processamento para 30 min. As proteínas-alvo são biotiniladas antecipadamente e detectadas por SA-HRP ( Figura 1B ). Uma ponta GlycoBIST padrão (ponta especializada para perfil automático de glicanos) com 15 lectinas selecionadas foi construída para obter o perfil versátil de glicoproteína glicano para abrangência analítica.
Protocol
Os detalhes dos reagentes e do equipamento usado para este estudo estão listados na Tabela de Materiais.
1. Preparação da ponta especializada para perfil automático de glicanos
- Levar as esferas secas fixadas em lectina, armazenadas a 4 °C num saco selado, para a bancada e deixá-las voltar à temperatura ambiente (~23 °C).
NOTA: Isso evita a condensação nas contas, o que pode diminuir sua qualidade. Este método de preparação de grânulos fixadores de lectina foi relatado anteriormente5. - Coloque um pano antiestático sobre a mesa e coloque o eliminador estático portátil de frente para o espaço de trabalho. Isso permite que o trabalho subsequente seja realizado na bancada com proteção antiestática (Figura 2A).
- Insira as contas na ponta vazia usando uma pinça antiestática, de modo que duas contas espaçadoras sejam colocadas entre as contas de fixação de lectina (Figura 1A).
- Aperte suavemente a parte superior da ponta com pinças para fechar a abertura, tomando cuidado para não cortar a ponta depois que todas as contas estiverem preenchidas (Figura 2B).
NOTA: Isso é para prender as contas dentro da ponta. Recomenda-se que a pinça seja trabalhada para evitar cortar a ponta (Figura 2C). - Endireite a ponta com a mão se estiver dobrada.
- Conservar a ponta preparada a 4 °C num saco selado com exsicante até ser necessária.
NOTA: Antes de usar a ponta, ela deve ser levada à temperatura ambiente antes de abrir o saco lacrado e retirá-lo. A umidade reduz drasticamente a reatividade das lectinas.
2. Preparação das substâncias a analisar
- Pré-embale o reagente de biotinilação para análise de rotina.
NOTA: Para análises de rotina, é útil pré-embalar o reagente de biotinilação (Biotina-(AC5)2 Sulfo-Osu, consulte a Tabela de Materiais) em pequenas porções da seguinte forma para uma análise rápida. Recomenda-se que o reagente de biotinilação seja seco e armazenado, pois seu éster succinimidílico é geralmente suscetível à hidrólise.- Dissolver o reagente de biotinilação em água desionizada.
- Dispensar 10 μg do reagente em tubos utilizando uma pipeta.
- Secar os tubos com reagente de biotinilação num concentrador de vácuo refrigerado e conservá-los com um exsicante a 4 °C num saco resistente à luz até à utilização.
- Adicionar a solução a analisar (200 ng de proteína eq.) ao tubo que contém o reagente de biotinilação desidratado preparado no passo 2.1.
NOTA: Esteja atento à composição do tampão usada para dissolver e diluir os analitos, dependendo das propriedades do reagente de biotinilação usado. Por exemplo, evite usar reagentes de amina primária como tampões para acoplamento de amina. Isso evitará uma redução na eficiência de biotinilação dos analitos. - Misture bem por vórtice e, em seguida, gire para baixo (1000 x g por 5 s à temperatura ambiente) em uma centrífuga de mesa para soltar o líquido da tampa no fundo do tubo.
- Incubar os tubos durante 1 h à temperatura ambiente, protegidos da luz.
- Dilua as amostras biotiniladas 10 vezes no tampão de sondagem (1% Triton X-100, 0,5 M Glycine, 1 mM CaCl2 e 1 mM MnCl2 em TBS) para inativar quaisquer reagentes de biotinilação não reagidos.
NOTA: No caso de reações por acoplamento de amina, o reagente de biotinilação não reagido é consumido pela reação com um tampão contendo um reagente de amina primário como Tris em TBS, que impede a biotinilação de lectinas e outras substâncias imobilizadas nas esferas na medição subsequente. - Incubar à temperatura ambiente, protegido da luz, durante 2 h.
- Avaliar a eficiência da biotinilação dos analitos através de Western blotting, utilizando estreptavidina marcada com HRP como sonda de detecção, se necessário. Consulte a literatura para métodos de Western blotting6.
3. Preparação de reagentes para perfil de glicano com o dispositivo automatizado de medição de reação
- Diluir a estreptavidina conjugada com HRP com a solução de bloqueio na proporção de 1:3000.
- Misture com volumes iguais de Substrato A e Substrato B (consulte a Tabela de Materiais). Use 160 μL de substrato misto por amostra.
- Dispense a solução de bloqueio, tampão de lavagem (0,1% Triton X-100 / TBS), TBS, solução de estreptavidina conjugada com HRP e cada substrato nos poços designados de um cartucho com 10 reservatórios de líquido.
NOTA: O volume recomendado e a colocação das soluções para encher cada poço do cartucho de reação são descritos abaixo. No entanto, as soluções e a disposição do poço podem ser alteradas no dispositivo de medição de reação automatizado conforme apropriado com antecedência.- Adicione 0,2 mL de estreptavidina marcada com HRP preparada na etapa 3.1. para o poço #2 usando uma pipeta. O poço é nomeado # 1 na frente do cartucho.
- Adicione 0,2 mL de solução de bloqueio ao poço #3 usando uma pipeta.
- Adicione 1 mL de TBS aos poços #4 e #6 usando uma pipeta.
- Adicione 1 mL de tampão de lavagem aos poços #7 e #8 usando uma pipeta.
- Adicione 0,16 mL de mistura de substrato ao poço #10 usando uma pipeta.
- Confirme se os poços #1, #5 e #9 estão vazios.
- Adicione os analitos preparados na etapa 2 a um microtubo de tampa de rosca com baixo teor de proteína de 2 mL.
- Abra a porta frontal do dispositivo de medição de reação automatizado.
- Definir os reservatórios de líquido preparados no passo 3.3, as pontas preparadas no ponto 1 e as substâncias a analisar preparadas no passo 3.4 nas posições designadas do dispositivo de medição de reacção automatizado (figura 2D) do seguinte modo. Cada medição é realizada em uma única direção de linha vertical.
- Coloque uma ponta preparada na etapa 1 em cada orifício na posição "a" símbolo amarelo na Figura 2D.
NOTA: Novamente, se refrigerado, leve as pontas à temperatura ambiente antes de abrir o saco lacrado. As lectinas perdem seu sinal drasticamente devido à umidade. - Retire a tampa do tubo que contém a substância a analisar preparada no passo 3.4 e introduza-a em cada orifício na posição "b" de forma semelhante.
- Insira o cartucho que contém o líquido preparado no passo 3.3, tomando cuidado para não derramá-lo na posição "c".
- Remova toda a "caixa de lixo" na Figura 2D quando estiver cheia, despeje o conteúdo na lixeira e retorne a "caixa de lixo" à sua posição original.
NOTA: As pontas reagidas são coletadas automaticamente na "caixa de resíduos" após a medição.
- Coloque uma ponta preparada na etapa 1 em cada orifício na posição "a" símbolo amarelo na Figura 2D.
4. Perfil de glicano com o dispositivo de medição de reação automatizado
- Ligue o interruptor de alimentação na lateral da unidade principal.
- Toque no botão "Iniciar diagnóstico" exibido na tela (Figura 3A). O programa de autodiagnóstico é iniciado automaticamente. Depois de um tempo, o programa de autodiagnóstico é concluído e a tela HOME aparece (Figura 3B).
NOTA: As seguintes operações podem ser realizadas tocando nos respectivos ícones. "Ensaio" para iniciar a medição. "Manutenção" para confirmar qualquer erro do sistema. "Histórico" para visualizar dados de medição anteriores. - Toque em "Ensaio" exibido na tela HOME para selecionar o protocolo (Figura 3B).
- Selecione o "BAssaySTD" do método de ensaio (Figura 3C) e pressione "Avançar" no canto inferior direito.
- Digite o nome da amostra (Figura 3D) para cada amostra conforme necessário e toque em "Avançar" no canto inferior direito.
- Digite o nome da ponta (Figura 3E) usado para cada amostra conforme necessário e toque em "Avançar" no canto inferior direito.
- Confirme as notas à direita (Figura 3F) e, se não houver problema, toque em "Iniciar" no canto inferior direito para medição.
- Confirme a mudança da tela para o modo de execução (Figura 3G) e aguarde até que a seta "finalizar" seja destacada. Se necessário, toque em "Break" para interromper o modo de execução.
NOTA: As operações controladas automaticamente pelo dispositivo automatizado de medição de reação são as seguintes: (1) Os bicos de micropipeta automática dentro do instrumento conectam no máximo oito pontas simultaneamente (consulte o símbolo amarelo "a" na Figura 2D); (2) Aspirar / descarregar repetidamente a solução no tubo que contém o analito por 5 min; (3) Transferir para a solução de bloqueio e aspirar/descarregar durante 5 min; (4) Transferir para TBS e aspirar/descarregar por 2 min; (5) Transferir para a solução de estreptavidina marcada com HRP e aspirar/descarregar 5 min; (6) Transfira para o tampão de lavagem e aspire / descarregue por 7 min; (7) Transferir para TBS e aspirar/descarregar por 2 min; (8) Transfira para a mistura de substrato e aspire imediatamente; (9) Escaneie a quimioluminescência de oito pontas simultaneamente na parte traseira do dispositivo para obter dados de pico; (10) Coloque as pontas usadas na caixa de resíduos; (11) O bico retorna à posição inicial. - Confirme se a tela mudou para o modo de resultado (Figura 3H) quando a medição for concluída. Os oito códigos de pontos são exibidos simultaneamente na tela para facilitar a verificação.
NOTA: Toque em "Histórico" no canto inferior esquerdo para exibir o gráfico de varredura (Figura 3I, J), gráfico de barras (Figura 3K, L), tabela (Figura 3M) e detalhes (Figura 3N) como resultado na tela. Visualize os resultados de outras faixas na lista suspensa "Pista". - Insira uma memória USB na porta USB na lateral da unidade principal e toque em "Exportar" no canto inferior direito do modo de resultado para salvar os dados de todas as pistas individualmente na memória USB.
- Exporte os dados brutos (Figura 3M) como um arquivo CSV usando uma porta USB para análise de dados adicional e gráficos individuais no Excel. "Valor" da tabela denota a intensidade do sinal de cada lectina. Os valores de intensidade representam o valor máximo dos dados de pico (Figura 3I,J) na varredura das pontas reagidas.
NOTA: O limite de extração de pico pode ser definido. Dependendo dos analitos, o sinal na porção do cordão espaçador pode ser maior no geral, caso em que o valor na parte do espaçador é subtraído como plano de fundo.
Representative Results
Primeiro, a distribuição do sinal é mostrada para cada lectina para entender as características da matriz de esferas de lectina. Uma biblioteca de contas fixadas em lectina (1.000 contas cada) foi preparada. Quinze contas foram escolhidas aleatoriamente entre as 1.000 e adicionadas a uma única ponta. Um gráfico típico é mostrado na parte inferior da Figura 1A, representando quase a mesma intensidade de sinal observada entre as 15 esferas que imobilizam a mesma lectina. Este procedimento de medição pode ser aplicado para qualificar a produção de grânulos de lectina feitos à mão antes do uso do perfil de glicano. Por exemplo, seis pontas são usadas para a medição simultânea para calcular o coeficiente de variação (CV) a partir dos sinais nas 90 contas. Um exemplo típico é mostrado na Tabela 1. Quando a biblioteca de grânulos fixados por lectina foi expandida para acomodar 28 lectinas diferentes, 25 das 28 lectinas demonstraram alta reprodutibilidade com um CV inferior a 10%.
Em seguida, os dados de uma matriz de esferas de lectina com 15 lectinas diferentes são mostrados. As 15 lectinas mostradas na Tabela 2 foram selecionadas para o arranjo de contas de lectina. Além disso, foram medidos 12 mono/di/trissacarídeos de poliacrilamida (PAA) listados na Tabela 3. Depois de avaliar a confiabilidade da medição usando uma tela de dotcoding na tela, os dados brutos foram exportados do dispositivo de medição de reação automatizado (consulte a etapa 4.11 do protocolo) e um gráfico foi criado no Excel usando os valores de pico de reação em cada sacarídeo (Figura 4A). Esses valores foram então usados para análise de componentes principais para visualizar a correlação entre as 15 lectinas selecionadas e os sacarídeos reagidos (Figura 4B,C). Isso resultou em uma diferença distinta no padrão de ligação de cada lectina contra os 12 açúcares PAA mostrados na Figura 4A. Aqui, a validade de cada sinal é mostrada para se referir a listas anteriores integradas de especificidades de lectina e sacarídeo 7,8 (consulte também essas especificidades na Tabela 2). À medida que o AAL se liga ao terminal α-Fuc, Sia-Lex e Lex, a análise automática de glicanos confirmou o reconhecimento do AAL de moléculas contendo fucose (Figura 4A). SSA e MAL (consulte a Tabela 2 para os nomes completos das lectinas e abreviaturas fornecidas) reconheceram Siaα2-6Gal / GalNAc e Siaα2-3Gal, respectivamente. O MAL na matriz de esferas também pode se ligar a 3'-O-sulfo-Galβ3GalNAc e LacNAc. Isso é consistente com resultados anteriores analisados usando aprendizado de máquina7, em que o MAL prefere o 3'-O-sulfo-Gal dominantemente, enquanto o LacNAc não é dominante9. RCA120, uma lectina de reconhecimento de Gal, reagiu fortemente com β-Gal terminal, incluindo Lac / LacNAc, e foi permitido se ligar a Siaα2-6Lac, mas não a Siaα2-3Lac. ECA teve reatividade com LacNAc muito maior do que Lac. WFA teve uma especificidade mais ampla, que reconheceu não apenas α/β-GalNAc, mas também β-Gal terminal, conforme relatado 7,10. O GSL II, que reconhece GlcNAc e N-glicanos tri/tetra-antenares galactosilados, não se ligou a nenhum sacarídeo, exceto por um único GlcNAc entre os analitos preparados. O GRFT, que reconhece altos N-glicanos do tipo manose, reagiu com o α-Man. ABA, Jacalina e MAH têm sido geralmente usados para detectar O-glicanos. Como o ABA prefere as estruturas Galβ1-3GalNAcα-Thr/Ser (T) e sialil-T, o ABA na matriz de esferas reagiu fortemente com 3'-O-sulfo-Galβ1-3GalNAc e fracamente ligado a α-GalNAc. A ABA também reconheceu β-GlcNAc, consistente com os relatórios anteriores sobre a ligação a N-glicanos agalactosilados7. A jacalina tem uma especificidade relativamente mais ampla e, portanto, reagiu com α-manose, β-GalNAc, βGal, α-GalNAc, 3'-O-sulfo-Galβ1-3GalNAc. O MAH reconheceu especificamente 3'-O-sulfato-Galβ1,3GalNAc conforme relatado anteriormente7. As quatro lectinas residuais, NPA, LCA, PHA-L e PHA-E, reconheceram a estrutura interna do N-glicano não incluído nos sacarídeos PAA preparados e, portanto, não tiveram afinidade com todos os analitos.
Usando os dados obtidos, a semelhança das lectinas que contribuem para a ligação a cada sacarídeo pode ser esclarecida plotando-as em um gráfico de análise de componentes principais (Figura 4B). Todos os sacarídeos que contribuíram para a variância na Figura 4B foram representados por autovetor, um método de análise estatística no qual a razão de contribuição é representada por um vetor (Figura 4C). Vale ressaltar aqui que os sacarídeos reagidos contribuem para cada lectina sem viés. As lectinas não exibem especificidade para apenas um tipo de sacarídeo. Por exemplo, a presença de glicanos de tipo complexo é determinada pelo reconhecimento parcial de alguns glicanos dentro de glicanos de tipo complexo em humanos. A jacalina, que reconhece os O-glicanos (ver as especificidades na Tabela 2), pode reconhecer a estrutura 3'suTF e o α-GalNAc entre os sacarídeos usados neste estudo e, portanto, o 3'suTF e o α-GalNAc foram plotados (Figura 4C) contra a direção da posição da Jacalina (ponto marrom) plotada na Figura 4B. A WFA pode reconhecer α/β-GalNAc entre os sacarídeos usados neste estudo; portanto, α/β-GalNAc na Figura 4C foi plotado na mesma direção que o WFA plotado na Figura 4B. Em contraste, α-GalNAc é plotado entre WFA e Jacalin porque é uma estrutura de sacarídeo à qual Jacalin é capaz de se ligar facilmente. Todos os 11 sacarídeos plotados neste estudo são consistentes com a especificidade (ligação ao sacarídeo) das lectinas relatadas anteriormente 7,8, indicando que o método proposto é um método de medição confiável. Além disso, os autovetores correspondentes a cada sacarídeo são dispersos, apoiando que as 15 lectinas foram selecionadas com viés mínimo para garantir uma cobertura abrangente na análise. Consequentemente, a ponta reabastecida com essas 15 lectinas foi definida como a ponta GlycoBIST padrão (Tabela 2).
No exemplo mais prático, os produtos proteicos purificados foram submetidos à medição da ponta (Figura 5). A tireoglobulina bovina, que possui um sítio de N-glicosilação e contém N-glicanos complexos, híbridos e com alto teor de manose11, foi analisada. A ponta padrão indicou aumento da reatividade de certas lectinas aos sacarídeos8 (consulte as especificidades na Tabela 2): PHA-E, que reconhece a bissetriz de GlcNAc; GRFT, reconhecendo N-glicanos do tipo manose; SSA, reconhecendo Siaα2-6Gal / GalNAc; RCA120, reconhecendo lactose e LacNAc; e AAL, reconhecendo fucose. Essas estruturas de glicanos estão presentes em N-glicanos complexos, híbridos e de alto tipo manose, sugerindo que a estrutura de N-glicanos da tireoglobulina11 pode ser efetivamente avaliada. Além disso, a tireoglobulina tratada com sialidase A, uma enzima que digere o ácido siálico, mostrou diminuição da reatividade com SSA, que reconhece o ácido siálico, e aumento da reatividade com RCA120, WFA e ECA, que são mais prontamente reconhecidos após a remoção dos ácidos siálicos. A presença de estruturas de O-glicanos na tireoglobulina bovina não foi relatada anteriormente. Como esperado, não houve reconhecimento de O-glicano na tireoglobulina com ou sem tratamento com sialidase na análise automática de glicanos.
Um teste de reprodutibilidade dentro da execução usando as 15 lectinas selecionadas é mostrado para entender a robustez da medição (Tabela 4). Para este teste, sete pontas padrão para analitos e uma ponta adicional para controle negativo foram preparadas, e todas as oito pontas foram medidas simultaneamente. Este procedimento foi repetido três vezes em um único dia. "Mix Analittes", uma combinação de analitos que levou à obtenção de sinais significativos em todas as lectinas, foram preparados para o controle de qualidade da matriz de esferas. Os analitos de mistura foram formulados misturando adequadamente eritropoietina (EPO) digerida por sialidase, EPO digerida por sialidase e galactosidase, IgA humana, metaloproteinase 3 da matriz (MMP3) e tireoglobulina. Cada glicoproteína (200 ng) foi marcada com 10 μg de reagente de biotinilação em PBS contendo Triton X-100. Se necessário, os tratamentos com sialidase e galactosidase foram realizados conforme descrito nas instruções. Após a digestão, os produtos foram incubados em um bloco de calor a 75 °C por 10 min para inativar a enzima. As quantidades apropriadas de glicoproteínas biotiniladas (3 ng de EPO, 8 ng de IgA humana, 5 ng de MMP3 e 15 ng de tireoglobulina) foram misturadas imediatamente antes da análise. Os resultados das medidas indicaram que o valor máximo de CV foi de 13,5% e o CV médio para as 15 lectinas foi de 8,2%, demonstrando alta reprodutibilidade.
Além disso, um teste de reprodutibilidade diário é mostrado para entender as variações devido à data de medição (Tabela 5). As pontas padrão e os analitos de mistura foram preparados com antecedência, e as medições de sete pontas foram repetidas diariamente por cinco dias consecutivos. Os resultados mostraram que a maioria das lectinas tinha um CV inferior a 10%. No entanto, algumas lectinas, como LCA e ECA, exibiram CVs mais altos. O CV médio das 15 lectinas por ciclo foi de até 7,7%, e os valores médios de CV em 5 dias foram inferiores a 10%, significando alta reprodutibilidade. Observou-se que a Jacalina, devido à autodigestão de MMP3 no Analito Mix, demonstrou baixa reprodutibilidade temporal. Portanto, um analito mais adequado deve ser identificado para substituir o MMP3.
Para entender a estabilidade dos grânulos fixados por lectina, um teste de estabilidade de longo prazo é mostrado usando esses grânulos fixados em lectina secos (Tabela 6). Pontas padrão, reagentes de ensaio e analitos de mistura foram preparados com antecedência e as medições foram realizadas após 12 meses de armazenamento. Os cinco tipos de analitos biotinilados acima mencionados foram armazenados individualmente e misturados antes da medição. Os resultados mostraram que o CV médio para as 15 lectinas foi inferior a 10%, mesmo até 12 meses após o armazenamento seco. Isso sugere a viabilidade de medições rápidas e precisas, armazenando várias pontas (até 1.000) do mesmo lote de uma só vez.
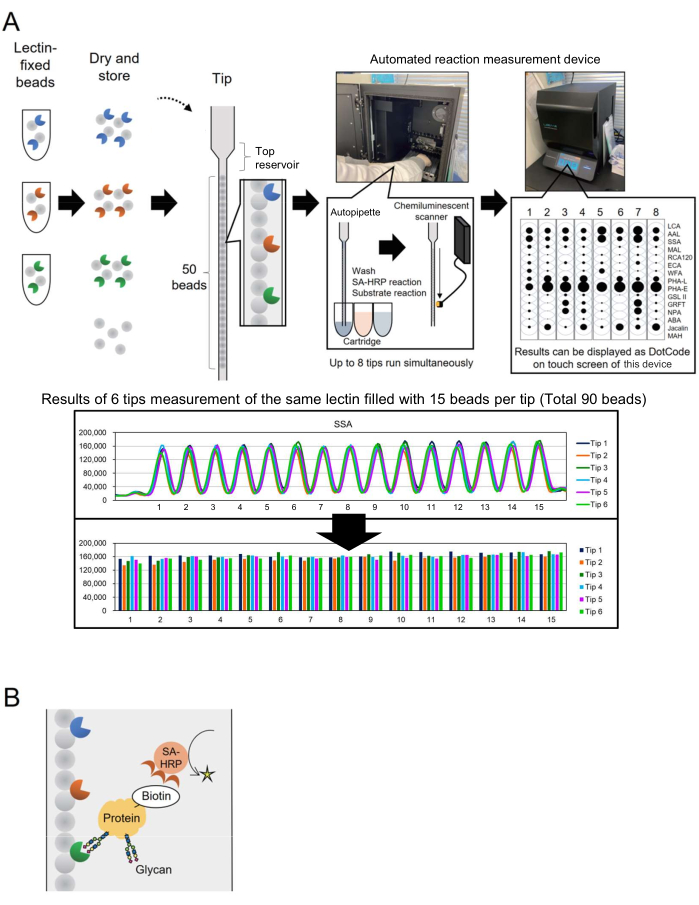
Figura 1: Esquemas do instrumento "bead array in a single tip" e método de análise. (A) Esquema do sistema automático de perfil de glicano. As esferas fixadas por lectina podem ser secas e armazenadas em pontas. Quando pontas e cartuchos contendo reagentes como o anticorpo antiestreptavidina marcado com HRP (SA-HRP) são colocados no instrumento de medição (dispositivo de medição de reação automatizado) e ativados, a ponta funciona como uma autopipeta. O scanner de detecção de quimioluminescência na parte traseira do instrumento quantifica os sinais de oito pontas simultaneamente. Os dados quantitativos são exibidos como códigos de pontos na tela sensível ao toque do instrumento para confirmação rápida dos resultados da medição. A seção inferior mostra os resultados da medição de 90 contas com a mesma lectina. Os resultados da medição são transportados do instrumento e representados graficamente pelo pesquisador individual. (B) Esquema do método de detecção usado neste experimento. As proteínas-alvo são pré-biotiniladas e detectadas usando SA-HRP. Clique aqui para ver uma versão maior desta figura.

Figura 2: Imagens de materiais e configuração para perfil automático de glicano com o dispositivo de medição de reação automatizado. (A) Representação de grânulos de fixação de lectina sendo embalados em uma ponta. Pegando as contas com pinças antiestáticas no tapete antiestático e enchendo a ponta com as contas. (B) Beliscar a ponta com uma pinça. Uma ligeira parte superior do cordão superior é espremida. (C) Uma pinça para cravar as pontas. O plástico (círculo amarelo) é comprimido para evitar que eles cortem completamente. (D) Disposição dos materiais usados no dispositivo automatizado de medição de reação. A: pontas, B: tubo contendo analitos e C: cartuchos contendo líquido devem ser inseridos no lugar, respectivamente. Na caixa de resíduos, as pontas reagidas são coletadas após a medição. A caixa pode ser removida. Clique aqui para ver uma versão maior desta figura.
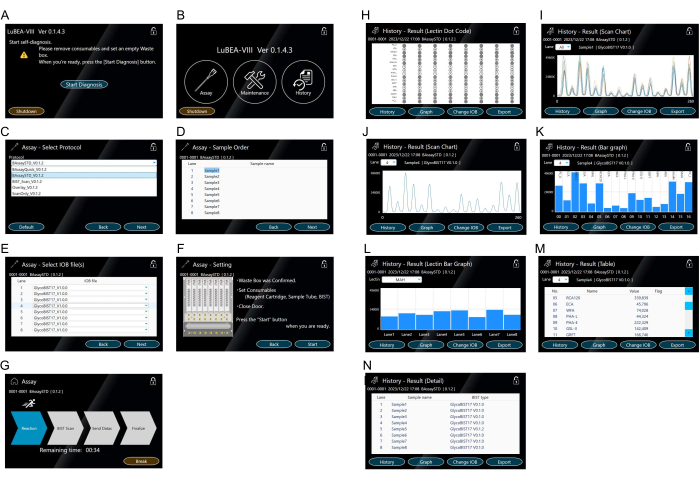
Figura 3: Tela sensível ao toque para manipulação da máquina. (A) O programa de autodiagnóstico inicia automaticamente quando a energia é ligada. (B) A tela HOME exibe "Ensaio", "Manutenção" e "Histórico". (CG) Operação do modo "Ensaio". Seleção do método de ensaio (C). Vários protocolos podem ser registrados com antecedência. Entrada da ordem da amostra (analito) (D). Selecionando o tipo de ponta (E). Confirmação das configurações do método de ensaio (F) e início do ensaio na tela "Iniciar ensaio" (G). (H-N) Representação do resultado em "História". Código de ponto da lectina (H) exibido após a conclusão do ensaio ou no modo "Histórico". Um gráfico de varredura de 15 lectinas é exibido na tela para todas as amostras (I) ou para cada amostra (J). O gráfico de varredura é exibido como um gráfico de barras (K). Os gráficos de barras para outras faixas podem ser selecionados na lista suspensa "Pista". Gráfico de barras comparando oito pistas de amostras por lectina (L). Outras lectinas podem ser selecionadas na lista suspensa "Lectina". Tabela mostrando valores quantitativos (M). Registro do método de medição executado (N). Clique aqui para ver uma versão maior desta figura.
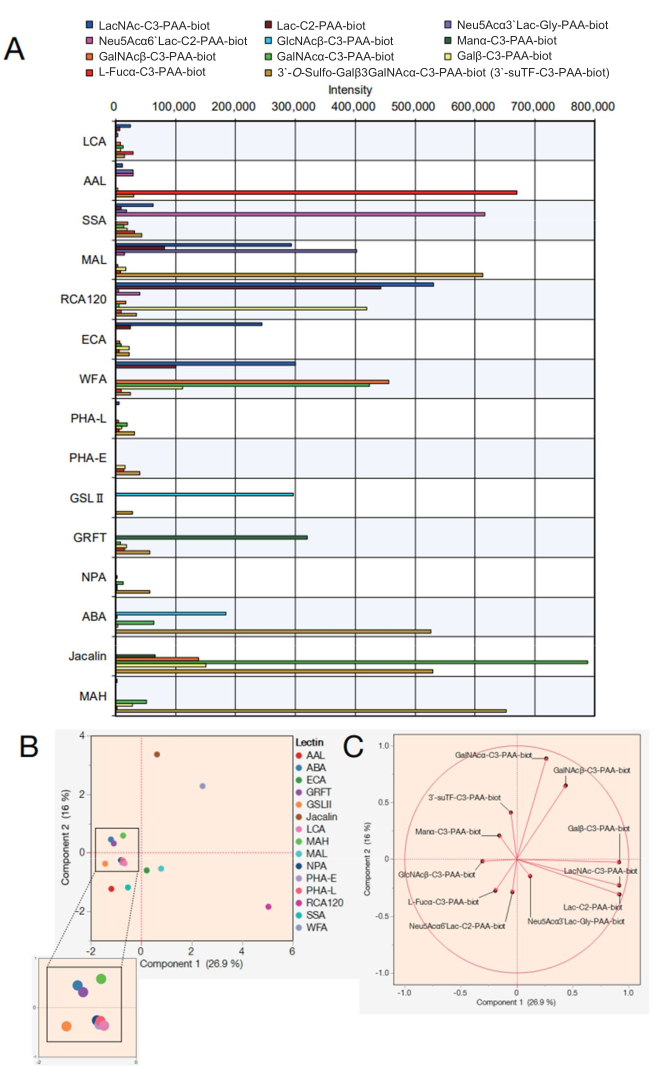
Figura 4: Avaliação da reatividade com sacarídeos PAA no sistema automático de perfil de glicanos. (A) Um gráfico representando a reatividade de lectinas com vários sacarídeos PAA foi ajustado para valores de controle negativos. (B) Análise de componentes principais com base nos dados em (A). (C) Vetores próprios da análise em (B). Software de análise compatível foi usado para as peças (B) e (C). Clique aqui para ver uma versão maior desta figura.
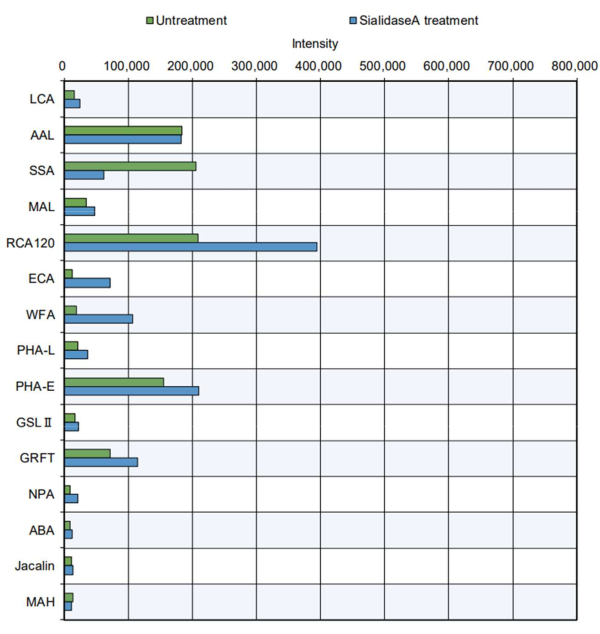
Figura 5: Verificação da reatividade do GlycoBIST usando proteína de tireoglobulina bovina purificada. A preparação da amostra sem tratamento foi realizada sob as mesmas condições de digestão que o tampão sozinho, excluindo a adição de sialidase A. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Teste de repetibilidade com grânulos fixados em lectina. A repetibilidade da reatividade para 28 lectinas foi avaliada. Os analitos reagiram com cada ponta. EPO: eritropoietina, PSA: antígeno prostático específico, Tf: transferrina, M2BP: proteína de ligação a Mac-2, hIgG: IgG humana, MMP3: metaloproteinase 3 da matriz, (Sia+): analitos digeridos pela sialidase A, (Sia+, Gal+): analitos digeridos pela sialidase A e galactosidase, Ab: anticorpo. DSA: aglutinina de Datura stramonium , HypninA2: aglutinina de Hypnea japonica , WGA: aglutinina de gérmen de trigo, UDA: aglutinina de Urtica dioica , BPL: lectina de Bauhinia purpurea , Orysata: lectina de Oryza sativa , LSL-N: domínio N-terminal da lectina de Laetiporus sulphureus , SNA: lectina de Sambucus nigra , BanLec: lectina de banana, MPA: aglutinina de Maclura pomifera , TxLC-I: aglutinina de Tulipa gesneriana , AOL: lectina de Aspergillus oryzae , ACG: Agrocybe cylindracea galectin. CV: coeficiente de variação. Clique aqui para baixar esta tabela.
Tabela 2: Lista de lectinas que constituem a ponta padrão para o perfil automático de glicanos. Clique aqui para baixar esta tabela.
Tabela 3: Sacarídeos PAA usados como padrões para o sistema automático de perfil de glicanos. Clique aqui para baixar esta tabela.
Tabela 4: Resultados do teste de reprodutibilidade intra-corrida. CV: coeficiente de variação. Clique aqui para baixar esta tabela.
Tabela 5: Resultados do teste de reprodutibilidade entre dias. CV: coeficiente de variação. Clique aqui para baixar esta tabela.
Tabela 6: Resultados do teste de estabilidade a longo prazo. CV: coeficiente de variação. Clique aqui para baixar esta tabela.
Discussion
Neste estudo, foi desenvolvida uma técnica de avaliação rápida para glicosilação empregando a tecnologia "bead array in a single tip". O presente estudo introduziu uma ponta GlycoBIST padrão, projetada para pesquisadores de glicociência e não glicociência, para facilitar a avaliação de glicosilação abrangente e de rotina. O microarranjo de lectinas, tipicamente empregando 20-100 lectinas12,13, tem sido amplamente utilizado em avaliações. No entanto, considerando as especificidades sobrepostas de algumas lectinas no microarray e a variedade relativamente limitada de glicoformas em uma glicoproteína alvo em comparação com a glicômica de amostras clínicas brutas, previu-se que 15 lectinas seriam suficientes para uma avaliação simplificada do perfil de glicanos focalizados.
A etapa crítica do protocolo é permitir que os grânulos ou pontas imobilizados com lectina preenchidos com esses grânulos retornem à temperatura ambiente após o armazenamento antes de usá-los para medições. Em particular, observou-se que a condensação enfraquece os sinais; portanto, é aconselhável nunca abrir o saco de armazenamento até que ele retorne à temperatura ambiente. Para o armazenamento de reagentes de biotinilação, também é essencial secar completamente antes do armazenamento. Um processo de secagem insuficiente diminui a eficiência da biotinilação em proteína devido à hidrólise do reagente de biotinilação14.
Em relação às modificações e solução de problemas, foi explorado um método para reduzir a variabilidade, especialmente no que diz respeito ao método de medição no dispositivo automatizado de medição de reação. Em particular, quando o volume de tampão no reservatório usado para análise é de 150 μL, as bolhas entram na ponta, levando à redução da eficiência da reação e resultando em uma grande variação nos valores. Portanto, recomenda-se que pelo menos 200 μL de tampão sejam colocados no reservatório.
A quantidade de líquido que preenche as pontas durante a pipetagem automática também é um fator importante. As pontas não podem expelir completamente o líquido à medida que ele se move para a próxima etapa de reação. Portanto, em grande quantidade, o tampão da etapa anterior é transportado para a próxima etapa e o tampão anterior permanece na área superior da solução inserida quando o novo tampão é aspirado. Portanto, o substrato e outras soluções devem ser suficientemente aspirados para encher o reservatório superior (ver Figura 1A). Dada a irreversibilidade das reações de anticorpos no instrumento, é crucial ter cautela e evitar erros durante a configuração inicial.
Uma limitação do método é que ele é restrito a proteínas solúveis derivadas de sobrenadantes de soro e cultura. No método atual, a lectina não é reticulada covalentemente ao grânulo, tornando-o incapaz de analisar amostras contendo altas concentrações de surfactantes, como extratos de células e tecidos. Consequentemente, melhorias futuras são necessárias para resolver essas limitações.
O significado da matriz de contas de lectina reside na intercambialidade e expansibilidade das espécies de lectina fixadas nas contas. Por exemplo, na análise diferencial em larga escala (>1.000 amostras) usando o sistema automatizado de perfil de glicano, os usuários podem adaptar uma linha de 15 lectinas na ponta com base no perfil preliminar de glicano baseado em microarray de lectina da glicoproteína alvo (<100 amostras). Além disso, a alta estabilidade dos grânulos fixados por lectina permite a experimentação imediata desde o desenho inicial da ponta até a medição com procedimentos de rotina. Não apenas as 25 das 28 lectinas que demonstraram alta confiabilidade (Tabela 1), mas quaisquer lectinas de interesse para o usuário podem ser usadas para criar um sistema automatizado de perfil de glicano personalizado para seus experimentos específicos, seguindo o teste de confiabilidade de rotina mencionado acima. Essa abordagem levou ao projeto de uma possível matriz de esferas de 120 lectinas de oito tipos diferentes de pontas para medição expandida em conjunto com o sistema de perfil de glicano padrão.
Um estudo anterior concentrou-se em estabelecer um procedimento de detecção para glicoproteínas marcadas com Cy3 de acordo com o microarray de lectina15. Este método foi limitado a 13 lectinas em paralelo na ponta devido à interferência da fluorescência. O método atual, usando rótulos de detecção HRP, acomoda 15 grânulos fixados por lectina e dois grânulos de controle (positivo e negativo) em uma ponta. Além disso, a biotinilação das amostras encurtou o processo de análise.
Este método analítico pode ser aplicado não apenas à pesquisa acadêmica, mas também à pesquisa médica e farmacêutica, alimentos, cosméticos e outros campos industriais. Embora as tecnologias de microarray de lectina e glicoproteômica estejam bem estabelecidas, a abordagem deste estudo permanece significativa por sua automação total e capacidade de análise rápida com menos etapas. No futuro, aprimoramentos nessa análise serão feitos em um método quantitativo, normalizando o sinal usando certas glicoproteínas, empregando grânulos fixados por anticorpos ao lado de grânulos fixados por lectina em uma ponta.
Disclosures
AO, TO, NT, HS, KK, MA, SY, TM, KN, OS e KS são funcionários da Precision System Science Co., Ltd.
Acknowledgements
Este trabalho foi apoiado pelo Programa de Transferência de Tecnologia Adaptável e Contínua por meio de P&D orientado a metas (A-STEP), financiado pela Agência Japonesa de Ciência e Tecnologia (JST), sob o número de concessão JPMJTR204A e em parte pela Sociedade Japonesa para a Promoção da Ciência (JSPS) KAKENHI Grant Number 23H02680 para AK. Gostaríamos de agradecer à Editage (www.editage.jp) por editar o idioma inglês.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2 mL 8 PCR tubes + flat cap | FastGene | FG-0028FC/SE | For dispensing and storage of biotin labeling reagents |
| 2.0 mL SC Micro Tube protein LB | SARSTEDT AG&Co. KG | 72.694.600 | For loading samples in LuBEA-VIII |
| Analysis software | SAS Institute Inc. | JMP 17 | |
| Anti-Static Ionizer. 110 volts | Plas-Labs | 800-AS/SPI | Use while inserting beads into BIST Tip. |
| Antistatic tweezers NK2A | RUBIS | 9-5681-01 | Use while inserting beads into BIST Tip. |
| Automated reaction measurement device | Precision System Science Co., Ltd. | LuBEA | All data in this study were obtained using LuBEA-VIII. While LuBEA is commercialized, LuBEA-VIII is currently in the development phase. The website for product details of LuBEA and LuBEA-VIII can be found at the following URL (https://www.pss.co.jp/english/technology/apit/bist02.html). |
| Biotin-(AC5)2 Sulfo-Osu | Dojindo | 341-06801 | As biotinilation reagent |
| BIST spacer bead- V-11 | Precision System Science Co., Ltd. | F4938000 | Diameter: 1 mm |
| BIST tip and sheath | Precision System Science Co., Ltd. | F4930-000 | Fill this empty tip with beads. |
| Calcium chloride | Wako | 039-00475 | For preparing buffer solutions |
| Can Get Signal Solution 2 | TOYOBO | NKB-301 | This blocking reagent is good for this method |
| CentriVap Benchtop Vacuum Concentrators | Labconco | 7810010 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Clarity Western ECL Substrate | Bio-Rad | 1705060 | For measuring the samples by LuVEA VIII |
| Cold trap CentriVap series | Labconco | 7460040 | For dry of biotinilation reagent. The substitution provided by other companies is possible. |
| Glycine | Wako | 077-00735 | For preparing buffer solutions |
| Manganese(II) Chloride Tetrahydrate | Wako | 139-00722 | For preparing buffer solutions |
| Milli-Q reference | MERCK | ZIQ7000T0C | As deionazed water for dilution of reagents and buffers |
| PBS | Wako | 162-19321 | For preparing buffer solutions |
| Peroxidase Streptavidin | Jackson | 016-030-084 | This antibody is good for GlycoBIST analysis. It is useful for the detection of the western blotting, although substitutions can be provided by other companies. |
| Probe bead | Precision System Science Co., Ltd. | FP4936000 | For preparing lectin-fixed beads. See Shimazaki, H. et al., Current Protocols in Protein Science, 2020 in reference section for detail. |
| Protein LoBind Tubes 1.5 mL | Eppendorf | 0030108442 | Use while preparing the samples such as biotinylation. |
| Silica gel (for desiccan) | Kanto chemical | 37039-02 | Use while storing the GlycoBIST tip and dried biotinylation reagent. |
| Static electricity removal sheet M | TRUSCO NAKAYAMA | SD5050 | Use while inserting beads into BIST Tip. |
| SV Cartridge II | Daido Chemical Industry | ED041 | For measuring the samples by LuVEA VIII |
| Table Microcentrifuge-CAPSULEFUGE | TOMY | PMC-060 | For spin-down of the mixture in each process |
| Triton X-100 | Nacalai tesque | 35501-15 | For preparing buffer solutions |
References
- Butler, M., Reichl, U. Animal cell expression systems. Adv Biochem Eng Biotechnol. 175, 1-36 (2021).
- Silva, M. L. S. Chapter One - Lectin biosensors in cancer glycan biomarker detection. Adv Clin Chem. 93, 1-61 (2019).
- Hiono, T., Nagai-Okatani, C., Kuno, A. 4.07 - Application of glycan-related microarrays. Comprehensive Glycoscience (Second Edition). , 134-148 (2021).
- Shimazaki, H., et al. Lectin bead array in a single tip facilitates fully automatic glycoprotein profiling). Anal Chem. 91 (17), 11162-11169 (2019).
- Shimazaki, H., et al. GlycoBIST: A System for automatic glycan profiling of a target protein using milli-bead array in a tip. Curr Protoc Protein Sci. 99 (1), e103 (2020).
- Mahmood, T., Yang, P. C. Western blot: Technique, theory, and trouble shooting. N Am J Med Sci. 4 (9), 429-434 (2012).
- Bojar, D., et al. A useful guide to lectin binding: machine-learning directed annotation of 57 unique lectin specificities. ACS Chem Biol. 17 (11), 2993-3012 (2022).
- Nagai-Okatani, C., et al. 2.0: An integrated visualization for lectin microarray-based mouse tissue glycome mapping data with lectin histochemistry. J Proteome Res. 20 (4), 2069-2075 (2021).
- Geisler, C., Jarvis, D. L. Effective glycoanalysis with Maackia amurensis lectins requires a clear understanding of their binding specificities. Glycobiology. 21 (8), 988-993 (2011).
- Noro, E., et al. N -glycan structures of Wisteria floribunda agglutinin-positive Mac2 binding protein in the serum of patients with liver fibrosis. Glycobiology. 31 (10), 1268-1278 (2021).
- Rawitch, A. B., Pollock, H. G., Yang, S. X. Thyroglobulin glycosylation: Location and nature of the N-Linked oligosaccharide units in bovine thyroglobulin. Arch Biochem Biophys. 300 (1), 271-279 (1993).
- Dang, K., Zhang, W., Jiang, S., Lin, X., Qian, A. Application of lectin microarrays for biomarker discovery. Chemistry Open. 9 (3), 285-300 (2020).
- Chen, S., Qin, R., Mahal, L. K. Sweet systems: Technologies for glycomic analysis and their integration into systems biology. Crit Rev Biochem Mol Biol. 56 (3), 301-320 (2021).
- Dwivedi-Agnihotri, H., Srivastava, A., Shukla, A. K. Reversible biotinylation of purified proteins for measuring protein-protein interactions. Methods Enzymol. 633, 281-294 (2020).
- Shimazaki, H., et al. Auto-lectin dotcoding by two octopuses: Rapid analysis of fluorescence-labeled glycoproteins by an 8-channel fully-automatic bead array scanner with a rolling-circle detector. Anal Chem. 95 (32), 11868-11873 (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved