È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Identificazione di cellule quiescenti in un modello di leucemia linfoblastica acuta a cellule T di pesce zebra mediante colorazione della proliferazione cellulare
In questo articolo
Riepilogo
Abbiamo utilizzato la colorazione della proliferazione cellulare per identificare le cellule quiescenti nel modello di leucemia linfoblastica acuta T di zebrafish. La colorazione viene trattenuta nelle cellule non in divisione e ridotta durante la proliferazione cellulare, consentendo la selezione delle cellule dormienti per ulteriori interrogatori. Questo protocollo fornisce uno strumento funzionale allo studio dell'autorinnovamento nel contesto della quiescenza cellulare.
Abstract
La quiescenza cellulare è uno stato di arresto della crescita o di proliferazione rallentata descritto nelle cellule staminali normali e tumorali (CSC). La quiescenza può proteggere le CSC dai farmaci chemioterapici antiproliferativi. Nei modelli murini di xenotrapianto di pazienti derivati da pazienti con leucemia linfoblastica acuta a cellule T (T-ALL), le cellule quiescenti sono associate alla resistenza al trattamento e alla staminalità. I coloranti per la proliferazione cellulare sono strumenti popolari per il monitoraggio della divisione cellulare. Il colorante fluorescente è ancorato in modo covalente in gruppi amminici sulla membrana e sulle macromolecole all'interno della cellula. Ciò consente il tracciamento delle cellule marcate per un massimo di 10 divisioni, che possono essere risolte mediante citometria a flusso.
In definitiva, le cellule con i più alti tassi di proliferazione avranno una bassa ritenzione del colorante, poiché verrà diluito ad ogni divisione cellulare, mentre le cellule dormienti, che si dividono più lentamente, avranno la ritenzione più alta. L'uso di coloranti di proliferazione cellulare per isolare le cellule dormienti è stato ottimizzato e descritto in modelli murini T-ALL. Complementare ai modelli murini esistenti, il modello T-ALL di zebrafish derivato da rag2:Myc offre un'eccellente sede per interrogare l'autorinnovamento nella T-ALL grazie all'alta frequenza di cellule staminali leucemiche (LSC) e alla convenienza del zebrafish per esperimenti di trapianto su larga scala.
Qui, descriviamo il flusso di lavoro per la colorazione delle cellule T-ALL di zebrafish con un colorante di proliferazione cellulare, l'ottimizzazione della concentrazione del colorante per le cellule di zebrafish, il passaggio di cellule colorate con successo in vivo e la raccolta di cellule con vari livelli di ritenzione del colorante mediante selezione di cellule vive da animali trapiantati. Data l'assenza di produttori di superfici cellulari ben consolidati per le LSC nella T-ALL, questo approccio fornisce un mezzo funzionale per interrogare le cellule quiescenti in vivo. Per ottenere risultati rappresentativi, descriviamo l'efficienza di attecchimento e la frequenza LSC delle cellule ad alta e bassa ritenzione del colorante. Questo metodo può aiutare a studiare ulteriori proprietà delle cellule quiescenti, tra cui la risposta ai farmaci, i profili trascrizionali e la morfologia.
Introduzione
Le cellule staminali adulte sono responsabili della rigenerazione di tipi cellulari differenziati in un dato organo e sono prevalentemente presenti in uno stato dormiente, non in divisione 1,2. Ad esempio, le cellule staminali ematopoietiche (HSC), che mantengono il sangue, rimangono in gran parte quiescenti e solo una piccola frazione entra nel ciclo cellulare per autorinnovarsi o differenziarsi per generare componenti del sangue maturo3. Allo stesso modo, nei tumori, una rara sottopopolazione di cellule chiamate cellule staminali tumorali (CSCs) possiede la capacità di auto-rinnovamento e sono responsabili del mantenimento a lungo termine della malignità4. Le cellule staminali tumorali esistono in vivo in uno stato di quiescenza o di crescita lenta, che può consentire loro di sfuggire ai trattamenti anti-proliferativi contro il cancro5, eludere l'eliminazione da parte del sistema immunitario6, ridurre lo stress ossidativo e migliorare le loro vie di riparazione del DNA7. Anche un basso numero di CSC rimaste dopo il trattamento può potenzialmente ripopolare il tumore, provocando una recidiva del paziente8. Di conseguenza, la comprensione della quiescenza cellulare è molto promettente per l'identificazione di potenziali vulnerabilità delle CSC e lo sviluppo di nuovi modi per colpirle.
I coloranti per la proliferazione cellulare, come la colorazione carbossifluoresceina succinimidil estere (CFSE) e i suoi derivati, sono comunemente usati per monitorare la frequenza delle divisioni cellulari9. Il colorante permea attraverso la membrana cellulare e, una volta all'interno della cellula, subisce l'attivazione da parte delle esterasi intracellulari in un prodotto fluorescente. Il composto fluorescente risultante viene trattenuto all'interno della cellula attraverso i legami ammidici covalenti formati tra la porzione succinimidilica e i gruppi funzionali amminici delle proteine intracellulari10. Ad ogni divisione cellulare, il composto fluorescente viene diviso equamente tra le due cellule risultanti, causando una doppia diluizione del segnale. Questo colorante consente di rilevare fino a 10 divisioni cellulari attraverso l'analisi della citometria a flusso11.
Questo approccio è stato precedentemente utilizzato per arricchire le popolazioni di CSC in vitro identificando popolazioni di cellule a ciclo lento con elevata ritenzione del colorante11,12. Nella T-ALL, CFSE è stata utilizzata per monitorare la crescita tumorale in vivo in xenotrapianti derivati da pazienti nei topi. Dopo la marcatura delle cellule e tre settimane di trapianto, l'analisi della citometria a flusso ha mostrato una rara popolazione di cellule che conservavano ancora la fluorescenza CFSE. Questa popolazione era associata a staminalità, resistenza al trattamento ed elevata somiglianza con le cellule che causano recidiva nei pazienti13. Di conseguenza, questo colorante fornisce uno strumento utile per lo studio dei fenotipi delle cellule staminali leucemiche (LSC) nella T-ALL.
Lo scopo del lavoro è quello di estendere l'applicazione del colorante di proliferazione cellulare per studiare la quiescenza in vivo utilizzando un modello T-ALL di zebrafish. In particolare, il modelloT-ALL 14 di zebrafish guidato da rag2:Myc fornisce un luogo eccellente per lo studio dell'auto-rinnovamento dovuto all'alta frequenza di LSC rispetto ai modelli murini e alle malattie umane15. Inoltre, l'uso del pesce zebra consente studi di trapianto su larga scala, che possono essere eseguiti a un costo di cura e manutenzione molto inferiore rispetto alle loro controparti topoche16. I pesci zebra sono eccellenti anche per le applicazioni di imaging dal vivo, poiché le cellule tumorali marcate in fluorescenza possono essere facilmente visualizzate utilizzando un semplice microscopio a fluorescenza per stimare il tasso di sviluppo del tumore16.
In questo protocollo, descriviamo il flusso di lavoro per la colorazione delle cellule T-ALL di zebrafish con colorante di proliferazione cellulare seguito dalla propagazione in vivo delle cellule colorate in zebrafish CG1 singenico. Dopo lo sviluppo della leucemia, descriviamo lo smistamento delle cellule che hanno conservato il colorante e il loro utilizzo per un successivo esperimento di trapianto di diluizione limitante per quantificare i tassi di auto-rinnovamento di LSC. Questo protocollo può essere esteso per ulteriori applicazioni, tra cui lo screening farmacologico in vivo di potenziali composti per il targeting di LSC quiescenti. Inoltre, le cellule raccolte possono essere utilizzate per diverse analisi a valle, come la profilazione trascrittomica, la proteomica e la metabolomica, offrendo informazioni uniche sul comportamento delle LSC quiescenti nella T-ALL.
Protocollo
In questo protocollo, stiamo utilizzando cellule T-ALL di pesce zebra marcate con GFP che sono state generate in precedenza nel ceppo CG1 e quindi possono essere iniettate direttamente nel pesce zebra CG1 singeneico ricevente15. In breve, la leucemia è stata generata mediante microiniezione di DNA di rag2:Myc e rag2:GFP in embrioni di zebrafish CG1 a cellula singola. Gli animali sono stati monitorati per lo sviluppo della leucemia a partire da 3 settimane dopo l'iniezione, utilizzando la microscopia a fluorescenza. Le cellule leucemiche GFP-positive sono state isolate FACS e trapiantate in serie in zebrafish CG1 ricevente per generare cloni con un'alta frequenza di LSC. I dettagli dell'intero protocollo sono descritti da Blackburn et al.15.
In alternativa, la T-ALL primaria derivata da rag2:Myc può essere generata mediante microiniezione di DNA di embrioni di zebrafish17. La microiniezione di DNA di rag2:Myc con un reporter fluorescente guidato da rag2 come la GFP può portare allo sviluppo di cellule B, cellule T e ALL18 miscelate. I cloni leucemici utilizzati in questo protocollo sono stati precedentemente verificati come T-ALL15. Tutte le procedure sperimentali che coinvolgono il pesce zebra sono state esaminate e approvate dal Comitato istituzionale per la cura e l'uso degli animali dell'Università del Kentucky, protocollo 2019-3399.
1. Propagazione delle cellule T-ALL di zebrafish in vivo
- Scongelare un flaconcino contenente 1 mL di cellule T-ALL di pesce zebra congelate marcate con GFP in un bagno d'acqua a 37 °C con agitazione delicata, fino allo scongelamento. Una volta scongelato, trasferire sul ghiaccio.
- Pipettare lentamente il contenuto della provetta in una provetta conica da 15 mL con 4 mL di terreno di coltura per pesci (0,9x PBS + 5% FBS) per diluire il mezzo di congelamento.
- Centrifugare per 5 minuti a 2.500 × g a 4 °C. Rimuovere il materiale dal pellet. Risospendere in 0,5 mL di terreno di coltura per pesci e mantenere le cellule in ghiaccio.
- Contare le cellule e, se necessario, diluirle a 100.000 cellule/5 μL.
- Anestetizzare il pesce zebra CG1 adulto aggiungendo 200 μL di anestetico da 4 mg/mL (MS-222) a 25 mL di acqua del sistema ittico in una capsula di Petri. Tenere il pesce CG1, con il lato ventrale rivolto verso l'alto e iniettare 5-6 μL nello spazio intraperitoneale (IP) utilizzando una siringa da microlitri di Hamilton. Riporta gli animali nelle loro vasche e monitora il ritorno al nuoto normale prima di rimetterli nel sistema.
NOTA: Per gli studi di trapianto descritti in questo protocollo sono stati utilizzati pesci zebra adulti, di 60 giorni o più. - Monitora la crescita della leucemia una volta alla settimana utilizzando un microscopio a fluorescenza. Si veda la Sezione 2 di seguito per i dettagli sulla procedura di screening.
2. Raccolta di cellule leucemiche marcate in fluorescenza
- Circa 21-28 giorni dopo il trapianto, anestetizzare il pesce zebra CG1 adulto aggiungendo 200 μL (4-5 gocce) di anestetico 4 mg/mL a 25 mL di acqua del sistema dei pesci in una capsula di Petri.
- Esaminare il CG1 zebrafish per il carico di leucemia valutando la percentuale di cellule leucemiche marcate con GFP nell'animale con un microscopio a epifluorescenza, utilizzando il filtro appropriato per rilevare la fluorescenza GFP. La leucemia GFP-positiva è pronta per essere raccolta se si è diffusa nel >70% dell'animale.
- Sopprimere l'animale aggiungendo 1 ml di anestetico da 4 mg/ml in una capsula di Petri contenente 9 ml di acqua del sistema dei pesci. Permettete che tutti i segni di vita cessino; Ad esempio, osservare i pesci per la cessazione del movimento opercolare (~5 min).
- Sposta il pesce zebra in una nuova capsula di Petri e aggiungi 1 ml di terreno di coltura per pesci che funga da tampone per le cellule. Usando una nuova lama di rasoio, prima decapitare il pesce zebra, quindi macerare il tessuto usando la stessa lama di rasoio. Pipet su e giù per rimuovere grandi grumi di cellule.
- Far passare le cellule attraverso un filtro cellulare da 40 μm in una provetta conica da 50 mL per dissociarle in una sospensione a cellula singola. Mantieni le celle sul ghiaccio.
3. Ottimizzazione della concentrazione del colorante di proliferazione cellulare (CellTrace Far Red) per la colorazione e la vitalità cellulare del pesce zebra
NOTA: Per questo protocollo, e poiché le cellule tumorali sono marcate con GFP, abbiamo utilizzato un colorante di proliferazione delle cellule rosse lontane (CellTrace Far Red) per evitare la sovrapposizione spettrale, che verrà indicato come CT-FR.
- Preparare una soluzione da 1 mM del colorante fluorescente immediatamente prima dell'uso aggiungendo il volume appropriato di DMSO secondo le raccomandazioni del produttore. Avere soluzioni di lavoro di 1 mM, 0,5 mM, 0,1 mM e 0,01 mM; Diluire secondo necessità in terreni di coltura per pesci per testare la vitalità cellulare dopo la colorazione.
- Contare le cellule e raccogliere 1 × 106 cellule in provette da microcentrifuga da 1,5 mL.
- Centrifugare a 2.500 × g a 4 °C per 5 min. Rimuovere il liquido e risospendere le cellule in 1 mL di PBS 0,9x.
- Dividere la sospensione cellulare in quattro provette da 250 μl ciascuna per il trattamento con CT-FR.
- Aggiungere 250 μl di PBS 0,9x per portare il volume fino a 500 μl ciascuno con circa 250.000 cellule/provetta.
- Aggiungere le quantità necessarie delle soluzioni di lavoro sopra descritte a ciascuna delle quattro provette per ottenere le seguenti concentrazioni finali di 1 μM, 0,5 μM, 0,1 μM e 0,05 μM. Incubare per 20 minuti a 37 °C, al riparo dalla luce.
- Pellettare le celle centrifugando a 2.500 × g per 5 minuti a temperatura ambiente. Rimuovere il liquido e lavare le celle con 500 μL di terreno di coltura per pesci per rimuovere il colorante in eccesso.
- Pellettare di nuovo, rimuovere il liquido e risospendere in 25 μL di terreno di coltura per pesci. Utilizzare 1 μl per contare le cellule in una diluizione 1:10 in blu di tripano al 50%.
- Esaminare le cellule al microscopio per verificarne la vitalità cellulare; le cellule vive non manterranno il blu di tripano e appariranno luminose al microscopio, mentre le cellule morte assorbiranno il colorante e appariranno scure. Esaminare prima le cellule sotto il canale di fluorescenza appropriato per identificare le cellule tumorali e quindi utilizzare il canale del campo chiaro per valutare la vitalità cellulare.
- Selezionare la concentrazione che consente di colorare con successo mantenendo la vitalità cellulare (>90% di cellule blu di tripano negative).
NOTA: Per le cellule T-ALL di zebrafish testate in questo esperimento, è stata utilizzata una concentrazione di 1 μM per una colorazione di successo e il >90% della vitalità cellulare.
4. Propagazione in vivo di cellule di pesce zebra T-ALL colorate con CT-FR
- Utilizzando la concentrazione ottimizzata di CT-FR della Sezione 3, colorare le cellule del pesce zebra. Lasciare alcune cellule tumorali non colorate per la generazione del tumore T-ALL marcato con GFP, come controllo positivo alla GFP durante lo smistamento FACS. Diluire le cellule colorate e non colorate a 50.000 cellule/5 μL per il trapianto.
- Anestetizzare il pesce zebra CG1 adulto aggiungendo 200 μL di anestetico da 4 mg/mL a 25 mL di acqua del sistema ittico in una capsula di Petri.
- Iniettare 5 μl della sospensione cellulare nella cavità IP del pesce zebra come descritto nella Sezione 1 sia per le cellule colorate che per quelle non colorate. Iniettare 5-6 zebrafish per gruppo per tenere conto della potenziale mortalità dopo l'iniezione.
- Esaminare gli animali mediante microscopia fluorescente sotto sedazione 2 volte alla settimana per lo sviluppo della leucemia.
NOTA: Dopo ~10-14 giorni, la leucemia dovrebbe occupare circa il 30-40% di questo corpo. Pianificare il prelievo della leucemia e il successivo smistamento entro questo lasso di tempo. Aspettare che il tumore si espanda a grandi dimensioni potrebbe comportare una maggiore diluizione del colorante cellulare fluorescente e difficoltà nel rilevare il suo segnale. Considerare l'ottimizzazione dei tempi di prelievo del tumore per il modello tumorale scelto. Inoltre, si raccomanda un monitoraggio più frequente del tumore per garantire la cattura della dimensione del tumore desiderata.
5. Preparazione per lo smistamento di cellule T-ALL di pesce zebra colorate con CT-FR
- Preparare i seguenti controlli per lo smistamento delle cellule: Nessun controllo del colore (non colorato, non transgenico, sospensione di una singola cellula, da un pesce zebra di tipo selvatico), cellule T-ALL marcate con GFP e cellule di pesce zebra CT-FR WT appena colorate (per impostare una soglia massima del segnale Far-Red).
- Preparazione di controlli non colorati e CT-FR
- Raccogliere cellule di pesce wild-type (WT) mediante eutanasia di un pesce zebra CG1 non trapiantato aggiungendo 1 mL di anestetico da 4 mg/mL in una capsula di Petri contenente 9 mL di acqua del sistema ittico.
- Sposta il pesce zebra in una nuova capsula di Petri e aggiungi 1 ml di terreno di coltura per pesci che funga da tampone per le cellule. Usa una nuova lama di rasoio per decapitare prima e poi macerare il pesce zebra usando la stessa lama di rasoio; Pipettare su e giù per rimuovere i grandi grumi di cellule.
- Far passare le cellule attraverso un filtro cellulare da 40 μm in una provetta conica da 50 mL per dissociarle in una sospensione a cellula singola. Mantieni le celle sul ghiaccio.
- Per il controllo CT-FR, trasferire 250.000 cellule WT in una provetta da microcentrifuga e pellettare le celle mediante centrifugazione a 2.500 × g per 5 minuti a temperatura ambiente. Rimuovere il terreno di coltura per pesci e aggiungere 500 μl di 0,9x PBS+ 1% FBS.
- Aggiungere 0,5 μl della soluzione madre di colorante CT-FR da 1 mM per una concentrazione finale di 1 μM. Pipettare delicatamente con 1.000 μl di pipetta per miscelare. Incubare a 37 °C per 20 minuti, al riparo dalla luce.
- Rimuovere il liquido e lavare le celle con 500 μL di terreno di coltura per pesci per rimuovere il colorante in eccesso.
- Pellettare nuovamente le celle mediante centrifugazione, rimuovere il liquido e risospendere le celle in 500 μl di 0,9x PBS+ 1% FBS e passare attraverso un tappo filtrante da 35 μm in una provetta FACS. Mantenere il ghiaccio.
- Preparazione della placca donatrice
NOTA: La piastra donatrice è una piastra con fondo a V a 96 pozzetti in cui verranno smistate le cellule T-ALL colorate con CT-FR del pesce zebra trapiantato. Ogni pozzetto della piastra contiene 5.000 cellule WT CG1 non colorate in 50 μL di terreno di coltura per pesci che fungono da vettore per aiutare a pellettare le cellule selezionate nelle fasi successive.- Contare 500.000 cellule WT CG1 e trasferirle in una provetta conica da 15 mL.
- Diluire questo stock cellulare a 100 cellule/μL aggiungendo 5 mL di terreno di coltura per pesci.
- Erogare 50 μl della sospensione cellulare in ciascun pozzetto della piastra a 96 pozzetti. Miscelare il tubo madre della sospensione cellulare dopo ogni fila per mantenere l'omogeneità della sospensione cellulare. Conservare la piastra a 4 °C fino alla cernita.
- Preparazione dei campioni di pesce trapiantati
- Selezionare i pesci trapiantati con un carico di T-ALL del 30-40%. Raccogliere i pesci trapiantati sia con le cellule colorate con CT-FR che con le cellule tumorali GFP non colorate.
- Raccogliere le celle come descritto nella Sezione 2.
NOTA: Poiché si prevede che le cellule che trattengono il colorante siano presenti in bassa abbondanza, prepararsi a selezionare un gran numero di cellule dal campione di GFP CT-FR. - Diluire in una concentrazione appropriata per la selezione di ~3-5 × 106 cellule/mL in 0,9x PBS + 1% FBS e passare attraverso un tappo filtrante da 35 μm in una provetta FACS. Aggiungere DAPI (1 mg/mL) alla diluizione 1:1.000 come colorante vitale per escludere le cellule morte. Mantenere il ghiaccio.
- Per le cellule tumorali GFP non colorate, trasferire 250.000 cellule in una provetta da microcentrifuga e pellettare a 2.500 × g per 5 minuti a temperatura ambiente. Rimuovere il materiale di scorta per pesci, aggiungere 500 μl di PBS e passare attraverso un tappo del filtro da 35 μm in una provetta FACS. Mantenere il ghiaccio.
6. Smistamento
NOTA: Tenere tutte le provette e la piastra donatrice sul ghiaccio per tutto il tempo, tranne quando vengono utilizzate per lo smistamento.
- Per l'ordinamento dei parametri e dei cancelli, iniziare applicando il gating di dispersione anteriore e laterale per identificare le celle di interesse in base alle loro dimensioni ed eliminare i detriti cellulari. Quindi, utilizzare l'altezza di dispersione in avanti rispetto all'area di dispersione in avanti per escludere le celle duplicate e mantenere solo i singoletti. Infine, utilizzare i controlli senza colore e monocolore per disegnare i quadranti per identificare le popolazioni cellulari GFP-positive e CT-FR-positive.
NOTA: Il controllo CT-FR dovrebbe essere molto più luminoso dei campioni per lo smistamento poiché la colorazione per il controllo è stata eseguita a fresco senza divisioni cellulari; si prevede che le cellule dei campioni di CT-FR GFP appaiano all'estremità inferiore del gate CT-FR positivo. - Ordinare il numero di cellule da trapiantare in ogni pesce in un pozzetto della piastra donatrice.
NOTA: Per questo esperimento, abbiamo utilizzato un saggio di diluizione limitante per stimare la frequenza delle cellule staminali leucemiche in CT-FR High e CT-FR Low. Questo test ha utilizzato una dose di 25 cellule, trapiantate in 3 animali, e una dose di 10 cellule, trapiantate in 10 animali. Un tipico saggio di diluizione limitante utilizzando il T-ALL derivato da rag2:Myc di zebrafish coinvolgerebbe un gruppo con un numero maggiore di cellule (500 o 1.000) poiché la frequenza di LSC in questo modello è stimata in 1 LSC in centinaia di cellule. Tuttavia, in questo saggio, stiamo utilizzando uno dei cloni di T-ALL precedentemente descritti da Blackburn et al.15. La frequenza LSC nel campione utilizzato qui è ~1 LSC in decine di celle; Prevediamo che la leucemia venga rilevata negli animali trapiantati nel gruppo delle 25 cellule. Quando si ottimizza questo test per il modello di malattia, è necessario prestare attenzione durante la selezione del gruppo con un numero di cellule più elevato per garantire l'attecchimento del tumore. - Dopo lo smistamento, conservare la piastra sul ghiaccio fino al momento del trapianto.
NOTA: Le procedure di selezione possono essere stressanti e influire sulla vitalità cellulare. Prima di eseguire l'esperimento di diluizione limitante vero e proprio, è stato fatto un esperimento pilota di smistamento. Le cellule selezionate sono state trapiantate e lo sviluppo della leucemia è stato monitorato per determinare se lo smistamento fosse dannoso per la vitalità cellulare. In alternativa, la vitalità cellulare può essere valutata colorando le cellule selezionate con il blu di tripano ed esaminandole al microscopio prima del trapianto.
7. Trapianto di cellule selezionate alla diluizione limitante e monitoraggio dell'insorgenza del tumore
- Centrifugare la piastra utilizzando una grande centrifuga da banco a 2.500 × g a 4 °C.
- Rimuovere con cautela 45 μl di surnatante da ciascun pozzetto, lasciando 5 μl di liquido e facendo attenzione a non toccare il pellet di cella sul fondo del pozzetto.
- Risospendere le cellule con una pipetta da 20 μl. Risospendere 2-3 pozzetti con la pipetta e quindi, utilizzando la siringa da microlitro di Hamilton, iniettare le sospensioni cellulari da ciascun pozzetto nel numero desiderato di pesce zebra CG1 tramite trapianto IP. Quindi, risospendere le cellule nel gruppo successivo di pozzetti per evitare il riassestamento delle cellule.
- Esaminare il pesce zebra mediante microscopia a fluorescenza sotto sedazione una volta alla settimana per lo sviluppo di T-ALL per almeno 6 settimane.
8. Determinazione della frequenza delle cellule staminali leucemiche
- Registrare il numero di animali positivi per dose cellulare e gruppo di trattamento a 6 settimane dopo il trapianto, considerando positivi gli animali con qualsiasi quantità di segnale GFP. Si consideri che gli animali hanno un carico significativo di leucemia quando il segnale GFP occupa il >50% del corpo.
- Utilizza il software statistico ELDA (Extreme Limiting Dilution Analysis) (http://bioinf.wehi.edu.au/software/elda/)19 basato sul web per calcolare la frequenza delle LSC all'interno di ciascuna popolazione cellulare.
NOTA: Nella Figura 1 viene fornita un'illustrazione schematica del flusso di lavoro.
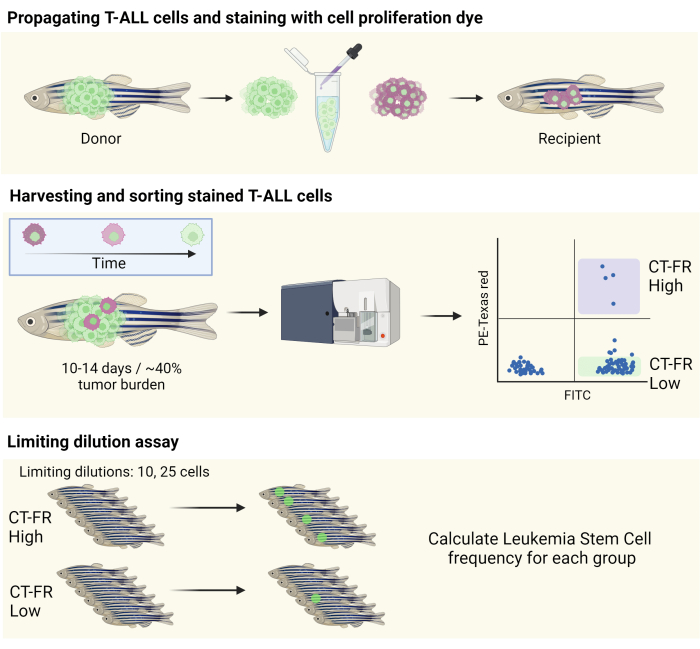
Figura 1: Flusso di lavoro per l'uso della colorazione di tracciamento cellulare per isolare le cellule quiescenti nel modello T-ALL del pesce zebra. Illustrazione schematica della colorazione delle cellule T-ALL di zebrafish con la colorazione di proliferazione cellulare, la propagazione in un zebrafish CG1 (pannello superiore) e l'ordinamento delle cellule in base alla ritenzione della colorazione di proliferazione cellulare (pannello centrale). Le cellule raccolte sono state utilizzate per un saggio di diluizione limitante per determinare la frequenza delle LSC (pannello inferiore). Abbreviazioni: T-ALL = leucemia linfoblastica acuta a cellule T; LSCs = cellule staminali leucemiche; CT-FR = CellTrace Lontano Rosso; FITC = isotiocianato di fluoresceina; PE = ficoeritrina. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
Abbiamo seguito il protocollo sopra descritto per selezionare le cellule che hanno trattenuto il colorante di proliferazione cellulare, CT-FR, e le abbiamo utilizzate per un saggio di diluizione limitante (LDA) per stimare la frequenza di LSC nelle popolazioni CT-FR High e CT-FR Low. Per impostare il gating per l'esperimento di citometria a flusso, abbiamo utilizzato un controllo senza fluoroforo (senza colore) oltre ai controlli a colore singolo (Figura 2A
Discussione
Le LSC sono note per essere resistenti ai trattamenti chemioterapici convenzionali e antiproliferativi e trovare terapie mirate contro queste cellule è molto promettente per ridurre l'insorgenza di recidive e migliorare la prognosi dei pazienti20. Ricerche precedenti hanno descritto l'uso di coloranti fluorescenti di proliferazione cellulare per identificare una piccola popolazione di cellule quiescenti associate alla resistenza ai farmaci e alla staminalità nei...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Il finanziamento per questa ricerca è stato fornito dal National Cancer Institute (R37CA227656 to JSB). Questa ricerca è stata supportata anche dalla citometria a flusso e dal monitoraggio immunitario delle risorse condivise del Markey Cancer Center (P30CA177558 dell'Università del Kentucky.
Materiali
| Name | Company | Catalog Number | Comments |
| 26 G/2” micro-syringe | Hamilton | 87930 | NA |
| 35 µm filter cap FACS tubes | Falcon | 352235 | NA |
| 40 µm cell strainer | CELLTREAT | 229482 | NA |
| 96-well skirted PCR plate | Thermo Fisher Scientific | AB0800 | NA |
| Cell sorter | Sony Biotechnology | SY3200 | NA |
| CellTrace Far Red | Thermo Fisher Scientific | C34564 | NA |
| Conical tubes | VWR | 10026-078 | NA |
| DAPI | Thermo Fisher Scientific | 62248 | NA |
| DMSO | Sigma-Aldrich | D4818 | NA |
| Dulbecco'sPhosphate-buffered saline (PBS) | Caisson Labs | 22110001 | NA |
| Epifluorescence stereo microscope | Nikon | SMZ25 | NA |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | 12306C | NA |
| Fish system water | N/A | N/A | 0.03-0.05% salinity, pH 6.5-8, buffered with sodium bicarbonate |
| Microcentrifuge tubes | Thermo Fisher Scientific | C2171 | NA |
| MS-222 | Pentaire | TRS-1 | tricaine mesylate, an anesthetic |
| Petri dishes | Corning | 07-202-011 | NA |
| Razor blades | American Line | 66-0089 | NA |
| Trypan Blue | Thermo Fisher Scientific | T10282 | NA |
Riferimenti
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414 (6859), 105-111 (2001).
- Arai, F., et al. Tie2/angiopoietin-1 signaling regulates hematopoietic stem cell quiescence in the bone marrow niche. Cell. 118 (2), 149-161 (2004).
- Li, L., Bhatia, R. Stem cell quiescence. Clin. Cancer Res. 17 (15), 4936-4941 (2011).
- Kreso, A., Dick, J. E. Evolution of the cancer stem cell model. Cell Stem Cell. 14 (3), 275-291 (2014).
- Chen, W., Dong, J., Haiech, J., Kilhoffer, M. -. C., Zeniou, M. Cancer stem cell quiescence and plasticity as major challenges in cancer therapy. Stem Cells Int. 2016, 1740936 (2016).
- Kleffel, S., Schatton, T. Tumor dormancy and cancer stem cells: Two sides of the same coin. Adv Exp Med Biol. 734, 145-179 (2013).
- Tuy, K., Rickenbacker, L., Hjelmeland, A. B. Reactive oxygen species produced by altered tumor metabolism impacts cancer stem cell maintenance. Redox Biol. 44, 101953 (2021).
- Zhou, B. -. B. S., et al. Tumour-initiating cells: Challenges and opportunities for anticancer drug discovery. Nat Rev Drug Discov. 8 (10), 806-823 (2009).
- Lyons, A. B., Blake, S. J., Doherty, K. V. Flow cytometric analysis of cell division by dilution of cfse and related dyes. Curr Protoc Cytom. 64 (1), 11-12 (2013).
- Lyons, A. B. Analysing cell division in vivo and in vitro using flow cytometric measurement of cfse dye dilution. J Immunol Methods. 243 (1-2), 147-154 (2000).
- Azari, H., Deleyrolle, L. P., Reynolds, B. A. Using carboxy fluorescein succinimidyl ester (cfse) to identify quiescent glioblastoma stem-like cells. Methods Mol Biol. 1686, 59-67 (2018).
- Deleyrolle, L. P., Rohaus, M. R., Fortin, J. M., Reynolds, B. A., Azari, H. Identification and isolation of slow-dividing cells in human glioblastoma using carboxy fluorescein succinimidyl ester (cfse). J Vis Exp. (62), e3918 (2012).
- Ebinger, S., et al. Characterization of rare, dormant, and therapy-resistant cells in acute lymphoblastic leukemia. Cancer Cell. 30 (6), 849-862 (2016).
- Langenau, D. M., et al. Myc-induced t cell leukemia in transgenic zebrafish. Science. 299 (5608), 887-890 (2003).
- Blackburn, J. S., et al. Clonal evolution enhances leukemia-propagating cell frequency in t cell acute lymphoblastic leukemia through akt/mtorc1 pathway activation. Cancer Cell. 25 (3), 366-378 (2014).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24 (3), 2288 (2023).
- Blackburn, J. S., Liu, S., Langenau, D. M. Quantifying the frequency of tumor-propagating cells using limiting dilution cell transplantation in syngeneic zebrafish. J Vis Exp. (53), e2790 (2011).
- Borga, C., et al. Simultaneous b and t cell acute lymphoblastic leukemias in zebrafish driven by transgenic myc: Implications for oncogenesis and lymphopoiesis. Leukemia. 33 (2), 333-347 (2019).
- Hu, Y., Smyth, G. K. Elda: Extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. J Immunol Methods. 347 (1-2), 70-78 (2009).
- Bhojwani, D., Pui, C. -. H. Relapsed childhood acute lymphoblastic leukaemia. Lancet Oncol. 14 (6), e205-e217 (2013).
- Zon, L. I., Peterson, R. T. In vivo drug discovery in the zebrafish. Nat Rev Drug Discov. 4 (1), 35-44 (2005).
- Al-Hamaly, M. A., et al. Zebrafish drug screening identifies erlotinib as an inhibitor of wnt/β-catenin signaling and self-renewal in t-cell acute lymphoblastic leukemia. Biomed Pharmacother. 170, 116013 (2024).
- Yan, C., et al. Visualizing engrafted human cancer and therapy responses in immunodeficient zebrafish. Cell. 177 (7), 1903-1914 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon