Quantificazione dei genomi virali adeno-associati in campioni di vettori purificati mediante reazione a catena della polimerasi a goccioline digitali
In This Article
Summary
La quantificazione precisa delle copie del genoma del vettore del virus adeno-associato (AAV) è fondamentale, ma un protocollo standardizzato deve ancora essere stabilito. Questo protocollo descrive un metodo convalidato per la preparazione di campioni di AAV purificati e per l'esecuzione di una reazione a catena della polimerasi (dd_PCR) digitale per quantificare in modo affidabile il titolo del genoma virale.
Abstract
Il virus adeno-associato (AAV) è un virus non patogeno utilizzato come veicolo di consegna per trasferire geni terapeutici nei pazienti. La quantificazione accurata del numero di copie del genoma AAV nelle preparazioni vettoriali è essenziale per l'ottimizzazione dei bioprocessi e il calcolo del dosaggio negli studi preclinici e clinici dei prodotti di terapia genica basati su AAV. Attualmente, manca un protocollo di consenso per la titolazione del genoma virale AAV. Qui presentiamo un protocollo digitale di PCR a goccioline (dd_PCR) per la quantificazione dei genomi virali in campioni di vettori purificati. I campioni vengono trattati con DNasi I per eliminare il DNA contaminante non confezionato. I campioni trattati con DNasi vengono quindi miscelati con un appropriato set di sonde primer (progettato in base al genoma AAV target) e reagenti PCR, quindi caricati in un generatore di goccioline. Le goccioline preparate vengono trasferite in una piastra PCR, dove viene eseguita e analizzata l'amplificazione PCR. Il titolo del genoma virale viene calcolato in base alla concentrazione (copie/μL), tenendo conto delle diluizioni del campione. Una misurazione riuscita mostra una netta separazione tra le nuvole di goccioline positive e negative, ha almeno 10.000 goccioline accettate, mostra un valore compreso tra 10 copie/μL e 10.000 copie/μL e ha un coefficiente di variazione (CV) tra le ripetizioni inferiore al 20%. Una titolazione affidabile del genoma virale aiuterà nello sviluppo di prodotti di terapia genica sicuri ed efficaci a base di AAV.
Introduction
La terapia genica è una modalità terapeutica comunemente utilizzata per trattare le malattie genetiche. La progettazione di una data terapia genica è specifica per la patologia associata all'indicazione target, ma tutte le terapie geniche comportano la somministrazione intracellulare di materiale genetico alle cellule bersaglio al fine di suscitare un effetto terapeutico1. La terapia genica può essere ulteriormente classificata in diverse categorie, tra cui la sostituzione genica per le mutazioni con perdita di funzione, il silenziamento genico per le anomalie del guadagno di funzione e le tecniche di editing genetico. Indipendentemente dalla strategia specifica impiegata, il materiale terapeutico dell'acido nucleico (indicato come transgene) deve essere impacchettato all'interno di un vettore per ottenere una consegna intracellulare mirata2.
Sebbene sia disponibile una varietà di sistemi vettoriali virali e non virali per lo sviluppo della terapia genica, i virus adeno-associati (AAV) sono spesso scelti a causa dell'ampio tropismo virale e della bassa immunogenicità associati a questo gruppo di virus 1,2. Ad oggi, sette terapie geniche che utilizzano l'AAV per la somministrazione terapeutica di geni hanno ottenuto l'approvazione da parte dell'Agenzia Europea per i Medicinali (EMA) o della Food and Drug Administration (FDA) per malattie che vanno dall'emofilia (ad esempio, Rotaviano) all'atrofia muscolare spinale (ad esempio, Zolgensma)3.
La produzione di terapie geniche basate sull'AAV deriva dalla comprensione dell'AAV wild-type stesso. L'AAV è un piccolo virus a DNA appartenente alla famiglia dei Parvoviridae che comprende 13 sierotipi principali (AAV1-13)3. Il genoma AAV comprende una molecola di DNA a singolo filamento di ~4,7 kb contenente due principali frame di lettura aperti (ORF) che codificano i geni virali essenziali necessari per la replicazione del genoma, l'assemblaggio del capside e l'impacchettamento (rep, cap). Il genoma virale è fiancheggiato sia alle estremità 5' che 3' da sequenze nucleotidiche palindromiche, denominate ripetizioni terminali invertite (ITR). Questi ITR formano strutture simili a forcine che svolgono un ruolo cruciale nella replicazione del genoma e nell'impacchettamento di genomi virali de novo in capsidi virali di nuova sintesi. L'AAV è un virus helper-dipendente e, pertanto, richiede l'espressione di geni ausiliari di altri virus, come il virus dell'herpes simplex (HSV) o l'adenovirus (AdV) per diventare competente in termini di replicazione1.
Al fine di produrre AAV, viene impiegato un opportuno sistema di espressione cellulare per facilitare l'espressione delle proteine del capside virale e il successivo assemblaggio in particelle virali de novo, seguito dall'incapsidazione di un transgene selezionato fiancheggiato da ITR (noto anche come genoma vettore). Questo processo utilizza comunemente un sistema a triplo plasmide, che comprende (1) un plasmide che ospita geni helper derivati da un virus helper, (2) un plasmide che codifica gli elementi virali essenziali (rep/cap) e (3) un plasmide che trasporta la cassetta di espressione terapeutica (comunemente indicato come plasmide di trasferimento)4. La presenza unica di segnali di impacchettamento a ripetizione terminale invertita (ITR) che affiancano la cassetta di espressione terapeutica nel plasmide di trasferimento garantisce un impacchettamento specifico del transgene, escludendo per lo più i geni virali presenti sugli altri plasmidi. La co-trasfezione di questo sistema a tre plasmidi in una piattaforma di espressione basata su cellule (tipicamente HEK293T cellule) porta alla produzione di particelle virali competenti in trasduzione e carenti di replicazione adatte per l'uso in applicazioni di terapia genica 3,4.
Esistono una serie di attributi critici di qualità (CQA) associati alla produzione di terapie geniche basate su AAV che devono essere valutati al fine di garantire la potenza, la purezza e la sicurezza del medicinale previsto4. Questi CQA includono il titolo del virus, il contenuto di capside e l'aggregazione. Il titolo virale stesso è una combinazione del numero di particelle virali (titolo del capside) e del numero di genomi vettori (titolo del genoma vettore) presenti in una data preparazione. Idealmente, il rapporto tra questi due titoli dovrebbe essere 1:1 poiché ogni particella virale dovrebbe contenere un genoma vettore, ma le inefficienze nell'impacchettamento del genoma vettoriale durante la biosintesi provocano la co-produzione di capsidi vuoti o parzialmente riempiti (quelli contenenti sequenze parziali del genoma vettoriale e/o sequenze del genoma non vettoriale)5. La presenza di tali impurezze può potenzialmente evocare risposte immunitarie ingiustificate e competere per i siti di legame del vettore, aumentando così il rischio di immunotossicità e riducendo il tasso di trasduzione dei capsidi completi6. Una quantificazione accurata dei genomi AAV è quindi essenziale per stabilire il titolo virale e il contenuto di capside. Ciò ha un impatto sia sulla ricerca di base che sull'industria della terapia genica, che richiede un dosaggio accurato per mantenere sia la sicurezza che l'efficacia dei medicinali.
La reazione a catena della polimerasi (dd_PCR) digitale è diventata strettamente associata alla quantificazione del titolo virale poiché può essere utilizzata per determinare il numero di genomi vettori presenti in una data preparazione7. La PCR digitale è stata introdotta per la prima volta negli anni '90 8,9 e dd_PCR è un miglioramento di questa tecnologia che consente l'elaborazione di campioni ad alta produttività 10,11. In dd_PCR, una reazione di PCR in tempo reale da 20 μL viene suddivisa in circa 20.000 goccioline racchiuse in olio, fornendo fino a 96 reazioni di questo tipo quando alloggiate su una piastra standard da 96 pozzetti. Rispetto alla PCR quantitativa convenzionale (qPCR), dd_PCR offre diversi vantaggi, tra cui una maggiore sensibilità, una maggiore precisione e una quantificazione più diretta e assoluta delle sequenze target senza la necessità di curve standard. Inoltre, l'elevato livello di partizionamento in dd_PCR riduce l'impatto degli inibitori della PCR e minimizza il potenziale di distorsione derivante dall'amplificazione preferenziale di alcuni modelli, rendendola un'opzione interessante per la misurazione analitica della titolazione del genoma del vettore.
Protocol
Il protocollo qui descritto è progettato per quantificare il titolo del genoma virale di un vettore AAV9 purificato e prodotto internamente con proteina fluorescente verde (GFP) come transgene12 con un alto livello di precisione (Tabella 1). Tuttavia, questo protocollo è applicabile a qualsiasi sierotipo AAV e a qualsiasi progetto di genoma vettoriale, a condizione che i set di primer/sonda siano progettati per colpire lo specifico genoma del vettore di interesse. I dettagli dei reagenti, dei primer, delle sonde e delle attrezzature utilizzate in questo studio sono elencati nella Tabella dei materiali.
1. Preparazione delle soluzioni madre
NOTA: Preparare tutte le soluzioni madre necessarie per la diluizione del campione in una stazione di lavoro PCR per evitare la contaminazione da DNA estraneo.
- Preparare 10 tamponi per il test PCR, che consistono in 500 mM di KCl, 100 mM di Tris-HCl, 15 mM di MgCl2 e 0,01 % (p/v) di BSA. Misurare il pH della soluzione e regolarlo a 8,3.
NOTA: Questa soluzione può essere conservata a temperatura ambiente per un massimo di 1 mese. - Preparare il tampone di diluizione AAV, che consiste in 1 tampone per test PCR, 0,2 ng/μL di DNA tranciato con spermatozoi di salmone (sss) e F-68 pluronico allo 0,1%. Preparare questa soluzione fresca ad ogni test e mantenerla a 4 °C.
NOTA: Se il pluronico F-68 non è disponibile, è possibile utilizzare un'alternativa come il tensioattivo non ionico Poloxamer 188.
2. Preparazione dei campioni - Trattamento con DNasi I e diluizione seriale
NOTA: Al fine di rimuovere i contaminanti del DNA che potrebbero ridurre l'accuratezza della titolazione del genoma del vettore, il DNA libero presente all'interno del campione AAV (incluso il DNA plasmidico rimanente o i genomi virali non incapsulati) può essere rimosso prima dell'amplificazione PCR utilizzando DNasi. Ciò è possibile poiché i genomi vettori sono incapsulati all'interno dei capsidi AAV e quindi non sono accessibili fino alla fase di denaturazione della reazione di PCR stessa (vedi passaggio 5). Inoltre, poiché il numero del genoma del vettore in un dato campione è sconosciuto, è necessario eseguire una diluizione seriale dei campioni per garantire che le misurazioni rimangano entro i limiti superiori e inferiori rilevabili. Eseguire tutte le manipolazioni dei campioni su una workstation PCR. Conservare tutti i campioni sul ghiaccio se non diversamente specificato.
- Miscelare il campione AAV selezionato per l'analisi agitando brevemente e quindi centrifugando per assicurarsi che tutto il liquido rimanga sul fondo della provetta contenente il campione.
- Per il trattamento con DNasi, aggiungere 45 μL di una soluzione contenente 1 tampone DNasi I, F-68 pluronico allo 0,1% e 0,04 U/μL DNasi I in acqua priva di DNasi in una provetta per PCR da 0,2 mL a 8 provette.
- Trasferire 5 μl di campione nella provetta contenente la soluzione di DNAsi. Agitare i campioni e centrifugare brevemente per assicurarsi che tutto il liquido rimanga sul fondo.
- In un termociclatore, incubare i campioni per 1 ora a 37 °C, quindi raffreddare a 4 °C. Mettere in ghiaccio il prima possibile dopo aver raggiunto i 4 °C.
- Utilizzando una striscia fresca di PCR da 0,2 mL a 8 provette, effettuare le diluizioni appropriate del campione trattato con DNasi nel tampone di diluizione AAV. Le diluizioni consigliate per i campioni dipendono dal titolo vg previsto (Tabella 2), ma puntare ad almeno 2 diverse diluizioni per campione ed eseguire ciascuna diluizione in duplicato. Assicurarsi di agitare rigorosamente (2.000-3.000 giri/min) i campioni per 5 s prima di estrarre qualsiasi volume.
- Dopo la diluizione in serie, agitare i campioni e centrifugare per assicurarsi che tutto il liquido rimanga sul fondo delle provette.
- Includere un controllo positivo di cui è noto il titolo vg e un controllo negativo (solo tampone di diluizione AAV) che è anche chiamato controllo senza modello (NTC).
NOTA: Il controllo positivo può essere un AAV prodotto internamente o un materiale di riferimento AAV commerciale, che include la sequenza target dei primer/sonda.
3. Preparazione di dd_PCR master mix
NOTA: Selezionare una sequenza target appropriata all'interno del genoma vettoriale e progettare un primer diretto, un primer inverso e sonde di idrolisi etichettate con fluorofori reporter FAM o HEX seguendo le linee guida pubblicate13. I primer che hanno come bersaglio il transgene sono preferiti rispetto ai primer che hanno come bersaglio gli ITR poiché il transgene è specifico per la progettazione del genoma del vettore e la struttura secondaria associata alla formazione a forcina degli ITR può ostacolare un efficiente legame del primer. Inoltre, i primer ITR potrebbero sovrastimare i titoli AAV se sono presenti capsidi parzialmente riempiti che contengono frammenti di genoma vettoriale troncato e ospitano ancora una delle sequenze ITR14. Preparare la dd_PCR master mix in una postazione di lavoro dedicata e separata (sala pre-PCR). Questa stanza deve essere separata da quella in cui vengono preparati i campioni per evitare contaminazioni incrociate.
- Lasciare che il primer, la sonda e dd_PCR reagenti supermix raggiungano la temperatura ambiente (20-25 °C). Successivamente, agitare tutti i reagenti e centrifugare brevemente le provette per raccogliere tutto il liquido sul fondo della provetta.
- Preparare un volume di dd_PCR master mix in una provetta da microcentrifuga separata in base al numero richiesto di reazioni combinando i primer diretti e inversi, la sonda dd_PCR supermix e l'acqua priva di DNasi secondo i volumi e le concentrazioni descritti nella Tabella 3.
- Agitare la miscela master e quindi centrifugare brevemente la provetta per assicurarsi che tutto il liquido rimanga sul fondo della provetta.
NOTA: Preparare sempre un volume di miscelazione master dd_PCR superiore a quello necessario (n + 1) per tenere conto della perdita di volume di pipettaggio.
- Agitare la miscela master e quindi centrifugare brevemente la provetta per assicurarsi che tutto il liquido rimanga sul fondo della provetta.
- Trasferire 19,8 μl della miscela master in ciascuna provetta su una striscia fresca da 0,2 mL di PCR da 8 provette.
4. Generazione di goccioline
NOTA: I campioni e dd_PCR master mix vengono miscelati separatamente prima di essere caricati in una cartuccia che genera goccioline. Eseguire tutte le manipolazioni su una workstation PCR, preferibilmente diversa da quella in cui sono stati preparati i campioni, per evitare la contaminazione da ampliconi. In alternativa, pulire accuratamente la workstation PCR dopo aver preparato i campioni.
- Trasferire 2,2 μl del campione diluito nel volume di 19,8 μl della miscela master dd_PCR in ciascuna provetta della striscia di 8 provette per PCR e mescolare bene. Agitare e centrifugare brevemente per assicurarsi che tutto il liquido rimanga sul fondo.
- Trasferire 20 μl di questa soluzione nei pozzetti contenuti nella fila centrale di "campione" di una cartuccia generatrice di goccioline. Evitare la formazione di bolle nella cartuccia che genera goccioline; Ciò può causare errori nello strumento e la mancata generazione di goccioline.
NOTA: Poiché le cartucce sono in formato a 8 pozzetti, così come le strisce di provette per PCR, si consiglia di utilizzare una pipetta a 8 canali. - Trasferire 60 μl di olio per la generazione di goccioline nei pozzetti contenuti nella fila inferiore di "olio" della cartuccia per la generazione di goccioline.
NOTA: Non lasciare mai vuoto un pozzetto della cartuccia generatrice di goccioline; Riempire il pozzetto del campione vuoto con dd_PCR controllo del tampone per sonde (con il volume corrispondente descritto sopra). - Posizionare una guarnizione di gomma sulla cartuccia che genera goccioline e posizionarla nel generatore di gocce.
- Dopo aver generato le goccioline, utilizzare una pipetta a 8 canali per trasferire lentamente la soluzione da 42,5 μl dal chip dd_PCR a una piastra PCR a più pozzetti.
NOTA: Pipettare lentamente e con un angolo di 45 gradi per evitare il taglio delle goccioline nel liquido durante il pipettaggio. Ispezionare visivamente la piastra in modo che tutti i pozzetti siano riempiti con la stessa quantità di liquido e che le goccioline siano visibili come uno strato opaco nel pozzetto. - Sigillare la piastra con un foglio di alluminio in una termosaldatrice per 5 s a 180 °C.
NOTA: Ispezionare visivamente che tutti i pozzetti siano sigillati. In caso contrario, ripetere altri 5 s a 180 °C. La piastra sigillata può essere conservata per un massimo di 4 ore a 4 °C prima di dd_PCR amplificazione in un termociclatore.
5. Amplificazione dd_PCR
NOTA: Il termociclatore deve essere collocato in una stanza separata dalla sala pre-PCR per separare spazialmente le attività pre-PCR dall'amplificazione PCR ed evitare risultati falsi positivi dovuti alla contaminazione da ampliconi.
- Posizionare la piastra in un termociclatore (con un modulo di reazione a 96 pozzetti profondi) per l'amplificazione PCR e chiuderla saldamente.
- Configurare il termociclatore in modo che funzioni alle condizioni descritte nella Tabella 4 e con un volume di reazione di 40 μL. Includere una fase di preriscaldamento nelle impostazioni del programma e assicurarsi che il coperchio sia riscaldato a 99 °C.
- Al termine del programma PCR, mantenere la piastra a 4 °C per almeno 15 minuti per assicurarsi che la piastra si sia completamente raffreddata.
NOTA: La piastra può essere conservata per 48-72 ore a 4 °C prima di dover leggere le goccioline.
6. Lettura delle goccioline
- Caricare la piastra in un lettore di gocce, inserire le seguenti informazioni nel software di sistema e continuare la lettura.
NOTA: Tipo di esperimento = Quantificazione diretta; Supermix = dd_PCR supermix per le sonde (no dUTP); Tipo di test = singolo bersaglio per canale; Informazioni sul target, canale del segnale 1 = canale FAM o canale HEX a seconda della sonda utilizzata. - Annotare i campioni in modo che la posizione di ciascun campione possa essere determinata e identificata in modo efficace dal file di dati.
7. Analisi dei dati
- Eseguire l'analisi dei dati utilizzando il software compatibile fornito dal produttore del lettore di gocce.
- Effettuare un test di idoneità del sistema per valutare le prestazioni complessive (e, quindi, l'affidabilità) del test.
- Controllare il conteggio degli eventi (numero di goccioline) misurato per ciascun pozzetto sulla piastra PCR. Idealmente, il numero di eventi è compreso tra 15.000 e 20.000. Se il conteggio degli eventi è <10.000, questo valore deve essere escluso dall'analisi.
- Valutare il grafico dell'ampiezza 1D o 2D e verificare la presenza di pioggia di goccioline.
- Vai alla scheda della tabella dati ed esporta i dati in un file Excel.
- Calcola il titolo vg seguendo i passaggi seguenti:
- L'intervallo di lavoro di dd_PCR è 10-10.000 copie/μL. Escludere dall'analisi qualsiasi valore che sia <10 copie/μL. L'intervallo consigliato per l'apparecchiatura dd_PCR utilizzata nel presente documento è di 25-5000 copie/μl.
NOTA: Il software di analisi dei dati fornirà un numero di genoma vettoriale in base al volume di reazione della PCR (40 μL), ma questo non prende in considerazione i fattori di diluizione applicati durante la preparazione del campione. - Calcolare il titolo vg del campione per volume (tipicamente espresso come vg/mL) tenendo conto dei vari fattori di diluizione durante la preparazione del campione utilizzando la seguente relazione:
NOTA: Concentrazione (valore ottenuto dall'analisi dei dati) x 10 (diluizione pre-trattamento DNasi I) x 10 (diluizione della master mix PCR) x 1000 (da μL a mL) diluizione del campione selezionato (in tampone di diluizione AAV - ad es. Tabella 2). - Calcolare il coefficiente di variazione (%CV) tra le repliche e tra le diverse diluizioni dello stesso campione.
NOTA: %CV si calcola dividendo la deviazione standard per la media. Se la %CV tra le diverse diluizioni è >20 %, il valore non deve essere considerato accurato. - Controllare i valori del controllo negativo e positivo. Il controllo negativo deve essere inferiore a 5 copie/μL.
- L'intervallo di lavoro di dd_PCR è 10-10.000 copie/μL. Escludere dall'analisi qualsiasi valore che sia <10 copie/μL. L'intervallo consigliato per l'apparecchiatura dd_PCR utilizzata nel presente documento è di 25-5000 copie/μl.
Representative Results
I risultati possono essere visualizzati utilizzando un software appropriato. Per ogni pozzetto, il grafico dell'ampiezza 1D mostra tutte le goccioline relative alla loro ampiezza. Ci si aspetta una netta separazione tra goccioline positive e negative. Se più del 10% delle goccioline totali si trova tra le nuvole di goccioline positive e negative (un fenomeno noto come pioggia di goccioline), è necessario rimisurare il campione (vedi Figura 1). Ulteriori informazioni sulla pioggia di goccioline sono disponibili nella discussione.
È possibile creare una tabella di dati per riassumere tutte le informazioni registrate, tra cui il nome del campione, il numero di goccioline accettate (conteggio degli eventi) e la concentrazione (copie/μL). Idealmente, il numero di eventi dovrebbe essere compreso tra 15.000 e 20.000 goccioline accettate. Se il numero di eventi per un pozzo è inferiore a 10.000, il punto dati deve essere escluso dall'analisi. Un esempio dei dati di output è fornito nella Tabella 5.
Il titolo vg può essere calcolato in base alla concentrazione (copie/μL), tenendo conto delle diluizioni del campione. L'intervallo di lavoro di dd_PCR è di 10-10.000 copie/μL. Valori inferiori a 10 copie/μL devono essere esclusi dall'analisi. Devono essere calcolati il titolo vg per i campioni, nonché i controlli positivi e negativi. Il valore misurato del controllo positivo deve avere un coefficiente di variazione (%CV) inferiore al 20% rispetto al valore teorico. Il controllo negativo deve essere inferiore a 5 copie/μL. Inoltre, deve essere calcolata la %CV tra le ripetizioni e le diverse diluizioni per ciascun campione. Se la percentuale di CV tra le diverse diluizioni supera il 20%, il valore può essere considerato impreciso e può essere necessario misurare nuovamente il campione.
Una misurazione di successo è caratterizzata da una netta separazione delle nuvole di goccioline positive e negative, almeno 10.000 goccioline accettate, un valore compreso tra 10 copie/μL e 10.000 copie/μL e una %CV tra le ripetizioni inferiore al 20%.
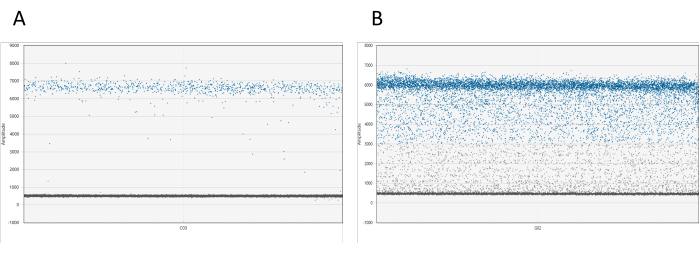
Figura 1: Visualizzazione delle goccioline dd_PCR. (A) Il grafico dell'ampiezza 1D mostra una chiara separazione delle goccioline positive e negative, indicando una partizione delle goccioline riuscita. (B) Il grafico mostra una scarsa separazione delle goccioline positive e negative, nota come pioggia di goccioline, che suggerisce una partizione non ottimale o potenziali problemi con il saggio. Clicca qui per visualizzare una versione più grande di questa figura.
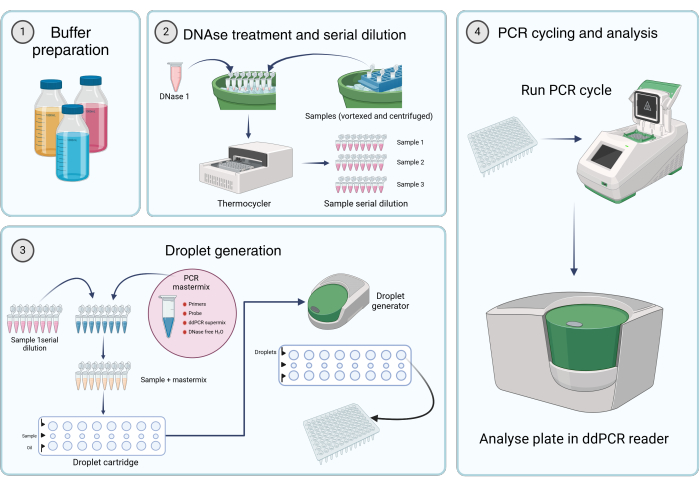
Figura 2: Flusso di lavoro per la quantificazione dei genomi virali mediante PCR digitale con goccioline. (1) I tamponi, i reagenti e le soluzioni vengono preparati secondo le istruzioni del produttore o la fase 1 del protocollo (Preparazione delle soluzioni stock). (2) 45 μl della soluzione di DNasi I vengono aliquotati in ciascuna provetta di una striscia per PCR da 8 provette. Dopo il vortice e la breve centrifugazione dei campioni, 5 μl di campione vengono aggiunti a una delle provette. La DNasi I e la striscia PCR a otto provette contenente campioni vengono incubate in un termociclatore per 1 ora a 37 °C, seguite da diluizioni seriali dei campioni. (3) La mastermix per PCR viene preparata come descritto e 19,8 μl vengono aliquotati in ciascuna provetta di una striscia per PCR da 8 provette. Le diluizioni seriali campione della fase 2 vengono aggiunte al mastermix. 20 μL di mastermix plus
La soluzione campione viene caricata nella fila centrale della cartuccia e 60 μL di olio generatore di goccioline vengono trasferiti ai pozzetti inferiori di una cartuccia generatrice di goccioline. La cartuccia viene quindi inserita nel generatore di goccioline e fatta funzionare secondo le condizioni specificate. Dopo la generazione di goccioline, 42,5 μl dalla fila superiore della cartuccia vengono trasferiti a una piastra PCR a più pozzetti. (4) La piastra PCR viene caricata in un termociclatore PCR ed eseguita secondo le condizioni fornite. La lastra viene analizzata utilizzando un lettore dd_PCR. Clicca qui per visualizzare una versione più grande di questa figura.
Tabella 1: Dati di precisione. Questa tabella presenta i dati di precisione provenienti da quattro serie di campioni di controllo qualità (QC), ciascuno con cinque concentrazioni (QC1-QC5). Ogni QC è stato misurato cinque volte. Viene mostrato il coefficiente di variazione (%CV) tra diverse esecuzioni, che rappresenta la ripetibilità (A), e tra diversi set di campioni, che rappresenta la precisione intermedia (B). Clicca qui per scaricare questa tabella.
Tabella 2: Diluizioni del campione. La tabella mostra le diluizioni raccomandate del campione in base al titolo del genoma virale previsto (vg/mL). Si raccomanda un totale di tre diluizioni per garantire che almeno due valori rientrino nell'intervallo di lavoro del test. Clicca qui per scaricare questa tabella.
Tabella 3: dd_PCR composizione del master mix. Questa tabella delinea la composizione della dd_PCR master mix, che include un primer diretto e inverso (909 nM), una sonda (227 nM) e dd_PCR supermix per sonde (senza dUTP, 1x). Clicca qui per scaricare questa tabella.
Tabella 4: Condizioni di termociclo. La tabella descrive in dettaglio il programma di PCR raccomandato, che include: (1) un'incubazione di 10 minuti a 95 °C per la distruzione del capside e l'attivazione enzimatica, (2) 40 cicli di 30 s a 94 °C per la denaturazione del DNA e 1 minuto a 60 °C per la ricottura e l'estensione, (3) un'incubazione di 10 minuti a 98 °C per la disattivazione degli enzimi, e (4) mantenimento a 4 °C. La temperatura di ricottura può richiedere un'ottimizzazione in base al set di primer/sonda utilizzato. Clicca qui per scaricare questa tabella.
Tabella 5: Esempio di dati di output da un'esecuzione AAV effettiva. La tabella fornisce un esempio di dati di output da un'esecuzione AAV effettiva, in cui un campione è stato misurato in due diverse diluizioni, ciascuna in duplicato. Il titolo del genoma virale (vg) (in vg/mL) viene calcolato utilizzando la seguente formula: 10 (pretrattamento con DNasi I) x 10 (diluizione nella miscela master) x 1.000 (μL in mL) x diluizione in tampone di diluizione AAV. STDEV rappresenta la deviazione standard e CV indica il coefficiente di variazione. Clicca qui per scaricare questa tabella.
Discussion
La quantificazione accurata delle copie del genoma del vettore AAV nelle preparazioni vettoriali è essenziale per lo sviluppo di prodotti di terapia genica basati su AAV. Esistono diversi metodi per determinare il titolo vg, con la PCR quantitativa (qPCR) e la dd_PCR che sono le due tecniche più utilizzate e accettate. dd_PCR è spesso preferita alla qPCR per la sua indipendenza dall'efficienza di amplificazione, la maggiore precisione e la maggiore robustezza15. In letteratura sono disponibili numerosi protocolli per la titolazione del genoma vettoriale mediante dd_PCR, ciascuno con i propri metodi di preparazione dei campioni 15,16,17. Tuttavia, manca un protocollo di consenso qualificato. Questo articolo presenta un protocollo di dd_PCR convalidato e idoneo allo scopo per la quantificazione dei genomi dei vettori AAV in campioni di vettori purificati (Figura 2).
Quando si esegue questo protocollo, è fondamentale prestare molta attenzione alla gestione dei campioni. La contaminazione incrociata può rappresentare una sfida significativa nella valutazione accurata dei titoli vg; pertanto, i campioni vengono elaborati al meglio con una workstation PCR per evitare la contaminazione con DNA estraneo. Inoltre, la corretta esecuzione dell'incubazione della DNasi è fondamentale per eliminare il DNA contaminante non confezionato senza interrompere il capside e potenzialmente digerire il genoma del vettore confezionato. Molti protocolli includono l'inattivazione del calore e il trattamento con proteinasi K 16,17,18. Tuttavia, durante lo sviluppo interno del metodo, è stato riscontrato che un riscaldamento eccessivo era dannoso per la titolazione vg e che il trattamento con proteinasi K non era necessario (dati non mostrati).
Poiché dd_PCR diventa sempre più popolare per quantificare i genomi virali, i produttori hanno pubblicato guide applicative dettagliate con linee guida sulla progettazione e l'ottimizzazione dei dd_PCR saggi19. Sono disponibili anche informazioni sulla risoluzione dei problemi. In pratica, quando un test è progettato correttamente, i problemi più frequenti includono la pioggia di goccioline e il basso numero di goccioline. La pioggia di goccioline è spesso causata dalla scarsa accessibilità dell'innesco/sonde all'amplicona, ma diverse strategie possono risolvere questo problema. Ad esempio, un esperimento sul gradiente di temperatura può aiutare a stabilire la temperatura di ricottura ottimale. Inoltre, la diluizione dei campioni per ridurre la quantità di DNA (si consiglia di essere inferiore a 66 ng) o l'esecuzione di una digestione di restrizione con enzimi specifici che tagliano al di fuori della regione dell'amplicone possono migliorare l'accessibilità del modello. Una raccomandazione di 10 U di enzima di restrizione per μg di DNA è in genere efficace. Il problema del basso numero di goccioline (meno di 10.000) è spesso dovuto a uno scarso pipettaggio del campione e dell'olio nelle cartucce. Si consiglia di pipettare lentamente con puntali appropriati per evitare il taglio delle goccioline. Sebbene il test dd_PCR sia robusto, una limitazione è il tempo prolungato per ottenere il risultato. Il processo dal trattamento del campione alla lettura delle goccioline richiede diverse ore, il che può rappresentare uno svantaggio quando i risultati sono necessari rapidamente.
La quantificazione accurata delle copie del genoma AAV nelle preparazioni vettoriali è necessaria in tutte le fasi del ciclo di vita della terapia genica AAV. In particolare, è importante per ottimizzare i processi di produzione e purificazione, condurre studi preclinici e determinare i dosaggi clinici per i prodotti di terapia genica a base di AAV. Il protocollo dd_PCR qui presentato è ampiamente applicabile e può essere utilizzato su prodotti AAV purificati con diversi sierotipi e transgeni.
Disclosures
IVH, BM ed EH sono co-fondatori di Tavira Therapeutics. Gli altri autori dichiarano di non avere conflitti di interesse.
Acknowledgements
Questa ricerca è stata resa possibile dalla sovvenzione "Flanders Resilience" del governo fiammingo, proveniente dal "Fondo europeo per la ripresa e la resilienza" (RRF) (VV021/13). La Figura 2 viene creata con Biorender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
References
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- . Planning droplet digital PCR experiments Available from: https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024)
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242 (2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570 (2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Droplet digital PCR applications guide. Bulletin 6407 Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024)
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved