Quantificação de genomas virais adeno-associados em amostras de vetores purificados por reação em cadeia da polimerase por gotículas digitais
In This Article
Summary
A quantificação precisa das cópias do genoma do vetor do vírus adeno-associado (AAV) é crítica, mas um protocolo padronizado ainda não foi estabelecido. Este protocolo descreve um método validado para preparar amostras de AAV purificadas e conduzir a reação em cadeia da polimerase de gotículas digitais (dd_PCR) para quantificar de forma confiável o título do genoma viral.
Abstract
O vírus adeno-associado (AAV) é um vírus não patogênico usado como veículo de entrega para transferir genes terapêuticos para os pacientes. A quantificação precisa do número de cópias do genoma do AAV em preparações de vetores é essencial para a otimização de bioprocessos e cálculo de dosagem em estudos pré-clínicos e clínicos de produtos de terapia gênica baseados em AAV. Atualmente, falta um protocolo de consenso para a titulação do genoma viral do AAV. Aqui, apresentamos um protocolo de PCR de gotículas digitais (dd_PCR) para a quantificação de genomas virais em amostras de vetores purificados. As amostras são tratadas com DNase I para eliminar o DNA contaminante não embalado. As amostras tratadas com DNase são então misturadas com um conjunto de primer-sonda apropriado (projetado de acordo com o genoma AAV alvo) e reagentes de PCR e, em seguida, carregados em um gerador de gotículas. As gotículas preparadas são transferidas para uma placa de PCR, onde a amplificação por PCR é realizada e analisada. O título do genoma viral é calculado com base na concentração (cópias/μL), contabilizando as diluições das amostras. Uma medição bem-sucedida mostra uma separação clara das nuvens de gotículas positivas e negativas, tem pelo menos 10.000 gotículas aceitas, mostra um valor entre 10 cópias/μL e 10.000 cópias/μL e tem um coeficiente de variação (CV) entre repetições inferior a 20%. A titulação confiável do genoma viral ajudará no desenvolvimento de produtos de terapia genética seguros e eficazes baseados em AAV.
Introduction
A terapia gênica é uma modalidade terapêutica comumente usada para tratar distúrbios genéticos. O desenho de qualquer terapia gênica é específico para a patologia associada à indicação alvo, mas todas as terapias gênicas envolvem a entrega intracelular de material genético às células-alvo para obter um efeito terapêutico1. A terapia gênica pode ser classificada em várias categorias, incluindo substituição de genes para mutações de perda de função, silenciamento de genes para anormalidades de ganho de função e técnicas de edição de genes. Independentemente da estratégia específica empregada, o material de ácido nucleico terapêutico (referido como transgene) deve ser empacotado dentro de um vetor para atingir a entrega intracelular direcionada2.
Embora uma variedade de sistemas de vetores virais e não virais esteja disponível para o desenvolvimento de terapia gênica, os vírus adeno-associados (AAVs) são frequentemente escolhidos devido ao amplo tropismo viral e à baixa imunogenicidade associada a esse grupo de vírus 1,2. Até o momento, sete terapias genéticas que utilizam AAV para entrega terapêutica de genes obtiveram aprovação da Agência Europeia de Medicamentos (EMA) ou da Food and Drug Administration (FDA) visando doenças que variam de hemofilia (por exemplo, Roctavian) a atrofia muscular espinhal (por exemplo, Zolgensma)3.
A produção de terapias genéticas baseadas em AAV decorre de uma compreensão do próprio AAV do tipo selvagem. O AAV é um pequeno vírus de DNA da família Parvoviridae que compreende 13 sorotipos principais (AAV1-13)3. O genoma AAV compreende uma molécula de DNA de fita simples de ~ 4,7 kb contendo dois quadros de leitura abertos principais (ORFs) que codificam os genes virais essenciais necessários para a replicação do genoma, montagem do capsídeo e empacotamento (rep, cap). O genoma viral é flanqueado nas extremidades 5 'e 3' por sequências de nucleotídeos palindrômicos, chamadas de repetições terminais invertidas (ITRs). Esses ITRs formam estruturas semelhantes a grampos que desempenham papéis cruciais na replicação do genoma e no empacotamento de genomas virais de novo em capsídeos virais recém-sintetizados. O AAV é um vírus dependente de auxiliares e, portanto, requer a expressão de genes auxiliares de outros vírus, como o vírus herpes simplex (HSV) ou o adenovírus (AdV), para se tornar competente em replicação1.
Para produzir AAVs, um sistema de expressão baseado em células adequado é empregado para facilitar a expressão das proteínas do capsídeo viral e subsequente montagem em partículas virais de novo, seguido pelo encapsulamento de um transgene selecionado flanqueado por ITR (também conhecido como genoma vetorial). Este processo geralmente utiliza um sistema de plasmídeo triplo, compreendendo (1) um plasmídeo que abriga genes auxiliares derivados de um vírus auxiliar, (2) um plasmídeo que codifica os elementos virais essenciais (rep / cap) e (3) um plasmídeo que carrega o de expressão terapêutica (comumente referido como plasmídeo de transferência) 4. A presença única de sinais de empacotamento de repetição terminal invertida (ITR) flanqueando o de expressão terapêutica no plasmídeo de transferência garante o empacotamento específico do transgene, excluindo principalmente os genes virais presentes nos outros plasmídeos. A co-transfecção deste sistema de três plasmídeos em uma plataforma de expressão baseada em células (normalmente HEK293T células) resulta na produção de partículas virais competentes em transdução e deficientes em replicação, adequadas para uso em aplicações de terapia gênica 3,4.
Há uma série de atributos críticos de qualidade (CQAs) associados à produção de terapias genéticas baseadas em AAV que devem ser avaliados para garantir a potência, pureza e segurança do medicamento pretendido4. Esses CQAs incluem título de vírus, conteúdo de capsídeo e agregação. O título do vírus em si é uma combinação do número de partículas virais (título do capsídeo) e do número de genomas vetoriais (título do genoma do vetor) presentes em qualquer preparação. Idealmente, a proporção entre esses dois títulos deve ser de 1:1, uma vez que cada partícula viral deve conter um genoma vetorial, mas as ineficiências no empacotamento do genoma vetorial durante a biossíntese resultam na coprodução de capsídeos vazios ou parcialmente preenchidos (aqueles contendo sequências parciais do genoma do vetor e/ou sequências do genoma não vetorial)5. A presença de tais impurezas pode potencialmente evocar respostas imunes injustificadas e competir por locais de ligação ao vetor, aumentando assim o risco de imunotoxicidade e reduzindo a taxa de transdução de capsídeos completos6. A quantificação precisa dos genomas do AAV é, portanto, essencial para estabelecer o título do vírus e o conteúdo do capsídeo. Isso afeta tanto a pesquisa básica quanto a indústria de terapia genética, que requer dosagem precisa para manter a segurança e a eficácia dos medicamentos.
A reação em cadeia da polimerase de gotículas digitais (dd_PCR) tornou-se intimamente associada à quantificação do título do vírus, uma vez que pode ser utilizada para determinar o número de genomas vetoriais presentes em qualquer preparação7. A própria PCR digital foi introduzida pela primeira vez na década de 1990 8,9 e dd_PCR é um aprimoramento dessa tecnologia que permite o processamento de amostras de alto rendimento10,11. Em dd_PCR, uma reação de PCR em tempo real de 20 μL é particionada em aproximadamente 20.000 gotículas envoltas em óleo, dando até 96 dessas reações quando acomodadas em uma placa padrão de 96 poços. Em comparação com a PCR quantitativa convencional (qPCR), a dd_PCR oferece várias vantagens, incluindo sensibilidade aprimorada, maior precisão e quantificação mais direta e absoluta das sequências-alvo sem a necessidade de curvas padrão. Além disso, o alto nível de particionamento em dd_PCR reduz o impacto dos inibidores de PCR e minimiza o potencial de viés da amplificação preferencial de certos modelos, tornando-o uma opção atraente para medição analítica da titulação do genoma do vetor.
Protocol
O protocolo descrito aqui é projetado para quantificar o título do genoma viral de um vetor AAV9 purificado e produzido internamente com proteína fluorescente verde (GFP) como o transgene12 com um alto nível de precisão (Tabela 1). No entanto, este protocolo é aplicável a qualquer sorotipo AAV e a qualquer projeto de genoma vetorial, desde que os conjuntos de primer / sonda sejam projetados para atingir o genoma vetorial específico de interesse. Os detalhes dos reagentes, primers, sondas e equipamentos usados neste estudo estão listados na Tabela de Materiais.
1. Preparação das soluções-mãe
NOTA: Prepare todas as soluções de estoque necessárias para a diluição da amostra em uma estação de trabalho de PCR para evitar a contaminação por DNA estranho.
- Prepare 10x tampão de ensaio de PCR, que consiste em 500 mM de KCl, 100 mM de Tris-HCl, 15 mM de MgCl2 e 0,01% (p/v) de BSA. Medir o pH da solução e ajustá-lo a 8,3.
NOTA: Esta solução pode ser mantida em temperatura ambiente por até 1 mês. - Prepare o tampão de diluição AAV, que consiste em 1x tampão de ensaio de PCR, 0,2 ng/μL de DNA cortado de esperma de salmão (sss) e 0,1% de F-68 plurônico. Fazer esta solução de frescor com cada doseamento e mantê-la a 4 °C.
NOTA: Se o F-68 plurônico não estiver disponível, uma alternativa como o surfactante não iônico Poloxamer 188 pode ser usada.
2. Preparação de amostras - tratamento com DNase I e diluição seriada
NOTA: Para remover contaminantes de DNA que podem diminuir a precisão da titulação do genoma do vetor, o DNA livre presente na amostra de AAV (incluindo DNA de plasmídeo remanescente ou genomas virais não encapsulados) pode ser removido antes da amplificação por PCR usando DNase. Isso é possível porque os genomas do vetor estão encapsulados nos capsídeos do AAV e, portanto, não são acessíveis até a etapa de desnaturação da própria reação de PCR (ver etapa 5). Além disso, como o número do genoma do vetor em qualquer amostra é desconhecido, é necessário realizar uma diluição em série das amostras para garantir que as medições permaneçam dentro dos limites superior e inferior detectáveis. Execute todas as manipulações de amostras em uma estação de trabalho de PCR. Mantenha todas as amostras no gelo, a menos que especificado de outra forma.
- Misturar a amostra de AAV seleccionada para análise por vórtice breve e centrifugação para garantir que todo o líquido permanece no fundo do tubo que contém a amostra.
- Para o tratamento com DNase, adicione 45 μL de uma solução contendo 1x tampão DNase I, 0,1% de F-68 plurônico e 0,04 U / μL de DNase I em água livre de DNase em um tubo de tira de 8 tubos de PCR de 0,2 mL.
- Transfira 5 μL de amostra para o tubo que contém a solução de DNAse. Vortex as amostras e centrifugue brevemente para garantir que todo o líquido permaneça no fundo.
- Em um termociclador, incubar as amostras por 1 h a 37 °C e depois esfriar até 4 °C. Coloque no gelo o mais rápido possível após atingir 4 °C.
- Usando uma nova tira de 8 tubos de PCR de 0,2 mL, faça diluições apropriadas da amostra tratada com DNase em tampão de diluição AAV. As diluições de amostra recomendadas dependem do título vg esperado (Tabela 2), mas tente pelo menos 2 diluições diferentes por amostra e execute cada diluição em duplicata. Certifique-se de rigorosamente (2.000-3.000 rpm) vórtice as amostras por 5 s antes de retirar qualquer volume.
- Após a diluição em série, vortex as amostras e centrifugue para garantir que todo o líquido permaneça no fundo dos tubos.
- Inclua um controle positivo cujo título vg é conhecido e um controle negativo (somente tampão de diluição AAV) que também é chamado de controle sem modelo (NTC).
NOTA: O controle positivo pode ser um AAV produzido internamente ou um material de referência AAV comercial, que inclui a sequência alvo dos primers/sonda.
3. Preparação de dd_PCR master mix
NOTA: Selecione uma sequência-alvo apropriada dentro do genoma do vetor e projete um primer direto, primer reverso e sondas de hidrólise marcadas com fluoróforos repórteres FAM ou HEX seguindo as diretrizes publicadas13. Os primers que têm como alvo o transgene são preferidos aos primers que têm como alvo os ITRs, uma vez que o transgene é específico para o projeto do genoma do vetor, e a estrutura secundária associada à formação de grampos de cabelo dos ITRs pode dificultar a ligação eficiente do primer. Além disso, os primers ITR podem superestimar os títulos de AAV se os capsídeos parcialmente preenchidos que contêm fragmentos do genoma do vetor truncado estiverem presentes e ainda abrigarem qualquer uma das sequências ITR14. Prepare o master mix dd_PCR em uma estação de trabalho separada e dedicada (sala de pré-PCR). Esta sala deve ser separada daquela onde as amostras são preparadas para evitar contaminação cruzada.
- Deixe o primer, a sonda e os reagentes de supermistura dd_PCR atingirem a temperatura ambiente (20-25 °C). Depois, vortex todos os reagentes e centrifugue brevemente os tubos para coletar todo o líquido no fundo do tubo.
- Preparar um volume de dd_PCR mistura principal num tubo de microcentrífuga separado, de acordo com o número necessário de reacções, combinando os primários directos e inversos, a sonda, a supermistura dd_PCR e a água isenta de DNase, de acordo com os volumes e concentrações descritos no quadro 3.
- Vortex a mistura principal e, em seguida, centrifugue brevemente o tubo para garantir que todo o líquido permaneça no fundo do tubo.
NOTA: Sempre prepare dd_PCR volume de mistura principal extra do que o necessário (n + 1) para levar em conta a perda de volume de pipetagem.
- Vortex a mistura principal e, em seguida, centrifugue brevemente o tubo para garantir que todo o líquido permaneça no fundo do tubo.
- Transfira 19,8 μL da mistura principal para cada tubo em uma nova tira de 8 tubos de PCR de 0,2 mL.
4. Geração de gotículas
NOTA: As amostras e dd_PCR master mix são misturadas separadamente antes de serem carregadas em um cartucho gerador de gotículas. Realizar todas as manipulações em uma estação de trabalho de PCR, de preferência diferente de onde as amostras foram preparadas, para evitar a contaminação do amplicon. Como alternativa, limpe bem a estação de trabalho de PCR após preparar as amostras.
- Transfira 2,2 μL da amostra diluída para o volume de 19,8 μL de dd_PCR mistura principal em cada tubo da tira de 8 tubos PCR e misture bem. Vortex e centrifugue brevemente para garantir que todo o líquido permaneça no fundo.
- Transferir 20 μL desta solução para os alvéolos contidos na fileira central de "amostras" de um cartucho gerador de gotículas. Evite bolhas no cartucho gerador de gotículas; Isso pode fazer com que o instrumento dê erros e as gotículas não sejam geradas.
NOTA: Como os cartuchos estão no formato de 8 poços, assim como as tiras de tubo de PCR, recomenda-se o uso de uma pipeta de 8 canais. - Transfira 60 μL do óleo de geração de gotículas para os poços contidos na fileira inferior de "óleo" do cartucho gerador de gotículas.
NOTA: Nunca deixe um poço do cartucho gerador de gotículas vazio; Encha bem a amostra vazia com dd_PCR controle tampão para sondas (com o volume correspondente descrito acima). - Coloque uma junta de borracha sobre o cartucho gerador de gotículas e coloque-o no gerador de gotículas.
- Depois que as gotículas forem geradas, use uma pipeta de 8 canais para transferir lentamente a solução de 42,5 μL do chip dd_PCR para uma placa de PCR de vários poços.
NOTA: Pipete lentamente e em um ângulo de 45 graus para evitar o cisalhamento de gotículas no líquido durante a pipetagem. Inspecione visualmente a placa para que todos os poços sejam preenchidos com a mesma quantidade de líquido e as gotículas sejam visíveis como uma camada opaca no poço. - Sele a placa com uma tampa de folha de alumínio em uma máquina de selagem a quente por 5 s a 180 °C.
NOTA: Inspecione visualmente se todos os poços estão selados. Caso contrário, repita mais 5 s a 180 °C. A placa selada pode ser mantida por até 4 h a 4 °C antes de dd_PCR amplificação em um termociclador.
5. Amplificação dd_PCR
NOTA: O termociclador deve ser colocado em uma sala separada da sala de pré-PCR para separar espacialmente as atividades de pré-PCR da amplificação de PCR e evitar resultados falso-positivos devido à contaminação do amplicon.
- Coloque a placa em um termociclador (com um módulo de reação de poço de 96 profundidades) para amplificação de PCR e feche-a com segurança.
- Configure o termociclador para funcionar nas condições descritas na Tabela 4 e em um volume de reação de 40 μL. Inclua uma etapa de pré-aquecimento nas configurações do programa e certifique-se de que a tampa esteja aquecida a 99 °C.
- Quando o programa de PCR terminar, mantenha a placa a 4 °C por pelo menos 15 min para garantir que a placa esteja totalmente resfriada.
NOTA: A placa pode ser mantida por 48-72 h a 4 °C antes que as gotas tenham que ser lidas.
6. Leitura de gotículas
- Carregue a placa em um leitor de gotículas, insira as seguintes informações no software do sistema e continue a leitura.
NOTA: Tipo de experimento = Quantificação direta; Supermix = dd_PCR supermix para sondas (sem dUTP); Tipo de ensaio = alvo único por canal; Informações do alvo, canal de sinal 1 = canal FAM ou canal HEX de acordo com a sonda usada. - Anote as amostras para que a posição de cada amostra possa ser determinada e identificada efetivamente a partir do arquivo de dados.
7. Análise dos dados
- Realize a análise dos dados usando o software compatível fornecido pelo fabricante do leitor de gotículas.
- Realize um teste de adequação do sistema para avaliar o desempenho geral (e, portanto, a confiabilidade) do ensaio.
- Verifique a contagem de eventos (número de gotículas) medida para cada poço na placa de PCR. Idealmente, a contagem de eventos está entre 15.000 e 20.000. Se a contagem de eventos for < 10.000, esse valor deverá ser excluído da análise.
- Avalie o gráfico de amplitude 1D ou 2D e verifique a presença de chuva de gotículas.
- Vá para a guia da tabela de dados e exporte os dados para um arquivo do Excel.
- Calcule o título vg seguindo os passos abaixo:
- A faixa de trabalho de dd_PCR é de 10 a 10.000 cópias/μL. Exclua qualquer valor que seja <10 cópias/μL da análise. A faixa recomendada para o equipamento dd_PCR usado aqui é de 25 a 5000 cópias/μL.
NOTA: O software de análise de dados fornecerá um número do genoma do vetor de acordo com o volume da reação de PCR (40 μL), mas isso não leva em consideração os fatores de diluição aplicados durante a preparação da amostra. - Calcular o título de vg da amostra por volume (normalmente expresso em vg/ml) tendo em conta os vários factores de diluição ao longo da preparação da amostra, utilizando a seguinte relação:
NOTA: Concentração (valor obtido a partir da análise de dados) x 10 (diluição pré-tratamento DNase I) x 10 (diluição da mistura principal de PCR) x 1000 (μL a mL) diluição da amostra selecionada (em tampão de diluição AAV - por exemplo, Tabela 2). - Calcular o coeficiente de variação (%CV) entre os replicados e entre as diferentes diluições da mesma amostra.
NOTA: %CV é calculado dividindo o desvio padrão pela média. Se o %CV entre as diferentes diluições for >20 %, o valor não deve ser considerado exacto. - Verifique os valores do controle negativo e positivo. O controlo negativo deve ser inferior a 5 cópias/μl.
- A faixa de trabalho de dd_PCR é de 10 a 10.000 cópias/μL. Exclua qualquer valor que seja <10 cópias/μL da análise. A faixa recomendada para o equipamento dd_PCR usado aqui é de 25 a 5000 cópias/μL.
Representative Results
Os resultados podem ser visualizados usando o software apropriado. Para cada poço, o gráfico de amplitude 1D exibe todas as gotículas relativas à sua amplitude. Espera-se uma separação clara entre gotículas positivas e negativas. Se mais de 10% do total de gotículas forem encontradas entre as nuvens de gotículas positivas e negativas (um fenômeno conhecido como chuva de gotículas), é necessário medir novamente a amostra (ver Figura 1). Informações adicionais sobre chuva de gotículas estão disponíveis na discussão.
Uma tabela de dados pode ser criada para resumir todas as informações registradas, incluindo o nome da amostra, número de gotículas aceitas (contagens de eventos) e concentração (cópias/μL). Idealmente, a contagem de eventos deve ficar entre 15.000 e 20.000 gotículas aceitas. Se a contagem de eventos para um poço estiver abaixo de 10.000, o ponto de dados deverá ser excluído da análise. Um exemplo dos dados de saída é fornecido na Tabela 5.
O título vg pode ser calculado com base na concentração (cópias/μL), levando em consideração as diluições da amostra. A faixa de trabalho de dd_PCR é de 10 a 10.000 cópias/μL. Valores abaixo de 10 cópias/μL devem ser excluídos da análise. Deve ser calculado o título vg para as amostras, bem como os controlos positivos e negativos. O valor medido do controlo positivo deve ter um coeficiente de variação (%CV) inferior a 20% em relação ao valor teórico. O controlo negativo deve ser inferior a 5 cópias/μl. Além disso, deve ser calculada a %CV entre repetições e diluições diferentes para cada amostra. Se o %CV entre diferentes diluições exceder 20%, o valor pode ser considerado impreciso e a amostra pode ter de ser novamente medida.
Uma medição bem-sucedida é caracterizada por uma separação clara das nuvens de gotículas positivas e negativas, pelo menos 10.000 gotículas aceitas, um valor entre 10 cópias/μL e 10.000 cópias/μL e um %CV entre repetições inferior a 20%.
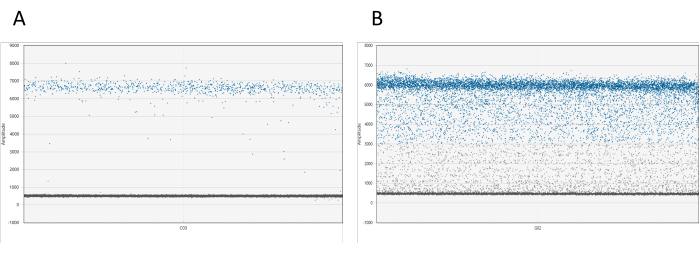
Figura 1: Visualização de dd_PCR gotículas. (A) O gráfico de amplitude 1D demonstra uma separação clara de gotículas positivas e negativas, indicando particionamento de gotículas bem-sucedido. (B) O gráfico mostra uma má separação de gotículas positivas e negativas, conhecida como chuva de gotículas, o que sugere partição abaixo do ideal ou possíveis problemas com o ensaio. Clique aqui para ver uma versão maior desta figura.
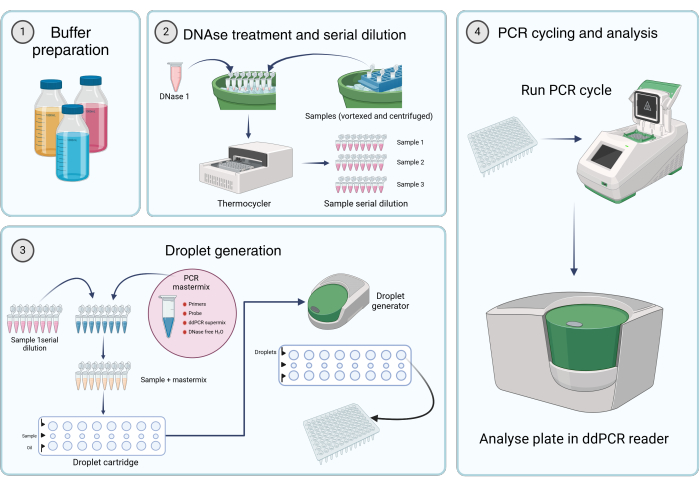
Figura 2: Fluxo de trabalho para quantificar genomas virais usando PCR de gotículas digitais. (1) Tampões, reagentes e soluções são preparados de acordo com as instruções do fabricante ou a etapa 1 do protocolo (Preparação de soluções de estoque). (2) 45 μL da solução de DNase I são aliquotados em cada tubo de uma tira de PCR de 8 tubos. Após o vórtice e a centrifugação breve das amostras, adicionam-se 5 μL da amostra a um dos tubos. A DNase I e a tira de PCR de oito tubos contendo amostra são incubadas em um termociclador por 1 h a 37 °C, seguidas de diluições seriadas das amostras. (3) O mastermix de PCR é preparado conforme descrito e 19,8 μL são aliquotados em cada tubo de uma tira de PCR de 8 tubos. Amostras de diluições seriais da etapa 2 são adicionadas ao mastermix. 20 μL do mastermix plus
A solução de amostra é carregada na fileira do meio do cartucho e 60 μL de óleo de geração de gotículas são transferidos para os poços inferiores de um cartucho gerador de gotículas. O cartucho é então colocado no gerador de gotículas e funciona de acordo com as condições especificadas. Após a geração de gotículas, 42,5 μL da linha superior do cartucho são transferidos para uma placa de PCR de vários poços. (4) A placa PCR é carregada em um termociclador PCR e executada de acordo com as condições fornecidas. A placa é analisada usando um leitor dd_PCR. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Dados de precisão. Esta tabela apresenta dados de precisão de quatro conjuntos de amostras de controle de qualidade (CQ), cada um com cinco concentrações (QC1-QC5). Cada CQ foi medido cinco vezes. O coeficiente de variação (%CV) entre diferentes ensaios, representando a repetibilidade (A), e entre diferentes conjuntos de amostras, representando precisão intermediária (B), é mostrado. Clique aqui para baixar esta tabela.
Tabela 2: Diluições da amostra. A tabela mostra as diluições de amostra recomendadas com base no título do genoma viral esperado (vg/mL). Um total de três diluições são recomendadas para garantir que pelo menos dois valores estejam dentro da faixa de trabalho do ensaio. Clique aqui para baixar esta tabela.
Tabela 3: dd_PCR composição da mixagem principal. Esta tabela descreve a composição do master mix dd_PCR, que inclui um primer direto e reverso (909 nM), sonda (227 nM) e dd_PCR supermix para sondas (sem dUTP, 1x). Clique aqui para baixar esta tabela.
Tabela 4: Condições de termociclagem. A tabela detalha o programa de PCR recomendado, que inclui: (1) uma incubação de 10 minutos a 95 ° C para interrupção do capsídeo e ativação enzimática, (2) 40 ciclos de 30 s a 94 ° C para desnaturação do DNA e 1 minuto a 60 ° C para recozimento e extensão, (3) uma incubação de 10 minutos a 98 ° C para desativação enzimática, e 4) a temperatura de 4 °C. A temperatura de recozimento pode exigir otimização com base no conjunto de primer/sonda usado. Clique aqui para baixar esta tabela.
Tabela 5: Exemplo de dados de saída de uma execução real do AAV. A tabela fornece um exemplo de dados de saída de uma execução real de AAV, em que uma amostra foi medida em duas diluições diferentes, cada uma em duplicata. O título do genoma viral (vg) (em vg/mL) é calculado usando a seguinte fórmula: 10 (pré-tratamento com DNase I) x 10 (diluição no master mix) x 1.000 (μL para mL) x diluição em tampão de diluição AAV. STDEV representa o desvio padrão e CV indica o coeficiente de variação. Clique aqui para baixar esta tabela.
Discussion
A quantificação precisa das cópias do genoma do vetor AAV em preparações de vetores é essencial para o desenvolvimento de produtos de terapia gênica baseados em AAV. Existem vários métodos para determinar o título de vg, sendo a PCR quantitativa (qPCR) e a dd_PCR as duas técnicas mais utilizadas e aceitas. dd_PCR é frequentemente preferido à qPCR devido à sua independência da eficiência de amplificação, maior precisão e maior robustez15. Numerosos protocolos para titulação do genoma do vetor por dd_PCR estão disponíveis na literatura, cada um com seus próprios métodos de preparação de amostras 15,16,17. No entanto, falta um protocolo de consenso qualificado. Este artigo apresenta um protocolo de dd_PCR validado para a quantificação de genomas de vetores AAV em amostras de vetores purificados (Figura 2).
Muita atenção ao manuseio de amostras é fundamental ao executar este protocolo. A contaminação cruzada pode representar um desafio significativo na avaliação precisa dos títulos de vg; portanto, as amostras são melhor processadas em uma estação de trabalho de PCR para evitar a contaminação com DNA estranho. Além disso, a execução adequada da incubação de DNase é crucial para eliminar o DNA contaminante não empacotado sem interromper o capsídeo e potencialmente digerir o genoma do vetor empacotado. Muitos protocolos incluem inativação por calor e tratamento com proteinase K 16,17,18. No entanto, durante o desenvolvimento do método interno, verificou-se que o aquecimento excessivo era prejudicial à titulação de vg e o tratamento com proteinase K era desnecessário (dados não mostrados).
À medida que dd_PCR se torna cada vez mais popular para quantificar genomas virais, os fabricantes publicaram guias de aplicação detalhados com diretrizes sobre como projetar e otimizar ensaios dd_PCR19. Informações sobre solução de problemas também estão disponíveis. Na prática, quando um ensaio é projetado corretamente, os problemas mais frequentes incluem chuva de gotículas e baixa contagem de gotas. A chuva de gotículas geralmente é causada pela baixa acessibilidade do primer/sondas ao amplicon, mas várias estratégias podem resolver esse problema. Por exemplo, um experimento de gradiente de temperatura pode ajudar a estabelecer a temperatura ideal de recozimento. Além disso, diluir amostras para diminuir a quantidade de DNA (recomendado para ser inferior a 66 ng) ou realizar uma digestão de restrição com enzimas específicas que cortam fora da região do amplicon pode melhorar a acessibilidade do modelo. Uma recomendação de 10 U de enzima de restrição por μg de DNA é tipicamente eficaz. O problema da baixa contagem de gotículas (menos de 10.000) é frequentemente devido à má pipetagem da amostra e do óleo nos cartuchos. Recomenda-se a pipetagem lenta com pontas apropriadas para evitar o cisalhamento das gotículas. Embora o ensaio dd_PCR seja robusto, uma limitação é o tempo prolongado para o resultado. O processo desde o tratamento da amostra até a leitura das gotículas leva várias horas, o que pode ser uma desvantagem quando os resultados são necessários rapidamente.
A quantificação precisa das cópias do genoma do AAV em preparações de vetores é necessária em todos os estágios do ciclo de vida da terapia gênica do AAV. Especificamente, é importante para otimizar os processos de produção e purificação, conduzir estudos pré-clínicos e determinar dosagens clínicas para produtos de terapia genética baseados em AAV. O protocolo dd_PCR apresentado aqui é amplamente aplicável e pode ser usado em produtos AAV purificados com diferentes sorotipos e transgenes.
Disclosures
IVH, BM e EH são co-fundadores da Tavira Therapeutics. Os demais autores declaram não haver conflito de interesses.
Acknowledgements
Esta investigação foi possível graças à subvenção «Resiliência da Flandres» do Governo flamengo, proveniente do «Mecanismo Europeu de Recuperação e Resiliência» (MRR) (VV021/13). A Figura 2 é criada com Biorender.com.
Materials
| Name | Company | Catalog Number | Comments |
| 8-channel pipette 10 µL | Eppendorf | 3,12,50,00,010 | |
| 8-channel pipette 200 µL | Eppendorf | 3,12,50,00,036 | |
| 8-channel pipette 300 µL | Eppendorf | 3,12,50,00,052 | |
| 8-well PCR strip | Sarstedt | 72.991.002 | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A9418 | |
| C1000 Touch Thermal Cycler with 96–Deep Well Reaction Module | Bio-Rad | 1851197 | |
| ddPCR Buffer Control for Probes 9 mL (2 x 4.5 mL) | Bio-Rad | 1863052 | |
| ddPCR Supermix for Probes (No dUTP) (2 x 1 mL) | Bio-Rad | 1863023 | |
| ddPCR 96-Well Plates (pkg of 25) | Bio-Rad | 12001925 | |
| ddPCR Droplet Reader Oil (2 x 1L) | Bio-Rad | 1863004 | |
| DG8 Cartridge Holder | Bio-Rad | 1863051 | |
| DG8 Cartridges for QX200/QX100 (pkg of 24) | Bio-Rad | 1864008 | |
| DG8 Gaskets for QX200/QX100 (pkg of 24) | Bio-Rad | 1863009 | |
| DNase I (10U/µL) + buffer | Roche | 4716728001 | |
| Droplet Generation Oil for Probes (10 x 7 mL) | Bio-Rad | 1863005 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 0.1-10 μL, 34 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078500 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 20-300 μL, 55 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078560 | |
| Eppendorf ep Dualfilter T.I.P.S. Filter Tip, 2-100 μL, 53 mm, Rack, PCR Clean, STERILE | Eppendorf | 30078543 | |
| Forward lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Forward primer: 5'-GAACGGCATCAAGGTGAACT-3' | |
| Lyopohilized probe and master stock at 100 µM | IDT | GFP as target sequence. PrimeTime Eco Probe: /56-FAM/CAAGATCCG/ZEN/CCACAACATCGAGGA/3IABkFQ/ | |
| Magnesium Chloride (MgCl2) | Chemlab Analytical | CL00.1381 | |
| Nuclease free water | IDT | 11-04-02-01 | |
| PCR Plate Heat Seal, foil, pierceable (pkg of 100) | Bio-Rad | 1814040 | |
| Pluronic F-68 non-ionic surfactant (100x) | Thermo Fisher Scientific | 24040032 | |
| Potassium Chloride (KCl) | Honeywell research chemicals | 31248 | |
| QX manager software | Bio-Rad | Software to analyse ddPCR data | |
| QX200 Droplet Generator | Bio-Rad | 1864002 | |
| QX200 Droplet Reader | Bio-Rad | 1864003 | |
| Reagent reservoir | VWR | 613-1181 | |
| Reverse lyophilized primers and respective master stocks at 100 mM | IDT | GFP as target sequence. Reverse primer: 5'-TGCTCAGGTAGTGGTTGTCG-3' | |
| SafeSeal reaction tube, 1.5 mL | Sarstedt | 72.706.200 | |
| Salmon Sperm DNA, sheared (10 mg/mL) | Thermo Fisher Scientific | AM9680 | |
| TE buffer | IDT | Accompanied by primers when ordering | |
| Tris hydrochloride (Tris-HCl) | Roche | 10812846001 |
References
- Balakrishnan, B., Jayandharan, G. R. Basic biology of adeno-associated virus (AAV) vectors used in gene therapy. Curr Gene Ther. 14 (2), 86-100 (2014).
- Srivastava, A. In vivo tissue-tropism of adeno-associated viral vectors. Curr Opin Virol. 21, 75-80 (2016).
- Pupo, A., et al. AAV vectors: The Rubik's cube of human gene therapy. Mol Ther. 30 (12), 3515-3541 (2022).
- Gimpel, A. L., et al. Analytical methods for process and product characterization of recombinant adeno-associated virus-based gene therapies. Mol Ther Methods Clin Dev. 20, 740-754 (2021).
- Brimble, M. A., et al. Preventing packaging of translatable P5-associated DNA contaminants in recombinant AAV vector preps. Mol Ther Methods Clin Dev. 24, 280-291 (2022).
- Wright, J. F. Product-related impurities in clinical-grade recombinant AAV vectors: Characterization and risk assessment. Biomedicines. 2 (1), 80-97 (2014).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, (2019).
- Sidransky, D., et al. Identification of ras oncogene mutations in the stool of patients with curable colorectal tumors. Science. 256 (5053), 102-105 (1992).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci USA. 96 (16), 9236-9241 (1999).
- Hindson, B. J., et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number. Anal Chem. 83 (22), 8604-8610 (2011).
- Pinheiro, L. B., et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification. Anal Chem. 84 (2), 1003-1011 (2012).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods Mol Biol. 1950, 51-83 (2019).
- . Planning droplet digital PCR experiments Available from: https://www.bio-rad.com/en-in/life-science/learning-center/introduction-to-digital-pcr/planning-ddpcr-experiments (2024)
- Dorange, F., Le Bec, C. Analytical approaches to characterize AAV vector production & purification: Advances and challenges. Cell Gene Ther Insights. 4 (2), 119-129 (2018).
- Lock, M., et al. Absolute determination of single-stranded and self-complementary adeno-associated viral vector genome titers by droplet digital PCR. Hum Gene Ther Methods. 25 (2), 115-125 (2014).
- Prantner, A., Maar, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18 (1), e0280242 (2023).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Front Microbiol. 10, 1570 (2019).
- Suoranta, T., Laham-Karam, N., Yla-Herttuala, S. Optimized protocol for accurate titration of adeno-associated virus vectors. Hum Gene Ther. 32 (19-20), 1270-1279 (2021).
- Droplet digital PCR applications guide. Bulletin 6407 Available from: https://www.bio-rad.com/webroot/web/pdf/lsr/literature/Bulletin_6407.pdf (2024)
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ABOUT JoVE
Copyright © 2025 MyJoVE Corporation. All rights reserved