Method Article
Hi-C: genomların Üç boyutlu Mimari Eğitim Bir Yöntem.
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Erratum Notice
Özet
Hi-C yöntemi kromatin etkileşimler (1) tarafsız, genom kimlik sağlar. Hi-C çiftler yakınlık ligasyonu ve güçlü paralel sıralama. Ortaya çıkan veriler farklı ölçeklerde genomik mimarlık eğitimi için kullanılabilir: ilk sonuçlar, kromozom toprakları, segregasyon, açık ve kapalı kromatin ve megabase ölçekte kromatin yapısı gibi özellikleri tespit.
Özet
Kromozom üç boyutlu katlama genom compartmentalizes ve yakın mekansal yakınlığı 2-6 içine rehberleri ve arttırıcılar gibi uzak fonksiyonel elemanları, getirebilir. Kromozom organizasyon ve genom aktivitesi arasında ilişki Deşifre transkripsiyon ve replikasyon gibi, genomik süreçleri anlamada yardımcı olacaktır. Ancak, kromozom kat hakkında çok az bilinir. Mikroskopi lokusların aynı anda yüksek çözünürlükte çok sayıda ayırt etmek mümkün. Bugüne kadar, kromozom konformasyon yakalama (3C) ve sonraki uyarlamalar kullanarak kromozom etkileşimleri tespit genom çalışmaları imkansız 7-10 yapma, hedef lokusların bir dizi seçim gereklidir.
Biz Hi-C, 3C tarafsız, genom moda uzun menzilli etkileşimleri belirleme yeteneğine sahip bir uzantısı. Hi-C, hücreleri etkileşim lokusların kovalent DNA-protein çapraz bağlantılar yoluyla birbirine bağlı olmayı neden, formaldehit ile sabittir. DNA daha sonra bir restriksiyon enzimi ile parçalanmış, bu lokuslar bağlı kalır. 5 'çıkıntılar yanında, künt uç ligasyonu, çapraz bağlı DNA parçaları arasında ligasyonu olayları lehine seyreltik koşullar altında yapılır doldurulur biotinlenmiş kalıntı kurulmuştur. Ligasyonu ürünlerin genom kütüphane Bu sonuç, çekirdeğinde birbirine yakın başlangıçta parçaları çiftleri karşılık. Her ligasyonu ürün kavşak yerinde biotin ile işaretlenmiş. Kütüphane makaslanmış ve kavşak streptavidin boncuk aşağı çekti. Saflaştırılmış kavşak sonradan etkileşim parçalarının bir katalog, yüksek verimli bir sequencer kullanarak analiz edilebilir.
Doğrudan temas matris analizi, kromozom topraklarının varlığı ve küçük zengin gen-kromozom tercihli dernek olarak, genomik organizasyonu sayısız özellikleri ortaya koymaktadır. Korelasyon analizinde, insan genomunun iki bölmeye ayrılmış olduğunu gösteren iletişim matris uygulanabilir: kapalı, ulaşılmaz ve etkin kromatin bölgeleri içeren, açık, ulaşılabilir ve aktif kromatin ve daha yoğun bir bölmesi içeren daha az yoğun paketlenmiş bölmesi. Son olarak, teorik türevleri ve hesaplama simülasyonları ile birleştiğinde topluluğu iletişim matris analizi, Hi-C megabase ölçekte bir fraktal küreciğin konformasyon ile tutarlı özellikler gösterdiği saptandı.
Protokol
Bu yöntem bildirilen araştırmada kullanılan Lieberman-Aiden ve ark. Science 326, 289-293 (2009) .
I. Crosslinking, Sindirim, DNA Ends işaretlenmesi ve künt uç ligasyon
- Hi-C, tüm 3C tabanlı yöntemler arasında ortak bir iş parçacığı hücreleri ile çapraz bağlanma ile başlar. Başlamak için, yapışkan ya da süspansiyon ve çapraz hücreleri ya da 2 x 10 7 ve 2.5 x 10 7 memeli hücreleri arasında büyümek. (Hücre ile çapraz bağlanma ilgili ayrıntılar için, lütfen bkz: 11
- Homojenizatör kullanarak (500 ul 10 mM Tris-HCl, pH 8.0, 10 mM NaCl,% 0.2 Igepal CA-630 ve 50 ul proteaz inhibitörleri) 550 ul lysisbuffer hücrelerin Lyse. Spin kromatin ve 5.000 rpm'de 500 ul 1x NEBuffer 2 ile iki kez pelet yıkayın.
- 5 numaralı tüp içine 1x NEBuffer 2, kısım kromatin yeniden süspanse edin ve son hacim 362 ul 1x NEBuffer 2 ekleyin. 38 ul% 1'lik SDS, 65 ° C'de 10 dakika boyunca iyice karıştırın ve inkübe edin. Inkübasyondan sonra hemen geri buz üzerinde tüpler yerleştirin.
- 44 ul Triton X-100 ve dikkatlice karıştırın ekleyerek SDS Quench. Dönerken ° C gecede 37 HindIII ve inkübe 400 Adet ekleyerek kromatin Digest.
- Sonraki adımlar Hi-C özgüdür ve DNA işaretleme, biotin ve çapraz parçaları küt uç ligasyonu performans ile biter içermektedir. Bu adım ligasyonu kavşak sonra arıtılmış olmasını sağlayacaktır. Tüp 1 biyotinilasyon adım geçmesi gerektiğini ve bunun yerine ayrı tutulmalı ve bu sindirim sağlamak için 3C bir kontrol olarak hizmet ve ligasyonu koşulları optimum.
- Kısıtlama parçası çıkıntılar doldurmak ve DNA kalan 4 tüpler biotin ile biter işareti, 1.5 ul 10 mM dATP, 1.5 ul 10 mM dGTP, 1.5 ul 10 mM dTTP, 37.5 ul 0.4 mM biotin-14-dCTP eklemek için tüpler 2-5 10 ul 5U/μl Klenow. ° C 37 ° dikkatlice karıştırın ve 45 dakika boyunca inkübe
- Tüpler buz üzerine yerleştirin. Enzimleri inaktive etmek için, 86 ul% 10 SDS tüpler 1-5 ekleyin. Tüpler 65 ° C tam 30 dakika inkübe edin ve hemen ardından buz koyun.
- Ligasyonu çapraz parçaları arasında ligasyonu olayları lehine için son derece seyreltik koşullar altında yapılır. Buz üzerinde çalışarak, 7.61 ml ligasyon karışımı [745 ul% 10 Triton X-100, 745 ul 10x ligasyonu tamponu (500 mM Tris-HCl, pH 7.5, 100 mM MgCl 2, 100 mM DTT), 80 ul 10 mg / ml BSA ekleyin 80 ul 100 mM ATP ve 5.96 ml su] her beş, 15 ml tüpler sayılı. Karşılık gelen 15 ml tüp sindirilir her kromatin karışımı aktarın.
- Düzenli 3C ligasyonu için, 1 tüp 10 ul 1U/μl T4 DNA ligaz ekleyin. Künt uç Hi-C ligasyonu için 50 ul 1U/μl T4 DNA ligaz, tüpler 2-5 ekleyin. Tersine tüpleri ile karıştırın ve 4 saat boyunca 5 tüp inkübasyon 16 ° C.
- Crosslinks ters ve protein tüp başına 50 ul 10 mg / ml proteinaz K ekleyerek ve tüpler gece 65 ° C'de inkübe düşer Ertesi gün, tüp başına ek bir 50 ul 10 mg / ml proteinaz K ekleyin ve 65 inkübasyon devam ° C 2 saat.
- Tepkime oda sıcaklığına kadar soğutulur ve beş 50 ml konik tüpler aktarabilirsiniz. DNA fenol çıkarma yaparak bu tüpler arındırın. 10 ml fenol pH 8.0 ve 2 dakika boyunca girdap ekleyin. 3500 devirde 10 dakika tüpler Spin ve dikkatli bir şekilde yeni bir 50 ml tüp kadar sulu faz mümkün olduğunca aktarmak.
- Fenol pH 8.0 kullanarak çıkarma tekrarla: kloroform (1:1) ve etanol kullanarak DNA hızlandırabilir. (DNA arıtma hakkında ayrıntılı bilgi için bakınız: 11
- Etanol santrifüj DNA çöktürülmüş sonra, her DNA pelet 450 ul 1x TE (10 mM Tris-HCl pH: 8.0, 1 mM EDTA) çözülür. DNA karışımı, 1.7 ml santrifüj tüpüne aktarın.
- Kloroform ekstraksiyon: 2 fenol yaparak arınma başka yuvarlak yapılır. Kloroform (1:1) ve 1 dakika için vorteks: 500 ul fenol pH 8.0 ekleyin. 14.000 rpm'de 5 dakika santrifüj ve tüpler sulu faz yeni bir tüpe transfer. Ikinci ekstraksiyon sonra, NaOAc 0.1x hacmi,% 100 etanol ve 30 dakika inkübe 2x hacmi -80 ° C ekleyerek DNA çökelek
- Çöktürülmüş DNA iplik sonra her DNA pelet,% 70 etanol ile yıkayın ve 25 ul 1x TE her DNA pelet tekrar süspansiyon haline getirin. 1 ul 1 mg / ml RNAse başına boru ekleme ve 37 tüpleri az 30 dakika süreyle inkübe mevcut olabilecek herhangi bir RNA ° C tonlama Havuz tüpler 2-5 Hi-C içeriği hala tüp 1 3C bir kontrol olarak ayrı tutmak.
- Şimdi Hi-C işareti ve ligasyonu verimliliği incelemek için iyi bir fırsat. Bu kontroller, başarılı olmak için bir Hi-C kütüphanesi olup olmadığını mükemmel göstergeleridir.
- Kütüphanelerinin nitelik ve nicelik kontrol etmek için, 2 ul ve 3C ve Hi-C kütüphaneleri 01:10 dilüsyonlarının% 0.8 agaroz jel üzerinde 6 ul alikotları çalıştırın. (Bkz. Şekil 2A)
- Hi-C işareti ve Hi-C ligasyonu verimliliği, PCR sindirmek yöntemi ile doğrulanır. Başarılı dolgu ve HindIII sitesi ligasyonu (AAGCTT) restriksiyon enzimi NheI (GCTAGC) için bir site oluşturur. PCR (3C 11 şablon olarak her kütüphane 0.2 ul kullanarak gibi iki yakındaki kısıtlama parçalardan oluşan özel ligasyonu ürün yükseltilir PCR ürünleri daha sonra HindIII, NheI veya her ikisi ile sindirilir.% 2 jel örnekleri çalıştırdıktan sonra 3C ve Hi-C ligasyonu olayların göreceli sayıda kesilmiş ve kesilmemiş bantların yoğunluğu (Şekil 2B) ölçülmesidir tahmin edilebilir.
- Bazı parçaları bağlandı olmaz, bunları daha sonra aşağı çekerek önlemek T4 DNA polimeraz eksonükleaz etkinliğini kullanarak bu unligated ucundan biotin kaldırmak için.
- Biotin-14-dCTP olmayan bağlanan DNA ucunda T4 DNA polimeraz eksonükleaz aktivitesi ile kaldırılır. 1 ul 10 mg / ml BSA, 10 ul 10x NEBuffer 2, 1 ul 10 mM dATP, 1 ul 10 mM dGTP ve 5 Adet toplam hacmi 100 ul T4 DNA polimeraz Hi-C kütüphanesi 5 mikrogram karıştırın ve inkübe az karışım 12 ° C'de 2 saat. Mümkünse, birden fazla 5 mg reaksiyonlar gerçekleştirilir.
- 2 ul 0,5 M EDTA pH 8.0 ekleyerek reaksiyon durdurulur.
- Kloroform (1:1) çıkarma etanol yağış takip yapılır: DNA, fenol pH 8.0 arındırmak için.
- Süpernatant atılır ve DNA pelet toplam hacmi 100 ul su yeniden bekletildi ve havuzlu.
II. Kesme ve Boyut Seçimi
- Biotinlenmiş DNA yüksek verimlilik sıralaması için uygun yapmak için, DNA Covaris S2 alet ile 300-500 basepairs (duty cycle 5, yoğunluk 5 devir / 200, 4 kür için 60 saniye zaman patlaması) bir boyut makaslanmış olmalıdır .
- Makaslanmış DNA biter onarmak için 14 ul 10x ligasyonu tampon, 14 ul 2.5 mM dNTP karışımı, 5 ul T4 DNA polimeraz, 5 ul T4 polinükleotid kinaz, 1 ul Klenow DNA polimeraz ve 1 ul su ekleyin. Oda sıcaklığında 30 dakika inkübe edin.
- Inkübasyon sonrasında, üreticinin tavsiyelerine göre DNA arındırmak için bir Qiagen MinElute sütun kullanabilirsiniz. 15 ul 1x Tris-Düşük-EDTA (TLE: 10 mM Tris, pH 8.0, 0.1 mM EDTA) ile iki kez DNA Zehir. Sonra, 10 ul 1 mM dATP, 2 ul su ve 3 ul Klenow (exo-) 5 ul 10x NEBuffer2 ekleyerek son tamir DNA 3 'ucuna bir dATP takın. Reaksiyon az 30 dakika boyunca inkübe 37 ° C.
- Klenow parçası inaktive için, 65 az 20 dakika reaksiyonlar inkübe ° C ve daha sonra buz üzerinde reaksiyonlar serin. Bir speedvac kullanarak, 20 ul reaksiyon hacmi azaltır.
- Sonra,% 1.5 agaroz jel 1X TAE DNA yük ve 80-90V 3,5 saat çalışacak. SYBR yeşil jel boyama sonra, bir DarkReader DNA görselleştirmek. Vergi DNA parçalarının, 300 ve 500 baz çifti ve arasında jel ağırlığına göre 2-4 sütunlarını kullanarak Qiagen jel ekstraksiyon kiti ile onları arındırmak. 50 ul 1x TLE DNA Zehir.
- Qiaquick sütunlarından Eluatlar birleştirin ve 1x TLE 300 ul son hacmi kadar getirmek. Son olarak, QuBit flüorometre kullanarak Quant-iT yöntemi ile DNA konsantrasyonu belirlemek ve toplam DNA miktarını hesaplamak.
III. Biotin Pull-down ve Eşleştirilmiş sonu Sıralama
- Protokolün Bu bölümde, ligasyonu kavşaklar, etkin bir şekilde tanımlanması için eşleştirilmiş sonu sıralama tarafından kromatin parçaları etkileşim izin DNA havuzu saflaştırılmış. DNA LoBind tüpler sonraki tüm adımları uygulayın.
- Açılan ul 150 400 ul Tween Buffer (5 mM Tris-HCl, pH 8.0, 0.5 mM EDTA, 1 M NaCl,% 0.05 Tween TB) ile iki kez yeniden süspanse manyetik streptavidin boncuk yıkama biotin boncuk hazırlayın.
Bu ve gelecek yıkar beş adımdan oluşmaktadır:- Boncuklar tampon ekle
- Karışımı yeni bir tüpe transfer
- Oda sıcaklığında 3 dakika boyunca örnek Döndür
- Bir manyetik parçacık konsantratör kullanarak boncuk düşürmemek
- Süpernatantı
- 300 ul 2x yok Tween Buffer (2x NTB: 10 mM Tris-HCl, pH 8.0, 1 mM EDTA, 2 M NaCl), boncuklar yeniden süspanse ve 300 ul Hi-C DNA ile birleşir. Hi-C DNA etiketli biotin, karışımın rotasyon ile 15 dakika oda sıcaklığında inkübe streptavidin boncuk bağlamak için izin verin.
- Manyetik parçacık Concentr streptavidin boncuk bağlı DNA düşürmemekator ve supernatant çıkarın. 400 100 ul 1x ligasyonu tampon takip ul 1x NTB (5 mM Tris-HCl, pH 8.0, 0.5 mM EDTA, 1 M NaCl), boncuk yıkayın. 50 ul 1x ligasyonu tampon boncuk süspanse edin ve karışımı yeni bir tüp transferi.
- Illumina Eşleştirilmiş sonu sıralama için DNA hazırlamak için adım 2.6 'da daha önce hesaplanan biotin açılan, girdi olarak kullanılan DNA toplam tutarı ve Hi-C DNA miktarını tahmin etmek için 20 bölmek aşağı çekti ve ligasyonu için kullanılabilir. Ligasyonu için Hi-C DNA mikrogram ortalama Illumina Eşleştirilmiş End adaptörleri 6 picomoles ekleyin. DNA adaptörleri Arter 1200 Üniteleri T4 DNA ligaz kullanın. Oda sıcaklığında 2 saat süreyle inkübe edin.
- Hi-C DNA bağlı boncuk geri ödemelerine ve 400 ul 1x TB ile iki kez boncuk yıkama olmayan bağlanan Eşleştirilmiş Sona adaptörleri çıkarın.
- 200 ul ve daha sonra 50 ul 1x NEBuffer 2 200 ul 1x NTB boncuk yıkayın. Son yıkamadan sonra, 50 ul 1x NEBuffer 2 boncuk tekrar süspansiyon ve yeni bir tüpe transfer.
- Sıralaması için yeterli PCR ürünü üretmek için gerekli döngü sayısını belirlemek için dört test PCR reaksiyonları, 6, 9, 12 veya 15 döngü kurdu. (PCR ayrıntılı bilgi için bakınız: 12,% 5 poliakrilamid jel ve SYBR Green ile boyanarak PCR reaksiyonları çalışan sahte bantları ve 400 arasında bir smear varlığında yokluğunda sağlanması optimal döngü sayısını belirler. adaptörleri ligasyonu sonra makaslanmış ürünlerin uzunluğu 600 baz çiftinden.
- PCR döngüleri optimal sayıda büyük ölçekli bir PCR Hi-C-kütüphanesi bağlı streptavidin boncuk geri kalanı yükseltin. Ayrı kuyulardan Havuzu PCR ürünleri ve boncuklar geri. Büyük ölçekli PCR ürünü% 1 bir jel üzerinde çalışan ve üretici tavsiyelerine göre 1.8x hacmi Ampure boncuk PCR ürününün kalan arındırmak için ayrı tutun.
- 50 ul 1x TLE tamponu ile DNA Zehir ve özgün PCR ürünü% 1 kısım% 5 poliakrilamid jel üzerinde Ampure boncuk saflaştırılmış PCR ürünü% 1 karşılaştırın, PCR primerleri başarılı bir şekilde kaldırılmasını sağlamak.
- Ayrıca klonlama 1 Hi-C kütüphanesi ul ve Sanger sıralama kullanarak yaklaşık 100 klonların ürün belirlenmesinde öneririz. Bu Hi-C PCR karışımı okur alignable göreli sayısı değerlendirmek için sağlayacaktır. Tipik bir sonuç için, Şekil 3B bakın.
- Illumina eşleştirilmiş sonunda sıralama ile Hi-C kütüphanesi sırası. Bağımsız etkileşim kromatin parçaları belirlemek için Maq (http://maq.sourceforge.net/) kullanarak her iki ucunda aynı hizaya getirin.
IV. Temsilcisi Hi-C Sonuçlar
- Hi-C protokolü kalite kontrol standartları, teknik olarak iyi yürütülür ve kabul edilebilir zaman aşağıdaki sonuçlar beklenmektedir.
- Kalite kontrol adımlar hem 3C ve Hi-C kütüphaneleri 10 kb daha büyük daha dar bantlar çalıştırdığınız açığa çıkarmalıdır. Bir DNA smear kötü ligasyonu verimlilik gösterir. Genellikle, ligasyon verimliliği Hi-C kütüphanesi 3C bir şablon (Bkz. Şekil 2A) kıyasla biraz daha düşük.
- Hi-C işareti ve ligasyonu verimliliği 3C primerler kullanılarak oluşturulan bir PCR ürününün sindirim tahmin edilebilir. 3C kavşak NheI HindIII tarafından kesilir ve. Hi-C kavşaklar Bunun tam tersi doğrudur. Bu PCR sindirmek tahlil verimli (Bkz. Şekil 2B) ligasyonu kavşak işaretlenmesi doğrulayan, Hi-C amplikonlarının% 70 NheI ve HindIII tarafından kesilmiş olduğunu göstermektedir.
- Sıralı okuma Analizi intrachromosomal ve interchromosomal etkileşimleri, mavi ve kırmızı çizgilerle belirtilen okur göstermelidir, rasgele oluşturulmuş bir HindIII kısıtlama siteleri karşılaştırıldığında önemli ölçüde yakın (Şekil 3A), yeşil gösterilen okur hizalamak.
- Başarılı bir deneyde,% 55 interchromosomal etkileşimleri alignable okuma çifti temsil ediyor. On beş ayrı parçaları 20 kb daha az ve% 30 arasında yüzde intrachromosomal etkileşimleri temsil eden 20'den fazla kb (Bkz. Şekil 3B) birbirinden intrachromosomal okunan çifti. Bu dağıtım, kalite kontrol biçimi olarak, yüksek verimli sıralama önce örneklenmiş olabilir; klonlama ve yaklaşık 100 klonların Sanger sıralama genellikle yeterlidir.
- Kromatin etkileşimleri, x-ve y-eksenleri genomik amacıyla lokusların temsil eder ve her bir piksel, aralarındaki gözlenen etkileşimlerin sayısını temsil eder İlgi haritası olarak görüntülenebilmekte. Tipik olarak, çok lineer genom birbirlerine yakın DNA parçalarının eğilimi sık sık birbirleri ile etkileşim olacaktır. Bu belirgin bir diyagonal olarak intrachromosomal heatmaps (Şekil 4A) görülür.
- Genom organizasyonu çeşitli düzeylerde ortaya çıkarmak için veri analiz aşağıdaki sonuçlar farklı şekillerde göstermektedir. Olasılık plotlamay karşı genomik mesafe (Bkz. Şekil 5A) temas olasılığını sonunda bir plato ulaşan genomik mesafenin bir fonksiyonu olarak azalır gösterir. Her mesafede, düz çizgi gösterilen intrachromosomal etkileşimleri,, kesik çizgi ile temsil interchromosomal etkileşimleri göreli zenginleştirilmiştir. Bu doğrudan kromozom topraklarının varlığı anlamına gelir.
- Tüm kromozom çiftleri arasındaki interchromosomal ilişkiler gözlenen / beklenen sayıda hesaplanması özellikle kromozom çiftleri arasındaki tercihli ilişki ortaya koymaktadır. Küçük zengin gen-kromozom tercihen parlak kırmızı renk (Bkz. Şekil 5B) tarafından belirtildiği üzere, birbirleri ile etkileşim.
- Bireysel kromozom da incelenebilir. Ham İlgi haritası gözlenen / beklenen İlgi haritası, genomik lokusların çiftleri arasındaki mesafe hesabı için beklenen bir İlgi haritası kullanılarak ayarlanabilir. Sonra, bir korelasyon matrisi gözlenen / beklenen heatmaps satırlar ve sütunlar ilişkilendirilerek üretilebilir. Korelasyon analizi ile gösterilmiştir iki bölmeye insan genomu ayıran. Bu korelasyon heatmaps ekose deseni (Bkz. Şekil 4A-D) tarafından gösterilmiştir. (Hi-C veri analizi ayrıntılı bilgi için bakınız: 1.
- Hi-C verilerini kullanarak, yeni anlayışlar megabase ölçekte kromatin katlama elde edildi. Polimer yoğunlaşma klasik model bir denge küreciğin içine kromatin paketleri göstermektedir. Uzaklığın bir fonksiyonu olarak plotlama temas olasılığı eğimi yaklaşık -1 'dir (Şekil 6A) genomik mesafe bir güç yasası olarak bu temas olasılığı ölçekleri göstermektedir. Bu bir denge küreciğin davranışı ile tutarlı değil, maç beklentileri fraktal küreciğin (Şekil 6B) olarak bilinen alternatif bir yapı yok.
- Burada, iki globüler yapıları gösterilmiştir. Renklendirme mavi mavi, yeşil, sarı, turuncu ve kırmızı (Şekil 6C top) arasında değişen, bir uç nokta mesafeye tekabül eder. Denge globülleri aksine, fraktal globülleri karışıklıklardan yoksundur. Fraktal küreciğin, kontur boyunca yakındaki lokusların (Şekil 6C orta) monokromatik blokların varlığı, 3D Yakındaki eğilimindedir. Bu bloklar denge küreciğin (Şekil 6C alt) bulunmaz.
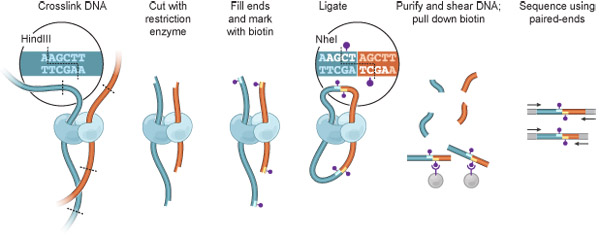
Şekil 1: Hi-C genel bakış. Hücreler arasındaki kovalent bağları mekansal bitişik kromatin kesimleri (Proteinler, bu tür etkileşimler arabuluculuk, açık mavi ve mavi gösterilen kırmızı koyu mavi, DNA parçaları), formaldehit ile çapraz bağlantılıdır. Kromatin bir kısıtlama enzim ile sindirilir (burada, HindIII kısıtlama sitesi: kesikli çizgi, içerlek bakınız). Çıkan yapışkan uçları biotinlenmiş biri nükleotid (mor nokta) ile doldurulur. Ligasyonu intramoleküler ligasyonu olayları lehine son derece seyreltik koşullar altında yapılır; HindIII sitesinde kaybolur ve bir NheI sitesi (inset) oluşturulur. Saflaştırılmış DNA ve makaslanmış ve biotinlenmiş kavşak streptavidin boncuk kullanılarak izole edilmiştir. Etkileşimde parçaları eşleştirilmiş sonu sıralama tarafından tespit edilir.

Şekil 2 Hi-C kütüphanesi kalite kontrolleri. (A) 3C kontrolü ve Hi-C kütüphanesi artan miktarda% 0.8 agaroz jel üzerinde çözüldü. Her ikisi kütüphane, daha dar bant 10 kb daha büyük bir olarak çalışan. Hi-C kütüphanesi Tipik ligasyonu verimliliği 3C şablon gözlenen olandan biraz daha düşük olup, Hi-C şerit smear ile gösterilir. (B) PCR sindirimi kontrol eder. Iki yakın parçalarından oluşan bir ligasyonu kavşak standart 3C PCR koşulları kullanılarak yükseltilir. Hi-C ligasyonu ürünleri ligasyonu sitenin sindirim konvansiyonel 3C üretilen ayırt edilebilir. Hi-C kavşak NheI, değil HindIII tarafından kesilir; ters 3C kavşaklar için de geçerlidir. Hi-C amplikonlarının% 70 ligasyonu kavşak işaretlenmesi verimli teyit NheI tarafından kesilmiştir. İki çoğaltır güvenilir kantifikasyon sağlamak için yapıldı.

Şekil 3 Hi-C kalite kontrolleri okundu. (A), hem de intrachromosomal (mavi) ve interchromosomal (kırmızı) etkileşimleri HindIII kısıtlama siteleri, rasgele oluşturulmuş bir okur (yeşil) ile karşılaştırıldığında önemli ölçüde yakın hizalamak karşılık gelen parçalardan okur. Intrachromosomal okur hem interchromosomal bir plato ~ 500 bp bir mesafe ulaşana kadar eğrileri hızla HindIII sitesi artışlardan mesafe olarak azaltmak okur. Bu sıralama için kullanılan maksimum parçası büyüklüğüne tekabül etmektedir. (B) Tipik olarak,% 55 alignable okuma çifti interchromosomal etkileşimleri temsil eder. Onbeş yüzde temsil parçaları en az 20 kb arasındaki intrachromosomal etkileşimlerapart ve% 30 fazla 20 kb dışında intrachromosomal okunan çifti. Bu dağıtım, kalite kontrol biçimi olarak, yüksek verimli sıralama önce örneklenmiş olabilir; klonlama ve yaklaşık 100 klonların Sanger sıralama genellikle yeterlidir.

Şekil 4 Korelasyon analizi çekirdeğin iki bölmeye ayrılmış olduğunu göstermektedir. (A), kromozom 14 üzerindeki intrachromosomal etkileşimler karşılık gelen İlgi Haritası. Her piksel 1 Mb lokus ve başka bir 1 Mb lokus arasındaki etkileşimi temsil eder; şiddeti okur toplam sayısı (aralık: 0-200 okuma) karşılık gelir. Onay işaretleri, her 10 Mb görünür. İlgi haritası, yoğun bir çapraz şeklinde ve büyük bloklar bir takımyıldızı alt yapı sergiler. (Kromozom 14 akrosentrik; kısa kol gösterilmez), belirli bir genomik mesafeden lokusların bir çift için ortalama bir kişi olasılığını hesaplamak için Hi-C veri kümesi kullanarak, bir beklenti matrisi (B) ne olacağını ilgili üretilen hiçbir uzun menzilli yapılar vardı gözlenen. Bu iki matrisler bölüm gözlenen / beklenen bir matris ise (C) tükenmesi, mavi ve kırmızı zenginleştirme [aralığı: 0.2 (mavi) 5 (kırmızı)] olarak gösterilir. Blok desen daha belirgin hale gelir. Her 14 kromozom boyunca lokusların çifti intrachromosomal etkileşim profilleri arasında korelasyon matrisi (D), korelasyon [-1 (mavi) 1 (kırmızı) aralığında] göstermektedir. Çarpıcı ekose deseni kromozomun içinde iki bölmeleri varlığını gösterir.

Şekil 5 kromozom topraklarının varlığı ve organizasyonu. (A) temas olasılığı sonunda ~ 90Mb (mavi) bir plato ulaşan, kromozom 1 genomik mesafenin bir fonksiyonu olarak azalır. Interchromosomal temas düzey (siyah çizgi) farklı kromozom çiftleri için farklıdır; 21 kromozom lokusların (kırmızı çizgi) ile etkileşime girmesi olası kromozom 10 lokusların (yeşil çizgi) ve en az 1 kromozom üzerinde lokusların ile etkileşim büyük olasılıkla. Interchromosomal etkileşimleri intrachromosomal etkileşimleri göreli olarak tükenmiş. (B) Gözlenen / beklenen kromozom çiftleri arasında interchromosomal kişilerin sayısı. Kırmızı zenginleştirme gösterir ve mavi tükenmesi gösterir [aralık: 2 (kırmızı) 0.5 (mavi)]. Küçük, zengin gen kromozom, birbirleri ile daha fazla etkileşim eğilimindedir.

Şekil 6 kromatin yerel ambalaj fraktal küreciğin davranışı ile tutarlı. (A) İletişim olasılık, genomik mesafenin bir fonksiyonu olarak (mavi) genom boyunca ortalama. Önde gelen bir güç hukuk ölçekleme 500kb ve -1,08 bir yamaç (camgöbeği olarak gösterilen uygun) 7MB (gölgeli bölge) arasında görülüyor. (B) denge (kırmızı) ve fraktal (mavi) globülleri mesafenin bir fonksiyonu olarak temas olasılığı için simülasyon sonuçları. Fraktal küreciğin eğim roman teorik öngörü 1 doğrulayan, çok yaklaşık -1 (mavi). Bir denge küreciğin eğim maçlar öncesinde teorik beklentiler, -3 / 2. Hi-C veri görülen bir denge küreciğin eğimi ise fraktal küreciğin eğim yakından Hi-C sonuçları gözlenen eğim andırıyor. (C) Top: gelişeceğini polimer zinciri, 4000 monomerlerin uzun. Renklendirilmesi, mavi, mavi, yeşil, sarı, turuncu, ve kırmızı arasında değişen, bir uç nokta mesafeye tekabül eder Orta: bizim topluluk alınan bir fraktal küreciğin tipik örneğidir. Fraktal globülleri karışıklıklardan yoksundur. Bir denge küreciğin: kontur boyunca yakındaki Loci yüzey ve kesit Bottom belirgin büyük monokromatik blokların varlığı, 3D Yakındaki olma eğilimindedirler. Yapısı oldukça dolaşmış; 3D kontur (benzer renk) boyunca yakındadır lokusların Yakındaki gerekmez.
Tartışmalar
Biz kromatin etkileşimleri, tarafsız bir genom şekilde haritalama, genomun 3 boyutlu mimari okuyan bir yöntem mevcut. Önceki çalışma dışında bu teknoloji ayarlar en kritik deneysel adım künt uç ligasyonu önce çapraz parçalarının biter kısıtlama biotinlenmiş nükleotidlerin eklenmesi. Bu adımı başarıyla derin tüm ligasyonu kavşak sıralama sağlar ve Hi-C, kapsamı ve güç verir.
Okunma sayısı sonuçta etkileşim haritalar kararı belirleyecektir. Burada, insan genomu için 1 Mb etkileşim harita ~ 30 milyon alignable okuma kullanarak sunulmaktadır. N bir faktör 'tüm amacı' çözümü artırmak amacıyla, okur sayısı n 2 bir faktör artış olmalıdır.
Hi-C tekniği kolayca kütüphane nesil sonra hibrid yakalama (genomun belirli bölümlerine hedef) ve bağlanmasından sonra kromatin immunoprecipitation (spesifik protein ile ilişkili bölgelerde kromatin çevreyi araştırmak) gibi diğer teknikleri ile kombine edilebilir.
Açıklamalar
Teşekkürler
AP Aiden, XR Bao, M. Brenner, D. Galas, W. Gosper, A. Jaffer, A. Melnikov, A. Miele, G. Giannoukos, C. Nusbaum, AJM Walhout; A. Kosmrlj tartışmalar ve kod için teşekkür ederim , L. Wood, K. Zeldovich tartışmaları, görselleştirme ile yardım ve L. Gaffney ve B. Wong.
Fannie ve John Hertz Vakfı lisansüstü burs, Milli Savunma Bilimi ve Mühendisliği yüksek lisans burs, NSF lisansüstü burs, Ulusal Uzay Biyomedikal Araştırma Enstitüsü, hiçbir hibe tarafından desteklenir. Ulusal İnsan Genomu Araştırma Enstitüsü (NHGRI) (EL) T32 HG002295; i2b2 (Biyoloji ve Hastabaşı entegre Bilişim), Brigham ve Kadın Hastanesi (LAM) Biyomedikal Computing NIH destekli Merkezi; hibe yok. NHGRI HG003143 ve Keck Vakfı ayırt genç bilim adamı ödülü (JD). Ham ve eşlenmiş Hi-C dizisi veri GEO veritabanı (tevdi edilmiştir www.ncbi.nlm.nih.gov/geo/ üyelik yok). GSE18199. Ek görselleştirme. http://hic.umassmed.edu .
Malzemeler
| Name | Company | Catalog Number | Comments |
| Protease inhibitors | Sigma-Aldrich | P8340-5ml | Step 1.2 |
| biotin-14-dCTP | Invitrogen | 19518-018 | Step 1.6 |
| Klenow | New England Biolabs | M0210 | Steps 1.6 and 2.2 |
| T4 DNA ligase | Invitrogen | 15224 | Step 1.9 |
| T4 DNA polymerase | New England Biolabs | M0203 | Steps 1.17 and 2.2 |
| 10x ligation buffer | New England Biolabs | B0202 | Steps 2.2 and 3.4 |
| T4 PNK | New England Biolabs | M0201 | Step 2.2 |
| Klenow (exo-) | New England Biolabs | M0212 | Step 2.3 |
| Dynabeads MyOne Streptavin C1 Beads | Invitrogen | 650.01 | Step 3.2 |
| T4 DNA ligase HC | Enzymatics | L603-HC-L | Step 3.5 |
| Phusion HF mastermix | New England Biolabs | F531 | Step 3.8 |
| Ampure beads | Beckman Coulter Inc. | A2915 | Step 3.9 |
Referanslar
- Lieberman-Aiden, E., Van Berkum, N. L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I., Lajoie, B. R., Sabo, P. J., Dorschner, M. O., Sandstrom, R., Bernstein, B., Bender, M. A., Groudine, M., Gnirke, A., Stamatoyannopoulos, J., Mirny, L. A., Lander, E. S., Dekker, J. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Kosak, S. T., Groudine, M. Form follows function: the genomic organization of cellular differentiation. Genes and Dev. 18, 1371-1384 (2004).
- Misteli, T. Beyond the sequence: cellular organization of genome function. Cell. 128, 787-800 (2007).
- Dekker, J. Gene Regulation in the Third Dimension. Science. 319, 1793-1794 (2008).
- Cremer, T., Cremer, C. Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat Rev Genet. 2, 292-301 (2001).
- Sexton, T., Schober, H., Fraser, P., Gasser, S. M. Gene regulation through nuclear organization. Nat Struct and Mol Biol. 14, 1049-1055 (2007).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing Chromosome Conformation. Science. 295, 1306-1311 (2002).
- Zhao, Z., Tavoosidana, G., Sjölinder, M., Göndör, A., Mariano, P., Wang, S., Kanduri, C., Lezcano, M., Sandhu, K. S., Singh, U., Pant, V., Tiwari, V., Kurukuti, S., Ohlsson, R. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1341-1347 (2006).
- Simonis, M., Klous, P., Splinter, E., Moshkin, Y., Willemsen, R., de Wit, E., van Steensel, B., de Laat, W. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nat Genet. 38, 1348-1354 (2006).
- Dostie, J., Richmond, T. A., Arnaout, R. A., Selzer, R. R., Lee, W. L., Honan, T. A., Rubio, E. D., Krumm, A., Lamb, J., Nusbaum, C., Green, R. D., Dekker, J. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Res. 16, 1299-1309 (2006).
- Miele, A., Dekker, J. Mapping Cis- and Trans Chromatin Interaction Networks Using Chromosome Conformation Capture (3C). Methods Mol Biol. 464, 105-121 (2009).
- Maccallum, I., Przybylski, D., Gnerre, S., Burton, J., Shlyakhter, I., Gnirke, A., Malek, J., McKernan, K., Ranade, S., Shea, T. P., Williams, L., Young, S., Nusbaum, C., Jaffe, D. B. ALLPATHS 2: small genomes assembled accurately and with high continuity from short paired reads. Genome Biol . 10, R103-R103 (2009).
Erratum
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır