Method Article
Belirli Genomik Bölgeler ve enChIP tarafından İlişkili Moleküllerin Tanımlanması İzolasyonu
Bu Makalede
Özet
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. This protocol describes procedures to perform engineered DNA-binding molecule-mediated chromatin imunoprecipitation (enChIP) for identification of proteins and RNAs associated with a specific genomic region.
Özet
The identification of molecules associated with specific genomic regions of interest is required to understand the mechanisms of regulation of the functions of these regions. To enable the non-biased identification of molecules interacting with a specific genomic region of interest, we recently developed the engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) technique. Here, we describe how to use enChIP to isolate specific genomic regions and identify the associated proteins and RNAs. First, a genomic region of interest is tagged with a transcription activator-like (TAL) protein or a clustered regularly interspaced short palindromic repeats (CRISPR) complex consisting of a catalytically inactive form of Cas9 and a guide RNA. Subsequently, the chromatin is crosslinked and fragmented by sonication. The tagged locus is then immunoprecipitated and the crosslinking is reversed. Finally, the proteins or RNAs that are associated with the isolated chromatin are subjected to mass spectrometric or RNA sequencing analyses, respectively. This approach allows the successful identification of proteins and RNAs associated with a genomic region of interest.
Giriş
Özel ilgi genomik bölgeleri ile ilişkili moleküllerin belirlenmesi gibi transkripsiyon ve epigenetik düzenleme gibi genomik fonksiyonların düzenlenmesinde mekanizmalarını anlamak için gereklidir. Çeşitli teknikler özel genomik bölgelere 1-7 biyokimyasal analizi için geliştirilmiş olmasına rağmen, bunlar, çünkü bu tür sınırlı bir uygulama (örneğin, sadece tekrarlar yüksek kopya sayısı lokusları veya lokus için) ve bunların iç sorunlar bu aşamada yaygın olarak kullanılmamaktadır çok fazla zaman ve çaba gerektiriyordu.
Kolayca belirli genomik bölgelerin biyokimyasal analizini gerçekleştirmek amacıyla, biz 14-17 iki lokus spesifik kromatin immunoprecipitation (ChIP) teknolojileri, yani sokma ChIP (iChip) 8-13 ve mühendislik DNA bağlayıcı molekül aracılı ChIP (enChIP) geliştirdik . IChip olarak, ilgi konusu bir lokus gibi Lex gibi dışsal bir DNA-bağlama proteini sokulması tanıma sekansları tarafından etiketlenmişA. lokusu sonra etiketli bir DNA-bağlama proteini kullanılarak afinite saflaştırma ile izole edilmektedir. EnChIP olarak, örneğin çinko parmak proteinleri, transkripsiyon aktivatörü gibi (TAL) protein gibi DNA-bağlama molekülleri, yapay ve kümelenmiş düzenli aralıklarla yerleştirilmişlerdir kısa palindromik tekrarları (CRISPR) kompleksleri, (Şekil 1) ilgi konusu bir lokusu etiketlemek için kullanılır. Daha sonra, genomik bölge ile işaretlenmiş DNA-bağlama moleküllerinin afinite saflaştırma ile izole edilmektedir.
IChip fazla enChIP avantajlarından biri bir ekzojen DNA-bağlama proteini tanıma sekansları sokulması gerekli değildir olmasıdır. Cas9 (dCas9) içindeki bir katalitik aktif olmayan biçimini kapsayan CRISPR kompleksleri ve bir kılavuz RNA (gRNA) ile lokusların Hedefleme TAL ve çinko-parmak proteinleri kullanılarak iChip veya enChIP bu bölgelerin hedeflemeyi çok daha kolaydır. Burada, kütle spektrometrisi ve RNA dizi analizi ile kombine enChIP için adım adım protokol açıklar (RNA-Seq) lokusa Associa tespit etmekted proteinleri ve RNA'lar sırasıyla.
Protokol
Hedef lokusun tanıma Engineered DNA-bağlama Moleküllerin 1. Tasarım
- CRISPR kompleksleri kullanılarak enChIP için, daha önce, 18 olarak tanımlanan ilgili genomik bölgenin aday gRNA hedef sekanslarını tespit etmek CRISPRdirect web aracı (http://crispr.dbcls.jp) kullanın. Bu web aracı formunun 23 bp genomik siteleri döndürür 5'-N 20 NGG-3 'hedef bölge içinde.
- U6 promotör sekansı ve 5'-N 20 sekansına ticari hizmetlerin kullanılarak 5'-N 20 NGG-3 'içeren bir gBlock sentezlenmesi 19,20 (bakınız Şekil 2). Amaçlara alt klonlama için gBlock dışındaki uygun kısıtlayıcı enzim sitelerinin içerir.
- Daha önce tarif edildiği gibi 16, uygun bir vektör içine gBlock yerleştirin. GBlocks için çeşitli retroviral vektörler (Malzeme) mevcuttur.
- TAL proteinler kullanılarak enChIP için Targ tanıyan TAL proteinleri tasarımet loci. Ticari hizmetlerini kullanarak TAL proteinleri kodlayan plazmid oluşturun. Daha önce tarif edildiği gibi 15 3 x FLAG bir veya daha fazla etiketleri ile kaynaşık TAL proteinleri ihtiva eden ifade vektörlerini oluşturmak.
EnChIP Analizi Hücre 2. kurulması
- 3 x FLAG-dCas9 ve gRNA (CRISPR bazlı prosedür) ya da Express 3, daha önce tarif edildiği gibi 14-17 x FLAG-TAL hücrelerinde (TAL protein bazlı prosedür) analiz edilecek.
- Transfeksiyon verimi yüksek ise geçici transfeksiyon kullanın. 3 x FLAG-dCas9 CMV promotörü ihtiva eden bir ekspresyon vektörü (Malzeme) mevcuttur.
- Geçici transfeksiyon verimliliği düşükse geleneksel yöntemler kullanılarak stabil transformantların kurulması düşünün. GBlock için yukarıda belirtilen retroviral vektörler ek olarak, 3 x FLAG-dCas9 retroviral vektörler (Malzeme) mevcuttur.
- , Daha önce tarif edildiği 14-17 gibi bir anti-FLAG antikoru (Ab) kullanılarak bağışıklık beneklenme analizi ile transfekte edilmiş hücrelerde 3xFLAG-dCas9 ya 3xFLAG-TAL proteininin ifadesini teyit edin.
Not: Bu etiketli proteinlerinin ekspresyonu, aynı zamanda, anti-FLAG-FITC ve daha sonraki bir FACS analizi ile hücre içi boyama ile teyit edilebilir. Hücre içi boyama için bir protokol bizim ana (http://www.biken.osaka-u.ac.jp/lab/microimm/fujii/iChIP_protocols/english.html) indirilebilir. - Standart RT-PCR tekniği ile gRNA ifadesini onaylayın.
- Ilgili genomik bölge ile etkileşime giren proteinlerin kantitatif belirlenmesi için, hücre kültürü (SILAC) enChIP (enChIP-SILAC) ile birleştirildi analizinde amino asitlerle stabil bir izotop işaretleme kullanımını dikkate.
Not: SILAC analizi için Media ticari satıcılardan temin edilebilir. SILAC belirli etkileşimleri tespit güçlüdür.- SILAC ağır ortam kültür hücreleri. Hücreler Cultu hazırlayınBir negatif kontrol olarak SILAC ışığı ortamı olarak verilmiştir. (Ağır veya hafif) her SILAC ortamı için en az 5 x 10 7 hücreleri kullanın. Etkili bir etiketleme için, her iki L-Lizin-2HCI, 13 ° C 6 ve L-Arginin-HCl, SILAC Ağır ortam içinde 13 ° C 6, 15 N 4 ekleyin. Not: en az beş hücre bölünmesi etiket protein için gereklidir.
- Örneğin, kararlı bir şekilde Lizin-2HCI artı L-Arginin-HCl (Işık ortamı) SILAC için DMEM ortam ve diyalize edilmiş fetal büyükbaş hayvan serumu, 37 ° C'de ve% 5 CO2 seviyesinde 3xFLAG-dCas9 ve gRNA ifade kültürün insan fibrosarkoma HT1080 hücreleri ya da 13C 6 L-Lizin-2HCI artı 13 Cı 6 15 N 4 L-Arginin-HCl (Ağır ortam) 16 (Malzeme). Ortamın 500 ml hafif ya da ağır bir L-Lizin-2HCI ve L-Arginin-HCl, 50 mg ekleyin.
- Hücreler üslü growt muhafaza böylece tripsinize ve replate hücreler konfluent hale gelmeden önceh.
- SILAC ağır ortam kültür hücreleri. Hücreler Cultu hazırlayınBir negatif kontrol olarak SILAC ışığı ortamı olarak verilmiştir. (Ağır veya hafif) her SILAC ortamı için en az 5 x 10 7 hücreleri kullanın. Etkili bir etiketleme için, her iki L-Lizin-2HCI, 13 ° C 6 ve L-Arginin-HCl, SILAC Ağır ortam içinde 13 ° C 6, 15 N 4 ekleyin. Not: en az beş hücre bölünmesi etiket protein için gereklidir.
Formaldehit ile Hücreler 3. çapraz bağlanması
- Süspansiyon kültüründe hücreler, transfer 2 x 10 7 3xFLAG-TAL proteinler veya 3xFLAG-dCas9 artı gRNA eksprese eden hücreler ve 50 ml'lik bir santrifüj tüpü içinde düzenli bir kültür ortamında 30 ml asılır. Daha fazla hücre kullanıldığında, orantılı reaktif hacimleri ve miktarlarını artırmak. SILAC deneyleri için, ağır ortam veya hafif ortamında (5 x 10 7 hücreleri her) kültüre hücrelerin karışımı ve 2 x 10 7 hücre ihtiva eden 5 tüp içine 1 x 10 8 hücre bölün.
- Hücre süspansiyonu (nihai konsantrasyon% 1) 30 ml% 37 formaldehit 810 ul ilave edin ve 5 dakika boyunca 37 ° C'de inkübe edin. Yapışık hücreler için, doğrudan kültür ortamında 30 ml% 37 formaldehit 810 ul ekleyerek kültür kapları içinde hücrelerin tespit.
- 1,25 M glisin çözeltisi (nihai konsantrasyon 127 mM) 3.1 ml ekleyerek çapraz bağlanmasını durdurmak ve10 dakika boyunca oda sıcaklığında inkübe edilir.
- Santrifüj (4 ° C 'de 5 dakika süre ile 300 x g) hücreleri toplamak. Dikkatle süpernatant dahil formaldehit atmak ve uygun bir atık şişe içinde saklayın.
- Yapışık hücreler, bir 50 ml tüp içinde, bir hücre kazıyıcı ile hasat hücreleri ayırmak ve bu aşamada tarif edildiği gibi hücreleri toplamak.
- SILAC deneyleri için, ağır, orta veya hafif orta (5 x 10 7 hücre her) kültüre müstakil hücrelerin karışımı 2 x 10 7 hücre içeren 5 tüpler içine 1 x 10 8 hücrelerinin toplam bölmek ve bu tarif edilen hücreleri toplamak adım.
- Tüp başına iki kez PBS ile 30 ml hücre pelletini yıkayın. Dikkatle formaldehit içeren süpernatant atın ve kimyasal güvenlik yönergelerine göre formaldehit atık bottle.Handle uygun bir atık saklayın. Sabitlenen hücreler dondurulmuş ve -80 ° C'de muhafaza edilebilir.
Kromatin 4. Hazırlama (başına 2 x 107 hücreleri)
- Hücre lisis tamponu, 10 ml (10 mM Tris (pH 8.0), 1 mM EDTA,% 0.5 IGEPAL CA-630 ve 1 x proteaz inhibitörleri) sabit süspanse edilir ve 10 dakika süreyle buz üzerinde kuluçkalayın.
- Santrifüj örneği (8 dakika boyunca 4 ° C'de 830 x g) ve dikkatli bir supernatant atın.
- Nükleer lizis tamponu 10 ml pelet Askıya (10 mM Tris (pH 8.0), 1 mM EDTA, 0.5 M NaCl,% 1 Triton X-100,% 0.5 sodyum deoksikolat,% 0.5 loroilsarkosin ve 1x proteaz inhibitörleri). 10 dakika süreyle buz üzerinde inkübe ve her 2-3 dakikada bir vorteks.
- (830 × numuneyi santrifüj 8 dakika boyunca 4 ° C'de g) ve dikkatli bir şekilde supernatant atın.
- 10 ml PBS pelet yıkayın. Pelet (kromatin fraksiyonu) sıvı azot içinde hızlı soğutma, -80 ° C'de saklanabilir.
Kromatin 5. Sonication (2 x 10 7 hücre başına)
- 800 ul kromatin fraksiyonu Askıyadeğiştirilmiş lisis tamponu 3 (10 mM Tris (pH 8.0), 1 mM EDTA, 150 mM NaCI,% 0.1 sodyum deoksikolat,% 0.1 SDS ve 1x proteaz inhibitörleri) ve 1,5 ml tüp içine aktarılır.
- Çıktı: (Malzeme) bir sonikatör kullanılarak kromatine ve aşağıdaki koşulları ses dalgalarına maruz 3; görev:% 100 (sürekli); ve saati: ücretsiz. 10 saniye boyunca, sonikasyon 10-18 döngü gerçekleştirmek ve 20 saniye süreyle buz üzerinde soğutuldu. Aşırı ısınmayı önlemek 2 dakika her 5-6 devir için buz üzerinde örnekleri inkübe. Köpük önlemek için, 0.5 cm tüpün tabanından sonikasyon probunun ucu konumunu tutun.
- Santrifüj örneği (10 dakika boyunca 4 ° C'de 16.000 x g) ve 1,5 ml tüp içine süpernatant aktarın. Sonike kromatin sıvı nitrojen içinde hızlı dondurma işleminin -80 ° C'de saklanabilir.
Kromatin Parçalanma 6. Değerlendirilmesi
- Distile su 85 ul parçalanmış kromatin 10 ul karıştırın.
- 4 ul ekleve 5 M NaCI, 65 ° CO / N inkübe edin.
- 10 mg / ml RNase A 1 ul ilave edin ve 45 dakika boyunca 37 ° C'de inkübe edin.
- 0.5 M EDTA (pH 8.0), 1 M Tris (pH 6.8) 4 ul, 20 mg 1 ul / ml proteinaz K 2 ul ekleyin ve daha sonra 1,5 saat süre ile 45 ° C'de inkübe edin.
- Örneğin SYBR Green I boyama boyalar içermeyen bir% 1 agaroz jeli içinde elektroforez ile numunenin ayrı ayrı 10 ul.
- Parçalanmış kromatin uzunlukları dağılımını değerlendirmek için jel leke. 0.5-2 kbp (0,2-4 kbp'lik aralığı) ortalama uzunluğu üreten şartlar tavsiye edilir.
- Fenol-kloroform ekstraksiyonu ile veya bir DNA çıkarma kiti (Malzeme) kullanarak, kalan örneklerinin DNA saflaştınlır. Saflaştırılmış DNA enChIP analiz verimlerini (8.11) tahmin etmek için, giriş DNA olarak kullanılabilir.
Antikorlar ile Dynabeads Konjuge 7. Hazırlama (2 x 10 7 hücre başına)
- Önİki 1,5 ml tüpler, normal fare IgG kullanılarak ön-temizleme için ve bir anti-FLAG Ab ile inkübasyon diğer pare. Her bir tüp, Protein G-konjuge manyetik boncuk 150 ul (Malzemeler bakınız) ekleyin.
- Bir mıknatıs standına tüpler koyun ve 3 dakika bekleyin. Pipetleme süpernatant atın.
- PBS% 0.01 Tween-20 (PBS-T) içeren 1 ml boncuk süspanse edin. Manyetik bir stand üzerinde tüpler koyun ve 2 dakika bekleyin. Pipetleme süpernatant atın ve sonra adımı tekrarlayın.
- % 0.1 BSA içeren PBS-T 1 ml boncuk süspanse edin.
- Normal fare IgG veya anti-FLAG Ab 15 ug ekleyin ve 4 ° CO / N dönmektedir.
- Daha sonra, kısa bir süre aşağı Spin bir mıknatıs standına tüpler koyun ve 3 dakika bekleyin. Pipetleme süpernatant atın.
- PBS-T 1 ml boncuk süspanse edin. Örnek birkaç kez ters çevirin ve kısaca aşağı doğru döndürün. Daha sonra pipetleme süpernatant atın, bir mıknatıs standında tüpler koyun ve 3 dakika bekleyin. Yıkama tekrarlayınPBS-T (üç yıkama adımları toplamı) ile iki kez daha.
8. Kromatin immünopresipitasyonu (2 x 10 7 hücre başına)
- Hacminin beşte biri% 5 Triton X-100 (% 1 Triton X-100 nihai konsantrasyon) ihtiva eden tadil edilmiş liziz tamponu 3 (yaklaşık 200 ul) ile 5.3 hazırlanan parçalanmış kromatin) (yaklaşık 800 ul) karıştırın.
- Normal fare IgG'si ile konjüge edilmiş Protein G-konjuge manyetik boncukları hazırlandı edildiği tüp kromatin solüsyonu ekleyin. 1 saat süre ile 4 ° C 'de döndürün.
- Bir mıknatıs stand üzerinde tüp yerleştirin ve 3 dakika bekleyin.
- Anti-FLAG Ab konjüge edilmiş Protein G-konjüge edilmiş manyetik boncukları hazırlandı edildiği tüp içine süpernatant aktarın. 4 ° CO / N at döndürün.
- Bir mıknatıs stand üzerinde tüp yerleştirin ve 3 dakika bekleyin. Pipetleme süpernatant atın.
- Düşük tuzlu tampon (20 mM Tris (pH 8.0), 2 mM EDTA, 150 mM NaCI, 1% Tri 1 ml boncuk Askıyatonluk X-100,% 0.1 SDS ve 1x proteaz inhibitörleri) ve 5 dakika boyunca 4 ° C'de döner. Bir mıknatıs stand üzerinde tüp yerleştirin ve 3 dakika bekleyin. Pipetleme Süpernatantı atın ve yıkama adımı tekrarlayın.
- Iki kez yüksek tuzlu tampon (20 mM Tris (pH 8.0), 2 mM EDTA, 500 mM NaCI,% 1 Triton X-100,% 0.1 SDS ve 1x proteaz inhibitörleri) ile boncuk yıkayın.
- Iki kez LiCI tampon maddesi (10 mM Tris (pH 8.0), 1 mM EDTA, 250 mM LiCI,% 0.5 IGEPAL CA-630,% 0.5 sodyum deoksikolat ve 1 x proteaz inhibitörleri) ile boncuk yıkayın.
- Bir kez TBS-IGEPAL CA-630 (50 mM Tris (pH 7.5), 150 mM NaCI,% 0.1 IGEPAL CA-630 ve 1 x proteaz inhibitörleri) ile boncuk yıkayın.
- Elüsyon tamponu 200 ul boncuk Askıya (50 mM Tris (pH 7.5), 150 mM NaCl,% 0.1 IGEPAL CA-630, 1 x proteaz inhibitörleri ve 500 ug / ml 3 x FLAG peptidi), ve 37 ° C'de inkübe edin 20 dakika. Bir mıknatıs stand üzerinde tüp yerleştirin ve 3 dakika bekleyin. Yeni 1.5 ml tüp içine süpernatant aktarın ve ELUT tekrariyon adım.
- Fenol-kloroform ekstraksiyonu ile veya bir DNA ekstraksiyon kiti kullanılarak sıyırma sıvısının DNA küçük bir kısmı (örn, 5%) saflaştınlır (Malzeme). Saflaştırılmış DNA spesifik primer enChIP daha önce tarif edildiği gibi 14,16 Aşama 6.7'de hazırlanan giriş DNA ile karşılaştırarak analiz verimlerini tahmin etmek için ayarlar, PCR için kullanılabilir.
9. SDS-PAGE, Boyama ve Kütle Spektrometrik Analizi
- 1 2-propanol ml 3M sodyum asetat (pH 5.2), 50 ul ve 20 mg / ml glikojen 5 ul eluat (400 ul) karıştırın. -20 ° CO / N de kromatin çöktürün.
- Santrifüj örneği (30 dakika boyunca 4 ° C'de 16.000 x g) ve süpernatan atılır. (10 dakika boyunca 4 ° C'de 16.000 x g),% 70 etanol ve santrifüj yeniden 1 ml pelet durulayın. Pipetle tamamen süpernatant atın.
- 2x numune tamponu (125 mM Tris (pH 6.8),% 10 2-merc 40 ul pelet Askıdaaptoethanol,% 4 SDS,% 10 sakroz ve% 0.004 bromofenol mavisi). 5 dakika vorteksleyin, tamamen pelet çözülür ve daha sonra proteinleri denatüre ve çapraz bağlantıyı ters döndürecek 30 dakika boyunca 100 ° C 'de inkübe etmek.
- Boya çok altında 1 cm ulaşana kadar SDS-PAGE için jel üzerinde numunenin 40 ul çalıştırın. Not: Genellikle% 4-20 gradyan jelleri, ancak diğer% jeller kullanılabilmektedir.
- Coomassie Brilliant Blue veya gümüş boyası ile jel Leke.
- Beş adet (2 mm yükseklik) içine jel kesti.
- Daha önce tarif edildiği gibi jel 13-16 sindirim ve kütle spektrometresi ile analiz gerçekleştirir.
- 5 x 10 7 hücreleri, her (1 x 10 8 hücrelerin toplam) ile SILAC deneyleri için, 2x numune tampon maddesi 40 ul, ilk beş borunun (o kadar büyütmek için gerekli değildir) pelet askıya.
RNA ve RNA-Seq Analizi 10. saflaştırılması
- EnChIP sonra RNA'yı saflaştırmak için RNaz inhibitörü 5 U / ml ilave (seTampon çözelti tüm e malzemeler), değiştirilmiş lisis tamponu 3 ve elüsyon tamponu dışında olan RNaz İnhibitörü 40 U / ml ekleyin.
- 5 M NaCI, 16 ul eluat (400 ul) karıştırın ve 2 saat süre ile 65 ° C'de inkübe edin.
- Örnek asit guanidinyum-fenol bazlı bir tepkime maddesi 1 ml (Malzemeler bakınız) ekleyin. Daha sonra 15 saniye vorteksleyin ve 5-15 dakika boyunca oda sıcaklığında inkübe edilir. 12.000 x g ve oda sıcaklığında 15 dakika boyunca santrifüjleyin.
- Yeni 1,5 ml tüp süpernatantı aktarın ve p-bromoanisol 5 ul ilave edin. Daha sonra 15 saniye vorteksleyin ve 3-5 dakika için oda sıcaklığında inkübe edilir. 12.000 x g ve oda sıcaklığında 10 dakika boyunca santrifüjleyin.
- Yeni 2 ml'lik bir tüp içine süpernatant (yaklaşık 1 mi) aktarın ve 2-propanol 1 ml. Tüp ters çevirin ve 10 dakika boyunca oda sıcaklığında inkübe edilir. (Malzeme) bir RNA saflaştırma kiti bir kolona karışımı yerleştirin. 12.000 × 1 dakika boyunca santrifüj sütun g RT.
- Yıkamabir RNA saflaştırma kiti RNA Yıkama Tamponu 400 ul (Malzeme) ile kolon.
- DNase I (1 U / ul), 10 x DNase I Reaksiyon Tamponu ve 8 ul, DNase / RNase içermeyen su 3 ul, ve RNA Yıkama Tamponu 64 ul içinde 5 ul DNaz I kokteyl 80 ul ekle (karışım sütun bir RNA arıtma kiti (Malzeme)). 15 dakika boyunca 37 ° C'de inkübe edin ve daha sonra numune 12.000 x g ve oda sıcaklığında 30 saniye boyunca santrifüj.
- Iki kez bir RNA saflaştırma kiti RNA Ön Yıkama Tamponu 400 ul (Malzeme) ile sütun yıkayın.
- DNase / RNase içermeyen su, 50 ul RNA Zehir. Elüt RNA, RNA sıralaması için kullanılabilir.
Sonuçlar
Genel olarak, bir hedef genomik bölgelerinden% 1-30. EnChIP kullanılarak arıtılmış, 3 enChIP verimlerini gösteren temsili verileri içermektedir Şekil edilebilir telomerlerin ve interferon (IFN), düzenleyici faktörü-1 (IRF-1) gen promotörü hedef analizleri. Tipik sonuçlar, Tablo 1, örnek olarak enChIP-SILAC, Tablo tarafından tanımlanan bir IFNy özgü bir şekilde IRF-1 promotör ile ilişkili proteinler 2 listeler ile kütle spektroskopisi enChIP tarafından tanımlanan telomer bağlayıcı proteinler (enChIP-MS) ve Tablo 3 enChIP-RNA-Sek tarafından belirlenen telomerlerin ilişkili RNA'lar.

EnChIP 1. Genel Bakış Şekil. enChIP kullanılarak CRISPR (A, B) ve TAL (C, D, E ) gösterilir. Ilgi odağı bir böyle bir TAL protein veya Cas9 (DCAS) bir katalitik inaktif formda oluşan CRISPR sistemi olarak tasarlanmış DNA bağlama molekülleri ile etiketlendi ve (gRNA) RNA rehberlik edilir. Moleküler etkileşimleri formaldehit ya da başka çapraz bağlayıcı maddelerin, gerektiğinde ile sabitlenir. Daha sonra, sabit kromatin sonication veya enzimatik sindirimi ile parçalanır. Etiketli genomik bölge afinite saflaştırma ile saflaştırılır. Son olarak, çapraz tersidir ve etkileşim molekülleri (genomik bölgelerde, RNA'lar ve proteinler) yeni nesil sıralama ve kütle spektrometresi kullanılarak tanımlanır. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.

GBlock Şekil 2. Sıra. U6 organizatörü (siyah), G nükleotid before kılavuz dizisi (mavi), kılavuz dizisi (kırmızı) (gRNA boşluk), (yeşil) iskele dizisi ve (turuncu) terminatör dizisi gösterilir.
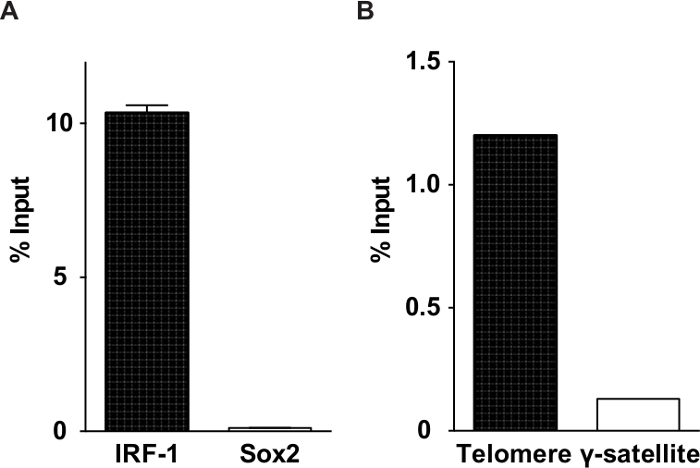
Temsili enChIP analizleri 3. Bir Getirisi Şekil. Hedefe IRF-1 odağı ve hedef olmayan Sox2 lokus için (A) Yüzde girişi. Hedef telomer ve hedef olmayan γ-uydular için (B) Yüzde girişleri. Rakamlar uyarlanmış ve önceki yayınlardan 15,16 den modifiye edilmiştir.
| Kategoriler | Proteinler |
| Transkripsiyon | DDX1, PARP1'i, CKAP4, Pescadillo homologu, PURβ, aktive edilmiş RNA polimeraz II transkripsiyonel aktivatör p15, BTF3, Myb bağlayıcıProtein 1A |
| Histon deasetilasyonu, corepressor bileşenleri | RBBP4, PA2G4, TBL3 |
| Asetiltransferaz | Protein arginin, N-metiltransferaz 1 |
| DNA topoizomeraz | DNA topoizomeraz 2α |
| Histonlar | Histon H2A.Z histon H3.2 |
Tablo 1:. EnChIP-SILAC tarafından tanımlanan bir IFNy-spesifik bir şekilde insan IRF-1 promotör bölgesi ile ilişkili proteinlerin örnekleri tablo önceki yayın 16 adapte edilmiştir.
| Kategoriler | Proteinler |
| Memeli telomer-bağlayıcı proteinler | PML, RPA, CDK1, PARP1'i, PCBP1 |
| Telomer-biMayada nding proteinler ya da diğer organizmalar | IMP4 |
| Ile etkileşim Proteinler telomer bağlayıcı proteinleri [ilişkili telomer bağlayıcı protein] | DNA polimeraz α (POLA1) [Cdc13p], ARMC6 [TRF2] CTBP1 [FoxP2-POT1], exportin-5 [TERT] GNL3L [TRF1], exportin-1 [TERT], 14-3-3 [TERT] |
| Heterokromatindir için lokalize Proteinler | BEND3 |
| Proteinler epigenetik işaretleri düzenleyen | KDM5C |
| Kimin mutasyonlar telomer fonksiyonunu etkileyen Proteinler | DNA polimeraz α (POLA1), HAT1, Nup133, CDK7, DPOE1, PRDX1, TYSY, Glutamat-sistein ligazı, glutaredoksin, SMRC1 |
Tablo 2:. EnChIP-MS yolu ile teşhis fare telomerlerin ilişkili proteinlerin örnekleri, Tablo adaptö olmuşturBir önceki yayın 15 den d.
| Kategoriler | RNA'lar |
| Telomeraz bileşenleri | Terc, Rmrp |
| Telomer RNA'lar | Terras |
| scaRNAs | Scarna6, Scarna10, Scarna13, Scarna2 |
| H / ACA snoRNAs | Snora23, Snora74a, Snora73b, Snora73a |
| C / D snoRNAs | Snord17, Snord15a, Snord118 |
| lncRNA | Neat1 |
Tablo 3:. EnChIP-RNA-Seq tablo önceki yayın 17 den adapte edilmiştir tarafından belirlenen fare telomerlerin ile ilişkili RNA'lar örnekleri.
Tartışmalar
Here, we describe the purification of specific genomic regions using engineered DNA-binding molecules such as the CRISPR system and TAL proteins, and the identification of proteins and RNAs bound to these genomic regions. Binding of engineered DNA-binding molecules to the genome may affect chromatin structure, including nucleosome positioning, and may abrogate genomic functions, as described in CRISPR interference experiments21. To avoid these potential aberrant effects, we propose specific guidelines for choosing target genomic regions. First, to avoid potential inhibition of the recruitment of RNA polymerases and transcription factors, as well as disruption of nucleosome positioning around the transcription start site, the target regions for analyses of promoter regions should be several hundred base pairs upstream of (5' to) the transcription start site. By contrast, when analyzing genomic regions with distinct boundaries, such as enhancers and silencers, genomic regions that are directly juxtaposed to these regions can be targeted because it is less likely that the binding of engineered DNA-binding molecules will affect their functions. Furthermore, it is best to avoid using target regions that are conserved among different species, because important DNA-binding molecules often bind to evolutionarily conserved regions and inhibition of their binding might disrupt the functions of the target genomic regions. In this regard, it is always necessary to check that the function of the target genomic region is maintained in the established cells used for enChIP analyses. Because multiple gRNAs can be tested easily and it is tedious and expensive to generate multiple versions of TAL or zinc-finger proteins recognizing different target genomic regions, enChIP using CRISPR is more advantageous than enChIP using other proteins.
It has been shown that dCas9 binds to off-target sites although affinity to those sites might be weaker than that to the target sites22-25. There are several ways to manage contamination of molecules bound to those off-target sites. First, the use of several, at least two, different gRNAs would be recommended. Those molecules commonly observed in enChIP using distinct gRNAs would be true positives. Second, comparison of different conditions for enChIP would be effective in cancelling contamination of non-specific molecules and molecules bound to off-target sites. Examples of those comparison sets would be (i) stimulation (-) and (+), or (ii) different cell types such as T cells vs. B cells. Finally, quantitative analysis of binding of candidate molecules should be performed to confirm their specific binding to the target sites. It is preferable to prepare cells expressing only dCas9 but not gRNA as a negative control.
Using enChIP analyses, we were able to successfully identify a number of known and novel molecules interacting with specific genomic regions (Tables 1-3)14-17. However, this technique failed to detect some other known proteins interacting with these regions. For example, STAT1 reportedly associates with the IRF-1 promoter upon IFNγ stimulation8, but our enChIP-SILAC analysis did not detect STAT1 as a protein induced to interact with this genomic region16. In addition, in the enChIP-MS analysis of telomeres, we did not detect shelterin proteins consisting of TRF-1 and TRF-215, which have been shown to interact with telomeres26. There are a few potential reasons for these discrepancies. First, the stoichiometry of binding of Stat1 to the IRF-1 promoter might be very low. It is reasonable that enChIP-MS, including enChIP-SILAC, detects proteins that are more abundantly associated with target genomic regions; hence, the analysis of more cells might be necessary to detect these proteins. Increases in the sensitivities of MS instruments would also contribute to the efficient detection of proteins with low stoichiometric binding. Second, some proteins, possibly including Stat1 and shelterins, might be difficult targets for MS analyses. Third, in our analysis of telomere-binding proteins15, the 3×FLAG-TAL proteins recognizing telomeres (3×FN-Tel-TAL) might have blocked the binding of shelterins to telomeres in a competitive fashion.
In contrast to the relative difficulty of detecting transcription factors binding to specific genomic regions using enChIP, we successfully identified epigenetic regulators such as histone modification enzymes using enChIP analyses. The success of this technique may be due to the fact that epigenetic regulators bind to a broad range of genomic regions; hence, more proteins per genomic region are available for MS. Because epigenetic regulators are increasingly recognized as important targets for drugs against intractable diseases such as cancer, enChIP would be a useful tool for the identification of epigenetic drug targets.
Açıklamalar
The authors have filed a patent on enChIP (Patent name: 'Method for isolating specific genomic regions using DNA-binding molecules recognizing endogenous DNA sequences'; Patent number: PCT/JP2013/74107). H.F. is a member of the Advisory Board of Addgene.
Teşekkürler
Bu çalışma Takeda Bilim Vakfı (TF) tarafından desteklenmiştir; Asahi Glass Vakfı; Uehara Memorial Foundation (HF); Kurata Memorial Hitachi Bilim ve Teknoloji Vakfı (TF ve HF); Grant-Aid Genç Bilginler (B) (# 25830131), Grant-Aid Bilimsel Araştırma (C) (# 15K06895) (TF) için; ve Hibe-Aid Yenilikçi Alanları 'Transkripsiyon Döngüsü' Bilimsel Araştırma (# 25118512 & # 15H01354), Grant-Aid Bilimsel Araştırma (B) (# 15H04329) için, Grant-Aid Keşif Araştırma ( # 26650059) ve Japonya Eğitim Bakanlığı, Kültür, Spor, Bilim ve Teknoloji dan 'Genom Desteği' (# 221S0002) (HF).
Malzemeler
| Name | Company | Catalog Number | Comments |
| gBlock synthesis service | Life Technologies | Gene Synthesis by GeneArt | |
| gBlock synthesis service | IDT (Integrated DNA Technologies) | gBlocks Gene Fragments | |
| pSIR-neo | Addgene | 51128 | |
| pSIR-GFP | Addgene | 51134 | |
| pSIR-DsRed-Express2 | Addgene | 51135 | |
| pSIR-hCD2 | Addgene | 51143 | |
| TAL synthesis service | Life Technologies | GeneArt Precision TALs | |
| 3xFLAG-dCas9/pCMV-7.1 | Addgene | 47948 | |
| 3×FLAG-dCas9/pMXs-puro | Addgene | 51240 | |
| 3×FLAG-dCas9/pMXs-IG | Addgene | 51258 | |
| 3×FLAG-dCas9/pMXs-I2 | Addgene | 51259 | |
| 3×FLAG-dCas9/pMXs-neo | Addgene | 51260 | |
| anti-FLAG M2 Ab | Sigma-Aldrich | F1804 | |
| FITC-conjugated anti-FLAG M2 | Sigma-Aldrich | F4049 | |
| DMEM medium for SILAC | Life Technologies | 89985 | Other medium can be purchased from Life Technologies |
| Dialyzed FBS for SILAC | Life Technologies | 89986 | |
| L-Lysine-2HCl for SILAC | Life Technologies | 89987 | For Light medium |
| L-Arginine-HCl for SILAC | Life Technologies | 89989 | For Light medium |
| L-Lysine-2HCl, 13C6 for SILAC | Life Technologies | 89988 | For Heavy medium |
| L-Arginine-HCl, 13C6, 15N4 for SILAC | Life Technologies | 89990 | For Heavy medium |
| Complete, mini, EDTA-free | Roche Diagnostics | 4693159 | |
| Ultrasonic Disruptor UD-201 | Tomy Seiko | ||
| ChIP DNA Clean & Concentrator | Zymo Research | D5205 | |
| Dynabeads-Protein G | Life Technologies | DB10004 | |
| RNasin Plus RNase Inhibitor | Promega | N2611 | |
| Isogen II | Nippon Gene | 311-07361 | |
| Direct-zol RNA Miniprep kit | Zymo Research | R2050 | |
| LTQ Orbitrap Velos | Thermo Fisher Scientific | A component of a nanoLC-MS/MS system for MS analysis | |
| nanoLC | Advance, Michrom Bioresources | A component of a nanoLC-MS/MS system for MS analysis | |
| HTC-PAL autosampler | CTC Analytics | A component of a nanoLC-MS/MS system for MS analysis |
Referanslar
- Zhang, X. Y., Horz, W. Analysis of highly purified satellite DNA containing chromatin from the mouse. Nucleic Acids Res. 10, 1481-1494 (1982).
- Workman, J. L., Langmore, J. P. Nucleoprotein hybridization: a method for isolating specific genes as high molecular weight chromatin. Biochemstry. 24, 7486-7497 (1985).
- Boffa, L. C., Carpaneto, E. M., Allfrey, V. G. Isolation of active genes containing CAG repeats by DNA strand invasion by a peptide nucleic acid. Proc. Natl. Acad. Sci. USA. 92, 1901-1905 (1995).
- Jaskinskas, A., Hamkalo, B. A. Purification and initial characterization of primate satellite chromatin. Chromosome Res. 7, 341-354 (1999).
- Griesenbeck, J., Boeger, H., Strattan, J. S., Kornberg, R. D. Affinity purification of specific chromatin segments from chromosomal loci in yeast. Mol. Cell Biol. 23, 9275-9282 (2003).
- Ghirlando, R., Felsenfeld, G. Hydrodynamic studies on defined heterochromatin fragments support a 30-µm fiber having six nucleosomes per turn. J. Mol. Biol. 376, 1417-1425 (2008).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136, 175-186 (2009).
- Hoshino, A., Fujii, H. Insertional chromatin immunoprecipitation: a method for isolating specific genomic regions. J. Biosci. Bioeng. 108, 446-449 (2009).
- Fujita, T., Fujii, H. Direct idenification of insulator components by insertional chromatin immunoprecipitation. PLoS One. 6, e26109 (2011).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions by insertional chromatin immunoprecipitation (iChIP) with a second-generation tagged LexA DNA-binding domain. Adv. Biosci. Biotechnol. 3, 626-629 (2012).
- Fujita, T., Fujii, H. Locus-specific biochemical epigenetics / chromatin biochemistry by insertional chromatin immunoprecipitation. ISRN Biochem. 2013, 913273 (2013).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions retaining molecular interactions by the iChIP system using recombinant exogenous DNA-binding proteins. BMC Mol. Biol. 15, 26 (2014).
- Fujita, T., Kitaura, F., Fujii, H. A critical role of the Thy28-MYH9 axis in B cell-specific expression of the Pax5 gene in chicken B cells. PLoS One. 10, (2015).
- Fujita, T., Fujii, H. Efficient isolation of specific genomic regions and identification of associated proteins by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP) using CRISPR. Biochem. Biophys. Res. Commun. 439, 132-136 (2013).
- Fujita, T., et al. Identification of telomere-associated molecules by engineered DNA-binding molecule-mediated chromatin immunoprecipitation (enChIP). Sci. Rep. 3, 3171 (2013).
- Fujita, T., Fujii, H. Identification of proteins associated with an IFNγ-responsive promoter by a retroviral expression system for enChIP using CRISPR. PLoS One. 9, e103084 (2014).
- Fujita, T., Yuno, M., Okuzaki, D., Ohki, R., Fujii, H. Identification of non-coding RNAs associated with telomeres using a combination of enChIP and RNA sequencing. PLoS One. 10, e0123387 (2015).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. , (2014).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nat. Methods. 10, 1116-1121 (2013).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173-1183 (2013).
- Wu, X., et al. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat. Biotechnol. 32, 670-676 (2014).
- Kuscu, C., Arslan, S., Singh, R., Thorpe, J., Adli, M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol. 32, 677-683 (2014).
- Cencic, R., et al. Protospacer adjacent motif (PAM)-distal sequences engage CRISPR Cas9 DNA target cleavage. PLoS One. 9, 109213 (2014).
- O'Green, H., Henry, I. M., Bhakta, M. S., Meckler, J. F., Segal, D. J. A genome-wide analysis of Cas9 binding specificity using ChIP-seq and targeted sequence capture. Nucleic Acids Res. 43, 3389-3404 (2015).
- de Lange, T. Shelterin: the protein complex that shapes and safeguards human telomeres. Genes Dev. 19, 2100-2110 (2005).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır