Method Article
Ham Virion ayıklama DNA analizi yeni virüs genleri keşfetmek için virüslü yaprakları gelen RNA analizi ile birleştirerek
Bu Makalede
Özet
Burada iki iplikçikli DNA genleri ile bitki virüsleri tanımlamak için yeni bir yaklaşım mevcut. DNA ve RNA virüslü yaprakları ayıklamak ve yeni nesil sıralama dışarı taşımak için standart yöntemleri kullanın. Bioinformatic araçları dizileri contigs monte, virüs genleri temsil eden contigs tanımlamak ve genleri taksonomik gruplara atamak.
Özet
Bu metagenome yaklaşım dairesel DNA genleri ile bitki virüs ve onların transkript tanımlamak için kullanılır. Ev sahibi olarak düşük titreleri meydana veya mekanik olarak başka bir ana bilgisayara aşılamak değil sık sık bitki DNA virüsler bulaşıcı malzeme büyük bir titresi elde etmek için yaymak zordur. En uygun pH ve çoğu bacilliform Para retrovirüsler arıtma için önerilen iyonik bileşimi ile hafif bir tampon toprağa virüslü yapraklar. Üre virions tuzak dahil cesetleri kırmaya ve hücresel bileşenleri dağıtmak için kullanılır. Fark Santrifüjü daha fazla virions ayrılması bitki kirletici üzerinden sağlar. Sonra İndinavir K tedavi capsids kaldırır. Sonra viral DNA konsantre ve yeni nesil sıralama (NGS) için kullanılır. NGS veri virüs dizileri oluşturulan veri kümesi kümesini tanımlamak için NCBI-BLASTn için hangi gönderilmektedir contigs birleştirmek için kullanılır. Paralel bir boru hattı, RNA bir standart sütunlara göre RNA ayıklama yöntemi kullanarak enfekte yapraklarından izole edilmiştir. MRNA ve virüs tutanaklar bir alt için zenginleştirmek için ribozom tükenmesi sonra gerçekleştirilir. Birleştirilmiş dizileri (RNA-seq) sıralama RNA türetilmiş virüs serilerinde bu veri kümesi kümesini tanımlamak için NCBI-BLASTn için sunuldu. Bizim çalışmada, iki veri kümeleri içinde iki ilgili tam uzunlukta badnavirus genleri tespit edilmiştir. Bu yöntem bitki virüs genom dizileri reconstitute için toplam nüfusun küçük RNA dizilerinin ayıklayan başka bir ortak yaklaşım tercih edilir. Bu ikinci metagenomic boru hattı kurtarır virüs ile ilgili diziler bu retro transkripsiyonu öğeleri bitki genom eklenir. Bu daha da aktif enfeksiyöz ajanlar ayırt etmek biyokimyasal veya moleküler deneyleri için birleştirilmiştir. Bu çalışmada, belgelenen yaklaşım dizileri büyük olasılıkla etkin virüs enfeksiyonu gösteren virüsler çoğaltılıyor temsilcisi kurtarır.
Giriş
Ortaya çıkan bitki hastalıklarının araştırmacılar doğru nedensel temsilcisi tanımlamak için yeni araçlar geliştirmek için sürücü. Yeni veya yinelenen virüs hastalıklarının başlangıç raporlarını mozaik ve yaprak, takas ven, cücelik, solan, lezyonlar, nekroz malformasyonlar gibi yaygın olarak karşılaşılan belirtiler veya diğer belirtiler üzerinde temel alır. Diğer bulaşıcı patojenler ayırmak için bir hastalık için nedensel ajan olduğu gibi yeni bir virüs raporlama için standart uygun konukçuda yaymak ve özgün ana bilgisayar türlerinin sağlıklı bitkiler aşı hastalık ürerler. Bu yaklaşımda bitki virüslerin birçok cins bir böcek ya da diğer vektörleri iletim uygun bir ana bilgisayar veya yeniden özgün ana türler için üzerine bağlıdır kısıtlamadır. Bu durumda, uygun vektör için arama uzun, Yöneyin laboratuar koloniler kurmak için zorluklar olabilir ve daha fazla çabaları bir protokol deneysel iletimi için hazırlamak gereklidir. Eğer başarılı laboratuar iletim çalışmaları için koşullar elde çalışma yeni bir virüs hastalık raporlama için standart kısa düşüyor. Çok düşük titreleri doğal onların ana oluşan virüs olarak araştırmacılar araştırma taşımak için yeterli bulaşıcı hisse senetleri korumak için alternatif hosts yayma için tanımlamanız gerekir. Sadece bir kaç bitki bulaştırmak virüs türler için bu da hisse senedi kültürler1büyüyen için bir engel olabilir.
Son yıllarda, bilim adamları daha sık yüksek üretilen iş NGS ve metagenomic yaklaşımlar için bilinen bir hastalık ilgisiz bulunabilir, ancak taksonomik tür ve cins atanabilir çevre, mevcut virüs dizileri ortaya çıkarmak için istihdam olduğunu 2 , 3 , 4. bulma ve ayrı bir ortamda genetik materyallerin sınıflandırılması böyle yaklaşımlar virüs çeşitlilik doğanın ya da varlığı belirli bir ekosistem tanımlamak için bir yol sağlar, ancak mutlaka tanımlamak için bir çerçeve için onaylamak değil nedensel maddeleri için belirgin bir hastalık.
Badnavirus cins Caulimoviridae pararetroviruses ailesine ait. Bu virüsler bacilliform şekli ile dairesel Çift Kişilik iplikçik DNA genleri yaklaşık 7-9 KB vardır. Tüm pararetroviruses bir RNA orta yineler. Pararetroviruses episomes mevcut ve bitki kromozom DNA5,6bağımsız çoğaltmak. Saha çalışmaları virüs nüfusun bu virüs nüfus genetiği karmaşık olduğunu gösteriyor. Buna ek olarak, bir bitki genom aralığında yüksek işlem hacmi sıralama tarafından elde edilen bilgileri gayri meşru tümleştirme olaylar tarafından bitki genleri eklenen badnavirus genom parçaları sayısız örnekleri ortaya. Bu endojen badnavirus dizilere mutlaka enfeksiyon7,8,9,10,11ile ilişkili değildir. Daha sonra yeni badnaviruses hastalığı nedensel ajan olarak tanımlamak için NGS kullanımı episomal genleri subpopulation çeşitliliği yanı sıra endojen dizileri12,13oluşumunu karmaşıktır.
Roman pararetrovirus genleri keşfi için değil bir en uygun potansiyel varken, bu virüs hastalığı için nedensel ajanı olarak tanımlamak için iki genel yaklaşım vardır. Virüslü yaprakları küçük RNA sıralarının zenginleştirmek ve virüs genome(s)14,15,16,17yeniden oluşturmak için bu dizilere araya bir yöntemdir. Çalışırken daire amplifikasyon (RCA) dairesel DNA virüs genleri18yükseltmek için başka bir yaklaşımdır. RCA başarısı yaş yaprak ve virüs titresi seçilen doku bağlıdır. RCA ürünleri kısıtlama sindirim için tabi ve plazmidler için19,20,21sıralama doğrudan içine klonlanmış.
Canna sarı mottle virüs (CaYMV) bir badnavirus olduğunu ve sadece bir 565 bp parçası genom virüslü cannas22daha önce izole olmasına rağmen canna, sarı mottle hastalığının etyolojik neden olarak tanımlanıyor. Çağdaş bir çalışma Alpinia purpurata (çiçekli zencefil; CaYMV tespit CaYMV-Ap)23. Bu çalışmanın amacı tam badnavirus genom dizileri virüslü canna lilyum kurtarmak için oldu. Biz virüs--dan bitki kirletici arındırıcı ve bu hazırlık üzerinden viral DNA izole eden bir protokol tanımlamak ve DNA kitaplığa NGS kullanmak için hazırlamak. Bu yaklaşım ara moleküler amplifikasyon adımları gereksinimini ortadan kaldırır. Biz de RNA-seq. NGS için virüslü bitkilerden mRNA izole olan RNA-seq içerir her nükleik asit hazırlık kullanılarak gerçekleştirilmiştir. Monte contigs Badnavirus takson nükleik asit (BLASTn) için Biyoteknoloji ve bilgi (NCBI) temel yerel hizalama arama aracı için Ulusal Merkezi kullanarak her iki DataSet'lerdeki ile arasında bir ilişki bulunamadı. Biz iki badnavirus türler24genleri tespit.
Protokol
1. genel virüs arıtma fark Santrifüjü Covey vd tarafından standart yöntemi kullanarak tarafından 25
- İlk olarak, 80-100 g hastalıklı bitki yapraklarından, kesmek ve eziyet 200 mL arabellek zımpara kullanarak 4 ° C'de WARING bir karıştırıcıda (0,5 M NaH2PO4, 0, 5 M Na2HPO4 (pH 7.2). ve %0,5 (w/v) Na2SO3). Bir laboratuvar kat ve bu yordamın tüm adımları için eldiven giymek.
- Ardından homogenate (300 mL) 1.0 L kabı aktarın. 18 g üre ve 25 mL % 10 Noniyonik deterjan (t-Ekim-C6H4-(OCH2CH2)9OH) homogenate içinde kimyasal bir başlık ekleyin.
Not: Bu adım için koruyucu gözlük ve kişisel koruma için basit nefes maske giymek en iyisidir. - Kısaca mahalledeki bir manyetik karıştırıcı ile karıştırın ve kabı folyo ile kaplayın. Sonra folyo kaplı kabı soğuk bir odaya transfer ve gecede 4 ° C'de bir manyetik karıştırıcı ile karıştırın
- Rotor şişe (250 mL konteynerler) ve 4 ° C'de 10 dakika için 4.000 x g de sabit açılı rotor santrifüje santrifüj kapasitesi homogenate aktarma Bir kimyasal duman başlık, süpernatant ve tülbent 4 kat filtreden kurtarmak.
- Homogenate 38,5 mL polipropilen santrifüj tüpleri ve santrifüj 40.000 x g 4 ° C'de de 2,5 h arasında bölmek Genellikle, bir yeşil Pelet tüpün dibinde ve tüp uzunluğu boyunca beyaz bir Pelet varlığı için kontrol edin. Her iki Pelet korumak ve süpernatant dökün; örnekleri buza koyun.
Not: Yeşil Pelet kloroplast, nişasta ve diğer organelleri içerir. - Kimyasal bir mahallede çalışma, bir lastik polis granül ayırmak için kullanın. GKD2O her rotor şişe 1 ml beyaz Pelet süspansiyonlar 4 ° C'de malzemeleri tam çözümde dağıtmak izin vermek için bir gecede korunarak 1-2 saat boyunca resuspend. Süspansiyon 6.000 g x ve 4 ° C için kalan enkaz kaldırmak 10 dk santrifüj kapasitesi.
- 136,000 x g 4 ° C'de virions cips için 2 h için de konsantre süspansiyon santrifüj kapasitesi. Granül 1 mL arabellek (50 mM Tris-HCl, pH 7.5, 5 mM MgCl2) resuspend.
Not: İsteğe bağlı bir adım virions DNaz ile tedavi etmektir ben (10 µg/mL) 37 ° C'de encapsidated DNA, kloroplast ve mitokondri DNA kirletici Yani, kaldırmak için 10 dakika. O zaman, DNaz devre dışı bırakın ben 1 mM EDTA ekleyerek. - Virions ile 2 µg/µL İndinavir K 15 dakika 37 ° C'de 40 µL bozabilir.
- Virion DNA tarafından organik ayıklama kurtarmak için kimyasal bir başlık içinde çalışmak. Potansiyel Akut sağlık etkileri karşı koruma için ayıklama sırasında yüz kalkan, eldiven ve bir laboratuvar ceket giymek. Fenol kloroform izoamil alkol (49:50:1) 1 birim için örnek eklemek ve 16.000 x g., 5 min kaldırmak için üst sulu faz ve yeni bir tüp aktarmak için oda sıcaklığında 20 s. santrifüj el ile çalkalanır. Bu ayıklama iki kez tekrarlayın. Organik aşaması için uygun kurumsal kimyasal atık26cam atık şişe koyarak atın.
- Etanol yağış kullanarak DNA konsantre. Sodyum asetat (pH 5.2) 0,3 M son konsantrasyonu kullanın ve %2.5 95 etanol hacimleri. Örnekleri-20 ° C-30-60 dk ve santrifüj 13.000 x g 10-20 dk DNA26cips de yerleştirin.
- Bir laboratuvar tezgah çalışma, DNA pelet 1 mL 0.1 mM TE arabellek (pH 8.0) resuspend. Filler üzerinden (normalde polimeraz zincir tepkimesi (PCR) temizlemek kullanılır) ticari jel filtrasyon sütun süspansiyon tuzları ve NGS engel düşük moleküler ağırlıklı malzeme ortadan kaldırmak için.
- Örnekleri arasında etidyum bromür hazırlıkları kalitesini görüntülemek için boyama kullanarak % 1'özel jel elektroforez tarafından analiz. DNA nanodrop spektrofotometre kullanarak kalitesini değerlendirmek.
Not: 260 λ ve 280 λ 1.85 ve 2.0 arasında örnek absorbans oranı genellikle hazırlık "temiz" kirleri ve istenen kaliteye sahip gösterir. - DNA kalitesini analiz (5 10 sayfaya kullanın ng) kapiler Elektroforez araç tabanlı bir çip kullanılarak.
Not: Temiz tepeler, DNA parçalarının boyutu bir x ekseni boyunca dağıtım şirketi temsil eden kaliteli çıkışı gösterir. En yüksek yükseklik bereket parça gösterir. Sivri tepeler kısmen bozulmuş parçaları veya kimyasal kirleticiler gösteriyor. Bir smear kalitesiz gösteren DNA'ın yuvarlak eğrileri temsil
2. Kütüphane hazırlık kullanma DNA ve klonal amplifikasyon (emPCR amplifikasyon) emülsiyon esaslı
Not: Kütüphane genellikle müşteri odaklı çalışma taşıyan bir NGS tesis tarafından hazırlanmıştır.
- DNA parçaları dönüştürür bir Nebulizatör kullanarak bir çözüm DNA (> 200 ng) kesme. Kitabın yönergeleri27göre ticari bağdaştırıcıları ligate.
- DNA örneği üreticinin yönergeleri28,29,30göre amplifikasyon emPCR yerine getirir. Üç kez yıkama adımı yineleyin ve her yıkama sonra bir minicentrifuge boncuk için 10 s. atma süpernatant her yıkama sonra cips.
Not: Yordamı çamaşır kit ile sağlanan ticari yıkama arabellek tarafından yakalama boncuk hazırlanması ile başlar. emPCR için şablon amplifikasyon NGS için yaygın olarak kullanılır. - Isı denatüre DNA veya RNA 95 ° C'de 2 min ve o zaman 4 ° C kadar kullanıma hazır. Kullanım 200 - milyon moleküllerinin DNA/RNA son hacmi 30 µL. 5 milyon yakalama boncuklar DNA/RNA örnek ve nükleik asit örnek hem de sahte örnek aşağıdaki adımları gerçekleştirmek yanında sahte bir örnek hazırlamak.
- Emulsiyon vortexing emülsiyon petrol 10 için tüp yerine getirilir s maksimum hızda sonra bir platform homogenizer ile uyumlu olan tüp karıştırarak bir plastik içine tüm içeriği (4 mL) dökün. Karıştırma tüp 5 min için 2.000 devirde emülsiyon karıştırmak için bir platform üzerine yerleştirin.
- Emülsiyon 100 µL aliquots 8-şerit kap tüpler veya bir 96-şey plaka dağıtmak. Tüpler kap veya plaka mühür ve üreticinin önerilen program28kullanarak emPCR taşımak.
Not: PCR tamamlandığında, emülsiyon sağlam olup olmadığını ve sonra devam etmek için kuyu olup olmadığını denetleyin. Emülsiyon çalışmıyorsa tüm şey atmak. - Önlük giymek ve güçlendirilmiş DNA boncuk (ADB) toplamak için kimyasal bir mahallede çalışma. Vakum kuyulardan emülsiyon Aspire edin ve bir 50 mL tüp boncuk toplamak. İsopropanol 100 µL iki kez Wells'le durulama ve aynı 50 mL tüp için durulama Aspire edin.
- Girdap toplanan emülsiyonlar ve ADB son hacmi 35 mL için isopropanol ile resuspend. ADB vasıl 930 x g 5 dk. Kaldır süpernatant cips ve arabellek geliştirme 10 mL ekleyin. Girdap ADB ve her yıkama ve iki kez yıkama adımı yineleyin sonra 40 mL son hacim santrifüj isopropanol ve atma süpernatant ekleme tarafından o zaman yıkama.
- Etanol isopropanol yerine kullanarak son yıkama yerine getirir. Eklemek 35 mL son hacim, girdap ve Pelet arabelleğe artırılması vasıl 930 x g 5 dakika süreyle boncuk süpernatant kaldırmayı ancak arabellek geliştirme 2 mL bırakmak.
- Süspansiyon microcentrifuge tüp aktarmak ve kısaca ADB cips santrifüj kapasitesi. Süpernatant discarding sonra ADB Pelet iki kez artırılması arabelleği 1 mL ile yıkayın. Santrifüj kapasitesi ve süpernatant her yıkama sonra atın.
- DNA Kütüphane boncuk zenginleştirme için hazırlamak için boncuk 1 mL 1 N NaOH ekleyin. Girdap ADB ve oda sıcaklığında 2 min için kuluçkaya. Santrifüj kapasitesi ve süpernatant atın. Bir kez bu yıkama tekrarlayın.
- Tavlama arabellek, sonra girdap ADB 1 mL ekleyin ve oda sıcaklığında 2 min için kuluçkaya. Kısaca santrifüj kapasitesi ve süpernatant atın. Tekrar 100 µL tavlama arabelleği kullanarak bu adımı yineleyin.
- DNA için sıralama astar tavlama Seq astar a 15 µL ve Seq astar kit ile sağlanan b 15 µL ekleyin. Kısaca vortexing tarafından mix ve 5 dk. 2 min için buz Transfer 65 ° C ısı bloğunda microcentrifuge tüpü yerleştirin.
- Üç kez 1.0 arabellek tavlama mL ile yıkayın. 5 s ve atma süpernatant her zaman için girdap.
- Sıralama daha önce bir ticari boncuk sayaç boncuk sayısını ölçmek. En az 500.000 zenginleştirilmiş boncuk olmalıdır.
Not: Boncuk sayaç bir sağlanan microcentrifuge tüp boncuk ölçer özel bir aygıttır.
3. genel mRNA yalıtım ve dsDNA sentez başlangıç ile enfekte Canna test yaprakları tarafından RT-PCR CaYMV kullanarak bildirilen tanılama astarlar için
- Bir laboratuvar kat ve lateks eldiven kişisel koruma için tüm sonraki adımlar giymek. Bir laboratuvar tezgah çalışma, yapraklarından 12 örnekler toplamak ve örnekleri sıvı azot Dalma. Bir boncuk değirmen homojenizasyon için kullanın. Toplam bitki RNA izolasyon için standart bir sütun tabanlı yöntem sağlar bir ticari seti kullanın. 20 için belgili tanımlık teçhizat zemin örnek ve sallamak için sağladığı guanidin-isothiocyanate lizis arabellek eklemek s.
- Etanol ve kit yönergelerine göre iyice karıştırın. Her homogenate RNA membran için bağlar bir spin sütun ekleyin. Üç kez yıkama ve RNA kurtarma tüp24elute.
- 260 λ ve % 1'özel jel elektroforez etidyum bromür ile lekeli kullanarak 280 λ. bütünlük doğrulama RNA absorbans oranı ölçmek için bir spektrofotometre kullanarak RNA ölçmek.
Not: 1.85 bir absorbans oranı 2.0 hazırlık istenen kalite olduğunu gösterir. RNA DNaz ile tedavi 37 ° C'de 10 dakika ben (10 µg/mL) Bir ticari spin sütun RNA RNase free su31' de konsantre için kullanın. Devam etmeden önce havuzu RNA örnekleri. - Bir rRNA kaldırma kiti bitki ribozomal RNA kaldırmak için kullanın. Microcentrifuge tüp ve iki kez yıkama aliquot manyetik boncuklar RNase free ile su. Girdap resuspend için aliquot tüp tüp manyetik bir stand üzerine yerleştirin ve sıvı temizlemek bekleyin. Süpernatant atın ve yerine koymak ile manyetik boncuk resuspension eriyik. Girdap resuspend ve RNase inhibitörü 1 µL eklemek için.
Not: Bu tür kitleri oligo-dT için mRNA melezlemek manyetik boncuklar bağlı kullanın. Transkript24kurtarmak için standart mıknatıs boncuk ayırma teknoloji yöntemini kullanır. - RNA, 1,25 µg birleştirmek 500 ng RNase free su ve reaksiyon arabellekleri kit tarafından sağlanan. Yer 50 ° C'de 10 dakika karışımı Kaldır ısı ve yıkanmış manyetik boncuklar RNAse free suya ekleyin. Kısaca girdap ve oda sıcaklığında 5 min için kullanýlýr.
- Manyetik bir stand üzerine yerleştirin ve sıvı temizlemek bekleyin. Süpernatant temiz microcentrifuge tüp aktarın. Buz üzerinde ayarlayın.
- Exosomes ve 200 zenginlik için bir çözüm tabanlı yakalama yöntemi kullanın ng cDNA Kütüphanesi hazırlamak için RNA'ın.
Not: Çift Kişilik strand cDNA Kütüphanesi genellikle müşteri odaklı çalışma taşıyan bir NGS tesis tarafından hazırlanmıştır. - Piyasada satılan bir RNA parçalanma solüsyonu (0.136 g ZnCl2 ve 100 mM Tris-HCl pH 7,0) kullanarak RNA parçası. Çözüm 2 µL RNA'ın 18 µL ekleyin (200 ng toplam). Microcentrifuge kısaca tüplerde spin, 70 ° C 30 s ve buz için transfer için örnekleri yerleştirin. 0, 5 M EDTA pH 8.0 ve 28 µL 10 mM Tris-HCl pH 7.5 2 µL kullanarak tepki bırak.
- Bağlama RNA, oda sıcaklığında 10 dakika süreyle karıştırma tarafından manyetik boncuklar için boncuk toplamak ve süpernatant atmak için manyetik yoğunlaştırıcı kullanın. Boncuk üç kez 200 µL % 70 etanol ile yıkayın. Her yıkama ve kuru hava atma pelleted boncuk 3 dakika süreyle oda sıcaklığında 10 mM Tris-HCl pH 7.5 19 µL içinde Resuspend.
- 70 ° c 10 dk Isıtma tarafından parçalanmış RNA rasgele astar tavlama ve tüp buz 2 dk. hazırla ilk strand ve bir standart ticari cDNA sentez kit kullanarak ikinci strand cDNA yerleştirin.
- Manyetik boncuk yoğunlaştırıcı kullanarak iki iplikçikli cDNA arındırmak. % 70 etanol 800 µL ile üç kez yıkayın. Her yıkama ve kuru hava atma granül 3 dakika süreyle oda sıcaklığında 10 mM Tris-HCl pH 7.5 16 µL içinde Resuspend. Manyetik boncuk yoğunlaştırıcı boncuk şimdi çözümdür çift iplikçikli cDNA ayırmak için kullanın. CDNA yeni 200 µL PCR tüp içine pipetting tarafından kaldırın.
- Taq polimeraz ve ticari Kütüphane hazırlık seti tarafından sağlanan deoxyribonucleotides karışımı kullanarak parça son onarım gerçekleştirmek. Ticari seti her iki ucuna ticari ligaz 25 ° C'de 10 dakikadır kullanarak iki iplikçikli cDNA eklemek için önceden seyreltilmiş bağdaştırıcılar sağlar.
4. NGS DNA kitaplığı hazırlanmış ham virüs hazırlık ve dsDNA mRNA kitaplığı hazırlanmış
- DNA dizilerinin doğrudan sonuçlar üretmek için tüm önerilen üreticileri protokolleri takip ve bir standart yüksek işlem hacmi pyrosequencing aleti kullanın. Ticari sıralama reaktifler, Fluorescently etiketli nükleotit dahil olmak üzere kullanın.
Not: için Ayrıntılar araç ile sağlanan üretici yönergelerine bakın. - Sonrası sıralama analiz otomatik olarak contigs ilk dizi < 700 bp. ortalama uzunluğu ile üretmek için okuma çeviren genom derleme yazılım kullanarak yürütmek kalite kontrol gerçekleştirir iPlant/CyVerse Web sitesinde FastQC yazılımlar ham sıralı veri32üzerinde denetimler. Serileri ile Phred puanları ≥ 30 haritalama ve amplicon yazılım kullanarak daha küçük sıra Okunma24 uzun serileri birleştirmeye devam etmek için seçin.
Not: için üreticinin yönerge ayrıntıları bakın. - MEGABLAST varsayılan modül hem de Viridplantae NCBI-BLASTn analiz için monte bu contigs gönderin (TaxID: 33090) ve virüsler (TaxID: 10239) organizmalar sınırlama33isimleri gibi. Toplamak için yüksek benzerliği göstermek contigs subpopulation Badnavirus genleri bir raporda bildirdi.
- Aday tam uzunlukta virüs genleri, bir veya daha fazla temsil birleştirilen iskele düzgün standart badnavirus genom olarak aynı örgüt-si olmak çerçeve diziler üretmek doğrulayın. Bunu yapmak için aday tam uzunlukta virüs genomu yazılım çizim bir plazmid girdi. Sonra ilk 15 nükleotit oluşur hangi badnaviruses arasında son derece korunmuş bir tRNAbir araya geldi (TGGTATCAGAGCGAG) onaylayın. Potansiyel Poliadenilasyon sinyal sonuna 3' genom bulun. İki küçük ORFs ve bir polyprotein kodlama bir büyük ORF varlığı tanımlamak için komple genom açıklama ekleyin. Sonra kullanım ExPASy portalı çevirme aracı ORF1, ORF2 ve ORF3 çeviri ürünler34badnavirus tespit etmek.
Not: Bu bilimsel yazılım ücretsiz ve dairesel DNA oluşturmak, tüm açık okuma tespit çerçeveler ve sıra tam uzunlukta dairesel DNA genom temsil ettiğini doğrulamak için bir acil çıkış sağlar. - Açık kaynak birden çok sıra karşılaştırma araçları, kas ve İLOGENETİK, DNA ve RNA analizleri35,36elde virüs genleri karşılaştırmak için kullanın.
- 30 badnavirus türlerin tam genom dizileri elde edilir ve .fasta biçiminde bir belge olarak dışa aktarın NCBI nükleotit veritabanında arama yapın. Diziler dizileri NGS tarafından elde edilen virüsü genom dizileri birlikte evrimsel Genetik Analizi yürütmektedir bir yazılım yükleyin. Birden fazla sıra hizalamaları ve maksimum olabilirlik ağaçlar kas37kullanarak oluşturun.
5. kalite De Novo sıralama PCR güçlendirme virüs genleri enfekte bitkiler tarafından değerlendirilmesi
- PCR astar38türetmek için ücretsiz çevrimiçi Primer3 araç içine yeni tanımlanan tam uzunlukta badnavirus genom dizileri (.fasta formatında) girdi. 1.000-1.500 BP virüs genome(s) tüm uzunluğu boyunca sendeleyerek ürünleri üretecek astar kümeleri tanımlar. Dizileri sentez ve PCR astar sağlayan bir servise göndermek.
Not: Çıkış yaygın ve kabul edilebilir erime sıcaklıkları ve hassas astar konumları tanıtılan sıraları boyunca kabul edilebilir astar çiftleri tanımlar. - Bir laboratuvar tezgah çalışma ve önlük ve eldiven giyiyor, DNA 5 µg bitki malzeme39 DNA izole etmek için standart paramagnetic selüloz parçacıklar içerir otomatik bir yöntem kullanarak virüs bulaşmış ve sağlıklı kontrol yapraklarından izole etmek. Bir microcentrifuge tüp sıvı azot yaprak malzeme (20-40 mg) donma ve Boncuk Değirmen kullanarak eziyet. Örnek bir microcentrifuge tüp lizis arabelleği ile birleştirmek ve her örnek için RNase A ekleyin. Girdap örnek 10-20 s ve kısa bir süre spin katı parçacıkları kaldırmak için örnek.
Not: Paramagnetic selüloz parçacıklar yüksek DNA'ya bağlanıcı kapasitesine sahip ve yüksek verim saf DNA'ın izole. Standart ticari silis sütun yöntemleri DNA izolasyonu için etkili bir şekilde DNA çeşitli bitki türleri arasında ayıklamak değil. Sonuç olarak, onlarca Yöntem değişiklikleri tek tek bitki türleri verimliliği artırmak için bu yordamlardan birini olan mevcut. 25'ten fazla otsu Gadidae türler40daha fazla ve daha yüksek kalitede DNA verimleri çünkü otomatik paramagnetic selüloz parçacık yöntemi seçilmiştir. - Ticari reaktif kartuşlarını otomatik paramagnetic DNA izolasyon için kullanın. Her ticari reaktif Kartuşu ve transfer bitki aynı kartuş ile lysate 300 µL nükleaz boş su ekleyin. Kartuş kartuş rafa yerleştirin, elüsyon Tube En yakın iyi bir dalgıç yerleştirin ve elüsyon arabellek elüsyon tüp içine yerleştirin. Kartuşları otomatik nükleik asit yalıtım makineye yükleyin ve bitki DNA izolasyon protokol41,42çalıştırın.
- Üst üste gelen PCR ürünleri bir dizi türetmek için PCR taşımak. Her ileriye ve geriye doğru astar 5 mikron PCR güçlendirme 35 döngüleri ile kullanın. Aşağıdaki Bisiklet koşullarını kullanın: denatürasyon 95 ° c 60 s, 7-10 dakika ortadan kaldırmak için bir hazır ambalajlı jel filtrasyon sütun kullanım tuzlar için 45 s ve uzantısına sahip bir son 72 ° c için 1-2 dk 72 ° C'de uzantısı için 50 ° C'de tavlama ve düşük moleküler ağırlıklı malzemesi olarak Adım 1.231' de.
- 50'ye ligate için PCR ürünü miktarını belirlemek için vektör için PCR ürünü 3:1 molar oranını hesaplamak ng Doğrusallaştırılmış pGEM plazmid43. Bir denetim ekleyin DNA d verimli bir biçimde çalışması olup olmadığını belirlemek için kullanın. Gecede 4 ° C'de T4 DNA ligaz (3 U/µL) kullanarak birleştirmesini gerçekleştirmek Sonra dönüşüm ticari olarak hazırlanan JM109 yetkili Escherichia coli hücreleri. Kullanımı verimli dönüştürme için pozitif kontrol olarak kesilmemiş plazmid DNA 100 pg kontrol. 100 µL üzerine LB-ağar kaplamalar dönüştürülmüş hücrelerin ve birleştirilmiş plazmid26kurtarmak için antibiyotik ve mavi/beyaz seçimle plaka. Kaplamalar, 16-24 h 37 ° C'de için kuluçkaya
Not: PGEM vektör β-galaktozidaz kodlar bir lacZ gen vardır. Bir plaka üzerinde yetiştirilen dönüştürülmüş bakteri içeren 100 µg/mL ampisilin, 0,5 mM IPTG, 80 µg/mL 5-bromo-4-chloro-3-indoyl-β-D-galactopyranosidase (X-gal) β-galaktozidaz etkinliği nedeniyle mavi dönecek. PGEM plazmid lacZ gen bozan bir şekilde Doğrusallaştırılmış. PCR ürünü ekler içeren koloniler lacZ gen bozabilir ve X-gal metabolize değil. Bu koloniler beyazdır. Böylece kolonileri bir ekleme ile o olmadan bir ekleme kolonisi (mavi beyaz)26renk tarafından ayrıştırılan. - Standart sütun tabanlı plasmid izolasyon kit39kullanarak üç kolonilerden DNA izole et. Dönüştürme ürünün üç plazmid sıra. Her DNA dizisi NGS tarafından üretilen monte de novo virüs genleri ile karşılaştırın. İLOGENETİK dizileri hizalamak için ve uygun bir şekilde sıralanır emin olmak için kullanın.
Sonuçlar
Bu değiştirilmiş virüs arıtma yöntemi virüs DNA'lar NGS ve Biyoinformatik iki virüs türü tanımlamak için yararlı bir zenginleştirme sağlanan. Homogenate 40.000 x g 2,5 h için de centrifuged sonra tüp altındaki yeşil bir pelet ve uzunluğu boyunca beyaz bir Pelet vardı. Yeşil Pelet bir microcentrifuge tüp içine resuspended ve beyaz Pelet iki microcentrifuge tüpler içine resuspended. PCR standart CaYMV PCR tanılama primerler kullanılarak gerçekleştirildiği ve ürünleri solubilized beyaz Pelet ve değil yeşil Pelet (Şekil 1A) tespit edildi. Ham hazırlama örneği transmisyon elektron mikroskobu tarafından incelenmiş ve 124-133 nm uzunluğundadır (Şekil 1B) ölçme bacilliform partikülleri görülmektedir. Çoğu badnaviruses öngörülen kalıcı uzunluğu içinde bu. DNA beyaz ve yeşil çökeltilerini çıkarılan ve ayrı ayrı resuspended. Şekil 1C, biz 5 µL yeşil çıkarılan DNA'ın yüklü ve beyaz Pelet örnek (yeşil kısmı için DNA'ın 1.6 µg) ve DNA 3,1 µg beyaz kısmı için % 0,8 özel Elektroforez jel ve etidyum bromür takip DNA analiz boyama. Beyaz kesir daha yüksek molekül ağırlıklı DNA, hem de daha düşük moleküler ağırlıklı DNA (Şekil 1C) iki grup üretilen ise yeşil kesir düşük moleküler ağırlıklı DNA içerdiği. Şekil 1' deC sunulan jel 100 V 40 min için çalıştırıldı ve jel gerilim daha net bantları üretmek için düşürülmelidir şeritte 3 smear öneriyor. Bu verileri beyaz Pelet virions için zenginleştirilmiş göstermektedir. Beyaz örneğinden çıkarılan DNA (0.6 µg/mL) konsantrasyonu düşük ama yeterli için en az 10 gerektirir NGS ng devam etmek için DNA'ın. Parçalanmış DNA'lar bir kütüphane NGS için hazırlamak için kullanıldı.
Buna paralel olarak, RNA virüslü canna bitkilerden (Şekil 1D) yüksek üretilen iş RNA-devamı için ayıklandı Standart bir iş akışı için Kütüphane hazırlık, contigs oluşturma ve viral genom dizileri (Şekil 1E) tanımlayan NGS yapılmıştır. DNA ve RNA malzemeleri başlangıç olarak kullanarak çıkış sonuçları karşılaştırıldı.
Biz 188,626 ham DNA okuma NGS tarafından ham virüs hazırlık izole DNA'yı kullanarak elde. Okuma 13,269 contigs monte ve BLASTn NCBI veri kümesi nükleotit dizileri aramak için kullanılan (Viridplantae TaxID kullanarak: 33090 ve virüs TaxID: 10239 sınırlayıcı organizmalar olarak) (resim 1E). NCBI-BLASTn sonuçları monte de novo contigs % 93 hücresel dizileri vardı, % 22 bilinmeyen ve virüs contigs (resim 2A) % 0,3 olduğunu ortaya koydu. Contigs hücresel dizileri olarak mitokondriyal tespit edilmiştir olarak kategorize veya kloroplast DNA çoğunluğu. Virüs contigs veri kümesi içinde virüs contigs % 32 (yani Badnavirus dizileri edilmiştir), üyelerine Caulimoviridae ilgili olduğunu ve bunların % 58'i için ile ilgili Badnavirus. Virüs contigs, % 29 yaşına geldiğinde son derece benzer (e < 1 x 10-30)'e CaYMV izole V17 ORF3 gen (EF189148.1), şeker kamışı bacilliform virüs izole Batavia D, komple genom (FJ439817.1), ve muz çizgi CA virüs genomu (tamamlamak KJ013511). Bu nüfus içinde iki tam uzunlukta genleri andıran uzun contigs vardı.
Yüksek üretilen iş RNA-seq üretilen 153,488 temizlenmiş bireysel sıra okuma okuma < 500 bp. dokunma derleme uzunluğu ortalama ile bu 8,243 contigs için azaltılmış. Bunlar NCBI-BLASTn için sunuldu (Viridplantae TaxID kullanarak: 33090 ve virüs TaxID: 10239 sınırlayıcı organizmalar olarak) contigs % 76'bitki hücre dizileri, % 23 kategoride yer çıkışlarını bilinmeyen ve % 0.1 virüs contigs () kategorize Resim 2 B). virüs contigs % 0.1 nüfusunun nüfusun daha yakından incelendiğinde belirlenen bunlar % 68'i Caulimoviridae için (Şekil 2B) ayrıldı. Bu nüfus içinde üç büyük contigs yüksek benzerliği ile tespit edildi (e < 1 X 10-30) CaYMV ayrı tut moduyla V17 ORF3 gen için (EF189148.1), şeker kamışı bacilliform virüs izole Batavia D, komple genom (FJ439817.1) ve muz çizgi CA virüs komple genom (KJ013511). Üç contigs incelenmesi, biz el ile bu bir tam uzunlukta virüs genomu üretmek için iki katıldı.
Biz iki tam uzunlukta virüs genleri varlığını doğrulamak için karşılıklı bir iskele olarak DNA ve RNA sıralama tarafından üretilen virüs genomu uzunluğu contigs göre. 6,966 bir tam uzunlukta virüs genomu bp Canna sarı mottle ilişkili virüs 1 (CaYMAV-1) (Şekil 3A) geçici olarak seçildi. İkinci genom 7,385 yapıldı bp ve Alpinia purpurata (CaYMV-Ap01) (Şekil 3A) hastalık CaYMV bir türevi.
Son olarak, her virüs klon ~ 1000 bp parçası için tasarlanmış PCR astar differentially nüfusu 9 ticari türü temsil eden 227 canna bitkiler her iki genleri tespit etmek için kullanılmıştır. Birçok durumda bireysel bitkiler ile her iki virüs bulaşmış. Biz 12 bitkilerde CaYMAV-1 ve CaYMV-Ap01 RT-PCR tespiti için bir örnek sağlar. Bunlardan üçüydü sadece CaYMV-Ap01 için olumlu ve dokuz her iki virüs (Şekil 3B) olumlu idi.

Resim 1 : Virüs nükleik asit ürünleri ve NGS iş akışı. (A)özel (% 1.0) Jel Elektroforez, 565 bp PCR parçacıkları CaYMV genleri. İki PCR ürünleri Beyaz Pelet (şerit 1, 2) hazırlanan örnekleri ama yeşil Pelet örnek (lane 3) tespit edildi. Pozitif kontrol (+) virüslü bitki standart paramagnetic selüloz parçacıkları içeren otomatik bir yöntem kullanarak izole DNA güçlendirilmiş bir PCR ürünü temsil eder. Lane L örnek yolları doğrusal DNA bantlarında boyutunu ölçmek için standart olarak kullanılan DNA merdiven içerir. (B) örneği virüs parçacık transmisyon elektron mikroskobu içinde virüslü canna yaprakları ham ayırma tarafından kurtarılan beyaz pelet tarafından görüldü. (C) özel (% 0.8) yeşil (lane 1) kurtarılan DNA'ın Elektroforez jel ve beyaz (lane 2) A. panelinde o test pozitif-PCR tarafından granül Lane 2 yanındaki kırmızı ve sarı nokta beyaz kesir ortaya iki yüksek molekül ağırlıklı DNA grup tanımlayın. (D) özel (% 1) jel elektroforez toplam RNA sütunlara göre RNA arıtma tarafından kurtarıldı. Lane L örnek Lanes doğrusal bantları boyutunu ölçmek için standart olarak kullanılan DNA merdiven içerir. Lane 1-6 dan havuza alınmış ribo tükenmesi ve nükleik asit izolasyonları, Kütüphane hazırlık, sıralama, dokunma derleme ve virüs genomu RNA-devamı (E) şematik boru hattı için tek bir örnek için virüslü canna yaprakları izole RNA içerir bir keşif. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
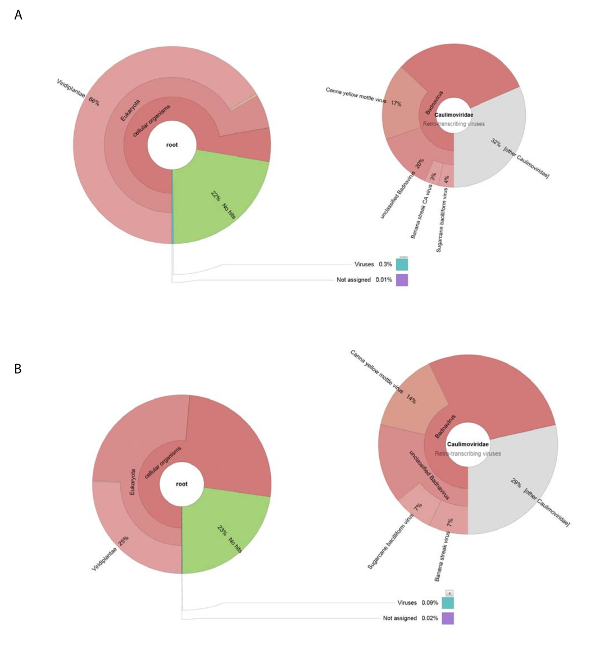
Resim 2 : Contigs taksonomik kategorilerini görselleştirme Kronu grafikler. (A) grafik üzerinde bereket ve contigs taksonomik dağıtımı ham virüs hazırlık monte sol gösterir. Doğru grafik Caulimoviridae Aile, Badnavirus cins ve üç yakından ilişkili tür ile ilişkili virüs contigs oranlarını gösteriyor. (B) sol panelde RNA-seq türetilmiş contigs bolluk taksonomik dağılımı üzerinde merkezli gösterir. Sağ tarafta contigs Caulimoviridae Aile, Badnavirus cins ve üç yakından ilişkili tür ile ilişkili virüs contigs nüfusu içinde bolluk gösteren grafiktir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
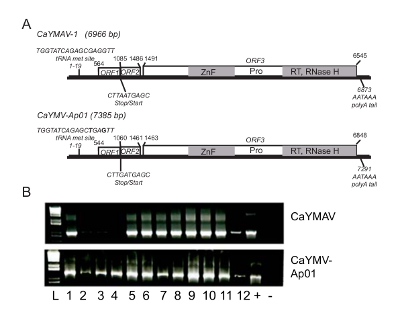
Şekil 3 . CaYMAV-1 ve CaYMV-Ap01 genleri karakterizasyonu. Canna sarı mottle ilişkilendirmek virüs 1 (CaYMAV) ve Canna sarı mottle virüs --dan izole genom benzer grafiksel gösterimi(a) Alpinia purpurata (CaYMV-Ap01). Nükleotit konumları 1-10 genom başlangıç olarak tanımlanır ve bir tRNAtanışmadan antikodon sitesi en badnavirus genleri tipik içerir. Açık okuma çerçevesi (ORF) 1 ve 2 çeviri için durdurma ve başlangıç pozisyonları bitişiktir. Bu proteinler bilinmeyen var işlevleri. ORF3 çinko parmak (ZnF), proteaz (Pro), ters transkriptaz (RT) ve RNAse H etki alanlarını içeren bir polyprotein var. 3' Poli(a) sinyal sıra her iki virüs genleri için korunmuş. (B) RT-PCR analiz virüs bulaşmış yaprakları ve CaYMAV ve CaYMV-Ap01 tespit astar izole RNA kullanılarak gerçekleştirildi. Kalan enfekte ise CaYMAV ve CaYMV-Ap01 ile 12 bitkiler aynı topluluk içinde CaYMV-Ap01 ile üç sadece, bulaşmış. (+) pozitif kontrol gösterir ve (-) negatif kontrol gösterir. Bu rakam çoğaltılamaz/değiştirilmiş Wijayasekara vd. 24 iznine sahip. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Tartışmalar
Son yıllarda çeşitli yöntemler istihdam bitki virüs biyoçeşitlilik virüs benzeri parçacıklar (VIP) ya da virüs belirli RNA veya DNA2,3,44için zenginleştirici içeren doğal ortamlarda çalışmaya, 45,46 . Bu yöntemler NGS ve bioinformatic analiz tarafından takip edilmektedir. Bu çalışmanın amacı ekili fabrikasında ortak bir hastalık nedensel Ajan bulmaktı. Hastalık bu sigara zarflı bacilliform parçacıklar vardır ve yalnızca bir 565 bp parçası klonlanmış47olduğu bilinmeyen bir virüs sonucu olduğu bildirildi. Bu bilgi kuramsal olarak Caulimoviridaeaile içinde cins Badnavirus virüs atamak önceden araştırmacılar için yeterli oldu. Önceki raporlar canna lilyum canna mottle hastalığında bu çalışmada, özetlenen metagenomics yaklaşımı kullanarak tek bir badnavirus sonucuydu onaylanmadığına karar ise hastalık iki geçici badnavirus türler24tarafından neden olduğu belirledi. Böylece, biz şimdi durumlar belirleyebilir bir hastalık nedensel Ajan keşfetmek için bir metagenome yaklaşımı kullanmanın gücü olduğunu yerde birden fazla nedeni olabilir.
DNA ve RNA sıralama veri birleştirerek yaklaşımımız kapsamlı olduğunu ve ayrıca iki yaklaşım kullanarak sonuçları tutarlı sonuçlar vermiştir ve iki ilgili virüs varlığını doğruladı. Biz caulimoviruses yalıtım için değiştirilmiş bir yordam istihdam ve bu ilişkili virüs nükleik asitler için zenginleştirilmiş ve bu virüs kapsid içinde korunan bir örnek üretti. Bir hizmet laboratuar DNA sıralama dışarı taşımak için sözleşme imzalamıştı. Temel de novo sıralama için bu DNA polimeraz nükleotit bir DNA şablon iplik DNA sentezi sıralı döngüsü sırasında etiketli floresan içerir bir kavramdır. Contigs monte takip NGS tarafından virüs contigs tespit edilmiştir birkaç contigs üreten bir bioinformatic iş akışı içine sunuldu. Doğrulanmadı iki virüs genleri10,24,48,49,50 bioinformatic Analizi ribo tükenmiş RNA ürünleri alınan RNA-seq veri ile elde edildi. Bir ilginç sonuç dizileri nüfus DNA ve RNA sıralama tarafından kurtarılan öğrenmek için çıktı bu benzer viral sigara ve viral nükleik asitleri dağılımları sağlanan. DNA ve RNA sıralama için < %0,5 sıralarının virüs kökenli idi. Virüs dizileri 78-%82 Caulimoviridaeailesine ait nüfus içinde. DNA ve RNA sıralama üzerinden birleştirilmiş virüs contigs karşılaştırarak, iki monte genleri her iki veri kümelerini meydana geldiğini doğruladı.
Yeni virüs genleri tanımlamak için tek DNA sıralama kullanarak bir endişe badnavirus genom açık bir dairesel DNA olmasıdır. Süreksizliklerin genom içinde üst üste dizileri contigs genom derleme için engel mevcut olabilir tahmin edebilmiş. DNA sıralama sonuçlarının ilk sınav iki benzer virüs genleri ortaya koydu. Biz bu genleri de değil okudu bir türün genetik çeşitlilik temsil olan veya24aynı ortak bulaşmasını temsil iki tür bitki. Bu nedenle, toplu bioinformatic analiz NGS DNA ve RNA sıralamanın, tarafından elde edilen veri kümeleri iki tam uzunlukta genomları varlığı teyit etkin.
VIP ve nükleik asitler bitki homogenates metagenomic araştırmalar, DNA dan karnabahar mozaik virüsü (CaMV; bir caulimovirus)3kurtarmak için yordamlar dayalı çıkarma için alternatif bir Yöntem geliştiren başka bir rapordur. Bu yaklaşım Roman RNA ve DNA virüs olmayan ekili bitkiler serilerinde tespit edilmiştir. Doğal olarak enfekte bitkiler24VIP ayıklamak için türetilmiş adımları aksine bir hastalık ekili bitkiler nedensel Ajan keşfetmek için bu çalışmada kullanılan caulimovirus yalıtım yordamla elde adımlar vardır. Değiştirilmiş her iki yöntem başarısı caulimovirus yalıtım framework yordamı genel bitki virüs metagenomic çalışmalar için değerli bir başlangıç noktası olabileceğini düşündürmektedir.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Araştırma bilim ilerleme ve teknoloji uygulanan araştırma programı faz II AR 132-053-2 için Oklahoma Merkezi tarafından finanse edildi; ve tarım özel bitkileri Oklahoma departmanı tarafından hibe programı araştırma. Biz Dr HongJin Hwang ve OSU Biyoinformatik çekirdek tesisi olan NSF (EOS-0132534) ve NIH (2P20RR016478-04, 1P20RR16478-02 ve 5P20RR15564 / 03) gelen hibe tarafından desteklenen teşekkür ederim.
Malzemeler
| Name | Company | Catalog Number | Comments |
| NaH2PO4 | Sigma-Aldrich St. Louis MO | S5976 | Grinding buffer for virus purification |
| Na2HPO4 | Sigma-Aldrich | S0751 | Grinding buffer for virus purification |
| Na2SO3 | Thermo-Fisher Waltham, MA | 28790 | Grinding buffer for virus purification |
| urea | Thermo-Fisher | PB169-212 | Homogenate extraction |
| Triton X-100 | Sigma-Aldrich | X-100 | Homogenate extraction |
| Cheesecloth | VWR Radnor, PA | 21910-107 | Filter homogenate |
| Tris | Thermo-Fisher | BP152-5 | Pellet resuspension& DNA resuspension buffers |
| MgCl2 | Spectrum, Gardena, CA | M1035 | Pellet resuspension buffer |
| EDTA | Spectrum | E1045 | Stops enzyme reactions |
| Proteinase K | Thermo-Fisher | 25530 | DNA resuspension buffer |
| phenol:chloroform:isoamylalcohol | Sigma-Aldrich | P2069 | Dissolve virion proteins |
| DNAse I | Promega | M6101 | Degrade cellular DNA from extracts |
| 95% ethanol | Sigma-Aldrich | 6B-100 | Virus DNA precipitation |
| Laboratory blender | VWR | 58984-030 | Grind leaf samples |
| Floor model ultracentrifuge &Ti70 rotor | Beckman Coulter, Irving TX | A94471 | Separation of cellular extracts |
| Floor model centrifuge and JA-14 rotor | Beckman Coulter | 369001 | Separation of cellular extracts |
| Magnetic stir plate | VWR | 75876-022 | Mixing urea into samples overnight |
| Rubber policeman | VWR | 470104-462 | Dissolve virus pellet |
| 2100 bioanalyzer Instrument | Agilent Genomics, Santa Clare, CA | G2939BA | Sensitive detection of DNA and RNA quality and quantity |
| 2100 Bioanalyzer RNA-Picochip | 5067-1513 | Microfluidics chip used to move, stain and measure RNA quality in a 2100 Bioanalyzer | |
| 2100 Bioanalyzer DNA-High Sensitive chip | 5067-4626 | Microfluidics chip used to move, stain and measure DNA quality in a 2100 Bioanalyzer | |
| Nanodrop spectrophotometer | Thermo-Fisher | ND-2000 | Analysis of DNA/RNA quality at intermediate steps of procedures |
| Plant total RNA isolation kit | Sigma-Aldrich | STRN50-1KT | Isolate RNA for RNA-seq |
| RNase-free water | VWR | 10128-514 | Resuspension of DNA and RNA for NGS |
| RNA concentrator spin column | Zymo Research, Irvine, CA | R1013 | Prepare RNA for RNA-seq |
| rRNA removal kit | Illumina, San Diego, CA | MRZPL116 | Prepare RNA for RNA-seq |
| DynaMag-2 Magnet | ThermoFisher | 12321D | Prepare RNA for RNA-seq |
| RNA enrichment system | Roche | 7277300001 | Prepare RNA for RNA-seq |
| Agarose | Thermo-Fisher | 16500100 | Gel analysis of DNA/RNA quality at intermediate steps of procedures |
| Ethidium bromide | Thermo-Fisher | 15585011 | Agarose gel staining |
| pGEM-T +JM109 competent cells | Promega, Madison, WI | A3610 | Clone genome fragments |
| pFU Taq polymerase | Promega | M7741 | PCR amplify virus genome |
| dNTPs | Promega | U1511 | PCR amplify virus genome |
| PCR oligonucleotides | IDT, Coralvill, IA | Custom order | PCR amplify virus genome |
| Miniprep DNA purification kit | Promega | A1330 | Plasmid DNA purification prior to sequencing |
| PCR clean-up kit | Promega | A9281 | Prepare PCR products for cloning |
| pDRAW32 software | ACAClone | Computer analysis of circular DNA and motifs | |
| MEGA6.0 software | MEGA | Molecular evolutionary genetics analysis | |
| Primer 3.0 | Simgene.com | ||
| Quant-iT™ RiboGreen™ RNA Assay Kit | Thermo-Fisher | R11490 | Fluorometric determination of RNA quantity |
| GS Junior™ pyrosequencing System | Roche | 5526337001 | Sequencing platform |
| GS Junior Titanium EmPCR Kit (Lib-A) | Roche | 5996520001 | Reagents for emulsion PCR |
| GS Jr EmPCR Bead Recovery Reagents | Roche | 5996490001 | Reagents for emulsion PCR |
| GS Junior EmPCR Reagents (Lib-A) | Roche | 5996538001 | Reagents for emulsion PCR |
| GS Jr EmPCR Oil & Breaking Kit | Roche | 5996511001 | Reagents for emulsion PCR |
| GS Jr Titanium Sequenicing kit* | Roche | 5996554001 | Includes sequencing reagents, enzymes, buffers, and packing beads |
| GS Jr. Titanium Picotiter Plate Kit | Roche | 5996619001 | Sequencing plate with associated reagents and gaskets |
| IKA Turrax mixer | 3646000 | Special mixer used with Turrax Tubes | |
| IKA Turrax Tube (specialized mixer) | 20003213 | Specialized mixing tubes with internal rotor for creating emulsions | |
| GS Nebulizers Kit | Roche | 5160570001 | Nucleic acid size fractionator for use during library preparations |
| GS Junior emPCR Bead Counter | Roche | 05 996 635 001 | Library bead counter |
| GS Junior Bead Deposition Device | Roche | 05 996 473 001 | Holder for Picotiter plate during centrifugation |
| Counterweight & Adaptor for the Bead Deposition Devices | Roche | 05 889 103 001 | Used to balance deposition device with picotiter plate centrifugation |
| GS Junior Software | Roche | 05 996 643 001 | Software suite for controlling the instrument, collecting and analyzing data |
| GS Junior Sequencer Control v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Run Processor v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS De Novo Assembler v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Reference Mapper v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Amplicon Variant Analyzer v. 3.0 | Roche | (Included in item 05 996 643 001 above) |
Referanslar
- Dijkstra, J., Jager, C. P. Practical Plant Virology : Protocols and Exercises. , Springer-Verlag. Berlin Heidelberg. 1 edn (1998).
- Roossinck, M. J. Plant virus metagenomics: biodiversity and ecology. Annu Rev Genet. 46, 359-369 (2012).
- Melcher, U., et al. Evidence for novel viruses by analysis of nucleic acids in virus-like particle fractions from Ambrosia psilostachya. J Virol Methods. 152 (1-2), 49-55 (2008).
- Stobbe, A. H., Schneider, W. L., Hoyt, P. R., Melcher, U. Screening metagenomic data for viruses using the e-probe diagnostic nucleic Acid assay. Phytopathology. 104 (10), 1125-1129 (2014).
- Borah, B. K., et al. Bacilliform DNA-containing plant viruses in the tropics: commonalities within a genetically diverse group. Mol Plant Pathol. 14 (8), 759-771 (2013).
- Bousalem, M., Douzery, E. J., Seal, S. E. Taxonomy, molecular phylogeny and evolution of plant reverse transcribing viruses (family Caulimoviridae) inferred from full-length genome and reverse transcriptase sequences. Arch Virol. 153 (6), 1085-1102 (2008).
- Geering, A. D., et al. Banana contains a diverse array of endogenous badnaviruses. J Gen Virol. 86, Pt 2 511-520 (2005).
- Kunii, M., et al. Reconstruction of putative DNA virus from endogenous rice tungro bacilliform virus-like sequences in the rice genome: implications for integration and evolution. BMC Genomics. 5, 80(2004).
- Laney, A. G., Hassan, M., Tzanetakis, I. E. An integrated badnavirus is prevalent in Figure germplasm. Phytopathology. 102 (12), 1182-1189 (2012).
- Gambley, C. F., Geering, A. D., Steele, V., Thomas, J. E. Identification of viral and non-viral reverse transcribing elements in pineapple (Ananas comosus), including members of two new badnavirus species. Arch Virol. 153 (8), 1599-1604 (2008).
- Gayral, P., et al. A single Banana streak virus integration event in the banana genome as the origin of infectious endogenous pararetrovirus. J Virol. 82 (13), 6697-6710 (2008).
- Lyttle, D. J., Orlovich, D. A., Guy, P. L. Detection and analysis of endogenous badnaviruses in the New Zealand flora. AoB Plants. 2011, 008(2011).
- Le Provost, G., Iskra-Caruana, M. L., Acina, I., Teycheney, P. Y. Improved detection of episomal Banana streak viruses by multiplex immunocapture PCR. J Virol Methods. 137 (1), 7-13 (2006).
- Singh, K., Talla, A., Qiu, W. Small RNA profiling of virus-infected grapevines: evidences for virus infection-associated and variety-specific miRNAs. Funct Integr Genomics. 12 (4), 659-669 (2012).
- Alfson, K. J., Beadles, M. W., Griffiths, A. A new approach to determining whole viral genomic sequences including termini using a single deep sequencing run. J Virol Methods. 208, 1-5 (2014).
- Kreuze, J. F., et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses. Virology. 388 (1), 1-7 (2009).
- Zheng, Y., et al. VirusDetect: An automated pipeline for efficient virus discovery using deep sequencing of small RNAs. Virology. 500, 130-138 (2017).
- James, A. P., Geijskes, R. J., Dale, J. L., Harding, R. M. Molecular characterisation of six badnavirus species associated with leaf streak disease of banana in East Africa. Annals of Applied Biology. 158 (3), 346-353 (2011).
- Baranwal, V. K., Sharma, S. K., Khurana, D., Verma, R. Sequence analysis of shorter than genome length episomal Banana streak OL virus like sequences isolated from banana in India. Virus Genes. 48 (1), 120-127 (2014).
- Sukal, A., Kidanemariam, D., Dale, J., James, A., Harding, R. Characterization of badnaviruses infecting Dioscorea spp. in the Pacific reveals two putative novel species and the first report of dioscorea bacilliform RT virus 2. Virus Res. 238, 29-34 (2017).
- BÖmer, M., Turaki, A. A., Silva, G., Kumar, P. L., Seal, S. E. A sequence-independent strategy for amplification and characterisation of episomal badnavirus sequences reveals three previously uncharacterised yam badnaviruses. Viruses. 8 (7), (2016).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Canna yellow mottle virus detected in Canna in Florida. Plant Health Progress. , August 2-4 (2004).
- Zhang, J., et al. Characterization of Canna yellow mottle virus in a new host, Alpinia purpurata, in Hawaii. Phytopathology. 107 (6), 791-799 (2017).
- Wijayasekara, D., et al. Molecular characterization of two badnavirus genomes associated with Canna yellow mottle disease. Virus Res. 243, 19-24 (2018).
- Covey, S. N., Noad, R. J., al-Kaff, N. S., Turner, D. S. Caulimovirus isolation and DNA extraction. Methods Mol Biol. 81, 53-63 (1998).
- Sambrook, J., Fritsch, E. F., Maniatis, T. Molecular cloning: A laboratory manual. 2nd edn. , Cold Spring Harbor Press. (1989).
- Radford, A. D., et al. Application of next-generation sequencing technologies in virology. J Gen Virol. 93, Pt 9 1853-1868 (2012).
- Kanagal-Shamanna, R. Emulsion PCR: Techniques and Applications. Methods Mol Biol. 1392, 33-42 (2016).
- Getts, D. R., et al. Targeted blockade in lethal West Nile virus encephalitis indicates a crucial role for very late antigen (VLA)-4-dependent recruitment of nitric oxide-producing macrophages. J Neuroinflammation. 9, 246(2012).
- van Dijk, E. L., Jaszczyszyn, Y., Thermes, C. Library preparation methods for next-generation sequencing: tone down the bias. Exp Cell Res. 322 (1), 12-20 (2014).
- Gel filtration principles and methods. GE Healthcare. , (2010).
- Goff, S., et al. The iPlant Collaborative: Cyberinfrastructure for Plant Biology. Frontiers in Plant Science. 2, (2011).
- Lin, Z., et al. Next-generation sequencing and bioinformatic approaches to detect and analyze influenza virus in ferrets. J Infect Dev Ctries. 8 (4), 498-509 (2014).
- Artimo, P., et al. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Res. 40, Web Server issue 597-603 (2012).
- Edgar, R. C. MUSCLE: a multiple sequence alignment method with reduced time and space complexity. BMC Bioinformatics. 5, 113(2004).
- Hung, J. H., Weng, Z. Sequence Alignment and Homology Search with BLAST and ClustalW. Cold Spring Harb Protoc. 2016 (11), (2016).
- Sohpal, V. K., Dey, A., Singh, A. MEGA biocentric software for sequence and phylogenetic analysis: a review. Int J Bioinform Res Appl. 6 (3), 230-240 (2010).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Res. 40 (15), 115(2012).
- Dhaliwa, A. DNA extraction and purification. Mater Methods. 3, 191(2013).
- Moeller, J. R., Moehn, N. R., Waller, D. M., Givnish, T. J. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Moeller, J. R., et al. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Grooms, K. Review: Improved DNA Yield and Quality from Diverse Plant Taxa. , (2015).
- Nishimori, A., et al. In vitro and in vivo antivirus activity of an anti-programmed death-ligand 1 (PD-L1) rat-bovine chimeric antibody against bovine leukemia virus infection. PLoS One. 12 (4), 0174916(2017).
- Rojas, M. R., Gilbertson, R. L. Plant Virus Evolution. Roossinck, M. J. 1, Springer-Verlag. 27-51 (2008).
- Roossinck, M. J. The big unknown: plant virus biodiversity. Curr Opin Virol. 1 (1), 63-67 (2011).
- Roossinck, M. J., Martin, D. P., Roumagnac, P. Plant Virus Metagenomics: Advances in Virus Discovery. Phytopathology. 105 (6), 716-727 (2015).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Plant Health Progress. , Online (2004).
- Eni, A., Hughes, J. D., Asiedu, R., Rey, M. Sequence diversity among badnavirus isolates infecting yam (Dioscorea spp.). Archives of Virology. 153 (12), Ghana, Togo, Benin and Nigeria. 2263-2272 (2008).
- Harper, G., et al. The diversity of Banana streak virus isolates in Uganda. Arch Virol. 150 (12), 2407-2420 (2005).
- Muller, E., Sackey, S. Molecular variability analysis of five new complete cacao swollen shoot virus genomic sequences. Arch Virol. 150 (1), 53-66 (2005).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır