0:01
Concepts
3:25
Immunoprecipitation Using Protein A/G Plus Agarose Beads
8:18
IP Verification Through Western Blot Analysis
12:36
Results
면역침전반응 기반 기술: 아가로스 비즈를 사용한 내인성 단백질의 정제
출처: 수잔나 C. 시슬러1,토냐 J. 웹1
1 미생물학 및 면역학학과, 메릴랜드 대학교 볼티모어, MD 21201
면역 침전물(IP, '풀다운' 분석이라고도 함)은 다양한 분야에서 응용 분야를 가지고 있는 널리 사용되는 기술이다. 1984년에 처음 구상된 이 요리는 1988년(1, 2)에 정제되었다. IP의 근본적인 목표는 그 단백질에 대하여 항체를 사용하여 특정 단백질의 정제 그리고 격리입니다. 단어 "면역"은 "강수량"이라는 단어가 용액에서 특정 물질을 당기는 것을 의미하는 동안 항체의 사용을 지칭한다. 표적 단백질은 내인성 또는 재조합일 지도 모릅니다. 대부분의 재조합 단백질에는 후속 정화를 단순화하기 위해 에피토프 태그(즉, myc 또는 flag)가 부착되어 있습니다. 전형적으로, 재조합 에피토프 태그에 대한 항체가 매우 강하고 효과적이기 때문에 재조합 단백질 IP를 최적화하는 것이 더 쉽습니다. 내인 성 단백질에 대한 항체는 매우 가변적 인 효능을 가지고 있어 이러한 IP를 최적화하기가 훨씬 더 어려워지게됩니다. 면역 침전 후 필요한 단계는 정화의 검증이다. 격리된 단백질은 SDS-PAGE를 사용하여 해결되고 그 후 서양 얼룩에 의한 순도를 위해 조사됩니다(그림 1). 중요한 대조군은 정확한 단백질의 당김을 확인하기 위해 서양 블롯 동안 다른 항체를 사용하는 것이다. IP와 후속 기술의 조합은 강력한 분석 도구입니다. 정제 후의 목표는 NMR, 질량 분광법 및 시험관 내 분석, 또는 단백질의 상호 작용 파트너(즉, 단백질, DNA, RNA)의 분석(즉, 단백질, DNA, RNA) (3, 4, 5)에 의해 단백질 자체의 특성화일 수 있다.
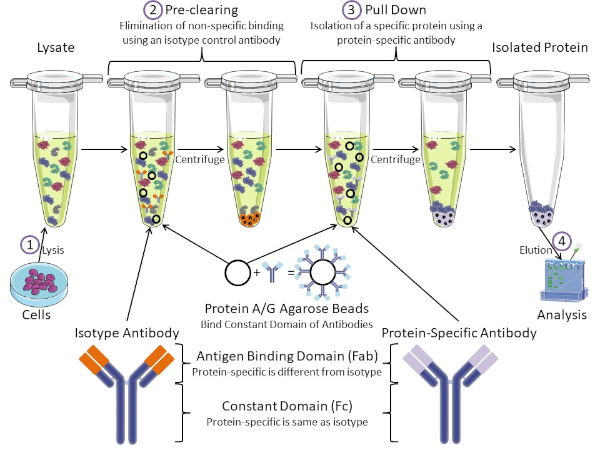
그림 1: 면역 침전 절차의 개요. 면역 침전은 항체를 사용하여 특정 단백질의 격리이다. 세포로부터 용재를 생산한 후, 두 가지 주요 단계-사전 정리 및 풀다운이 있습니다. 사전 클리어링 단계 동안, 세포-lysates는 isotype 대조군 항체를 사용하여 비특이적으로 항체에 결합하는 단백질의 미리 지워집니다. 당기면, 표적 단백질은 단백질 특이적 항체를 사용하여 아래로 당겨졌다. 고립 된 단백질은 서양 얼룩에 의해 분석됩니다. 동형 항체 및 단백질 특이적 항체는 동일한 상수 도메인을 가지지만, 상이한 항원 결합 도메인이 있다. 이 프로토콜의 핵심 성분은 항체의 일정한 도메인을 결합하는 단백질 A/G 아가로즈 구슬입니다 - 표적 단백질의 면역 침전을 허용합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
항체는 다른 형태의 단백질 정제(즉, 니켈 친화 컬럼 정제)와 구별되는 면역 침전의 핵심 성분이다. 항체는 특정 단백질 에피토프를 인식할 수 있는 B 세포에 의해 만들어진 분자입니다. 항체에는 상수(Fc) 및 항원 결합(Fab)(도 1)의 두 가지 영역이 있습니다. 상수 도메인은 항체의 유형을 식별하고 생체 내에서기능을 지시한다. 일반적으로 IP에 사용되는 항체의 상수 영역은 마우스, 쥐 또는 토끼 IgG입니다. 항체의 항원 결합 부는 특정 단백질의 특정 에피토프를 인식한다. 항체는 단백질이 변성될 때 존재하지 않을 수 있는 접힌 단백질에 대한 전형체를 인식할 수 있고 그 반대의 경우도 마찬가지입니다. 따라서, 에피토프의 가용성은 단백질 접이식에 달려 있습니다 - IP에 대한 항체 및 조건을 선택할 때 고려해야 할 중요한 요소를 식별합니다.
대핵계와 진핵계에는 항체 결합 단백질이 있습니다. 진핵 계통에서, 목적은 근생 계통에 있는 동안 박테리아로부터 면역 보호입니다, 목적은 면역 계통으로부터 보호입니다. 항체 결합 단백질은 두 가지 방법으로 IP 방법론에 영향을 미칩니다. 첫째, 항체를 결합하는 단백질의 용액을 제거하는 데 필요한 사전 정리 단계(도 1)가 있어 최종 제품에 비특이적 결합을 줄입니다. 이 단계는 단백질 특이적 항체와 는 다른 항체 결합 도메인과 동일한 상수 영역을 가지고 있는 동종형 항체를 사용합니다. 세균성 항체 결합 단백질은 이 방법의 두 번째 핵심 성분이다. 단백질 특이적 항체가 표적 단백질을 결합한 후, 항체: 단백질 복합체는 아래로 당겨져야 한다(도 1). 단백질 A, G 및 L은 항체의 일정한 도메인을 결합하는 세균성 단백질입니다. 박테리아는 면역 계통을 전복하기 위하여 이것을 사용하는 동안, 연구원은 쉬운 항체 정화를 위한 이 시스템을 공동 선택했습니다, 그리고 사전 정리 및 풀다운 단계 둘 다 도중 이용됩니다. 이 단백질은 다른 종 및 다른 일정한 도메인 하위형에 대한 상이한 결합 친화성을 가지고 있습니다 - IP를 위한 조건을 선택할 때 고려해야 할 또 다른 요인. 많은 회사에서 단백질 A/G 라벨아가로즈 구슬(그림 1), 미리 만들어진 스핀 컬럼 또는 수지를 판매하여 컬럼을 만듭니다. 일반적으로 구슬과 스핀 컬럼은 더 작은 샘플 크기에 사용되는 반면 수지는 대량 정화에 사용됩니다.
이 실험실 운동에서, 우리는 단백질 A/G 플러스 아가로즈 구슬 기지를 둔 기본적인 면역 침전 기술을 사용하여, 1 차적인 뮤린 흉구에서 내인성 단백질 c-myc를 정화하는 방법을 보여줍니다. 프로토콜은 세포 용해 준비에서 시작하고 서양 얼룩 분석을 사용하여 성공적인 단백질 풀다운의 검증으로 끝납니다.
위에 자세히 설명된 절차의 결과는 그림 2에 나와 있습니다. 왼쪽에서 오른쪽으로, 차선에는 대조군(isotype), 시험 군(c-myc), 미리 지워진 용해(lysate), 분자량 사다리(사다리)가 포함됩니다. 25 kDa 래더 밴드가 표시됩니다. ~25 kDa 및 50 kDa의 두 개의 눈에 띄는 밴드는 결합 항체의 빛과 무거운 사슬이며, 각각 IP 또는 샘플에 특이적이지 않습니다. c-myc 단백질은 서양 얼룩에서 약 6...
요컨대, 면역 침전은 항체를 이용한 특정 단백질의 분리이다. 이 예에서, 면역 침전의 결과는 순도를 평가하기 위해 서양 블롯에 의해 분석되었다. 고립 된 단백질은 단백질 구조를 위한 NMR, 아미노산 서열에 대한 질량 분광법 또는 효소 특성화를 위한 체외 분석 등 여러 가지 응용 분야에서 사용될 수 있습니다. IP는 또한 단백질의 상호 작용 파트너를 특성화할 수 있습니다. 예를 들면, 격리...
- Olliver, C. L. and Boyd, C. D. (1984). Immunoprecipitation of In Vitro Translation Products with Protein A Bound to Sepharose. In J. M. Walker (eds), Nucleic Acids. Methods in Molecular Biology (pp. 157-160). New Jersey: Humana Press.
- Thurston, C. F. and Henley, L. F. (1988). Direct Immunoprecipitation of Protein. In J. M. Walker (eds), New Protein Techniques. Methods in Molecular Biology (pp. 149-158). New Jersey: Humana Press.
- Anderson, N. G. (1998). Co-immunoprecipitation: Identification of Interacting Proteins. In R. A. Clegg (eds), Protein Targeting Protocols.Methods in Molecular Biology (pp. 35-45). New Jersey: Humana Press.
- Jackson, D. I. and Dickson, C. (1999). Protein Techniques: Immunoprecipitation, In Vitro Kinase Assays, and Western Blotting. In P.T. Sharpe and I. Mason (eds), Molecular Embryology. Methods in Molecular Biology (pp. 699-708). New Jersey: Humana Press.
- Trieu, E. P. and Targoff, I. N. (2015). Immunoprecipitation: Western Blot for Proteins of Low Abundance. In B. Kurien and R. Scofield (eds), Western Blotting. Methods in Molecular Biology (pp. 327-342). New York, NY: Humana Press.
Tags
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved