0:01
Concepts
3:25
Immunoprecipitation Using Protein A/G Plus Agarose Beads
8:18
IP Verification Through Western Blot Analysis
12:36
Results
Técnicas Baseadas em Imunoprecipitação: Purificação de Proteínas Endógenas Usando Esferas de Agarose
Fonte: Susannah C. Shissler1, Tonya J. Webb1
1 Departamento de Microbiologia e Imunologia, Universidade de Maryland, Baltimore, MD 21201
Imunoprecipitação (IP, também conhecido como ensaio 'pull-down') é uma técnica amplamente utilizada que tem aplicações em uma variedade de campos. Concebido pela primeira vez em 1984, foi refinado em 1988 (1, 2). O objetivo fundamental do IP é a purificação e isolamento de uma proteína específica usando um anticorpo contra essa proteína. A palavra "imuno" refere-se ao uso de um anticorpo, enquanto a palavra "precipitação" refere-se a retirar uma substância específica de uma solução. A proteína alvo pode ser endógena ou recombinante. A maioria das proteínas recombinantes tem uma tag de epítope (ou seja, myc ou flag) anexada a elas para simplificar a purificação subsequente. Normalmente, é mais fácil otimizar a proteína recombinante IP porque os anticorpos contra tags de epitope recombinantes são muito fortes e eficazes. Anticorpos contra proteínas endógenas têm eficácia extremamente variável - tornando muito mais difícil otimizar esses IPs. Um passo necessário após a imunoprecipitação é a verificação da purificação. A proteína isolada é resolvida usando SDS-PAGE e posteriormente sondada para pureza por manchas ocidentais (Figura 1). Um controle importante é o uso de um anticorpo diferente durante a mancha ocidental para verificar a retirada da proteína correta. A combinação de IP com técnicas subsequentes é uma poderosa ferramenta de análise. O objetivo após a purificação pode ser a caracterização da proteína em si por NMR, espectrometria de massa e ensaios in vitro, ou análise dos parceiros interativos da proteína (ou seja, proteína, DNA, RNA) (3, 4, 5).
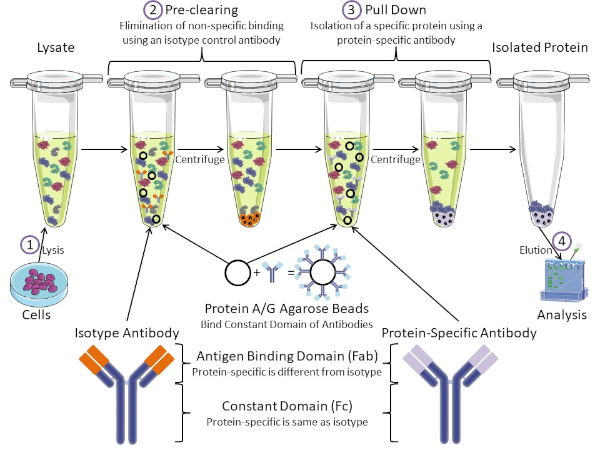
Figura 1: Visão geral do Procedimento de Imunoprecipitação. Imunoprecipitação é o isolamento de uma proteína específica usando um anticorpo. Após a produção de lise das células, há dois passos principais: pré-limpar e puxar para baixo. Durante a etapa de pré-limpeza, os lysatos celulares são pré-eliminados de proteínas que se ligam a anticorpos não especificamente usando um anticorpo de controle de isótipo. Em pull down step, a proteína alvo é puxada para baixo usando um anticorpo específico de proteína. A proteína isolada é então analisada pela mancha ocidental. Anticorpos isótipos e anticorpos específicos de proteína têm o mesmo domínio constante, mas diferentes domínios de ligação de antígeno. Um componente-chave deste protocolo são as contas de agarose da Proteína A/G que ligam o domínio constante dos anticorpos, permitindo a imunoprecipitação da proteína alvo. Clique aqui para ver uma versão maior desta figura.
Os anticorpos são o componente-chave de uma imunoprecipitação que o diferencia de outras formas de purificação de proteínas (ou seja, purificação da coluna de afinidade de níquel). Anticorpos são moléculas feitas por células B que podem reconhecer epítopos proteicos específicos. Os anticorpos possuem dois domínios: ligação constante (Fc) e antígeno (Fab) (Figura 1). O domínio constante identifica o tipo de anticorpo e dita a função in vivo. Normalmente, os domínios constantes dos anticorpos usados para IP são mouse, rat ou rabbit IgG. A porção de ligação de antígeno do anticorpo reconhece um epítope específico de uma proteína específica. Anticorpos podem reconhecer epítopos em proteínas dobradas que podem não existir quando a proteína é desnaturada e vice-versa. Portanto, a disponibilidade do epítope depende da dobra de proteínas - identificando um fator importante a considerar na escolha de anticorpos e condições para IP.
Ambos os sistemas procarióticos e eucarióticos têm proteínas de ligação de anticorpos. Nos sistemas eucarióticos, o objetivo é a proteção imune contra bactérias enquanto em sistemas procarióticos, o objetivo é a proteção do sistema imunológico. Proteínas de ligação de anticorpos afetam a metodologia IP de duas maneiras. Em primeiro lugar, há uma etapa de pré-compensação necessária (Figura 1) para livrar o lise de proteínas que ligam anticorpos - reduzindo assim a vinculação não específica no produto final. Esta etapa usa um anticorpo isótipo que tem o mesmo domínio constante que, mas um domínio de ligação de anticorpos diferente do seu anticorpo específico para proteínas. Proteínas bacterianas de ligação de anticorpos são o segundo componente-chave deste método. Depois que o anticorpo específico da proteína liga a proteína alvo, o anticorpo: complexo proteico deve ser puxado para baixo (Figura 1). Proteínas A, G e L são proteínas bacterianas que ligam o domínio constante dos anticorpos. Embora as bactérias usem isso para subverter o sistema imunológico, os pesquisadores têm cooptado este sistema para uma fácil purificação de anticorpos, e ele é usado durante os passos pré-clareira e pull-down. Essas proteínas têm diferentes afinidades de ligação para diferentes espécies e diferentes subtipos de domínio constantes - outro fator a considerar ao escolher condições para IP. Muitas empresas vendem contas de agarose rotuladas protein A/G (Figura 1), colunas de spin pré-feitas ou resinas para fazer colunas. Em geral, contas e colunas de spin são usadas para tamanhos amostrais menores, enquanto resinas são usadas para purificação a granel.
Neste exercício de laboratório, demonstramos como purificar a proteína endógena c-myc, dos timócitos murinas primários, utilizando contas de proteína A/G Plus baseadas em imunoprecipitação básica. O protocolo começa a partir da preparação de lisecelulares e termina com a verificação de retirada de proteínas bem sucedidas usando a análise de manchas ocidentais.
1. Imunoprecipitação usando contas de agarose de proteína agarose
Preparação de lysato celular
- Centrifugar 108 timócitos em uma microcentrifuuagem a 13.000 rpm por 3 min e remover o supernatante.
Nota: O número celular variará dependendo dos níveis de expressão da proteína desejada e do tipo celular escolhido. - Suspenda-as as células em RIPA tampão de 500 μL com PMSF.
- Interromp...
Os resultados do procedimento detalhado acima são mostrados na Figura 2. Da esquerda para a direita, as pistas contêm o grupo controle (isótipo), o grupo de teste (c-myc), o liseto pré-limpo (lysate) e a escada de peso molecular (escada). As bandas de escada de 25 e 75 kDa estão marcadas. As duas bandas proeminentes a ~25 kDa e 50 kDa são a cadeia leve e pesada do anticorpo de ligação, respectivamente e não são específicas para o IP ou as amostras. proteína c-myc que corre em ...
Em suma, a imunoprecipitação é o isolamento de uma proteína específica usando um anticorpo. Neste exemplo, os resultados da imunoprecipitação foram analisados pela mancha ocidental para avaliar a pureza. A proteína isolada poderia ser usada em uma série de aplicações posteriores, incluindo: NMR para estrutura proteica, Espectrometria de Massa para sequência de aminoácidos, ou ensaios in vitro para caracterização enzimática. Os IPs também podem caracterizar os parceiros interajantes das proteína...
- Olliver, C. L. and Boyd, C. D. (1984). Immunoprecipitation of In Vitro Translation Products with Protein A Bound to Sepharose. In J. M. Walker (eds), Nucleic Acids. Methods in Molecular Biology (pp. 157-160). New Jersey: Humana Press.
- Thurston, C. F. and Henley, L. F. (1988). Direct Immunoprecipitation of Protein. In J. M. Walker (eds), New Protein Techniques. Methods in Molecular Biology (pp. 149-158). New Jersey: Humana Press.
- Anderson, N. G. (1998). Co-immunoprecipitation: Identification of Interacting Proteins. In R. A. Clegg (eds), Protein Targeting Protocols.Methods in Molecular Biology (pp. 35-45). New Jersey: Humana Press.
- Jackson, D. I. and Dickson, C. (1999). Protein Techniques: Immunoprecipitation, In Vitro Kinase Assays, and Western Blotting. In P.T. Sharpe and I. Mason (eds), Molecular Embryology. Methods in Molecular Biology (pp. 699-708). New Jersey: Humana Press.
- Trieu, E. P. and Targoff, I. N. (2015). Immunoprecipitation: Western Blot for Proteins of Low Abundance. In B. Kurien and R. Scofield (eds), Western Blotting. Methods in Molecular Biology (pp. 327-342). New York, NY: Humana Press.
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved