0:01
Concepts
3:27
Preparation of Media and Plates
6:09
Streak Plating
8:28
Results and Data Analysis
纯培养和条纹电镀:从混合样品中分离单一细菌菌落
资料来源:蒂尔德·安徒森1号,罗尔夫·卢德1
1临床科学隆德系,感染医学系,生物医学中心,隆德大学,221 00 隆德,瑞典
似乎无法确定,微生物生物多样性确实令人震惊,估计有一万亿种物种并存(1,2)。虽然气候特别恶劣,如人类胃的酸性环境(3)或南极洲的冰下湖泊(4),可能由特定物种主导,但细菌通常存在于混合培养物中。由于每种菌株都可能影响另一种(5)的生长,分离和培养"纯"(仅包含一种类型)菌落的能力在临床和学术环境中都变得至关重要。纯培养能进一步进行遗传(6)和蛋白细胞学检查(7),分析样品纯度,也许更值得注意的是,从临床样本中鉴定和鉴定传染性病原体。
细菌具有广泛的生长要求,有许多类型的营养介质,旨在维持不苛求和挑剔的物种 (8)。生长介质可以以液体形式(作为肉汤)或以典型的琼脂基(一种从红藻中提取的凝胶剂)固体形式制备。直接接种到肉汤有产生基因多样性甚至混合细菌群的风险,电镀和再循环创造了一种更纯净的培养,每个细胞都有高度相似的遗传组成。条纹板技术基于样品的逐行稀释(图1),目的是将单个细胞彼此分离。任何由介质和指定环境维持的存活细胞(以下简称细胞形成单元,CFU)随后可以通过二元裂变找到孤立的子细胞群落。尽管细菌群落内突变率很快,但通常认为该细胞群为克隆细胞。因此,采集和重新采集这一种群可确保后续工作只涉及一种细菌类型。
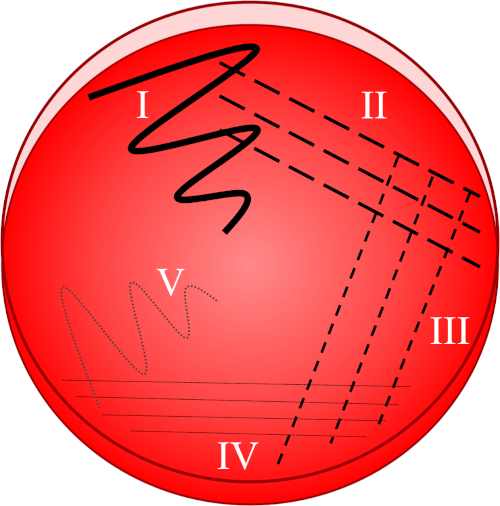
图1:条纹板基于原始样品的逐行稀释。I)接种最初使用锯齿形运动进行分散,从而形成细菌相对密集的区域。II-IV)条纹从前面的区域绘制,每次使用无菌接种循环,直到达到第四个象限。V)最后一个指向板块中间的锯齿状运动形成了一个区域,其中接种已明显稀释,使菌落彼此分开出现。
条纹板技术也可以与选择性和/或差分介质的使用相结合。选择性培养基会抑制某些生物体的生长(例如通过添加抗生素),而差别培养基将仅有助于区分另一种(例如,通过血液琼脂板上的溶血)。
微生物学的所有工作的基础是使用无菌(无菌)技术。每种细菌培养物都应被视为潜在的致病性,因为存在危险菌株意外生长、气溶胶形成和设备/人员污染的风险。为了尽量减少这些风险,所有介质、塑料、金属和玻璃器皿通常在使用前后通过高压灭菌进行灭菌,使其在 121°C 左右受到高压饱和蒸汽的影响,从而有效清除任何残留的细胞。工作空间通常在使用之前和之后使用乙醇进行消毒。在与传染性病原体一起工作时,始终佩戴实验室外套和手套。
- The Human Microbiome Project C. Structure, Function and Diversity of the Healthy Human Microbiome. Nature. 486:207-214. (2012)
- Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences. 113 (21) 5970-5975 (2016)
- Skouloubris S, Thiberge JM, Labigne A, De Reuse H. The Helicobacter pylori UreI protein is not involved in urease activity but is essential for bacterial survival in vivo. Infection and Immunity. 66:4517-21. (1998)
- Mikucki JA, Auken E, Tulaczyk S, Virginia RA, Schamper C, Sørensen KI, Doran PT, Dugan H, Foley N. Deep groundwater and potential subsurface habitats beneath an Antarctic dry valley. Nature Communications. 6:6831. (2015)
- Mullineaux-Sanders C, Suez J, Elinav E, Frankel G. Sieving through gut models of colonization resistance. Nature Microbiology. 3:132-140. (2018)
- Fournier PE, Drancourt M, Raoult D. Bacterial genome sequencing and its use in infectious diseases. Lancet Infectious Diseases. 7:711-23 (2007)
- Yao Z, Li W, Lin Y, Wu Q, Yu F, Lin W, Lin X. Proteomic Analysis Reveals That Metabolic Flows Affect the Susceptibility of Aeromonas hydrophila to Antibiotics. Scientific Reports. 6:39413 (2016)
- Medina D, Walke JB, Gajewski Z, Becker MH, Swartwout MC, Belden LK. Culture Media and Individual Hosts Affect the Recovery of Culturable Bacterial Diversity from Amphibian Skin. Frontiers in Microbiology. 8:1574 (2017)
Tags
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved