0:01
Concepts
3:27
Preparation of Media and Plates
6:09
Streak Plating
8:28
Results and Data Analysis
Reinkulturen und Streak-Plating: Isolierung einzelner Bakterienkolonien aus einer gemischten Probe
Quelle: Tilde Andersson1, Rolf Lood1
1 Institut für Klinische Wissenschaften Lund, Abteilung für Infektionsmedizin, Biomedical Center, Universität Lund, 221 00 Lund, Schweden
Die mikrobielle Biodiversität scheint unmöglich zu bestimmen, und die mikrobielle Artenvielfalt ist mit schätzungsweise einer Billion koexistierender Arten (1,2) wirklich erstaunlich. Obwohl besonders raue Klimazonen, wie die saure Umgebung des menschlichen Magens (3) oder die subglazialen Seen der Antarktis (4), von einer bestimmten Art dominiert werden können, sind Bakterien typischerweise in Mischkulturen zu finden. Da jede Sorte das Wachstum einer anderen (5) beeinflussen kann, ist die Fähigkeit, "reine" (nur aus einem Typ bestehende) Kolonien zu trennen und zu kultivieren, sowohl im klinischen als auch im akademischen Umfeld unerlässlich geworden. Reine Kulturen ermöglichen weitere genetische (6) und proteomische Untersuchungen (7), die Analyse der Probenreinheit und, vielleicht bemerkenswerter, die Identifizierung und Charakterisierung von Infektionserregern aus klinischen Proben.
Bakterien haben eine breite Palette von Wachstumsanforderungen und es gibt zahlreiche Arten von Nährstoffmedien, die entwickelt wurden, um sowohl die unanspruchsvollen als auch die anspruchsvollen Arten zu erhalten (8). Wachstumsmedien können entweder in flüssiger Form (als Brühe) oder in einer typisch engarbasierten (einem gelierenden Mittel aus Rotalgen) feste Form hergestellt werden. Während die direkte Impfung in die Brühe das Risiko birgt, eine genetisch vielfältige oder sogar gemischte Bakterienpopulation zu erzeugen, schafft die Beschichtung und Neuung eine reinere Kultur, in der jede Zelle ein sehr ähnliches genetisches Make-up hat. Die Streifenplattentechnik basiert auf der progressiven Verdünnung einer Probe (Abbildung 1), mit dem Ziel, einzelne Zellen voneinander zu trennen. Jede lebensfähige Zelle (im Folgenden als koloniebildende Einheit, KBE bezeichnet), die von den Medien und der bezeichneten Umgebung getragen wird, kann anschließend eine isolierte Kolonie von Tochterzellen durch binäre Spaltung finden. Trotz der schnellen Mutationsraten innerhalb bakterieller Gemeinschaften wird diese Zellgruppe allgemein als klonal betrachtet. Die Ernte und Erneute Verwesung dieser Population stellt somit sicher, dass die nachfolgende Arbeit nur einen einzigen bakteriellen Typ umfasst.
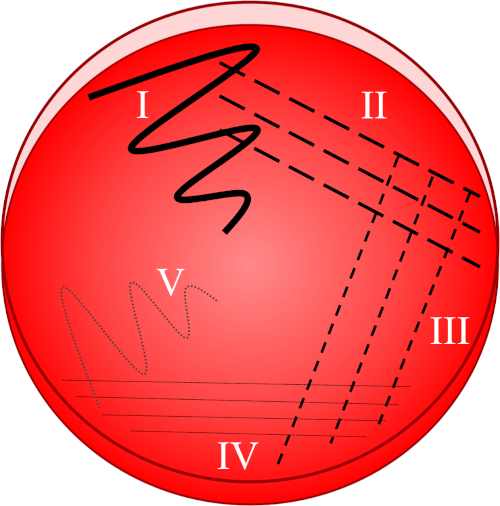
Abbildung 1: Eine Streifenplatte basiert auf der progressiven Verdünnung der Originalprobe. I) Das Inokulum wird zunächst mit einer Zick-Zack-Bewegung verteilt, wodurch ein Gebiet mit einer relativ dichten Bakterienpopulation entsteht. II-IV) Streifen werden aus dem vorhergehenden Bereich gezogen, wobei jedes Mal eine sterile Impfschleife verwendet wird, bis der vierte Quadrant erreicht ist. V) Eine letzte Zick-Zack-Bewegung, die auf die Mitte der Platte gerichtet ist, bildet einen Bereich, in dem das Inokulum deutlich verdünnt wurde, so dass Kolonien getrennt voneinander erscheinen können.
Die Streifenplattentechnik kann auch mit dem Einsatz selektiver und/oder differentialer Medien kombiniert werden. Ein selektives Medium hemmt das Wachstum bestimmter Organismen(z. B. durch Zugabe von Antibiotika), während ein Differenzmedium nur dazu beiträgt, sich voneinander zu unterscheiden(z. B. durch Hämolyse auf Blutagarplatten).
Allen Arbeiten in der Mikrobiologie liegt der Einsatz aseptischer (steriler) Techniken zugrunde. Jede Bakterienkultur sollte als potenziell pathogen betrachtet werden, da die Gefahr eines unbeabsichtigten Wachstums tückischer Stämme, Aerosolbildung und Kontamination von Geräten/Personal besteht. Um diese Risiken zu minimieren, werden alle Medien-, Kunststoff-, Metall- und Glaswaren in der Regel durch Autoklavieren vor und nach dem Gebrauch sterilisiert und mit hochdruckgesättigtem Dampf bei etwa 121°C belastet, der alle verweilenden Zellen effektiv auslöscht. Der Arbeitsbereich wird in der Regel mit Ethanol sowohl vor als auch nach der Verwendung desinfiziert. Labormantel und Handschuhe werden immer bei der Arbeit mit Infektionserregern getragen.
1. Einrichten
- Alle Mikroben sollten so behandelt werden, als wären sie gefährlich. Tragen Sie immer einen Labormantel und Handschuhe, binden Sie lange Haare zurück und sorgen Sie dafür, dass alle Wunden besonders gut geschützt sind.
- Bereiten Sie den Arbeitsbereich bereit, indem Sie ihn mit 70% Ethanol sterilisieren.
- Stellen Sie sicher, dass Agarplatten, Probenlösung(en) und entweder eine Schachtel mit vorsterilisierten Kunststoff-Impfschlaufen oder eine Metallschlaufe ...
Die anfängliche Streifenplatte kann Kolonien enthalten, die aus Zellen mit unterschiedlicher genetischer Zusammensetzung oder (je nach Probenreinheit) von verschiedenen Bakterienarten stammen (Abbildung 2A).
Durch die nachfolgende Isolierung einer einzelnen Kolonie, bei der alle Einheiten aus einer gemeinsamen Mutterzelle abgeleitet sind, erzeugt das zweite Streifenverfahren eine r.
Die Fähigkeit, eine reine Bakterienkolonie zu erhalten und zu kultivieren, ist sowohl im klinischen als auch im akademischen Umfeld unerlässlich. Die Streak-Beschichtung ermöglicht die Isolierung einer relativ klonalen Zellpopulation, die aus einer gemeinsamen KBE stammt und während der Diagnose oder für eine zusätzliche Charakterisierung des Isolats von besonderem Interesse sein kann. Eine Probe wird auf ein geeignetes Nährmedium auf Agarbasis gestreift und inkubiert, bis Kolonien sichtbar werden. Eine isolierte ...
- The Human Microbiome Project C. Structure, Function and Diversity of the Healthy Human Microbiome. Nature. 486:207-214. (2012)
- Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences. 113 (21) 5970-5975 (2016)
- Skouloubris S, Thiberge JM, Labigne A, De Reuse H. The Helicobacter pylori UreI protein is not involved in urease activity but is essential for bacterial survival in vivo. Infection and Immunity. 66:4517-21. (1998)
- Mikucki JA, Auken E, Tulaczyk S, Virginia RA, Schamper C, Sørensen KI, Doran PT, Dugan H, Foley N. Deep groundwater and potential subsurface habitats beneath an Antarctic dry valley. Nature Communications. 6:6831. (2015)
- Mullineaux-Sanders C, Suez J, Elinav E, Frankel G. Sieving through gut models of colonization resistance. Nature Microbiology. 3:132-140. (2018)
- Fournier PE, Drancourt M, Raoult D. Bacterial genome sequencing and its use in infectious diseases. Lancet Infectious Diseases. 7:711-23 (2007)
- Yao Z, Li W, Lin Y, Wu Q, Yu F, Lin W, Lin X. Proteomic Analysis Reveals That Metabolic Flows Affect the Susceptibility of Aeromonas hydrophila to Antibiotics. Scientific Reports. 6:39413 (2016)
- Medina D, Walke JB, Gajewski Z, Becker MH, Swartwout MC, Belden LK. Culture Media and Individual Hosts Affect the Recovery of Culturable Bacterial Diversity from Amphibian Skin. Frontiers in Microbiology. 8:1574 (2017)
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved