0:01
Concepts
3:27
Preparation of Media and Plates
6:09
Streak Plating
8:28
Results and Data Analysis
Cultivos puros y siembra por estrías: aislamiento de colonias bacterianas únicas de una muestra mixta
Fuente: Tilde Andersson1, Rolf Lood1
1 Departamento de Ciencias Clínicas Lund, División de Medicina de Infecciones, Centro Biomédico, Universidad de Lund, 221 00 Lund, Suecia
Aparentemente imposible de determinar, la biodiversidad microbiana es realmente sorprendente con un billón de especies coexistentes (1,2). Aunque climas particularmente duros, como el ambiente ácido del estómago humano (3) o los lagos subglaciales de la Antártida (4), pueden estar dominados por una especie específica, las bacterias se encuentran típicamente en cultivos mixtos. Como cada cepa puede influir en el crecimiento de otra (5), la capacidad de separar y cultivar colonias "puras" (que consisten sólo en un tipo) las colonias se ha vuelto esencial tanto en entornos clínicos como académicos. Los cultivos puros permiten más exámenes genéticos (6) y proteómicos (7), análisis de la pureza de la muestra y, tal vez más notable, la identificación y caracterización de agentes infecciosos a partir de muestras clínicas.
Las bacterias tienen una amplia gama de requisitos de crecimiento y hay numerosos tipos de medios nutritivos diseñados para sostener tanto a las especies poco exigentes como a las fastidiosas (8). Los medios de crecimiento se pueden preparar en forma líquida (como caldo) o en una forma sólida típicamente a base de agar (un agente gelificante derivado de algas rojas). Mientras que la inoculación directa en el caldo conlleva el riesgo de generar una población bacteriana genéticamente diversa o incluso mixta, el revestimiento y el re-streaking crea un cultivo más puro donde cada célula tiene una composición genética muy similar. La técnica de la placa de rayas se basa en la dilución progresiva de una muestra(Figura 1),con el objetivo de separar las células individuales entre sí. Cualquier célula viable (en lo sucesivo, una unidad formadoa de colonias, CFU) sostenida por los medios de comunicación y el entorno designado puede posteriormente encontrar una colonia aislada de células hijas a través de la fisión binaria. A pesar de las rápidas tasas de mutación dentro de las comunidades bacterianas, este grupo celular es generalmente considerado como clonal. La recolección y re-streaking de esta población garantiza que el trabajo posterior implica sólo un solo tipo de bacteria.
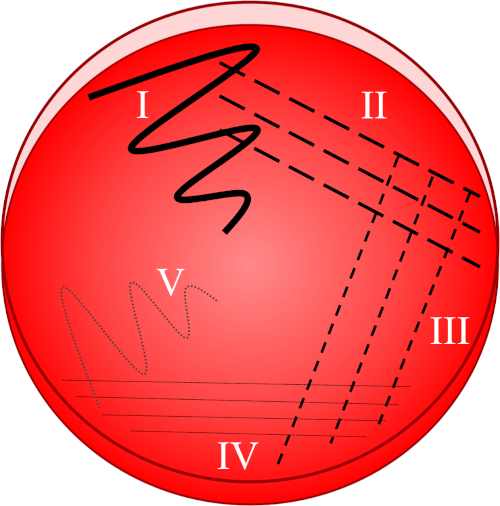
Figura 1: Una placa de rayas se basa en la dilución progresiva de la muestra original. I) El inóculo se dispersa inicialmente usando un movimiento en zig-zag, creando un área con una población bacteriana relativamente densa. II-IV) Las rayas se extraen del área anterior, utilizando un bucle de inoculación estéril cada vez, hasta que se alcanza el cuarto cuadrante. V) Un movimiento final en zig-zag dirigido hacia el centro de la placa forma una región donde el inóculo se ha diluido notablemente, permitiendo que las colonias aparezcan separadas unas de otras.
La técnica de la placa de rayas también se puede combinar con el uso de medios selectivos y/o diferenciales. Un medio selectivo inhibirá el crecimiento de ciertos organismos(por ejemplo, mediante la adición de antibióticos), mientras que un medio diferencial sólo ayudará a distinguirse unos de otros(por ejemplo, a través de la hemólisis en placas de agar en sangre).
Detrás de todo el trabajo en microbiología es el uso de técnicas asépticas (estériles). Cada cultivo bacteriano debe considerarse potencialmente patógeno, ya que existe un riesgo de crecimiento involuntario de cepas traicioneras, formación de aerosoles y contaminación de equipos/personal. Para minimizar estos riesgos, todos los medios, plásticos, metales y vidrio-ware se esterilizan típicamente a través de autoclave antes y después de su uso, sometiéndolos a vapor saturado de alta presión a alrededor de 121 oC que elimina eficazmente las células persistentes. El espacio de trabajo generalmente se desinfecta con etanol antes y después del uso. La capa de laboratorio y los guantes siempre se usan durante el trabajo con agentes infecciosos.
1. Configurar
- Todos los microbios deben tratarse como si fueran peligrosos. Use siempre una capa de laboratorio y guantes, ate el cabello largo y asegúrese de que las heridas estén particularmente bien protegidas.
- Preparar el espacio de trabajo esterilizando con 70% de etanol.
- Asegúrese de que las placas de agar, la solución(s) de muestra y una caja de bucles de inoculación de plástico preesterilizados o un bucle de metal más una llama Bunsen, estén cerca. Los bucles...
La placa de rayas inicial puede contener colonias originarias de células con diferente composición genética o (dependiendo de la pureza de la muestra) de diferentes especies bacterianas(Figura 2A).
A través del aislamiento posterior de una sola colonia, donde todas las unidades se derivan de una célula madre común, el segundo procedimiento de rayado genera una población bacte...
La capacidad de obtener y cultivar una colonia bacteriana pura es esencial, tanto en entornos clínicos como académicos. El revestimiento de rayas permite el aislamiento de una población celular relativamente clonal, originada a partir de una CFU compartida, que puede ser de particular interés durante el diagnóstico o para la caracterización adicional del aislado. Una muestra se extiende sobre un medio nutritivo a base de agar adecuado y se incuba hasta que las colonias se hacen visibles. Una colonia aislada es post...
- The Human Microbiome Project C. Structure, Function and Diversity of the Healthy Human Microbiome. Nature. 486:207-214. (2012)
- Locey KJ, Lennon JT. Scaling laws predict global microbial diversity. Proceedings of the National Academy of Sciences. 113 (21) 5970-5975 (2016)
- Skouloubris S, Thiberge JM, Labigne A, De Reuse H. The Helicobacter pylori UreI protein is not involved in urease activity but is essential for bacterial survival in vivo. Infection and Immunity. 66:4517-21. (1998)
- Mikucki JA, Auken E, Tulaczyk S, Virginia RA, Schamper C, Sørensen KI, Doran PT, Dugan H, Foley N. Deep groundwater and potential subsurface habitats beneath an Antarctic dry valley. Nature Communications. 6:6831. (2015)
- Mullineaux-Sanders C, Suez J, Elinav E, Frankel G. Sieving through gut models of colonization resistance. Nature Microbiology. 3:132-140. (2018)
- Fournier PE, Drancourt M, Raoult D. Bacterial genome sequencing and its use in infectious diseases. Lancet Infectious Diseases. 7:711-23 (2007)
- Yao Z, Li W, Lin Y, Wu Q, Yu F, Lin W, Lin X. Proteomic Analysis Reveals That Metabolic Flows Affect the Susceptibility of Aeromonas hydrophila to Antibiotics. Scientific Reports. 6:39413 (2016)
- Medina D, Walke JB, Gajewski Z, Becker MH, Swartwout MC, Belden LK. Culture Media and Individual Hosts Affect the Recovery of Culturable Bacterial Diversity from Amphibian Skin. Frontiers in Microbiology. 8:1574 (2017)
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved