0:01
Concepts
3:13
gDNA Isolation and Quality Check
3:43
Isolation of gDNA and gDNA Quality Check
5:52
Amplification and Purification of 16S rRNA Gene by PCR
7:20
Analysis of the DNA Sequences
9:02
Sequence Assembly and Database Search
Secuenciación del ARNr 16s: Una técnica basada en PCR para identificar especies bacterianas
Fuente: Ewa Bukowska-Faniband1, Tilde Andersson1, Rolf Lood1
1 Departamento de Ciencias Clínicas Lund, División de Medicina de Infecciones, Centro Biomédico, Universidad de Lund, 221 00 Lund, Suecia
El planeta Tierra es un hábitat para millones de especies bacterianas, cada una de las cuales tiene características específicas. La identificación de especies bacterianas se utiliza ampliamente en la ecología microbiana para determinar la biodiversidad de muestras ambientales y microbiología médica para diagnosticar pacientes infectados. Las bacterias se pueden clasificar utilizando métodos de microbiología convencionales, como la microscopía, el crecimiento en medios específicos, las pruebas bioquímicas y serológicas y los ensayos de sensibilidad a los antibióticos. En las últimas décadas, los métodos de microbiología molecular han revolucionado la identificación bacteriana. Un método popular es la secuenciación del gen del ARN ribosomal 16S (ARN RRNA). Este método no sólo es más rápido y preciso que los métodos convencionales, sino que también permite la identificación de cepas que son difíciles de cultivar en condiciones de laboratorio. Además, la diferenciación de las cepas a nivel molecular permite la discriminación entre bacterias fenotípicamente idénticas (1-4).
El ARNR 16S se une a un complejo de 19 proteínas para formar una subunidad 30S del ribosoma bacteriano (5). Está codificado por el gen 16S rRNA, que está presente y altamente conservado en todas las bacterias debido a su función esencial en el ensamblaje ribosoma; sin embargo, también contiene regiones variables que pueden servir como huellas dactilares para determinadas especies. Estas características han hecho del gen rRNA 16S un fragmento genético ideal para ser utilizado en la identificación, comparación y clasificación filogenética de bacterias (6).
La secuenciación del gen rRNA 16S se basa en la reacción en cadena de la polimerasa (PCR) (7-8) seguida de la secuenciación del ADN (9). La PCR es un método de biología molecular utilizado para amplificar fragmentos específicos de ADN a través de una serie de ciclos que incluyen:
i) Desnaturalización de una plantilla de ADN de doble cadena
ii) Recocido de imprimaciones (oligonucleótidos cortos) que son complementarios a la plantilla
iii) Extensión de imprimaciones por la enzima ADN polimerasa, que sintetiza una nueva cadena de ADN
En la Figura 1se muestra una descripción esquemática del método.
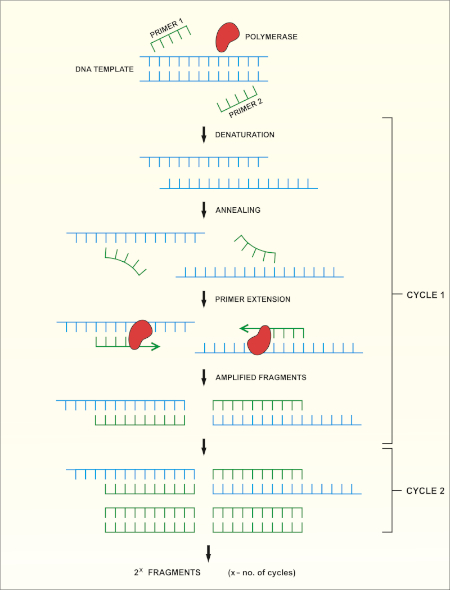
Figura 1: Descripción esquemática de la reacción PCR. Haga clic aquí para ver una versión más grande de esta figura.
Hay varios factores que son importantes para una reacción de PCR exitosa, uno de los cuales es la calidad de la plantilla de ADN. El aislamiento del ADN cromosómico de las bacterias se puede realizar utilizando protocolos estándar o kits comerciales. Se debe tener especial cuidado para obtener ADN que esté libre de contaminantes que puedan inhibir la reacción de PCR.
Las regiones conservadas del gen 16S rRNA permiten el diseño de pares de imprimación universales (uno hacia adelante y otro inverso) que pueden unirse y amplificar la región objetivo en cualquier especie bacteriana. La región de destino puede variar en tamaño. Mientras que algunos pares de imprimación pueden amplificar la mayor parte del gen rRNA 16S, otros amplifican sólo partes de él. En la Tabla 1 se muestran ejemplos de imprimaciones de uso común y sus sitios de enlace se muestran en la Figura 2.
| Primer nombre | Secuencia (5'-3') | Adelante/retroceso | Referencia |
| 8F b) | AGAGTTTGATCCTGGCTCAG | Adelante | -1 |
| 27F | AGAGTTTGATCMTGGCTCAG | Adelante | -10 |
| 515F | GTGCCAGCMGCCGCGGTAA | Adelante | -11 |
| 911R | GCCCCCGTCAATTCMTTTGA | Marcha atrás | -12 |
| 1391R | GACGGGCGGTGTARCA | Marcha atrás | -11 |
| 1492R | GGTTACCTTGTTACGACTT | Marcha atrás | -11 |
Tabla 1: Ejemplos de oligonucleótidos estándar utilizados en la amplificación de genes 16S rRNA a).
a) Las longitudes esperadas del producto PCR generado utilizando las diferentes combinaciones de imprimación se pueden estimar calculando la distancia entre los sitios de unión para la imprimación delantera y la inversa (véase la figura 2), por ejemplo, el tamaño del PCR producto con el par de imprimación 8F-1492R es de 1500 bp, y para el par de imprimación 27F-911R a 900 bp.
b) también conocido como fD1

Figura 2: Figura representativa de la secuencia de rRNA 16S y los sitios de enlace de imprimación. Las regiones conservadas se colorean en gris y las regiones variables se rellenan con líneas diagonales. Para permitir la resolución más alta, la imprimación 8F y 1492R (nombre basado en la ubicación en la secuencia rRNA) se utilizan para amplificar toda la secuencia, permitiendo la secuencia de varias regiones variables del gen. Haga clic aquí para ver una versión más grande de esta figura.
Las condiciones de ciclismo para pcR(es decir, la temperatura y el tiempo necesarios para que el ADN sea desnaturalizado, recocido con imprimadores y sintetizado) dependen del tipo de polimerasa que se utiliza y de las propiedades de las imprimaciones. Se recomienda seguir las pautas del fabricante para una polimerasa en particular.
Una vez completado el programa PCR, los productos son analizados por electroforesis de gel de agarosa. Un PCR exitoso produce una sola banda de tamaño esperado. El producto debe purificarse antes de la secuenciación para eliminar las imprimaciones residuales, los desoxirribonucleótidos, la polimerasa y el tampón que estaban presentes en la reacción de PCR. Los fragmentos de ADN purificados generalmente se envían para la secuenciación a servicios de secuenciación comercial; sin embargo, algunas instituciones realizan la secuenciación del ADN en sus propias instalaciones principales.
La secuencia de ADN se genera automáticamente a partir de un cromatograma de ADN por una computadora y debe ser cuidadosamente revisada para la calidad, ya que a veces se necesita la edición manual. Siguiendo este paso, la secuencia genética se compara con las secuencias depositadas en la base de datos 16S rRNA. Se identifican las regiones de similitud y se entregan las secuencias más similares.
1. Configurar
- Mientras se manipulan microorganismos, se requiere seguir buenas prácticas microbiológicas. Todos los microorganismos, especialmente las muestras desconocidas, deben tratarse como patógenos potenciales. Siga la técnica aséptica para evitar contaminar las muestras, los investigadores o el laboratorio. Lávese las manos antes y después de manipular las bacterias, use guantes y use ropa protectora.
- Llevar a cabo una evaluación de riesgos para el protocolo experimenta...
La identificación de especies bacterianas es importante para diferentes investigadores, así como para aquellos en la atención médica. La secuenciación de arNM 16S fue utilizada inicialmente por los investigadores para determinar las relaciones filogenéticas entre las bacterias. Con el tiempo, se ha implementado en estudios metagenómicos para determinar la biodiversidad de muestras ambientales y en laboratorios clínicos como un método para identificar patógenos potenciales. Permite una identificación rápida y ...
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved