0:01
Concepts
3:53
Preparation of Donor and Recipient Strains
6:01
Conjugation
9:27
Data Analysis by Polymerase Chain Reaction (PCR)
11:29
Results
Conjugaison : méthode de transfert de la résistance à l'ampicilline du donneur à l'hôte E. coli
Source: Alexander S. Gold1, Tonya M. Colpitts1
1 Département de microbiologie, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
Découverte pour la première fois par Lederberg et Tatum en 1946, la conjugaison est une forme de transfert horizontal de gènes entre bactéries qui repose sur un contact physique direct entre deux cellules bactériennes (1). Contrairement à d'autres formes de transfert de gènes, comme la transformation ou la transduction, la conjugaison est un processus naturel dans lequel l'ADN est sécrété d'une cellule de donneur à une cellule recevable d'une manière unidirectionnelle. Cette directionnalité et la capacité de ce processus d'augmenter la diversité génétique des bactéries a donné à la conjugaison la réputation comme une forme d'« accouplement » bactérien, qui aurait grandement contribué à l'augmentation récente de la résistance aux antibiotiques. bactéries (2, 3). En utilisant des pressions sélectives, par exemple l'utilisation d'antibiotiques, la conjugaison a été manipulée pour une utilisation en laboratoire, ce qui en fait un outil puissant pour le transfert horizontal de gènes entre les bactéries, et dans certains cas des bactéries à la levure, les plantes et les animaux cellules (4). Outre les applications en laboratoire, le transfert de gène bactérierium-eucaryote par conjugaison est une avenue passionnante de transfert d'ADN avec une multitude d'applications biotechniques possibles et des implications naturelles (5).
On pense que la conjugaison fonctionne par un « mécanisme en deux étapes » (6). Tout d'abord, avant que l'ADN puisse être transféré, la cellule du donneur doit établir un contact direct de cellule à cellule avec le receveur. Ce processus a été caractérisé le mieux dans les bactéries gram-négatives, dont la plus étudiée est Escherichia coli. Le contact cellule-cellule est établi par la présence d'un réseau complexe de filaments extracellulaires sur le donneur connu sous le nom de pilus sexuel, un élément conjugal codé par le gène transférable connu sous le nom de facteur F (fertilité) (7, 8). En plus d'établir le contact entre le donneur et le receveur, plusieurs protéines sont transportées par le pilus sexuel au cytoplasme receveur, formant un conduit de système de sécrétion de type IV (T4SS) entre les deux cellules, une structure nécessaire pour la deuxième étape de conjugaison, transfert d'ADN (6). En combinant cette fonction du pilus sexuel avec la réplication du cercle roulant de l'ADN, la cellule du donneur est capable de transférer l'ADN sous la forme d'un élément transposable, tel qu'un plasmide ou un transposon, au destinataire par un modèle de « tirer et pomper » (6). Dans ce cas, le « tir » est le transport de la protéine pilote, avec l'ADN lié, par le T4SS dans la cellule récepteur, et le « pompage » est le transport actif de l'ADN au destinataire, un processus dépendant du T4SS et catalysé par des protéines de couplage (6). La machinerie utilisée dans ce processus est composée d'une origine de séquence de transfert (oriT), qui doit être fournie par l'ADN dans les gènes cis et trans, qui codent une relaxase, complexe de formation de paire de compagnon, et le type IV protéine de couplage, et peuvent être présents en cis ou trans (9). Cette relaxase fend le site nic dans la séquence oriT et se fixe de façon covalente à l'extrémité 5' du brin transféré pour produire le relaxosome, un complexe d'ADN-relaxase à brin unique avec d'autres protéines auxiliaires (9). Une fois formé, le relaxosome se connecte au complexe de formation de paires d'accouplement, via la protéine de couplage de type IV, qui permet le transfert du complexe ssDNA-relaxase en cellules réceptrices par le T4SS (10). Une fois dans le cytoplasme du receveur, l'ADN peut s'intégrer dans le génome du receveur ou exister séparément sous la forme d'un plasmide, qui permettent l'expression de ses gènes.
Dans cette expérience, la souche de donneur de conjugaison largement utilisée E. coli WM3064 a été utilisée pour transférer l'encodage génétique pour la résistance à l'ampicilline à la souche bénéficiaire E. coli J53. Tandis que les deux souches des bactéries gram-négatives étaient résistantes à la tétracycline, seulement la souche de distributeur WM3064 a eu le gène pour la résistance d'ampicilline, codépour dans le vecteur de navette de pWD2-oriT, et était auxotrophique à l'acide diaminomiélique (DAP) (11-13). Cette expérience consistait en deux étapes principales, la préparation des souches du donneur et du receveur, suivie du transfert du gène de résistance à l'ampicilline du donneur au receveur par conjugaison (figure 1).
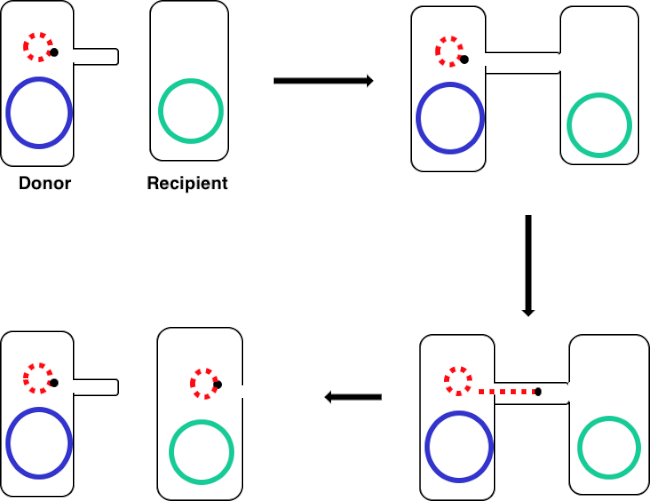
Figure 1 : Schéma de conjugaison. Ce schéma montre le transfert réussi d'un plasmide, un seul exemple d'un élément d'ADN transposable, d'une cellule de donneur à une cellule receveure utilisant la conjugaison. Au contact de la cellule réceptrice par la cellule du donneur par l'intermédiaire du pilus sexuel, le plasmide se reproduit par la réplication du cercle roulant, se déplace à travers le complexe multiprotéiné en joignant les deux cellules, et forme un nouveau plasmide complet dans la cellule réceptrice.
En couvant un mélange de cellules de donneur et de receveur, puis en placageant successivement ces cellules en présence de la tétracycline et du DAP, cela a permis le transfert réussi du gène de résistance à l'ampicilline. Les cellules de placage successivement cultivées à partir de ce mélange en présence de tétracycline et d'ampicilline, enlevé toutes les cellules de donneur en raison de l'absence de DAP et toutes les cellules receveuses qui peuvent ne pas avoir gagné le gène de résistance à l'ampicilline, donnant strictement destinataire J53 souche bactéries qui ont acquis une résistance à l'ampicilline (figure 2). Une fois effectué, le transfert réussi du gène de résistance d'ampicilline a été confirmé par PCR. Depuis que la conjugaison a été réussie, la souche J53 de E. coli a contenu pWD2-oriT et était résistante à l'ampicilline, et l'encodage de gène pour cette résistance est détectable par PCR. Cependant, en cas d'échec, il n'y aurait pas eu de détection du gène de résistance à l'ampicilline et l'ampicilline fonctionnerait toujours comme un antibiotique efficace contre la souche J53.
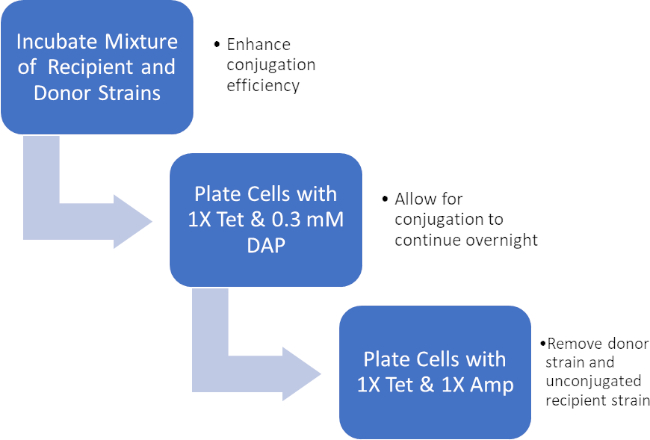
Figure 2 : Schéma de protocole. Ce schéma montre un aperçu du protocole présenté.
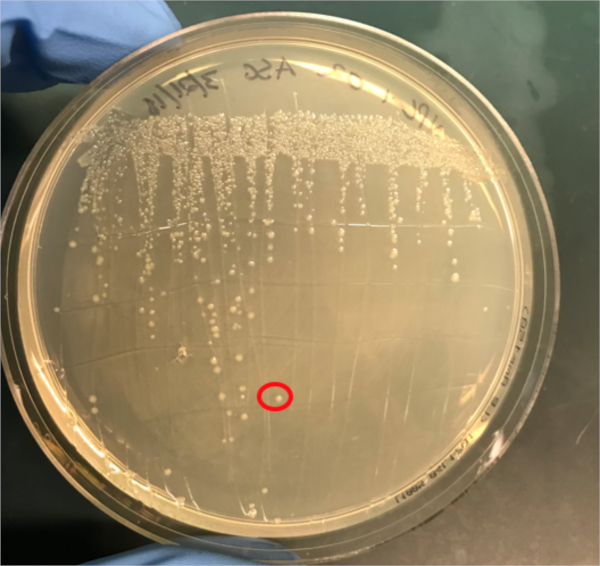
Figure 3A : Confirmation d'une conjugaison réussie par PCR. A) Les stocks de congélation des échantillons témoins conjugués et négatifs ont été striés sur des plaques d'agar et une colonie a été sélectionnée (rouge) pour l'isolement de l'ADN.
1. Mise en place
- Autoclave environ 1L du milieu de Luria-Bertani (LB). Ce LB stérile sera utilisé pour fabriquer environ 5 ml de LB contenant 0,3 mM d'acide diaminopiélique (DAP).
- Recueillir les plaques suivantes: plaques d'agar LB avec 1X Tet et 0,3 mM DAP, plaques d'agar LB avec 1X Tet seulement, et plaques d'agar LB avec 1X Amp / Tet seulement.
- Assurez-vous qu'un peu de glycérol et une boîte de pointes de pipette en plastique pré-stérilisée sont à portée de main...
Si la conjugaison a été couronnée de succès, un produit PCR de la taille d'une paire de 500 de base sera observé dans le puits où la réaction PCR 1 a été chargée (bien #2 à la figure 3B), alors qu'aucune bande ne sera observée dans le puits où la réaction PCR 3 a été chargée (bien #4 à la figure 3B). La présence de cette bande confirme le transfert réussi du gène de résistance à l'ampicilline, conférant ainsi une résistance à l'ampicilline à la souche J53 d'...
La conjugaison est un processus naturel de transfert horizontal de gènes qui repose sur le contact direct cellule-cellule d'une cellule donneuse et d'une cellule recevable. Ce processus est partagé entre toutes sortes de bactéries et a joué un rôle déterminant dans l'évolution bactérienne, notamment la résistance aux antibiotiques. En laboratoire, la conjugaison peut être utilisée comme une méthode efficace de transfert de gènes qui est beaucoup moins perturbateur par rapport à d'autres techniques. En dehor...
- Lederberg J, Tatum, E.L. Gene recombination in Escherichia coli Nature. 1946;158:558.
- Holmes R.K. J, M.G. Genetics: Exchange of Genetic Information. 4th Edition ed. Baron S, editor. Galveston, TX: University of Texas Medical Branch at Galveston; 1996.
- Cruz F, Davies, J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends in Microbiology. 2000;8:128-33.
- Llosa M, Cruz, F. Bacterial conjugation: a potential tool for genomic engineering. Ressearch in Microbiology. 2005;156:1-6.
- Lacroix B, Citovsky, V. Transfer of DNA from Bacteria to Eukaryotes. mBio. 2016;7(4):1-9.
- Llosa M, et al. Bacterial conjugation: a two-step mechanism for
- DNA transport. Molecular Microbiology. 2002;45:1-8.
- Grohmann E, Muth, G., Espinosa, M. Conjugative Plasmid Transfer in Gram-Positive Bacteria. Microbiology and Molecular Biology Reviews. 2003;67:277-301.
- Firth N, Ippen-Ihler, K, Skurray, RA. Structure and function of the F factor and mechanism of conjugation. Escherichia coli and salmonella: cellular and molecular biology. 1996;2:2377-401.
- Smillie C, Garcillan-Barcia MP, Francia MV, Rocha EPC, De La Cruz F. Mobility of Plasmids. Microbiology and Molecular Biology Reviews. 2010;74(3):434-52.
- Cascales E. Definition of a Bacterial Type IV Secretion Pathway for a DNA Substrate. 2004;304(5674):1170-3.
- Wang P, Yu Z, Li B, Cai X, Zeng Z, Chen X, et al. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas. Microbial Cell Factories. 2015;14(1):11.
- Yi H, Cho YJ, Yong D, Chun J. Genome Sequence of Escherichia coli J53, a Reference Strain for Genetic Studies. Journal of Bacteriology. 2012;194(14):3742-3.
- Baumann RLB, E. H.; Wiseman, J. S.; Vaal, M.; Nichols, J. S. Inhibition of Escherichia coli Growth and Diaminopimelic Acid Epimerase by 3-Chlorodiaminopimelic Acid. Antimicrobial Agents and Chemotherapy 1988;32:1119-23.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
Tags
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved