0:01
Concepts
3:53
Preparation of Donor and Recipient Strains
6:01
Conjugation
9:27
Data Analysis by Polymerase Chain Reaction (PCR)
11:29
Results
結合:アンピシリン耐性をドナーからレシピエント大腸菌に移す方法
ソース: アレクサンダー S. ゴールド1, トーニャ M. コルピッツ1
1ボストン大学医学部微生物学科、国立新興感染症研究所、ボストン、マサチューセッツ州
1946年にレダーバーグとテイタムによって最初に発見された共役は、2つの細菌細胞間の直接的な物理的接触に依存する細菌間の水平遺伝子伝達の一形態である(1)。形質転換やトランスダクションなどの他の形態の遺伝子導入とは異なり、結合はDNAがドナー細胞からレシピエント細胞に一方向的に分泌される自然発生プロセスです。この方向性と細菌の遺伝的多様性を高めるこのプロセスの能力は、抗生物質耐性の最近の上昇に大きく貢献していると考えられている細菌「交配」の一形態としての評判を共役に与えました。バクテリア(2、3)。選択的圧力(例えば抗生物質の使用)を用いることで、実験室での使用のためにコンジュゲーションが操作され、細菌間の水平遺伝子移動、場合によっては細菌から酵母、植物、動物への強力なツールとなっています。セル (4)。実験室での応用とは別に、結合による細菌・ユーカリオテ遺伝子の伝達は、多くの可能なバイオテクノロジーアプリケーションと自然に起こる影響を持つDNA伝達のエキサイティングな手段です(5)。
コンジュゲーションは「2段階機構」(6)によって働くと考えられている。まず、DNAを移送する前に、ドナー細胞はレシピエントと直接細胞間接触を行わなければならない。このプロセスは、グラム陰性細菌で最もよく特徴付けられており、その中で最も研究されているのは大腸菌である。細胞間接触は、性ピラスとして知られているドナー上の細胞外フィラメントの複雑なネットワークの存在によって確立され、F(不妊)因子(7,8)として知られる転写可能な遺伝子によってコードされる共役元素である。ドナーとレシピエントとの接触を確立することに加えて、いくつかのタンパク質は、レシピエント細胞質に性ピラスを介して輸送され、2つの細胞間のIV分泌系(T4SS)導管を形成し、第2工程に必要な構造である。DNA伝達(6)。この性ピルスの機能とDNAの転がり円複製を組み合わせることにより、ドナー細胞は、プラスミドまたはトランスポゾンなどのトランスポーザブル要素の形でDNAを「シュートアンドポンプ」モデル(6)によってレシピエントに移すことができる。この場合、「シューティング」は、パイロットタンパク質の輸送であり、連結DNAを用いて、T4SSによってレシピエント細胞に送られ、「ポンピング」はレシピエントへのDNAの活性輸送であり、T4SSに依存し、結合タンパク質によって触媒されるプロセスである(6)。このプロセスで使用される機械は、リラクサーゼ、合体対形成複合体、およびIV型結合タンパク質をコードするシスおよびトランス遺伝子のDNAによって提供されなければならない転移配列(oriT)の起源から構成され、シスまたはトランス(9)に存在することができます。このリラクサーゼは、oriT配列内のnic部位を切断し、転移した鎖の5'末端に生有して、他の補助タンパク質との一本鎖DNAリラクサーゼ複合体であるリラクソームを産生する(9)。いったん形成されると、リラクソームは交配対形成複合体に接続し、IV型結合タンパク質を介して、SsDNA-relaxase複合体をT4SS(10)によってレシピエント細胞に移植することを可能にする。レシピエントの細胞質に入ると、DNAはレシピエントゲノムに統合することも、プラスミドの形で別々に存在することも、その遺伝子の発現を可能にする。
この実験では、広く使用されている共役ドナー株大腸菌WM3064を、レシピエント株大腸菌J53に対するアンピシリン耐性をコードする遺伝子コードを伝達するために用いられた。グラム陰性菌の両方の株はテトラサイクリンに耐性であったが、ドナー株WM3064のみがアンピシリン耐性の遺伝子を持ち、pWD2-oriTシャトルベクターでコードされ、ジアミノピメル酸(DAP)に対して下方栄養性であった(11-13)。この実験は、ドナーおよびレシピエント株の調製、続いてコンジュゲーションによるドナーからレシピエントへのアンピシリン耐性遺伝子の移植の2つの主要なステップからなる(図1)。
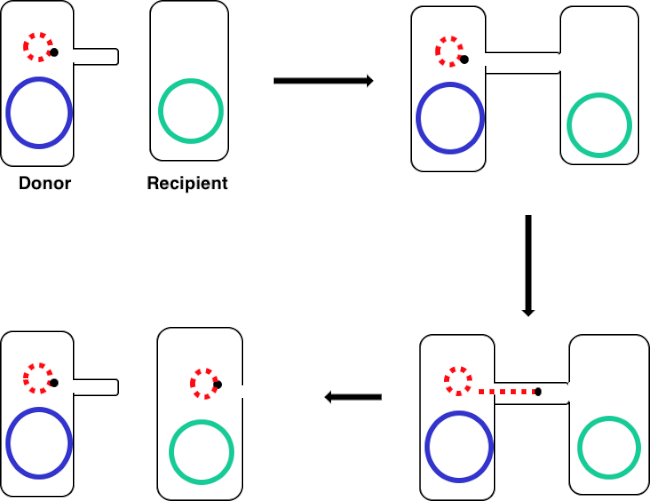
図 1: コンジュゲーション回路図。この回路図は、プラスミドの正常な転移を示し、転移可能なDNA要素の一例に過ぎず、共役を用いてドナー細胞からレシピエント細胞へ送り込む。性ピラスを介してドナー細胞によってレシピエント細胞と接触すると、プラスミドは転がり円複製によって複製し、2つの細胞を結合する多タンパク質複合体を通って移動し、レシピエント細胞に新しい全長プラスミドを形成する。
ドナー細胞とレシピエント細胞の混合物をインキュベートすることにより、テトラサイクリンおよびDAPの存在下でこれらの細胞を連続的にめっきし、これはアンピシリン耐性遺伝子の正常な転写を可能にした。テトラサイクリンとアンピシリンの存在下でこの混合物から増殖した細胞を連続的にめっきし、DAPの欠如とアンピシリン耐性遺伝子を得ていない可能性のある任意のレシピエント細胞の欠如のためにすべてのドナー細胞を除去し、厳密にレシピエントJ53株を得たアンピシリン耐性を獲得した細菌(図2)。一旦行われると、アンピシリン耐性遺伝子の正常な転写がPCRによって確認された。大腸菌のJ53株はpWD2-oriTを含み、アンピシリンに対して耐性があり、この耐性をコードする遺伝子はPCRによって検出可能であった。しかし、もし失敗した場合、アンピシリン耐性遺伝子の検出はなく、アンピシリンはJ53株に対する有効な抗生物質として依然として機能するだろう。
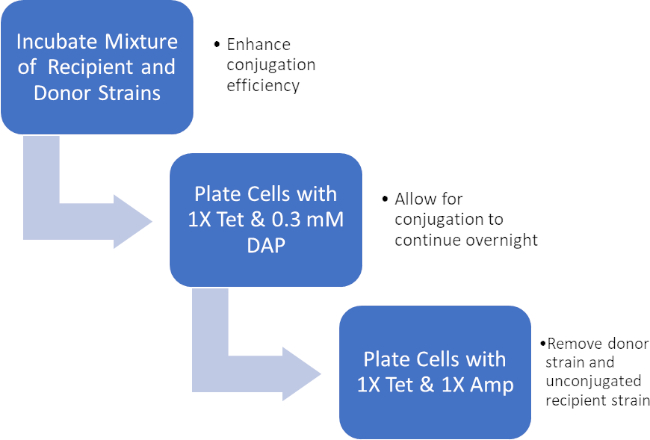
図 2: プロトコルの回路図。この回路図は、提示されたプロトコルの概要を示しています。
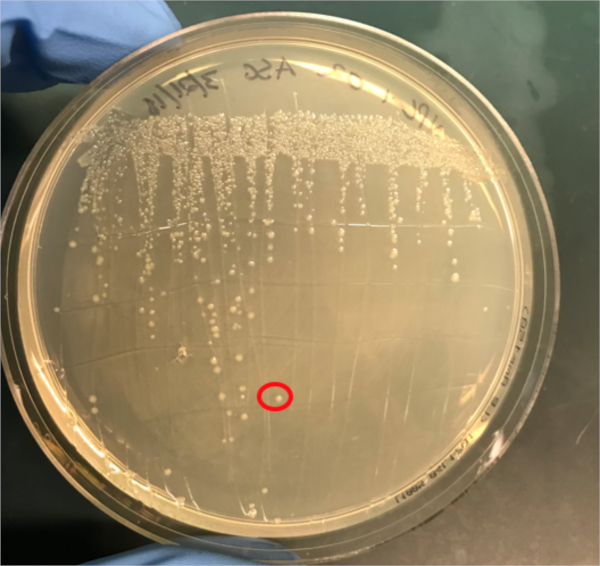
図3A:PCRによるコンジュゲーションの成功の確認A)共役および陰性対照試料の冷凍庫ストックを寒天プレート上にストリークアウトし、DNA分離のためにコロニー(赤色)を選択した。
- Lederberg J, Tatum, E.L. Gene recombination in Escherichia coli Nature. 1946;158:558.
- Holmes R.K. J, M.G. Genetics: Exchange of Genetic Information. 4th Edition ed. Baron S, editor. Galveston, TX: University of Texas Medical Branch at Galveston; 1996.
- Cruz F, Davies, J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends in Microbiology. 2000;8:128-33.
- Llosa M, Cruz, F. Bacterial conjugation: a potential tool for genomic engineering. Ressearch in Microbiology. 2005;156:1-6.
- Lacroix B, Citovsky, V. Transfer of DNA from Bacteria to Eukaryotes. mBio. 2016;7(4):1-9.
- Llosa M, et al. Bacterial conjugation: a two-step mechanism for
- DNA transport. Molecular Microbiology. 2002;45:1-8.
- Grohmann E, Muth, G., Espinosa, M. Conjugative Plasmid Transfer in Gram-Positive Bacteria. Microbiology and Molecular Biology Reviews. 2003;67:277-301.
- Firth N, Ippen-Ihler, K, Skurray, RA. Structure and function of the F factor and mechanism of conjugation. Escherichia coli and salmonella: cellular and molecular biology. 1996;2:2377-401.
- Smillie C, Garcillan-Barcia MP, Francia MV, Rocha EPC, De La Cruz F. Mobility of Plasmids. Microbiology and Molecular Biology Reviews. 2010;74(3):434-52.
- Cascales E. Definition of a Bacterial Type IV Secretion Pathway for a DNA Substrate. 2004;304(5674):1170-3.
- Wang P, Yu Z, Li B, Cai X, Zeng Z, Chen X, et al. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas. Microbial Cell Factories. 2015;14(1):11.
- Yi H, Cho YJ, Yong D, Chun J. Genome Sequence of Escherichia coli J53, a Reference Strain for Genetic Studies. Journal of Bacteriology. 2012;194(14):3742-3.
- Baumann RLB, E. H.; Wiseman, J. S.; Vaal, M.; Nichols, J. S. Inhibition of Escherichia coli Growth and Diaminopimelic Acid Epimerase by 3-Chlorodiaminopimelic Acid. Antimicrobial Agents and Chemotherapy 1988;32:1119-23.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
Tags
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved