0:02
Concepts
3:01
Preparation of Donor Phage Lysate
4:50
Transduction
8:39
Data Analysis by Quantitative Polymerase Chain Reaction
10:01
Results
Transducción de fagos: Un método para transferir resistencia a ampicilina de un donante una E. coli receptora
Fuente: Alexander S. Gold1, Tonya M. Colpitts1
1 Departamento de Microbiología, Escuela de Medicina de la Universidad de Boston, Laboratorios Nacionales de Enfermedades de Infecciones Emergentes, Boston, MA
La transducción es una forma de intercambio genético entre bacterias que utiliza bacteriófagos, o fagos, una clase de virus que infecta exclusivamente organismos procaarios. Esta forma de transferencia de ADN, de una bacteria a otra a través de un fago, fue descubierta en 1951 por Norton Zinder y Joshua Ledererg, quienes denominaron el proceso "transducción" (1). Los bacteriófagos fueron descubiertos por primera vez en 1915 por el bacteriólogo británico Frederick Twort, luego descubiertos de nuevo de forma independiente en 1917 por el microbiólogo franco-canadiense Felix d'Herelle (2). Desde entonces, la estructura y función de estos fagos se han caracterizado ampliamente (3), dividiendo estos fagos en dos clases. La primera de estas clases son los fagos líticos que tras la infección se multiplican dentro de la bacteria huésped, alterando el metabolismo bacteriano, lysing la célula, y liberando fago progenie (4). Como resultado de esta actividad antibacteriana y la creciente prevalencia de bacterias resistentes a los antibióticos, estos fagos líticos han demostrado ser útiles recientemente como tratamiento sustituto de los antibióticos. La segunda de estas clases son los fagos lisogénicos que pueden multiplicarse dentro del huésped a través del ciclo lítico o entrar en un estado de reposo en el que su genoma se integra en el del huésped (Figura 1), un proceso conocido como lisógeno, con la capacidad de fago producción que se inducirá en varias generaciones posteriores (4).
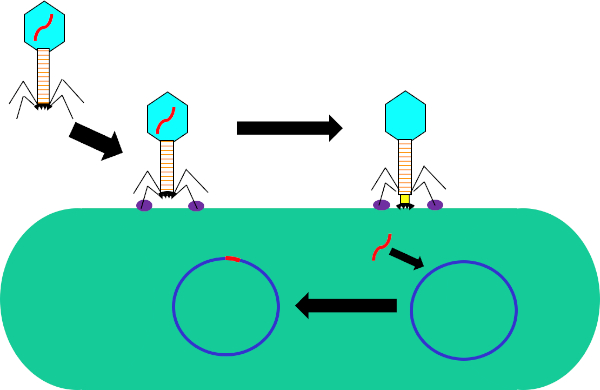
Figura 1: Infección de la célula huésped por bacteriófago. Adsorción por el fago a la pared celular bacteriana a través de interacciones entre las fibras de cola y el receptor (púrpura). Una vez en la superficie celular, el fago se une irreversiblemente a la célula bacteriana utilizando la placa base (negro) que se mueve a la pared celular por la vaina contráctea (amarillo). El genoma del fago (rojo) entra en la célula y se integra en el genoma de la célula huésped.
Mientras que la transducción bacteriana es un proceso natural, utilizando tecnología moderna ha sido manipulada para la transferencia de genes a bacterias en el entorno de laboratorio. Al insertar genes de interés en el genoma de un fago lisogénico, como el fago, uno es capaz de transferir estos genes a los genomas de las bacterias y, en consecuencia, expresarlos dentro de estas células. Mientras que otros métodos de transferencia de genes, como la transformación, utilizan un plásmido para la transferencia y expresión de genes, la inserción del genoma del fago en el de la bacteria receptora no sólo tiene el potencial de conferir nuevos rasgos a esta bacteria, sino que también permite mutaciones naturales y otros factores del entorno celular para alterar la función del gen transferido.
En comparación con otros métodos de transferencia de genes horizontales, como la conjugación, la transducción es bastante flexible en los criterios requeridos para las células donantes y receptoras. Cualquier elemento genético que pueda caber dentro del genoma del fago que se está utilizando puede transferirse de cualquier cepa de bacterias donantes a cualquier cepa de bacterias receptoras, siempre y cuando ambas sean permisivas para el fago, requiriendo la expresión de los receptores de fago necesarios en el superficies celulares. Una vez que este gen se mueve fuera del genoma del donante y se empaqueta en el fago, se puede transferir al receptor. Después de la transducción, es necesario seleccionar para las bacterias receptoras que contienen el gen de interés que deben seleccionarse. Esto podría hacerse mediante el uso de un marcador genético, como una etiqueta FLAG o una etiqueta de polihistidina, para marcar el gen de interés, o la función intrínseca del gen, en el caso de genes que codifican para la resistencia a los antibióticos. Además, la PCR podría utilizarse para confirmar aún más la transducción correcta. Mediante el uso de imprimaciones para una región dentro del gen de interés y la comparación de la señal con un control positivo, bacterias que tiene el gen de interés, y un control negativo, bacterias que se sometieron a los mismos pasos que la reacción de transducción sin fago. Si bien la transducción bacteriana es una herramienta útil en biología molecular, ha desarrollado y sigue desempeñando un papel importante en la evolución de las bacterias, particularmente en lo que respecta al reciente aumento de la resistencia a los antibióticos.
En este experimento, la transducción bacteriana se utilizó para transferir la codificación genética para la resistencia a la ampicilina antibiótica de la cepa W3110 de E.coli a la cepa J53 a través del bacteriófago P1 (5). Este experimento consistió en dos pasos principales. En primer lugar, la preparación de fago P1 que contiene el gen de resistencia a la ampicilina de la cepa del donante. En segundo lugar, la transferencia de este gen a la cepa receptora por transducción con el fago P1 (Figura 1). Una vez realizada, la transferencia exitosa del gen de resistencia a la ampicilina podría determinarse por qPCR (Figura 2). Si la transducción fuera exitosa, la cepa J53 de E. coli sería resistente a la ampicilina, y el gen que confiere esta resistencia es detectable por qPCR. De no tener éxito, no habría detección del gen de resistencia a la ampicilina y la ampicilina seguiría funcionando como un antibiótico eficaz contra la cepa J53.
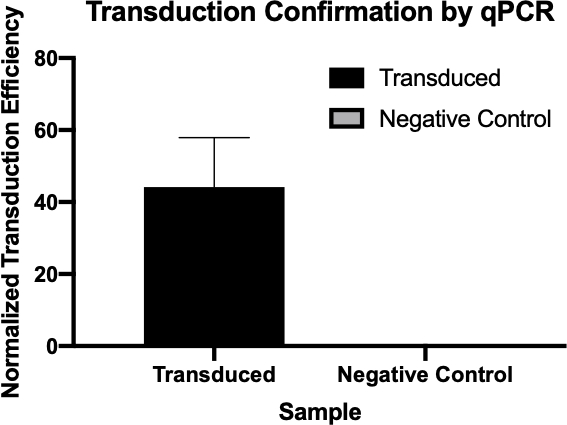
Figura 2: La confirmación de la transducción exitosa por qPCR. Al comparar los valores Cq detectados para el gen de interés de la reacción de transducción y la reacción de control negativa, y normalizar estos valores contra un gen de limpieza, se pudo confirmar que la transducción bacteriana fue exitosa.
1. Configuración
- Antes de empezar cualquier trabajo que involucre microbios, esterilice el espacio de trabajo con 70% de etanol. Utilice siempre el EPP necesario (abrigo de laboratorio y guantes).
- Asegúrese de que las placas de agar LB con 1x ampicilina, solución de lysato de fago P1 disponible en el uso comercial, cloroformo, citrato de sodio 1 M, glicerol y una caja de puntas de pipeta de plástico preesterilizadas y esparcidores celulares están cerca.
- Preparar el LB e...
La transferencia de genes hacia y desde bacterias por bacteriófago, mientras que un proceso natural, ha demostrado ser extremadamente útil para una multitud de propósitos de investigación. Mientras que otros métodos de transferencia de genes como la transformación y la conjugación son posibles, la transducción utiliza de manera única los bacteriófagos; no sólo permite la integración génica en el genoma del huésped, sino también la entrega de genes a múltiples bacterias que no son susceptibles a otros mét...
- Lederberg J, Lederberg E.M., Zinder, N.D., et al. Recombination analysis of bacterial heredity. Cold Spring Harbor symposia Quantitative Biol. 1951;16:413-43.
- Duckworth DH. "Who Discovered Bacteriophage?". Bacteriology Reviews. 1976;40:793-802.
- Yap ML, Rossman, M.G. Structure and Function of Bacteriophage T4. Future Microbiol. 2014;9:1319-27.
- Sulakvelidze A, Alavidze, Z., Morris, J. G. Bacteriophage Therapy Antimicrobial Agents and Chemotherapy 2001;45(3):649-59.
- Moore S. Sauer:P1vir phage transduction 2010 [Available from: https://openwetware.org/wiki/Sauer:P1vir_phage_transduction].
- Kobayashi A, et al. Growth Phase-Dependent Expression of Drug Exporters in
- Escherichia coli and Its Contribution to Drug Tolerance. Journal of Bacteriology. 2006;188(16):5693-703.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.
- Pálffy R. et al. Bacteria in gene therapy: bactofection versus alternative gene therapy. Gene Ther. 2006 13:101-5.
- O'Neill JM, et al. Intestinal delivery of non-viral gene therapeutics: physiological barriers and preclinical models. Drug Discovery Today. 2011;16:203-2018.
ABOUT JoVE
Copyright © 2024 MyJoVE Corporation. All rights reserved