A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
عزل وتوصيف الجراثيم الطبيعية للنموذج الديدان الخيطية Caenorhabditis elegans
In This Article
Summary
Caenorhabditis elegans هو أحد الأنواع النموذجية الرئيسية في علم الأحياء ، ومع ذلك يتم إجراء جميع الأبحاث تقريبا في غياب الميكروبات المرتبطة بها بشكل طبيعي. ستساعد الطرق الموضحة هنا على تحسين فهمنا لتنوع الميكروبات المرتبطة بها كأساس لأبحاث C. elegans الوظيفية المستقبلية.
Abstract
تتفاعل الديدان الخيطية Caenorhabditis elegans مع مجموعة كبيرة ومتنوعة من الكائنات الحية الدقيقة في الطبيعة. بشكل عام ، توجد C. elegans بشكل شائع في المواد النباتية الفاسدة ، وخاصة الفواكه الفاسدة مثل التفاح أو على أكوام السماد. كما أنه يرتبط ببعض مضيفات اللافقاريات مثل الرخويات وقمل الخشب. هذه الموائل غنية بالميكروبات ، والتي تعمل كغذاء ل C. elegans والتي يمكنها أيضا استعمار الأمعاء الخيطية باستمرار. حتى الآن ، لم يتم فهم التنوع الدقيق والاتساق لميكروبات C. elegans الأصلية عبر الموائل والمواقع الجغرافية بشكل كامل. هنا ، نصف نهجا مناسبا لعزل C. elegans عن الطبيعة وتوصيف الكائنات الحية الدقيقة للديدان. يمكن عزل الديدان الخيطية بسهولة عن مواد السماد أو التفاح المتعفن أو الرخويات أو جذبها عن طريق وضع التفاح على أكوام السماد. وقت الذروة للعثور على C. elegans في نصف الكرة الشمالي هو من سبتمبر حتى نوفمبر. يمكن غسل الديدان من مواد الركيزة المجمعة عن طريق غمر الركيزة في محلول عازل ، يليها جمع الديدان الخيطية ونقلها إلى وسط نمو الديدان الخيطية أو مخزن PCR المؤقت للتحليل اللاحق. نوضح كذلك كيف يمكن استخدام العينات لعزل وتنقية الكائنات الحية الدقيقة المرتبطة بالديدان ومعالجة الديدان لتحليل الحمض النووي الريبي الريبوسومي 16S لتكوين مجتمع الميكروبات. بشكل عام ، قد تحفز الطرق الموصوفة أبحاثا جديدة حول توصيف الكائنات الحية الدقيقة C. elegans عبر الموائل والمواقع الجغرافية ، مما يساعد على الحصول على فهم شامل لتنوع واستقرار ميكروبات الديدان الخيطية كأساس للبحث الوظيفي في المستقبل.
Introduction
في الطبيعة ، توجد C. elegans بشكل شائع في المواد النباتية الفاسدة ، وخاصة الفواكه الفاسدة مثل التفاح أو على أكوام السماد1. كما أنه يرتبط ببعض مضيفات اللافقاريات مثل الرخويات وقمل الخشب 2,3. هذه الموائل غنية بالميكروبات ، والتي لا تعمل فقط كغذاء للدودة ، ولكنها قد تشكل أيضا ارتباطات مستقرة معها. تم نشر معلومات عن تنوع الكائنات الحية الدقيقة المرتبطة بشكل طبيعي فقط في عام 20164،5،6. منذ ذلك الحين ، كشفت هذه الدراسات وعدد قليل فقط من الدراسات الحديثة أن C. elegans مرتبطة بمجموعة متنوعة من البكتيريا والفطريات ، بما في ذلك البكتيريا من جنس Pseudomonas ، Enterobacter ، Ochrobactrum ، Erwinia ، Comamonas ، Gluconobacter ، والعديد من الدراسات الأخرى6،7،8. يمكن للعديد من البكتيريا المرتبطة بها استعمار أمعاء الدودة بثبات ، على الرغم من أنها ليست كلها6،9،10،11،12. من المحتمل أن تكون ذات أهمية رئيسية لفهمنا لبيولوجيا C. elegans لأنها يمكن أن توفر التغذية ، وتحمي من مسببات الأمراض وربما الضغوطات الأخرى ، وتؤثر على سمات تاريخ الحياة المركزية مثل معدل التكاثر أو التطور أو الاستجابات السلوكية.
على سبيل المثال ، يمكن للعزلات المرتبطة بشكل طبيعي من أجناس Pseudomonas و Ochrobactrum وأيضا Enterobacter أو Gluconobacter أن تحمي الدودة من العدوى الممرضة والقتل بطرق مميزة5،6،11،13،14. تؤثر عزلة محددة من جنس Comamonas على الاستجابة الغذائية للديدان الخيطية وتطورها وعمرها وخصوبتها15،16،17. تنتج بكتيريا بروفيدنسيا التيرامين المعدل العصبي وبالتالي تعدل نشاط الجهاز العصبي المضيف والاستجابات السلوكية الناتجة18. تم إثبات أن مجموعة من البكتيريا المختلفة المرتبطة بشكل طبيعي تؤثر على معدل النمو السكاني والخصوبة والاستجابات السلوكية5،6،9،11،19.
حتى الآن ، لم يتم فهم التنوع والاتساق الدقيق لميكروبات C. elegans الأصلية عبر الموائل والمواقع الجغرافية بشكل كامل ، ولا يزال يتعين الكشف عن المزيد من الارتباطات بين الدودة والميكروبات من بيئتها. استخدمت العديد من الدراسات السابقة سلالات بكتيرية معزولة من بعض بيئة التربة ، أو موائل C. elegans الطبيعية ، أو من تجارب mesocosm (أي البيئات القائمة على المختبر التي تعيد إنشاء الموائل الطبيعية) مع سلالات مختبر C. elegans 4،5،20. على الرغم من أن هذه الدراسات حصلت على رؤى جديدة حول تأثير الميكروبات على سمات الديدان الخيطية المحددة (على سبيل المثال ، استقلاب الديدان الخيطية21) ، فإن أهمية هذه التفاعلات لبيولوجيا C. elegans في الطبيعة غير واضحة. لذلك ، تصف هذه المخطوطة طرق عزل C. elegans مباشرة عن الطبيعة وعزل الميكروبات المرتبطة بشكل طبيعي وتوصيفها لاحقا من كل من الديدان المفردة ومجموعات الديدان. الطرق الموصوفة هي نسخة محدثة ومحسنة من الإجراءات المستخدمة سابقا لعزل وتوصيف C. elegans الطبيعية والميكروبات الأصلية2،6،7. بالنظر إلى أن C. elegans موجود على نطاق واسع في المواد النباتية المتحللة في جميع أنحاء العالم (خاصة في الفواكه المتعفنة والمناطق المعتدلة وفي الخريف)1،2،22،23،24،25 ، يمكن تطبيق هذا البروتوكول من قبل أي مختبر كلما كان هناك اهتمام بربط C. elegans سمات الميكروبات المرتبطة بشكل طبيعي وبالتالي سياق أكثر صلة بشكل طبيعي. هذا الأخير محوري لفهم كامل لبيولوجيا الديدان الخيطية لأنه من المعروف من مجموعة متنوعة من الأنظمة المضيفة الأخرى أن الجراثيم المرتبطة بها يمكن أن تؤثر على خصائص تاريخ الحياة المتنوعة26 ، وهو جانب يتم إهماله حاليا إلى حد كبير في العديد من دراسات C. elegans عبر جميع تخصصات علوم الحياة تقريبا.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. إعداد المخازن المؤقتة والوسائط
- قم بإعداد S-buffer بإضافة 5.85 جم من كلوريد الصوديوم ، و 1.123 جم من K 2 HPO 4 ، و 5.926 جم من KH 2 PO4 ، و 1 لتر من H2O منزوع الأيونات إلى دورقوأوتوكلاف لمدة 20 دقيقة عند 121 درجة مئوية.
- تحضير وسط لزج عن طريق إضافة S-buffer يحتوي على 1.2٪ (وزن / حجم) هيدروكسي ميثيل سلولوز (المادة المسببة لزوجة الوسط) ، 5 ملغ / مل من الكوليسترول ، 1 ملي مول MgSO4 ، 1 ملي مول CaCl2 ، و 0.1٪ (v / v) أسيتون. الأوتوكلاف وتحريك الوسط اللزج حتى يصبح متجانسا تماما.
ملاحظة: قد يستغرق هذا عدة ساعات. أيضا ، يمكن تحضير S-buffer مباشرة وإضافته إلى الوسط اللزج دون تعقيم مسبق. - قم بإعداد M9-buffer بإضافة 3 جم من KH 2 PO 4 ، و 6 جم من NA 2 HPO4 · 2 H 2 O ، و 5 جم من NaCl ، و 1 L من H2O منزوع الأيونات إلى دورق 1 لتر. الأوتوكلاف الحل ، وبعد التبريد ، إضافة 1 مل من 1 M MgSO 4 (123.24 غرام من MgSO4 · 7H 2 O في 500 مل من H2O منزوع الأيونات ، مرشح معقمة).
- قم بإعداد محلول مخزون Triton X-100 بنسبة 10٪ (v / v) عن طريق خلط 5 مل من Triton X-100 مع 45 مل من M9-buffer. قم بتصفية وتعقيم المحلول باستخدام مرشح 0.2 ميكرومتر.
- قم بإعداد M9-buffer باستخدام Triton X-100 (M9-T) عن طريق إضافة 2.5 مل من محلول مخزون Triton X-100 بنسبة 10٪ (v / v) إلى 1 لتر من M9-buffer بعد التعقيم للحصول على 0.025٪ (v / v) M9-T.
- تحضير 30٪ (v / v) من الجلسرين في S-buffer عن طريق خلط 15 مل من الجلسرين المعقم بنسبة 100٪ و 35 مل من المخزن المؤقت S المعقم في أنبوب سعة 50 مل.
2. إعداد العينات البيئية (الشكل 1)
- اجمع عينات بيئية مثل السماد أو الفواكه الفاسدة وضع كل عينة في كيس بلاستيكي فردي أو أنبوب أو حاوية نظيفة أخرى.
ملاحظة: من أجل جذب الديدان الخيطية ، يمكن وضع التفاح على السماد قبل عدة أسابيع من أخذ العينات. - انشر قطعا من العينة البيئية بالتساوي في طبق بتري فارغ ومعقم بطول 9 سم.
ملاحظة: من المرجح أن تحتوي العينات ذات المستويات الأعلى من الاضمحلال على C. elegans. اختياريا ، يمكن استخدام طبق بتري مملوء بوسط أجار خال من الببتون (PFM) لزيادة التباين. - قم بتغطية العينة بعناية بحوالي 20 مل من الوسط اللزج المعقم.
ملاحظة: تطفو الديدان الخيطية على السطح في غضون 1-2 ساعة. يبطئ الوسط اللزج حركة الديدان ويسهل أخذ عينات منها. يمكن استخدام المخزن المؤقت M9 المعقم كبديل ، ومع ذلك ، ستكون حركة الدودة أسرع.
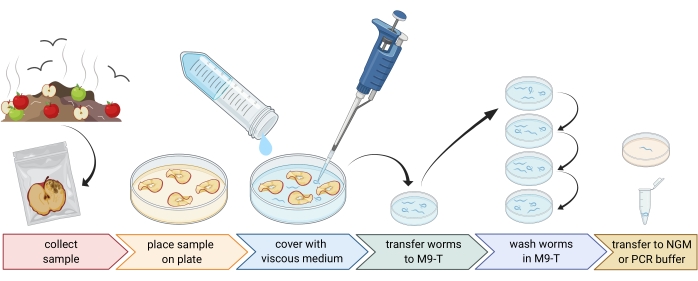
الشكل 1: عزل الديدان الخيطية من الركائز. توضع عينات الركيزة في أطباق بتري فارغة ومغطاة بوسط لزج لطرد الديدان الخيطية. يتم نقل الديدان الخيطية إلى M9-T وغسلها بشكل متكرر لإزالة البكتيريا من الخارج. يمكن استخدام الديدان الخيطية الفردية لعزل الحمض النووي ، أو عزل البكتيريا المرتبطة بها ، أو وضعها على ألواح أجار لمجموعات دودة الثقافة. الشكل الذي تم إنشاؤه باستخدام BioRender.com. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
3. عزل نيماتودا Caenorhabditis (الشكل 1)
- ابحث عن الديدان الخيطية Caenorhabditis باستخدام مجهر تشريح ، باتباع الإرشادات الواردة في فصل WormBook حول عزل C. elegans والديدان الخيطية ذات الصلة بواسطة Barrière و Félix27.
ملاحظة: Caenorhabditis خنثى / الإناث لديها أمعاء من اللون البني الفاتح (تحت الإضاءة المرسلة). علاوة على ذلك ، تحتوي خلايا الأمعاء على نوى خلايا كبيرة ، والتي يمكن رؤيتها كنقاط بيضاء. على النقيض من ذلك ، غالبا ما يكون لأنواع الديدان الخيطية الأخرى أمعاء بنية داكنة وقد تظهر عدم تناسق أمامي خلفي في شدة الصباغ وعادة لا توجد نوى مرئية. تم العثور على الفرج من البالغين Caenorhabditis خنثى / الإناث في منتصف الحيوان. لا يظهر توطين الفرج هذا دائما من قبل أصناف الديدان الخيطية الأخرى. تمتلك نيماتودا Caenorhabditis اثنين من المصابيح البلعومية ، وكلاهما مرئي مع تكبير كاف وإضاءة منقولة وتباين مع العديد من أصناف الديدان الخيطية الأخرى. الديدان الخيطية Caenorhabditis لها ذيل مدبب ، في حين أن بعض الديدان الخيطية الأخرى لها ذيل مستدير. - جمع الديدان الخيطية Caenorhabditis تحت المجهر تشريح في أقل قدر ممكن من السائل باستخدام ماصة 20-100 ميكرولتر. انقل الديدان الخيطية المجمعة مباشرة إلى 1-3 مل من M9-T المعقم في طبق بتري معقم 3 سم لغسل الديدان لإزالة الميكروبات غير المرفقة (الخطوة 3.3) أو ، بدلا من ذلك ، نقل الديدان الفردية إلى طبق به وسط نمو الديدان الخيطية (NGM) لإنشاء مجموعة من الديدان (الخطوة 3.4).
- اغسل الديدان الخيطية لإزالة الميكروبات من خارج الديدان الخيطية.
ملاحظة: يثري هذا البروتوكول بكتيريا الأمعاء ولكنه لا يقضي تماما على البكتيريا الملتصقة ببشرة الدودة.- احتضان الديدان الخيطية لمدة 10-15 دقيقة في M9-T.
- ماصة الديدان الخيطية في أقل قدر ممكن من السائل في 1-3 مل من M9-T الطازجة في طبق بتري معقم جديد 3 سم.
- كرر الحضانة ونقل الديدان الخيطية إلى M9-T الطازجة مرتين أخريين.
ملاحظة: يمكن القيام بالخطوات التالية إما مع الديدان الفردية أو مع مجموعات الديدان. يمكن الآن استخدام الديدان لتوصيف C. elegans والميكروبات المرتبطة بها ، وكذلك عزل البكتيريا (الخطوتان 4 و 5). بدلا من ذلك ، يمكن نقلها بشكل فردي إلى لوحات NGM لإنشاء مجموعة من الديدان (الخطوة 3.4).
- للحصول على مجموعة من الديدان ، ماصة نيماتودا فردية إلى لوحة NGM.
ملاحظة: فقط الديدان الخيطية الخنثى مثل C. elegans أو C . briggsae هي القادرة على إنتاج ذرية من الديدان المفردة. ومع ذلك ، قد تستمر الديدان المفردة من الأنواع الأخرى في إنتاج ذرية إذا كانت قد تزاوجت بالفعل.- عادة ما تحتوي الديدان الخيطية المعزولة بشكل طبيعي على بكتيريا في أمعائها ، والتي تسقطها على ألواح NGM ، حيث تنمو هذه البكتيريا وتكون متاحة كغذاء ل C. elegans. لا تضيف كائنات غذائية منفصلة مثل كائن الغذاء المختبري القياسي ، سلالة الإشريكية القولونية OP50.
ملاحظة: تؤثر تربية الديدان على الصفائح على تكوين المجتمع البكتيري المرتبط ، ومع ذلك لا يزال التكوين مشابها لتكوين عزلات C. elegans الطبيعية 6,7. - اسمح للديدان الخيطية بالتكاثر لمدة تصل إلى 10 أيام عند درجة الحرارة المناسبة (على سبيل المثال ، 15-20 درجة مئوية للمواقع المعتدلة). قم بتجميد هذه الديدان الخيطية (الخطوة 3.5) أو استخدمها لتوصيف C. elegans والميكروبات المرتبطة بها وكذلك عزل الميكروبات (الخطوتان 4 و 5).
- عادة ما تحتوي الديدان الخيطية المعزولة بشكل طبيعي على بكتيريا في أمعائها ، والتي تسقطها على ألواح NGM ، حيث تنمو هذه البكتيريا وتكون متاحة كغذاء ل C. elegans. لا تضيف كائنات غذائية منفصلة مثل كائن الغذاء المختبري القياسي ، سلالة الإشريكية القولونية OP50.
- تجميد الديدان الخيطية للتخزين طويل الأجل
- اترك الديدان الخيطية على الأطباق حتى تختفي بكتيريا الطعام ، وهناك مراحل يرقات صغيرة بشكل أساسي على اللوحات. اغسل الديدان من الألواح في 1.5 مل من S-buffer.
- امزج 500 ميكرولتر من S-buffer المحتوي على الديدان جيدا مع 500 ميكرولتر من 30٪ (v / v) من الجلسرين في S-buffer في أنبوب معقم سعة 2 مل. قم بتجميد الأنابيب على الفور عند -80 درجة مئوية للتخزين طويل الأجل ، وإلا فقد يضر الجلسرين بالديدان الخيطية.
4. تحضير الديدان للتعرف الجزيئي على C. elegans والميكروبات
- لتحديد غير متحيز للميكروبات المرتبطة بالديدان الخيطية ، قم بإعداد صفيحة 96 بئرا بثلاث حبات معقمة 1 مم ، و 19.5 ميكرولتر من محلول تفاعل البوليميراز المتسلسل ، و 0.5 ميكرولتر من بروتيناز K (20 مجم / مل) لكل بئر. ماصة فرد ، وغسل الديدان الخيطية إلى كل بئر ، ونقل أقل قدر ممكن من السائل.
- يمكن أيضا استخدام مجموعات الديدان. لهذا ، اغسل الديدان من الألواح باستخدام M9-buffer وانقل ~ 300 ميكرولتر من M9-buffer المحتوي على الديدان إلى أنابيب 2 مل مع 10-15 حبة.
- قم بتفتيت الديدان الخيطية باستخدام خالط الخرز (على سبيل المثال ، ضرب الخرز لمدة 3 دقائق عند 30 هرتز). قم بالطرد المركزي للوحة أو الأنابيب لفترة وجيزة لإيصال السائل إلى القاع (على سبيل المثال ، لمدة 10 ثوان عند 8000 × جم في درجة حرارة الغرفة [RT]).
- تحديد C . ايليجانس
- عزل الحمض النووي للديدان الخيطية الفردية عن طريق تسخين العينات في دورة PCR لمدة 1 ساعة عند 50 درجة مئوية و 15 دقيقة عند 95 درجة مئوية. عزل الحمض النووي لمجموعات الديدان باستخدام أي طريقة عزل مفضلة (مثال بروتوكولات طرق العزل المختلفة باستخدام مجموعات تجارية 7,9). قم بتجميد الحمض النووي عند -20 درجة مئوية للتخزين طويل الأجل.
- لتحديد C. elegans ، استخدم الحمض النووي والزوج التمهيدي nlp30-F (الجدول 1 ، 5'-ACACATACAACTGATCACTCA-3') و nlp30-R (الجدول 1 ، 5'-TACTTTCCCCATCCGTATC-3') في تفاعل البوليميراز المتسلسل ، باتباع تعليمات مورد Taq المفضل.
- استخدم شروط تفاعل البوليميراز المتسلسل التالية: خطوة التمسخ الأولية عند 95 درجة مئوية لمدة 2 دقيقة ، تليها 35 دورة من 95 درجة مئوية لمدة 45 ثانية ، و 55 درجة مئوية لمدة 30 ثانية ، و 72 درجة مئوية لمدة 1 دقيقة ، وخطوة استطالة نهائية عند 72 درجة مئوية لمدة 5 دقائق. تنتج C. elegans منتج PCR بقوة 154 نقطة أساس.
- قم بتوصيف البكتيريا المرتبطة بالديدان الخيطية عبر تسلسل 16S amplicon لمنطقة V3-V4 ، باستخدام الحمض النووي المعزول.
- قم بإعداد مكتبة 16S باستخدام البادئات 16S المفضلة واتبع بروتوكول مجموعة أدوات إعداد المكتبة. أحد الخيارات هو استخدام البادئات 341F (الجدول 1 ، 5'-CCTACGGGNGGCWGCAG-3') و 806R (الجدول 1 ، 5'-GACTACHVGGGTATCTAATCC-3') التي تغطي منطقة V3-V4 من جين 16S rRNA ، مما ينتج عنه تسلسلات يمكن تصنيفها بقواعد بيانات قياسية بدقةجيدة 7.
ملاحظة: كمية الحمض النووي المدخلات أمر بالغ الأهمية في هذه الخطوة. سيكون الحمض النووي المكتسب من الديدان المفردة أقل بكثير من الحمض النووي الذي تم الحصول عليه من مجموعات الديدان. بالنسبة للديدان المفردة ، قد يكون من الضروري زيادة كمية الحمض النووي المدخلات أو زيادة كمية دورات تفاعل البوليميراز المتسلسل7. - يمكن تسلسل المكتبات على منصة تسلسل باستخدام مجموعة تسلسل مناسبة.
ملاحظة: هنا ، يتم استخدام منصة MiSeq مع مجموعة كاشف MiSeq المناسبة. يتم تحسين الكواشف باستمرار ويجب اختيارها وفقا لأحدث المعايير.
- قم بإعداد مكتبة 16S باستخدام البادئات 16S المفضلة واتبع بروتوكول مجموعة أدوات إعداد المكتبة. أحد الخيارات هو استخدام البادئات 341F (الجدول 1 ، 5'-CCTACGGGNGGCWGCAG-3') و 806R (الجدول 1 ، 5'-GACTACHVGGGTATCTAATCC-3') التي تغطي منطقة V3-V4 من جين 16S rRNA ، مما ينتج عنه تسلسلات يمكن تصنيفها بقواعد بيانات قياسية بدقةجيدة 7.
5. عزل وزراعة البكتيريا المرتبطة بالديدان الخيطية (الشكل 2)
- لعزل البكتيريا ، قم بإعداد صفيحة 96 بئرا بثلاث خرزات معقمة 1 مم ، و 20 ميكرولتر من M9-buffer لكل بئر ، وماصة للديدان الخيطية المغسولة الفردية إلى كل بئر ، ونقل أقل قدر ممكن من السائل.
ملاحظة: بدلا من ذلك ، يمكن غسل مجموعات الديدان من الألواح باستخدام M9-buffer و ~ 300 ميكرولتر من M9-buffer المحتوي على الديدان يمكن نقلها إلى أنابيب 2 مل مع 10-15 حبة. في جميع الحالات ، يجب أن تأتي الديدان من مزرعة تم تحديدها على أنها C. elegans باستخدام الخطوات 4.1-4.4. - قم بتفتيت الديدان الخيطية باستخدام خالط الخرز (على سبيل المثال ، ضرب الخرز لمدة 3 دقائق عند 30 هرتز). قم بالطرد المركزي للوحة أو الأنابيب لفترة وجيزة لإيصال السائل إلى القاع (على سبيل المثال ، لمدة 10 ثوان عند 8000 × جم عند RT).
ملاحظة: أدى استخدام هذه الطريقة إلى عزل الأصناف البكتيرية المشابهة لتلك التي كشف عنها تسلسل amplicon 16S rDNAamplicon 7 ، مما يشير إلى أن ضرب الخرزة الموصوف يترك معظم الخلايا البكتيرية سليمة. - اجمع المادة الطافية ، وقم بتخفيفها بشكل متسلسل في الساعة 1:10 ، وقم بصفيحة تصل إلى 100 ميكرولتر على ألواح أجار 9 سم.
- لضمان إمكانية زراعة معظم البكتيريا ، استخدم مجموعة متنوعة من الوسائط البديلة ذات التركيبات الغذائية المختلفة ، بما في ذلك أجار الصويا المخفف (TSA ، تخفيف 1:10) ، أو أجار MacConkey ، أو أجار الجلوكوز Sabouraud ، أو أجار سكر العنب في البطاطس ، أو أجار سكر العنب الخميرة الببتون.
- احتضان الألواح في متوسط ظروف درجة الحرارة لموقع أخذ العينات (على سبيل المثال ، درجات حرارة تتراوح بين 15-20 درجة مئوية للمواقع المعتدلة) لمدة 24-48 ساعة.
- استخدم تقنية الخطوط الثلاثة القياسية وحلقة معقمة للحصول على مزارع بكتيرية نقية (الشكل 2).
- اختر مستعمرة واحدة من صفيحة باستخدام حلقة معقمة أو عود أسنان وقم بخطها على صفيحة أجار جديدة تحتوي على نفس وسط الأجار المستخدم للتنقية. تأكد من استخدام ما يقرب من 1/3 فقط من اللوحة.
- إما استخدام حلقة معقمة جديدة أو تعقيم حلقة قابلة لإعادة الاستخدام واسحبها خلال الخط الأول لإنشاء خط ثان على 1/3 آخر من نفس اللوحة.
- كرر هذه الخطوة عن طريق سحب حلقة معقمة خلال الخط الثاني.
- احتضان اللوحة في نفس ظروف النمو المستخدمة للعزل. يجب أن تؤدي هذه التقنية إلى مستعمرات مفردة تنمو في منطقة الخط الثالث.
ملاحظة: قد يكون من الضروري تكرار خطوة التنقية عدة مرات لأن العزلات الطبيعية تميل إلى تكوين الأغشية الحيوية و / أو الركام.
- قم بزراعة المستعمرات النقية في وسط سائل (من نفس نوع وسط أجار) باستخدام نفس درجة الحرارة ووقت النمو على النحو الوارد أعلاه (الخطوة 5.4)
- تحضير مخزون البكتيريا بإضافة 300 ميكرولتر من المزرعة البكتيرية إلى 200 ميكرولتر من 86٪ (v / v) من الجلسرين (في وسط النمو المعني ، على سبيل المثال ، TSB) والماصة لأعلى ولأسفل للخلط بشكل صحيح. بدلا من ذلك ، قم بإعداد مخزون DMSO عن طريق خلط 50 ميكرولتر من الثقافة البكتيرية مع 50 ميكرولتر من 7٪ (v / v) DMSO. تجمد عند -80 درجة مئوية للتخزين طويل الأجل.
- توصيف البكتيريا باستخدام تسلسل جين الحمض النووي الريبوزي الريبوسومي الكامل 16S
- استخراج الحمض النووي البكتيري من الثقافات السائلة النقية باستخدام تقنية مناسبة (على سبيل المثال ، مجموعة استخراج الحمض النووي ؛ من التجربة ، يعمل بروتوكول الاستخراج القائم على CTAB بشكل جيد للغاية22).
- تضخيم جين 16S rRNA باستخدام البادئات 27F (الجدول 1 ، 5'-GAGAGTTTGATCCTGGCTCAG-3') و 1495R (الجدول 1 ، 5'-CTACGGCTACCTTGTTACGA -3')28 وشروط تفاعل البوليميراز المتسلسل التالية: 95 درجة مئوية ، 2 دقيقة ، 22x (95 درجة مئوية ، 30 ثانية ؛ 55 درجة مئوية ، 30 ثانية ؛ 72 درجة مئوية ، 100 ثانية) ، وفترة تمديد نهائية عند 72 درجة مئوية ، 5 دقائق.
- من أجل الحصول على التسلسلات الكاملة ، استخدم أيضا اثنين من بادئات التسلسل الداخلي ، مثل 701F (الجدول 1 ، 5'-GTGTAGCGGTGAAATGCG-3') و 785R (الجدول 1 ، 5'-GGATTAGATACCCTGGTAGTCC-3')6.
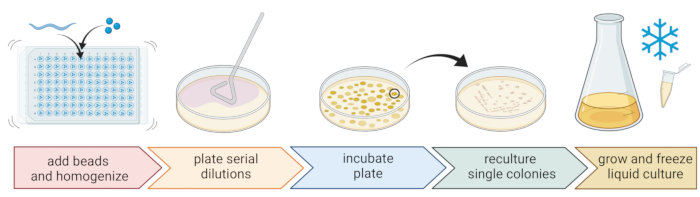
الشكل 2: تحديد الأنواع وعزل البكتيريا الفردية. يتم تقسيم الديدان الخيطية الفردية باستخدام خالط حبة ، ويتم عزل الحمض النووي لتحديد الأنواع عن طريق تفاعل البوليميراز المتسلسل أو التسلسل. بدلا من ذلك ، يتم تخفيف مادة الديدان الخيطية المكسورة بشكل متسلسل ومطلية على ألواح متوسطة النمو. يتم تحضين الصفائح حتى تظهر المستعمرات البكتيرية ، ويتم ربط المستعمرات المفردة بألواح جديدة للحصول على ثقافات نقية. تستخدم المستعمرات المفردة من المزارع النقية لزراعة مزارع بكتيرية سائلة لإعداد مخزون بكتيري للتخزين طويل الأجل عند -80 درجة مئوية. الشكل الذي تم إنشاؤه باستخدام BioRender.com. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Access restricted. Please log in or start a trial to view this content.
النتائج
كثيرا ما توجد الديدان الخيطية C. elegans في الفواكه المتحللة ، مثل التفاح ، وكذلك عينات السماد. في شمال ألمانيا ، تم العثور على C. elegans وكذلك الأنواع الجنيسة (خاصة C. remanei ولكن أيضا C. briggsae) بشكل أساسي من سبتمبر حتى2 نوفمبر. توجد الديدان الخيطية بشكل شائع في المواد ال?...
Access restricted. Please log in or start a trial to view this content.
Discussion
الديدان الخيطية Caenorhabditis elegans هي واحدة من أكثر الكائنات النموذجية التي تمت دراستها بشكل مكثف في البحوث البيولوجية. تم تقديمه من قبل سيدني برينر في 1960s ، في الأصل لفهم تطور ووظيفة الجهاز العصبي29. منذ ذلك الحين ، أصبحت C. elegans نموذجا قويا لدراسة العمليات الأساسية في جميع ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
نعلن أنه ليس لدينا تضارب في المصالح.
Acknowledgements
نحن نعترف بالدعم المالي من مؤسسة العلوم الألمانية (المشروعان A1.1 و A1.2 لمركز الأبحاث التعاونية 1182 حول أصل ووظيفة الكائنات الحية). نشكر أعضاء مختبر شولنبورغ على نصائحهم ودعمهم.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
References
- Schulenburg, H., Félix, M. -A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4(2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19(2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485(2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728(2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604(2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24(2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59(2012).
- Barrière, A., Félix, M. -A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and related nematodes. WormBook. , ed. The C elegans research community 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670(2016).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved