Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Aislamiento y caracterización de la microbiota natural del nematodo modelo Caenorhabditis elegans
En este artículo
Resumen
Caenorhabditis elegans es una de las principales especies modelo en biología, sin embargo, casi todas las investigaciones se realizan en ausencia de sus microbios naturalmente asociados. Los métodos descritos aquí ayudarán a mejorar nuestra comprensión de la diversidad de microbios asociados como base para futuras investigaciones funcionales de C. elegans.
Resumen
El nematodo Caenorhabditis elegans interactúa con una gran diversidad de microorganismos en la naturaleza. En general, C. elegans se encuentra comúnmente en materia vegetal podrida, especialmente frutas podridas como manzanas o en montones de compost. También se asocia con ciertos huéspedes invertebrados como y cochinillas. Estos hábitats son ricos en microbios, que sirven como alimento para C. elegans y que también pueden colonizar persistentemente el intestino del nematodo. Hasta la fecha, la diversidad exacta y la consistencia de la microbiota nativa de C. elegans en todos los hábitats y ubicaciones geográficas no se comprende completamente. Aquí, describimos un enfoque adecuado para aislar C. elegans de la naturaleza y caracterizar la microbiota de los gusanos. Los nematodos pueden aislarse fácilmente del material de compost, manzanas podridas, o atraídos colocando manzanas en montones de compost. El mejor momento para encontrar C. elegans en el hemisferio norte es de septiembre a noviembre. Los gusanos se pueden lavar del material de sustrato recolectado sumergiendo el sustrato en una solución tampón, seguido de la recolección de nematodos y su transferencia al medio de crecimiento de nematodos o tampón de PCR para su posterior análisis. Además, ilustramos cómo las muestras se pueden utilizar para aislar y purificar los microorganismos asociados al gusano y para procesar gusanos para el análisis de ARN ribosómico 16S de la composición de la comunidad de microbiota. En general, los métodos descritos pueden estimular nuevas investigaciones sobre la caracterización de la microbiota de C. elegans en hábitats y ubicaciones geográficas, ayudando así a obtener una comprensión integral de la diversidad y estabilidad de la microbiota del nematodo como base para futuras investigaciones funcionales.
Introducción
En la naturaleza, C. elegans se encuentra comúnmente en materia vegetal podrida, especialmente frutas podridas como manzanas o en montones de compost1. También se asocia con ciertos huéspedes invertebrados como y cochinillas 2,3. Estos hábitats son ricos en microbios, que no solo sirven como alimento para el gusano, sino que también pueden formar asociaciones estables con él. La información sobre la diversidad de microorganismos asociados naturalmente solo se publicó en 2016 4,5,6. Desde entonces, estos y solo unos pocos estudios más recientes han revelado que C. elegans se asocia con una variedad de bacterias y hongos, más comúnmente incluyendo bacterias del género Pseudomonas, Enterobacter, Ochrobactrum, Erwinia, Comamonas, Gluconobacter y varios otros 6,7,8. Varias bacterias asociadas pueden colonizar de manera estable el intestino del gusano, aunque no todas 6,9,10,11,12. Es probable que sean de importancia clave para nuestra comprensión de la biología de C. elegans porque pueden proporcionar nutrición, proteger contra patógenos y posiblemente otros factores estresantes, y afectar rasgos centrales de la historia de vida, como la tasa reproductiva, el desarrollo o las respuestas conductuales.
Como ejemplo, los aislados naturalmente asociados de los géneros Pseudomonas, Ochrobactrum, y también Enterobacter o Gluconobacter pueden proteger al gusano de la infección y muerte de patógenos de distintas maneras 5,6,11,13,14. Un aislado específico del género Comamonas influye en la respuesta dietética de los nematodos, el desarrollo, la esperanza de vida y la fertilidad15,16,17. Las bacterias de Providencia producen el neuromodulador tiramina y, por lo tanto, modulan la actividad del sistema nervioso del huésped y las respuestas conductuales resultantes18. Se demostró que un conjunto de diferentes bacterias naturalmente asociadas afectan la tasa de crecimiento de la población, la fertilidad y las respuestas conductuales 5,6,9,11,19.
Hasta la fecha, la diversidad exacta y la consistencia de la microbiota nativa de C. elegans en todos los hábitats y ubicaciones geográficas no se comprenden completamente, y aún no se han descubierto otras asociaciones entre el gusano y los microbios de su entorno. Varios estudios previos utilizaron cepas bacterianas aisladas de algún ambiente del suelo, hábitats naturales de C. elegans o de experimentos de mesocosmos (es decir, ambientes de laboratorio que recrean hábitats naturales) con cepas de laboratorio de C. elegans 4,5,20. A pesar de que estos estudios obtuvieron nuevos conocimientos sobre la influencia de los microbios en rasgos específicos de nematodos (por ejemplo, el metabolismo de los nematodos21), la relevancia de estas interacciones para la biología de C. elegans en la naturaleza no está clara. Por lo tanto, este manuscrito describe los métodos para aislar directamente C. elegans de la naturaleza y para aislar y posteriormente caracterizar los microbios naturalmente asociados tanto de gusanos individuales como de grupos de gusanos. Los métodos descritos son una versión actualizada y mejorada de los procedimientos utilizados anteriormente para el aislamiento y caracterización de C. elegans natural y su microbiota nativa 2,6,7. Teniendo en cuenta que C. elegans se encuentra ampliamente en la materia vegetal en descomposición en todo el mundo (especialmente en frutas podridas, regiones templadas y en otoño)1,2,22,23,24,25, este protocolo puede ser aplicado por cualquier laboratorio siempre que haya interés en relacionar C. elegans rasgos a microbios naturalmente asociados y, por lo tanto, un contexto más naturalmente relevante. Este último es fundamental para una comprensión completa de la biología del nematodo porque se sabe por una diversidad de otros sistemas huéspedes que la microbiota asociada puede afectar diversas características de la historia de vida26, un aspecto que actualmente se descuida en gran medida en la multitud de estudios de C. elegans en casi todas las disciplinas de ciencias de la vida.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación de buffers y medios
- Preparar el tampón S añadiendo 5,85 g de NaCl, 1,123 g de K 2 HPO 4, 5,926 g de KH2PO4 y 1 L deH2Odesionizado a un matraz y autoclave durante 20 min a 121 °C.
- Prepare un medio viscoso agregando tampón S que contenga 1.2% (p/v) de hidroximetilcelulosa (la sustancia que causa la viscosidad del medio), 5 mg / ml de colesterol, 1 mM de MgSO4, 1 mM de CaCl2 y 0.1% (v / v) de acetona. Autoclave y agita el medio viscoso hasta que sea completamente homogéneo.
NOTA: Esto puede tardar varias horas. Además, el tampón S se puede preparar directamente y agregar al medio viscoso sin esterilización previa. - Preparar el tampón M9 añadiendo 3 g de KH 2 PO 4, 6 g de NA 2 HPO4·2 H2O, 5 g de NaCl y1 L deH2Odesionizado a un matraz de 1 L. Autoclave la solución, y después de enfriar, añadir 1 mL de 1 MMgSO4 (123,24 g de MgSO4·7H2O en 500 mL deH2Odesionizado, filtro esterilizado).
- Prepare una solución madre de Triton X-100 al 10% (v/v) mezclando 5 ml de Triton X-100 con 45 ml de tampón M9. Filtrar-esterilizar la solución con un filtro de 0,2 μm.
- Prepare el tampón M9 con Triton X-100 (M9-T) agregando 2,5 ml de la solución madre Triton X-100 al 10% (v/v) a 1 L de tampón M9 después del autoclave para obtener M9-T al 0,025% (v/v).
- Prepare glicerol al 30% (v/v) en tampón S mezclando 15 ml de glicerol estéril al 100% y 35 ml de tampón S estéril en un tubo de 50 ml.
2. Preparación de muestras ambientales (Figura 1)
- Recoja muestras ambientales como compost o frutas podridas y coloque cada muestra en una bolsa de plástico individual, tubo u otro recipiente limpio.
NOTA: Para atraer nematodos, las manzanas se pueden colocar en compost varias semanas antes del muestreo. - Extienda los trozos de la muestra ambiental uniformemente en una placa de Petri vacía y estéril de 9 cm.
NOTA: Las muestras con niveles más altos de descomposición tienen más probabilidades de contener C. elegans. Opcionalmente, se puede usar una placa de Petri llena de medio de agar sin peptona (PFM) para aumentar el contraste. - Cubra la muestra cuidadosamente con aproximadamente 20 ml de medio viscoso estéril.
NOTA: Los nematodos flotan a la superficie dentro de 1-2 h. El medio viscoso ralentiza el movimiento de los gusanos y facilita su muestreo. El tampón M9 estéril se puede utilizar como alternativa, sin embargo, el movimiento del gusano será más rápido.
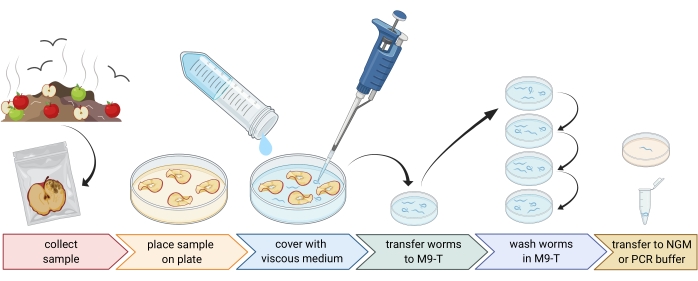
Figura 1: Aislamiento de nematodos de sustratos. Las muestras de sustrato se colocan en placas de Petri vacías y se cubren con medio viscoso para eliminar los nematodos. Los nematodos se transfieren a M9-T y se lavan repetidamente para eliminar las bacterias del exterior. Los nematodos individuales se pueden usar para el aislamiento del ADN, el aislamiento de bacterias asociadas o colocarse en placas de agar para cultivar poblaciones de gusanos. Figura creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
3. Aislamiento de nematodos Caenorhabditis (Figura 1)
- Búsqueda de nematodos Caenorhabditis utilizando un microscopio de disección, siguiendo las pautas presentadas en el capítulo de WormBook sobre el aislamiento de C. elegans y nematodos relacionados por Barrière y Félix27.
NOTA: Caenorhabditis hermafroditas/hembras tienen un intestino de color marrón claro (bajo iluminación transmitida). Además, las células intestinales tienen núcleos celulares grandes, que son visibles como puntos blancos. Por el contrario, otras especies de nematodos a menudo tienen un intestino marrón oscuro y pueden mostrar asimetría anteroposterior en la intensidad del pigmento y generalmente sin núcleos visibles. La vulva de Caenorhabditis hermafroditas/hembras adultas se encuentra en el centro del animal. Esta localización de la vulva no siempre es mostrada por otros taxones de nematodos. Los nematodos Caenorhabditis poseen dos bulbos faríngeos, que son visibles con suficiente aumento y transmiten iluminación y contraste con muchos otros taxones de nematodos. Los nematodos Caenorhabditis tienen una cola puntiaguda, mientras que algunos otros nematodos tienen una cola redonda. - Recoger los nematodos Caenorhabditis bajo un microscopio de disección en el menor líquido posible utilizando una pipeta de 20-100 μL. Transfiera los nematodos recolectados directamente a 1-3 ml de M9-T estéril en una placa de Petri estéril de 3 cm para lavar los gusanos para eliminar los microbios no adheridos (paso 3.3) o, alternativamente, transfiera gusanos individuales a una placa con medio de crecimiento de nematodos (NGM) para establecer una población de gusanos (paso 3.4).
- Lave los nematodos para eliminar los microbios del exterior del nematodo.
NOTA: Este protocolo enriquece las bacterias intestinales, pero no elimina completamente las bacterias que se adhieren a la cutícula del gusano.- Incubar los nematodos durante 10-15 min en M9-T.
- Pipetear los nematodos en el menor líquido posible en 1-3 ml de M9-T fresco en una nueva placa de Petri estéril de 3 cm.
- Repita la incubación y transfiera los nematodos a M9-T fresco dos veces más.
NOTA: Los siguientes pasos se pueden realizar con gusanos individuales o con poblaciones de gusanos. Los gusanos ahora se pueden usar para la caracterización de C. elegans y microbios asociados, así como para el aislamiento de bacterias (pasos 4 y 5). Alternativamente, pueden transferirse individualmente a placas NGM para establecer una población de gusanos (paso 3.4).
- Para obtener una población de gusanos, pipetear un nematodo individual a una placa NGM.
NOTA: Sólo los nematodos hermafroditas como C. elegans o C. briggsae son capaces de producir descendencia a partir de gusanos individuales. Sin embargo, los gusanos individuales de otras especies aún pueden producir descendencia si ya estaban apareados.- Los nematodos aislados naturalmente generalmente contienen bacterias en su intestino, que arrojan a las placas NGM, donde estas bacterias crecen y están disponibles como alimento para C. elegans. No agregue organismos alimentarios separados como el organismo alimenticio estándar de laboratorio, E. coli cepa OP50.
NOTA: La cría de gusanos en placas influye en la composición de la comunidad bacteriana asociada, sin embargo, la composición sigue siendo comparable a la de los aislados naturales de C. elegans 6,7. - Permita que los nematodos proliferen hasta 10 días a la temperatura adecuada (por ejemplo, 15-20 °C para lugares templados). Congelar estos nematodos (paso 3.5) o utilizarlos para la caracterización de C. elegans y microbios asociados, así como para el aislamiento de microbios (pasos 4 y 5).
- Los nematodos aislados naturalmente generalmente contienen bacterias en su intestino, que arrojan a las placas NGM, donde estas bacterias crecen y están disponibles como alimento para C. elegans. No agregue organismos alimentarios separados como el organismo alimenticio estándar de laboratorio, E. coli cepa OP50.
- Congelación de nematodos para almacenamiento a largo plazo
- Deje los nematodos en las placas hasta que las bacterias de los alimentos hayan desaparecido, y haya principalmente pequeñas etapas larvales en las placas. Lave los gusanos de las placas en 1,5 ml de tampón S.
- Mezcle bien 500 μL de tampón S que contiene gusanos con 500 μL de glicerol al 30% (v/v) en tampón S en un tubo estéril de 2 ml. Congele los tubos rápidamente a -80 °C para su almacenamiento a largo plazo, de lo contrario el glicerol puede dañar los nematodos.
4. Preparación de los gusanos para la identificación molecular de C. elegans y microbios
- Para la identificación imparcial de microbios asociados a nematodos, prepare una placa de 96 pocillos con tres perlas estériles de 1 mm, 19,5 μL de tampón de PCR y 0,5 μL de proteinasa K (20 mg/ml) por pocillo. Pipetear un nematodo individual lavado a cada pocillo, transfiriendo la menor cantidad de líquido posible.
- También se pueden utilizar poblaciones de gusanos. Para esto, lave los gusanos de las placas con M9-buffer y transfiera ~300 μL de membrujo M9 que contiene gusanos a tubos de 2 mL con 10-15 perlas.
- Rompa los nematodos usando un homogeneizador de cuentas (por ejemplo, batir cuentas durante 3 minutos a 30 Hz). Centrifugar la placa o los tubos brevemente para que el líquido llegue al fondo (p. ej., durante 10 s a 8000 x g a temperatura ambiente [RT]).
- Identificación de C. elegans
- Aislar el ADN de nematodos individuales calentando las muestras en un ciclador de PCR durante 1 h a 50 °C y 15 min a 95 °C. Aislar el ADN de las poblaciones de gusanos utilizando cualquier método de aislamiento de elección (protocolos de ejemplo de diferentes métodos de aislamiento utilizando kits comerciales 7,9). Congelar el ADN a -20 °C para su almacenamiento a largo plazo.
- Para la identificación de C. elegans, utilice el ADN y el par de cebadores nlp30-F (Tabla 1, 5'-ACACATACAACTGATCACTCA-3') y nlp30-R (Tabla 1, 5'-TACTTTCCCCATCCGTATC-3') en una PCR, siguiendo las instrucciones de un proveedor de Taq de elección.
- Utilice las siguientes condiciones de PCR: etapa de desnaturalización inicial a 95 °C durante 2 min, seguida de 35 ciclos de 95 °C durante 45 s, 55 °C durante 30 s, 72 °C durante 1 min, y una etapa final de elongación a 72 °C durante 5 min. C. elegans produce un producto de PCR de 154 pb.
- Caracterizar las bacterias asociadas a nematodos mediante la secuenciación del amplicón 16S de la región V3-V4, utilizando el ADN aislado.
- Prepare una biblioteca 16S con los cebadores 16S de su elección y siga el protocolo del kit de preparación de la biblioteca. Una opción es utilizar los cebadores 341F (Tabla 1, 5'-CCTACGGGNGGCWGCAG-3') y 806R (Tabla 1, 5'-GACTACHVGGGTATCTAATCC-3') cubriendo la región V3-V4 del gen 16S rRNA, lo que da como resultado secuencias que pueden clasificarse con bases de datos estándar con buena resolución7.
NOTA: La cantidad de ADN de entrada es crítica en este paso. El ADN adquirido de gusanos individuales va a ser mucho menor que el obtenido de las poblaciones de gusanos. Para gusanos individuales, podría ser necesario aumentar la cantidad de ADN de entrada o aumentar la cantidad de ciclos de PCR7. - Las bibliotecas se pueden secuenciar en una plataforma de secuenciación utilizando un kit de secuenciación adecuado.
NOTA: Aquí, la plataforma MiSeq se utiliza con un kit de reactivos MiSeq adecuado. Los reactivos se mejoran constantemente y deben elegirse según los estándares más nuevos.
- Prepare una biblioteca 16S con los cebadores 16S de su elección y siga el protocolo del kit de preparación de la biblioteca. Una opción es utilizar los cebadores 341F (Tabla 1, 5'-CCTACGGGNGGCWGCAG-3') y 806R (Tabla 1, 5'-GACTACHVGGGTATCTAATCC-3') cubriendo la región V3-V4 del gen 16S rRNA, lo que da como resultado secuencias que pueden clasificarse con bases de datos estándar con buena resolución7.
5. Aislamiento y cultivo de bacterias asociadas a nematodos (Figura 2)
- Para aislar las bacterias, prepare una placa de 96 pocillos con tres perlas estériles de 1 mm, 20 μL de tampón M9 por pocillo y pipete un nematodo lavado individual a cada pocillo, transfiriendo la menor cantidad de líquido posible.
NOTA: Alternativamente, las poblaciones de gusanos se pueden lavar de las placas con tampón M9 y ~ 300 μL de tampón M9 que contiene gusanos se pueden transferir a tubos de 2 ml con 10-15 perlas. En todos los casos, los gusanos deben provenir de un cultivo que se identificó como C. elegans siguiendo los pasos 4.1-4.4. - Rompa los nematodos usando un homogeneizador de cuentas (por ejemplo, batir cuentas durante 3 minutos a 30 Hz). Centrifugar la placa o los tubos brevemente para que el líquido llegue al fondo (por ejemplo, durante 10 s a 8000 x g en RT).
NOTA: El uso de este método condujo al aislamiento de taxones bacterianos similares a los revelados por la secuenciación del amplicón de ADNr 16S7, lo que sugiere que el batido de cuentas descrito deja intactas la mayoría de las células bacterianas. - Recoger el sobrenadante, diluirlo en serie a 1:10 y colocar hasta 100 μL en placas de agar de 9 cm.
- Para asegurarse de que la mayoría de las bacterias se puedan cultivar, use una variedad de medios alternativos con diferentes composiciones de nutrientes, incluido el agar de soja tripticasa diluido (TSA, dilución 1:10), agar MacConkey, agar de glucosa Sabouraud, agar dextrosa de papa o agar dextrosa peptona de levadura.
- Incubar las placas a las condiciones de temperatura media del lugar de muestreo (por ejemplo, temperaturas entre 15-20 °C para lugares templados) durante 24-48 h.
- Utilice la técnica estándar de tres rayas y un asa estéril para obtener cultivos bacterianos puros (Figura 2).
- Elija una sola colonia de una placa usando un asa estéril o palillo de dientes y estézcala en una nueva placa de agar que contenga el mismo medio de agar que se usa para la purificación. Asegúrese de usar solo aproximadamente 1/3 del plato.
- Use un nuevo bucle estéril o esterilice un bucle reutilizable y arrástrelo a través de la primera raya para crear una segunda raya en otro 1/3 de la misma placa.
- Repita este paso arrastrando un bucle estéril a través de la segunda raya.
- Incubar la placa en las mismas condiciones de crecimiento utilizadas para el aislamiento. Esta técnica debe dar lugar a colonias individuales que crezcan en el área de la tercera raya.
NOTA: Puede ser necesario repetir el paso de purificación varias veces, ya que los aislados naturales tienden a formar biopelículas y / o agregados.
- Cultivar las colonias puras en un medio líquido (del mismo tipo que el medio de agar) utilizando la misma temperatura y tiempo de crecimiento que el anterior (paso 5.4)
- Prepare las reservas de bacterias agregando 300 μL del cultivo bacteriano a 200 μL de glicerol al 86% (v / v) (en el medio de crecimiento respectivo, por ejemplo, TSB) y pipetear hacia arriba y hacia abajo para mezclar correctamente. Alternativamente, prepare las existencias de DMSO mezclando 50 μL de cultivo bacteriano con 50 μL de DMSO al 7% (v / v). Congelar a -80 °C para su almacenamiento a largo plazo.
- Caracterización de bacterias mediante la secuenciación del gen completo del ARN ribosómico 16S
- Extraer el ADN bacteriano de cultivos líquidos puros utilizando una técnica adecuada (por ejemplo, un kit de extracción de ADN; por experiencia, un protocolo de extracción basado en CTAB funciona muy bien22).
- Amplificar el gen 16S rRNA utilizando los cebadores 27F (Tabla 1, 5'-GAGAGTTTGATCCTGGCTCAG-3') y 1495R (Tabla 1, 5'-CTACGGCTACCTTGTTACGA -3')28 y las siguientes condiciones de PCR: 95 °C, 2 min, 22x (95 °C, 30 s; 55 °C, 30 s; 72 °C, 100 s), y un período de extensión final a 72 °C, 5 min.
- Para adquirir las secuencias completas, utilice adicionalmente dos cebadores de secuenciación internos, como 701F (Tabla 1, 5'-GTGTAGCGGTGAAATGCG-3') y 785R (Tabla 1, 5'-GGATTAGATACCCTGGTAGTCC-3')6.
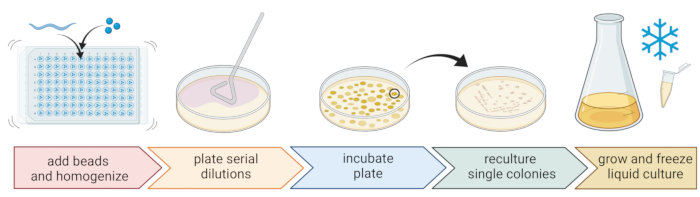
Figura 2: Identificación de especies y aislamiento de bacterias individuales. Los nematodos individuales se rompen utilizando un homogeneizador de perlas, y el ADN se aísla para la determinación de especies mediante PCR o secuenciación. Alternativamente, el material del nematodo roto se diluye en serie y se coloca en placas de medio de crecimiento. Las placas se incuban hasta que aparecen colonias bacterianas, y las colonias individuales se rayan a nuevas placas para obtener cultivos puros. Las colonias individuales de cultivos puros se utilizan para cultivar cultivos bacterianos líquidos para la preparación de reservas bacterianas para su almacenamiento a largo plazo a -80 °C. Figura creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
El nematodo C. elegans se encuentra con frecuencia en frutas en descomposición, como manzanas, y también muestras de compost. En el norte de Alemania, C. elegans, así como especies congenéricas (particularmente C. remanei pero también C. briggsae) se encuentran principalmente desde septiembre hasta el2 de noviembre. Los nematodos se encuentran más comúnmente en la materia vegetal en descomposición, especialmente en frutas podridas como manzanas o peras, ...
Access restricted. Please log in or start a trial to view this content.
Discusión
El nematodo Caenorhabditis elegans es uno de los organismos modelo más intensamente estudiados en la investigación biológica. Fue introducido por Sydney Brenner en la década de 1960, originalmente para comprender el desarrollo y la función del sistema nervioso29. Desde entonces, C. elegans se ha convertido en un poderoso modelo para estudiar procesos fundamentales en todas las disciplinas biológicas, incluida la biología del comportamiento, la neurobiología, el envejecimi...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Declaramos que no tenemos ningún conflicto de intereses.
Agradecimientos
Reconocemos el apoyo financiero de la Fundación Alemana de Ciencia (proyectos A1.1 y A1.2 del Centro de Investigación Colaborativa 1182 sobre el Origen y la Función de los Metaorganismos). Agradecemos a los miembros del laboratorio Schulenburg por sus consejos y apoyo.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| COMSOL | COMSOL | multiphysics simulation software |
Referencias
- Schulenburg, H., Félix, M. -A. The natural biotic environment of Caenorhabditis elegans. Genetics. 206 (1), 55-86 (2017).
- Petersen, C., Dirksen, P., Prahl, S., Strathmann, E. A., Schulenburg, H. The prevalence of Caenorhabditis elegans across 1.5 years in selected North German locations: the importance of substrate type, abiotic parameters, and Caenorhabditis competitors. BMC Ecology. 14 (1), 4(2014).
- Petersen, C., et al. Travelling at a slug's pace: possible invertebrate vectors of Caenorhabditis nematodes. BMC Ecology. 15 (1), 19(2015).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Félix, M. -A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences. 113 (27), 3941-3949 (2016).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14 (1), 38(2016).
- Johnke, J., Dirksen, P., Schulenburg, H. Community assembly of the native C. elegans microbiome is influenced by time, substrate and individual bacterial taxa. Environmental Microbiology. 22 (4), 1265-1279 (2020).
- Zhang, F., et al. Caenorhabditis elegans as a model for microbiome research. Frontiers in Microbiology. 8, 485(2017).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans microbiome resource. G3 Genes|Genomes|Genetics. 10 (9), 3025-3039 (2020).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2019).
- Kissoyan, K. A. B., et al. Exploring effects of C. elegans protective natural microbiota on host physiology. Frontiers in Cellular and Infection Microbiology. 12, 775728(2022).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Berg, M., et al. TGFβ/BMP immune signaling affects abundance and function of C. elegans gut commensals. Nature Communications. 10 (1), 604(2019).
- Kissoyan, K. A. B., et al. Natural C. elegans microbiota protects against infection via production of a cyclic lipopeptide of the viscosin group. Current Biology. 29 (6), 1030-1037 (2019).
- Watson, E., MacNeil, L. T., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Integration of metabolic and gene regulatory networks modulates the C. elegans dietary response. Cell. 153 (1), 253-266 (2013).
- Watson, E., et al. Interspecies systems biology uncovers metabolites affecting C. elegans gene expression and life history traits. Cell. 156 (4), 759-770 (2014).
- MacNeil, L. T., Watson, E., Arda, H. E., Zhu, L. J., Walhout, A. J. M. Diet-induced developmental acceleration independent of TOR and insulin in C. elegans. Cell. 153 (1), 240-252 (2013).
- O'Donnell, M. P., Fox, B. W., Chao, P. -H., Schroeder, F. C., Sengupta, P. A neurotransmitter produced by gut bacteria modulates host sensory behaviour. Nature. 583 (7816), 415-420 (2020).
- Snoek, B. L., et al. A multi-parent recombinant inbred line population of C. elegans allows identification of novel QTLs for complex life history traits. BMC Biology. 17 (1), 24(2019).
- Avery, L., Shtonda, B. B. Food transport in the C. elegans pharynx. Journal of Experimental Biology. 206 (14), 2441-2457 (2003).
- Zhang, J., et al. A delicate balance between bacterial iron and reactive oxygen species supports optimal C. elegans development. Cell Host & Microbe. 26 (3), 400-411 (2019).
- Petersen, C., et al. Ten years of life in compost: temporal and spatial variation of North German Caenorhabditis elegans populations. Ecology and Evolution. 5 (16), 3250-3263 (2015).
- Félix, M. -A., Duveau, F. Population dynamics and habitat sharing of natural populations of Caenorhabditis elegans and C. briggsae. BMC Biology. 10 (1), 59(2012).
- Barrière, A., Félix, M. -A. Temporal dynamics and linkage disequilibrium in natural Caenorhabditis elegans populations. Genetics. 176 (2), 999-1011 (2007).
- Dolgin, E. S., Félix, M. -A., Cutter, A. D. Hakuna Nematoda: genetic and phenotypic diversity in African isolates of Caenorhabditis elegans and C. briggsae. Heredity. 100 (3), 304-315 (2008).
- Douglas, A. E. Simple animal models for microbiome research. Nature Reviews Microbiology. 17 (12), 764-775 (2019).
- Barrière, A., Félix, M. -A. Isolation of C. elegans and related nematodes. WormBook. , ed. The C elegans research community 1-19 (2014).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Brenner, S. The genetics of Caenorhabditis elegans. Genetics. 77 (1), 71-94 (1974).
- Crombie, T. A., et al. Local adaptation and spatiotemporal patterns of genetic diversity revealed by repeated sampling of Caenorhabditis elegans across the Hawaiian Islands. Molecular Ecology. 31 (8), 2327-2347 (2022).
- Haber, M. Evolutionary history of Caenorhabditis elegans inferred from microsatellites: Evidence for spatial and temporal genetic differentiation and the occurrence of outbreeding. Molecular Biology and Evolution. 22 (1), 160-173 (2004).
- Watson, E., et al. Metabolic network rewiring of propionate flux compensates vitamin B12 deficiency in C. elegans. eLife. 5, 17670(2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados